1株假单胞菌oprD基因缺失和回补载体的构建
朱艳容 刘志新 杨靖 韩韵 陈果 徐祥


摘要 [目的]构建假单胞菌毒性基因(oprD)缺失突变株,为进一步探讨其编码的毒力因子功能奠定基础。[方法]利用pEX18Gm质粒作为基因敲除的载体,构建假单胞菌oprD 基因重组自杀质粒,通过同源重组并激活自杀基因方式,筛选到目的基因缺失的突变株,再利用pBBR1MCS-2质粒作为目的基因回补的载体,构建回补载体。[结果]同源重组后,经过庆大霉素和氯霉素双抗平板、蔗糖平板筛选和PCR鉴定,成功获得了oprD基因缺失突变株,类似操作获得了回补菌株。[结论]基因缺失突变株的成功构建可为下一步的毒性机理研究奠定基础。
关键词 假单胞菌;毒力因子;基因敲除;基因回补
中图分类号 S 188文献标识码 A文章编号 0517-6611(2022)02-0109-03
doi:10.3969/j.issn.0517-6611.2022.02.029
开放科学(资源服务)标识码(OSID):
Construction of oprD Gene Deletion and Complement Vector of Pseudomonas sp.
ZHU Yan-rong1,2,LIU Zhi-xin1,YANG Jing1 et al(1. Basic Medical College, Hubei University of Medicine, Shiyan, Hubei 442000; 2.Hanjiang River Bureau of Hydrology and Water Resources Survey, Xiangyang, Hubei 441000)
Abstract [Objective]To construct Pseudomonas virulence gene (oprD) deletion mutant,and lay the foundation of function for the virulence factor encoded.[Method]We constructed Pseudomonas oprD using pEX18GM plasmid as the vector of gene knockout.The mutant strain with target gene deletion was screened by homologous recombination and activation of suicide gene.The plasmid pBBR1MCS-2 was used as the vector of target gene complement,and the complement vector was constructed.[Result]The results showed that the mutant strain was subjected to gentamicin and chloramphenicol double resistance plate.Through sucrose plate screening and PCR identification,the oprD gene deletion mutant strain was successfully obtained.Similar operation obtained the complement strain gene deletion mutant strain.[Conclusion]The successful construction laid a foundation for further study of toxicity mechanism.
Key words Pseudomonas;Virulence factor;Knockout;Complement
基金项目 湖北省教育厅指导性项目(B2016113)。
作者简介 朱艳容(1985—),女,湖北咸宁人,高级工程师,硕士,从事水环境监测研究。*通信作者,讲师,硕士,从事病原微生物研究。
收稿日期 2021-05-12;修回日期 2021-06-15
假单胞菌属(Pseudomonas)细菌是杆状或稍弯的革兰染色阴性杆菌,菌体大小约为(0.5~1.0)μm ×(1.5~4.0)μm,不形成芽胞,有荚膜;某些假单胞菌株能产生荧光色素,如红、蓝、黄等水溶性色素,有些则具有较强的分解有机物的能力[1-3]。假单胞菌是广泛存在的环境微生物,在土壤、空气、水、动植物体内均有分布,该属中有许多致病菌,能引发动植物疾病[4-7];另外,假单胞菌属细菌在生物技术应用领域也有着巨大的潜力[8-10]。
该试验所用细菌是从东湖水域中筛选获得高产铁载体假单胞菌,其产铁载体的机制与其他假单胞菌不同,前期研究表明,其发挥毒性作用而导致线虫死亡。由于其產铁载体的机制与其他已知的假单胞菌不同,故其可能有特殊的毒性机制[11-13]。利用转座子插入突变株的构建和毒性相关基因的多次筛选,得到一些与细菌毒性相关的基因,以其中1个oprD为研究对象,扩增目的基因上下游片段,再利用同源重组交换方法,构建oprD基因缺失载体,同时构建回补菌株,从分子生物学方向揭示HYS菌株的毒性作用机制。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒。高产铁载体假单胞菌、大肠杆菌E.coli DH5α感受态细胞、克隆载体pEX18Gm、回补载体pBBR2由武汉大学谢志雄教授惠赠。
1.1.2 主要试剂。甘油(BIOSHARP)、氯霉素、庆大霉素、卡那霉素(普博欣);基因组提取试剂盒、胶回收试剂盒(天根生物);内切酶、T4 DNA连接酶、2×PCR Taq Mix(Takara 公司);Gold View(赛百盛生物);三氯甲烷、异丙醇、乙醇、蔗糖、氯化钠(国药集团);蛋白胨、酵母提取物、琼脂粉(美国BD公司)。
1.1.3 引物及测序。引物根据 NCBI 序列设计(表1),由生工生物工程合成。 该试验所有相关 DNA 测序工作均由华大基因完成。
1.1.4 主要仪器。PCR 仪(Veriti,Applied Biosystem 公司);全自动凝胶成像系统(美国Protein Simple公司);水平电泳仪(Bio-Rad 公司);移液器、离心机(Eppendorf 公司);恒温摇床、恒温培养箱(上海世平实验设备有限公司)。
1.2 试验方法
1.2.1 假单胞菌基因组提取。将活化后的HYS菌液按1%转接到新的LB中培养6 h离心去上清;用天根生物试剂公司的基因组试剂盒抽提假单胞菌基因组DNA,-20 ℃保存。
1.2.2 基因敲除菌株的构建。
1.2.2.1 待敲除基因上下游片段的扩增与回收。根据已知的HYS基因序列,定位oprD基因上下游长度约350 bp大小的序列通过软件Primer Premier 5.0设计引物扩增。反应体系50 μL:去离子水20 μL;2×Taq mix 25 μL; Primer 1 2 μL;Primer 2 2 μL; HYS DNA 1 μL。反应程序105 ℃预变性5 min;94 ℃变性30 s;58 ℃退火30 s;72 ℃延伸30 s,循环30次;72 ℃延伸10 min。PCR反应后,用天根生物试剂公司的胶回收试剂盒对目的条带回收。
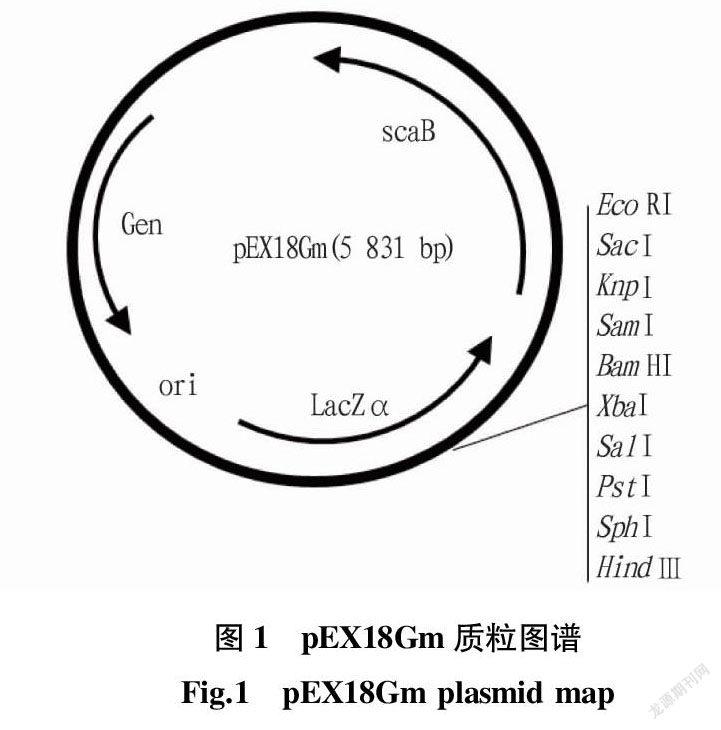
1.2.2.2 基因上下游片段与敲除载体的连接。胶回收的上游基因片段用Xba Ⅰ 和Sac Ⅰ 双酶切,下游片段用Sac Ⅰ 和Eco RⅠ 双酶切,酶切体系20 μL,其中去离子水6 μL,10×FastDigest Buffer,2 μL,PCR purified (0.2 μg)10 μL,FastDigest enzyme 1 1 μL,FastDigest enzyme 2 1 μL。质粒pEX18Gm用Xba Ⅰ 和 Eco RⅠ 双酶切(图1)。酶切体系20 μL,其中,去离子水13 μL,10×FastDigest Buffer 2 μL,plasmid DNA(1 μg)3 μL,FastDigest enzyme Xba I 1 μL,FastDigest enzyme Eco RI 1 μL。然后加T4连接酶放入16 ℃恒温箱孵育3 h。
1.2.2.3 转化过程及转化子鉴定。将孵育好的20 μL连接体系加入100 μL感受态细胞混匀,冰浴30 min后42 ℃热激90 s,立即置于冰上,2 min后补加LB液体培养基,37 ℃水浴。离心去上清后用LB悬浮细胞并涂布抗性平板。37 ℃孵育过夜。选取平板上的转化子进行PCR鉴定,反应体系10 μL,其中,去離子水4.0 μL,2×Taq max 5.0 μL,M13F 0.5 μL,M13R 0.5 μL,菌落(转化子)1个。反应程序:105 ℃预变性5 min;94 ℃变性30 s;55 ℃退火90 s;72 ℃延伸30 s,循环30次;72 ℃延伸10 min。PCR反应后,凝胶电泳并观测所得条带是否正确。正确条带的菌株送公司测序。
1.2.2.4 假单胞菌与转化子的同源交换。取过夜培养的HYS菌液和鉴定正确的转化子菌液分别稀释后等体积混合均匀,滴入LB后放入30 ℃恒温箱孵育36~48 h,用20%甘油将LB平板上的菌苔洗脱下来记为100,然后进行梯度稀释至10-6。取后3个稀释度的菌液涂布庆大霉素和氯霉素双抗平板,对照做同样处理培养24 h,从平板上挑单个菌落接种到同样双抗的LB液体培养基中,振荡培养12 h,然后转接到没有抗性的LB液体培养基中培养12 h,梯度稀释菌液至10-6,并取最后3个稀释度下的菌液涂布5%蔗糖平板,12 h后PCR鉴定单菌落是否为敲除株。
1.2.3 基因的回补菌株及其过表达菌株的构建。
1.2.3.1 回补基因表达质粒的构建。
以HYS为模板,设计引物将目的基因扩增后用Eco RⅠ 和 Xho Ⅰ 双酶切并回收,再与用相同内切酶处理的质粒载体pBBR1MCS-2进行连接,并通过转化进入大肠杆菌中,鉴定正确后得到带有重组表达质粒的菌株。具体操作参照敲除质粒的构建(图2)。
1.2.3.2 假单胞菌的基因回补。通过接合使重组表达质粒进入已构建好的基因敲除菌株和野生型HYS菌株中,接合时间为12 h,洗脱后,将菌液稀释至10-4~10-6涂布到有双抗性(卡那霉素和氯霉素)的筛选平板。同时设置对照组构建带有质粒载体pBBR1MCS-2的基因敲除菌株和HYS菌株。
2 结果与分析
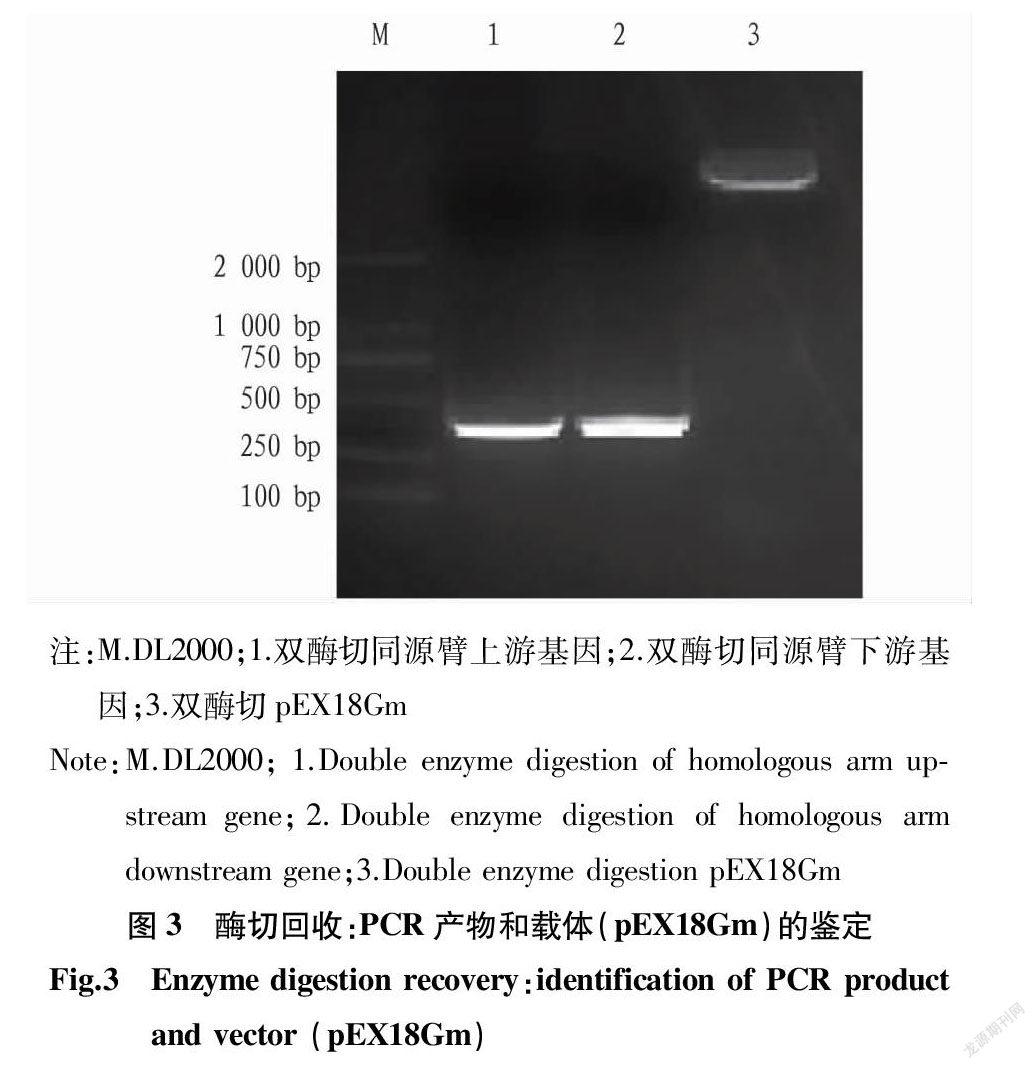
2.1 目的基因片段克隆 根据HYS基因组上oprD基因上下游序列设计引物,得到与预期大小一致的条带,上下游均约为350 bp。双酶切上下游片段和pEX18Gm(5 831 bp)载体,琼脂糖凝结结果见图3。
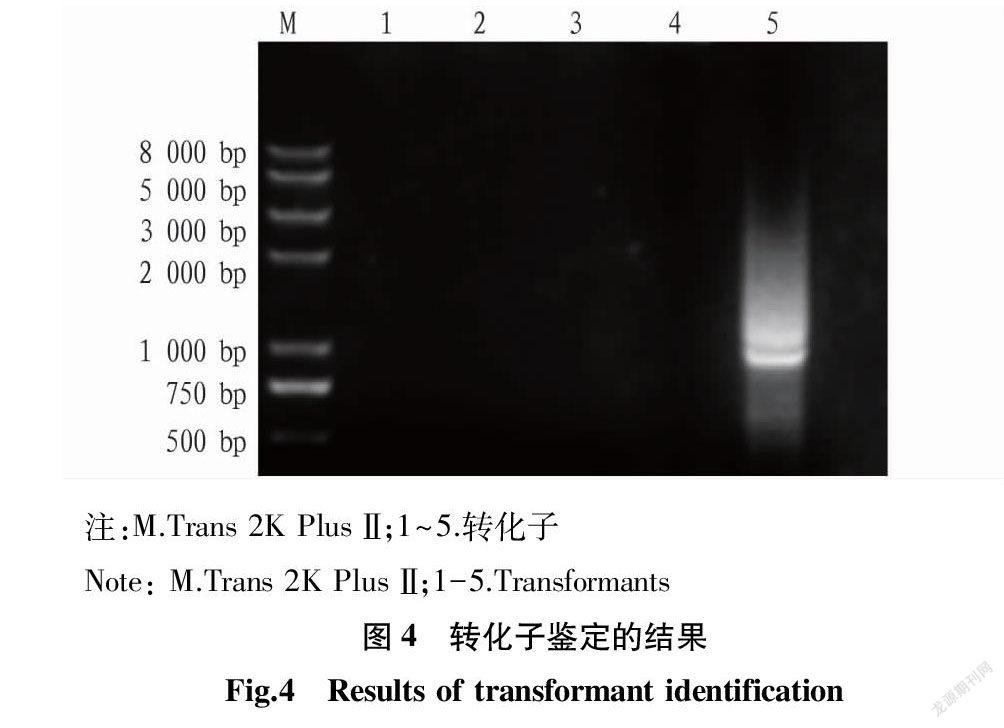
2.2 基因缺失载体的构建 将双酶切回收后的片段与载体进行三段酶连,所得产物转大肠杆菌感受态,涂布于抗性平板。所得转化子5个,以通用引物进行PCR鉴定,所得结果见图4。只有一个转化子在800 bp处有条带与预期相符。测序结果显示,序列同源性99%以上为阳性克隆。
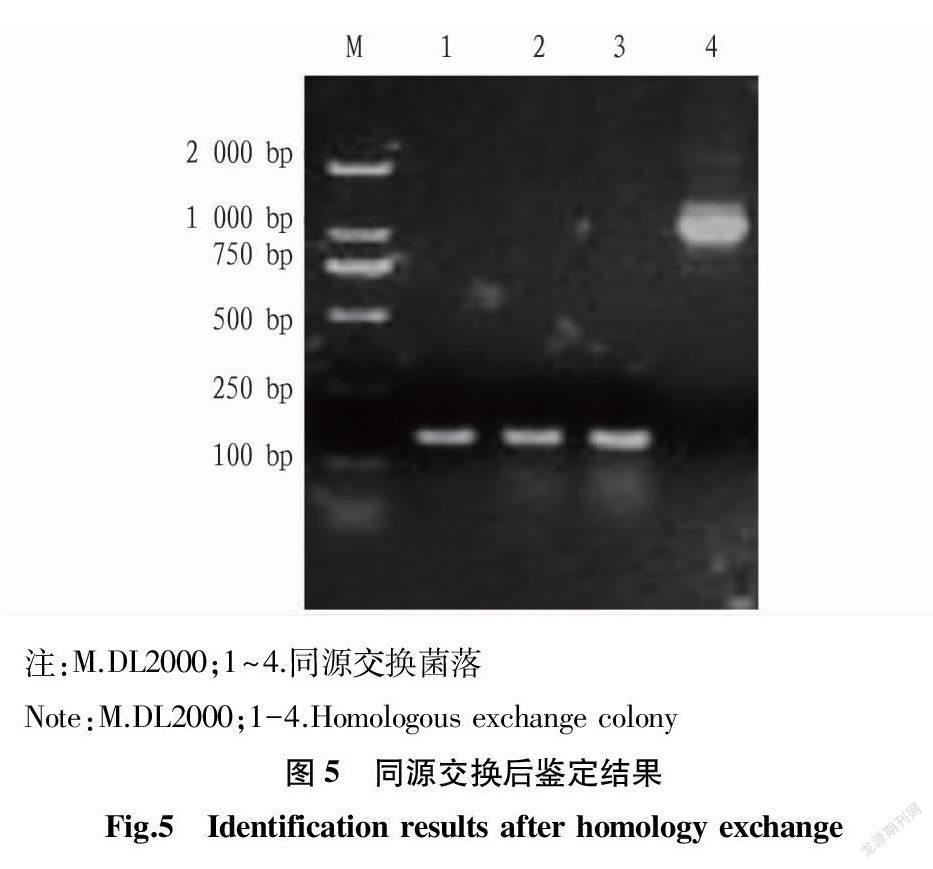
2.3 HYS同源交换 将HYS菌株与转化子混合均匀,孵育后洗脱,梯度稀释后选择合适的稀释梯度涂布庆大霉素和氯霉素双抗平板。将所选择菌落接种于同样双抗的LB液体培养基中12 h,转接到没有抗性的LB液体培养基12 h,梯度稀释后选择合适稀释度涂布5%蔗糖平板,以HYS基因组为模板,设计引物对长出的4个菌落PCR进行鉴定,结果表明有一个条带相符,公司测序结果符合为同源交换阳性株(图5)。
因质粒pEX18Gm不能在假单胞菌中独立复制,只可以通过整合后复制。故通过庆大霉素和氯霉素双抗选择和无抗选择,可以得到敲除株与野生株,利用5%蔗糖平板筛选出敲除株。
2.4 目的基因回补菌株的构建
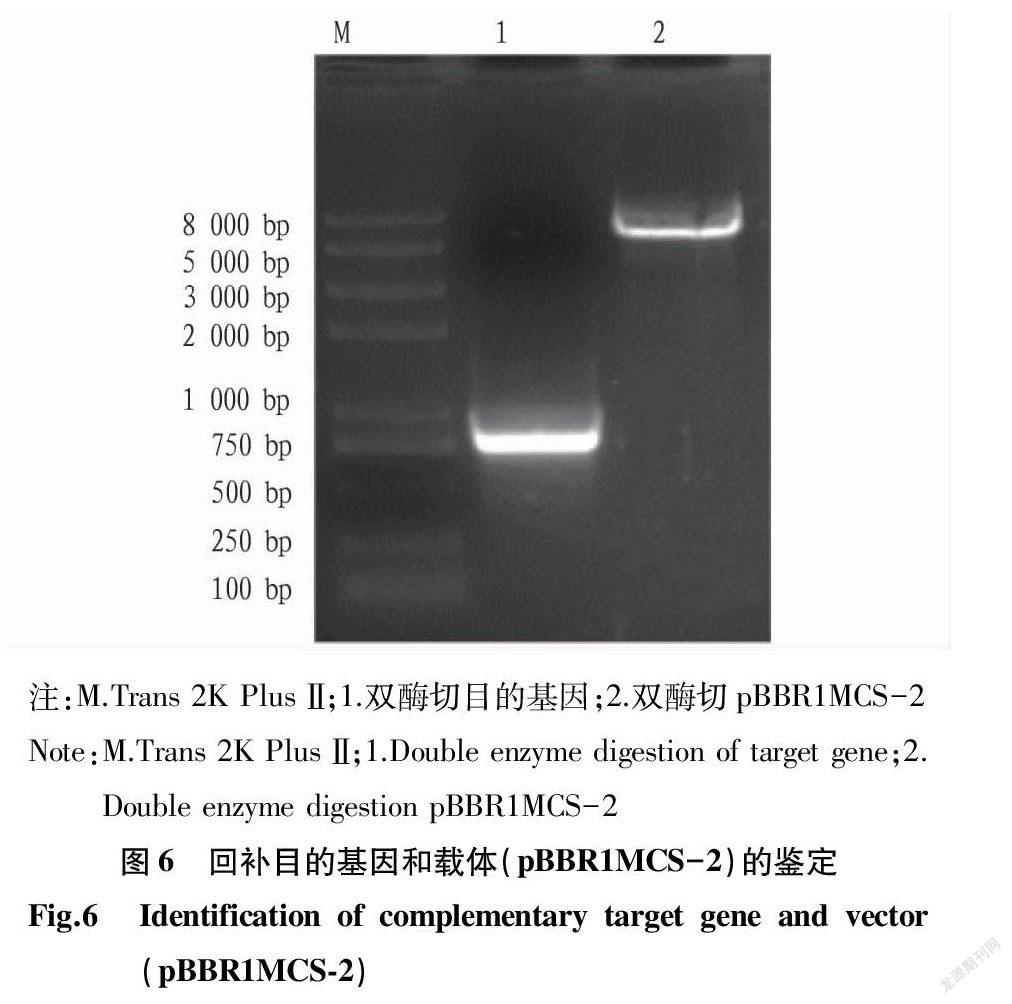
以HYS为模板,设计引物将目的基因扩增(750 bp)后双酶切,用相同内切酶处理的质粒载体pBBR1MCS-2(5 144 bp)胶回收(图6),酶连并转化,鉴定正确后得到带有重组表达质粒的菌株。测序结果正确,回补载体构建完成。
3 讨论该研究所用的假单胞菌,因产铁载体与其他假单胞菌不同[11,13],以该细菌喂食线虫,有明显的线虫致死效应。利用转座子插入突变的方式,多次筛选得到多个毒性相关基因。但目前对这些基因的毒性作用机理却知之甚少。
在对病原微生物毒性基因的研究过程中[14-15],目的基因的缺失是确定其功能、阐明毒性机理的重要方法。利用同源重组交换的方式敲除待研究基因是研究其功能的重要手段[16-20]。
利用分子生物學的手段,以同源重组为基础构建了假单胞菌潜在毒性基因oprD的缺失载体和回补载体。利用氨苄抗性筛选缺失基因转化子。在同源交换过程中,通过庆大霉素和氯霉素双抗筛选双交换敲除株和野生株,最终通过激活蔗糖致死性基因排除野生株,得到敲除株,并以测序的方式验证其正确性。另外,用类似的方法对目的基因进行回补,为下一步研究oprD基因的毒性功能以及为其他潜在的毒性基因功能研究奠定基础。
参考文献
[1]
高芸.生防芽孢杆菌及假单胞菌拮抗植物微生物病害研究进展[J].北方园艺,2021(2):131-136.
[2] GAO J W,XIE G F,PENG F,et al.Pseudomonas donghuensis sp.nov,exhibiting high-yields of siderophore[J].Antonie van leeuwenhoek,2015,107(1):83-94.
[3] 蒋海霞,周莲,何亚文.铜绿假单胞菌生防菌株抑菌代谢产物及其生防应用研究进展[J].微生物学通报,2015,42(7):1338-1349.
[4] PARKINS M D,SOMAYAJI R,WATERS V J.Epidemiology,biology,and impact of clonal Pseudomonas aeruginosa infections in cystic fibrosis[J].Clin Microbiol Rev,2018,31(4):1-38.
[5] KESWANI C,SINGH H B,GARCA-ESTRADA C,et al.Antimicrobial secondary metabolites from agriculturally important bacteria as next-generation pesticides[J].Appl Microbiol Biotechnol,2020,104(3):1013-1034.
[6] 陈磊.假单胞菌在烟草病虫害防治中的应用[J].安徽农业科学,2021,49(2):5-7.
[7] 徐志伟,魏云林,季秀玲.假单胞菌噬菌体基因组学研究进展[J].遗传,2020,42(8):752-759.
[8] 崔莹,宋凯,何亚文.假单胞菌代谢产物藤黄绿菌素化学结构、生物合成途径、调控机制及应用研究进展[J].中国生物防治学报,2021,37(1):52-59.
[9] FERN NDEZ M,PORCEL M,DE LA TORRE J,et al.Analysis of the pathogenic potential of nosocomial Pseudomonas putida strains[J].Front microbiol,2015,6:1-11.
[10] AL-WRAFY F,BRZOZOWSKA E,GRSKA S,et al.Pathogenic factors of Pseudomonas aeruginosa-the role of biofilm in pathogenicity and as a target for phage therapy[J].Postepy Hig Med Dosw (Online),2017,71:78-91.
[11] SHI H M,HUANG X Q,WANG Z,et al.Improvement of pyoluteorin production in Pseudomonas protegens H78 through engineering its biosynthetic and regulatory pathways[J].Appl Microbiol Biotechnol,2019,103(8):3465-3476.
[12] SULOCHANA M B,JAYACHANDRA S Y,KUMAR S K A,et al.Antifungal attributes of siderophore produced by the Pseudomonas aeruginosa JAS-25[J].J Basic Microbiol,2014,54(5):418-424.
[13] BANO N,MUSARRAT J.Characterization of a new Pseudomonas aeruginosa strain NJ-15 as a potential biocontrol agent[J].Curr Microbiol,2003,46(5):324-328.
[14] SAWADA H,FUJIKAWA T,NISHIWAKI Y,et al.Pseudomonas kitaguniensis sp.nov,a pathogen causing bacterial rot of Welsh onion in Japan[J].Int J Syst Evol Microbiol,2020,70(5):3018-3026.
[15] XIE G F,ZENG M,YOU J,et al.Pseudomonas donghuensis HYS virulence towards Caenorhabditis elegans is regulated by the Cbr/Crc system[J].Sci Rep,2019,9(1):1-12.
[16] 蒋成辉,曾巧英,王萌,等.CRISPR/Cas9 构建 srtA 基因敲除的金黄色葡萄球菌[J].生物技术通报,2020,36(9):253-265.
[17] YU X Y,CHEN M,JIANG Z,et al.The two-component regulators GacS and GacA positively regulate a nonfluorescent siderophore through the Gac/Rsm signaling cascade in high-siderophore-yielding Pseudomonas sp.strain HYS[J].J Bacteriol,2014,196(18):3259-3270.
[18] 石雨鹭,郑瑞,靳亚军,等.水稻 OsDTH13 基因编辑突变体的创制与分析[J].天津师范大学学报(自然科学版),2020,40(3):22-27.
[19] 徐涵,潘超,黄竞,等.铜绿假单胞菌waaL突变体构建及糖基化系统的建立[J].生物技术通讯,2020,31(6):634-640.
[20] 李明奇,王小凤,郭嘉,等.布鲁菌DK63_426基因缺失株的构建及生物学特性研究[J].石河子大学学报(自然科学版),2020,38(1):31-36.

