思南黄牛体重和体尺性状的GWAS分析
袁 扬,赵 恬,姚 丹,张定红,王胤晨*,吴鸿友,汪 勇
(1.贵州省农科院畜牧兽医研究所,贵州贵阳 550005;2.贵州省贵阳市南明区农业农村局,贵州贵阳 550005;3.贵州省罗甸县农业农村局,贵州罗甸 550100)
近年来全基因组关联分析(Genome-Wide Associate Analysis,GWAS)较多应用于家畜的遗传分析和辅助育种。大多数家畜的GWAS研究都与其经济性状相关,如猪的雄烯酮水平、毛被颜色、脂肪性状等相关GWAS研究,马的肌肉表型的研究,绵羊羊角形状研究,鸡的体重影响因素研究和产蛋研究。大多数农作物和家畜主要性状是多基因共同作用的,遗传结构较复杂,难以预测出对性状有重要影响的单个基因。GWAS技术能够在全基因组水平上搜索突变位点,综合性状给出性状相关的位点突变和性状候选基因,极大地促进对家畜复杂性状遗传结构的理解,改善家畜育种计划。
关于牛的体重性状、体尺性状GWAS研究非常多。Sorbolini等人使用混合线型模型对马其顿牛生长性状和胴体性状进行分析,根据SNP位置和连锁不平衡确定了影响性状的目的区域,搜索发现和3个基因上出现了影响脂质代谢、骨骼和肌肉的有意义位点。Ede等使用混合线性模型分析SNP芯片发现与韩国牛体重、胴体性状相关的多个SNP位点。Adoligbe等研究了中国本土牛中基因的SNP及其与体尺性状的关联,研究显示GDF-10存在SNP位点11471(A/G),该突变在秦川牛、郏县红牛和南阳牛中显著影响体尺性状,SNP位点G142A对郏县红牛臀部宽度、胸宽和胸围都有显著影响,认为基因可能是郏县红牛繁育筛选过程中体尺性状的重要遗传标记。李天科等分析甘南牦牛基因多态性和生产性状相关性,发现12116(G/A)、12152(C/T)和13041(T/C)3个SNP位点,结果表明3个位点之间存在弱连锁平衡,不同突变位点基因型与体斜长、体高、胸围、体重差异显著或极显著,与管围差异不显著。郭宪等研究南德温杂交肉牛发现1113(G/A)、9569(G/A)和9628(G/A)3个SNP位点与南德温杂交肉牛的体高、体斜长、胸围和管围显著相关,对肉牛体尺性状有影响。
思南黄牛是中国贵州本土优良黄牛品种,体质紧凑、四肢强健、善于爬山,适于山区耕作放牧,肉用性能良好。本研究以思南黄牛、西门塔尔牛以及二者杂交改良后代为研究对象,从基因组水平对牛体重和体尺性状进行关联分析,为肉牛新品种的选育提供技术参考与借鉴。
1 材料与方法
1.1 实验材料 实验动物群体选自贵州省石阡县聚凤乡彭寨村肉牛养殖合作社,在相同条件下饲养1年,对其中112头思南黄牛(12月龄~18月龄)、17头西门塔尔牛(12月龄~18月龄)以及19头本地杂交牛(18月龄~24月龄)共计148头牛,测量样本的体重、体高、体长、胸宽、胸围、管围等体重和体尺性状数据(测量方法参考DB52/T 1257.2-2017),采集148头牛耳静脉血样本并提取DNA。
1.2 实验仪器与试剂 磁珠法血液基因组DNA提取试剂盒(北京天根生化科技有限公司 DP329),通用台式冷冻离心机(epServices 5805)。样品送至北京诺禾致源生物信息科技有限公司完成全基因组关联分析。
1.3 血液基因组提取及GBS文库构建及测序
1.3.1 血液基因组DNA提取 使用磁珠法血液基因组DNA提取试剂盒(DP348-03)对血液样品的基因组DNA进行提取,操作步骤按照试剂盒说明书进行。
1.3.2 血液基因组DNA质量检测 DNA 3.5 μL(2 μL DNA 样品,1.5 μL 6×Loading Buffer,混匀)上样于1%琼脂糖凝胶,120 V电泳18 min,凝胶成像系统观察结果。
1.3.3 GBS文库构建及测序 检测合格的DNA样品经限制性内切酶对基因组进行酶切,加上带有barcode的接头后,对每个样品进行扩增,然后对样品进行混合,选择需要的片段进行文库构建,利用Illumina HiSeq测序平台进行双端150测序(北京诺禾致源生物信息科技有限公司)。步骤如下:
1)酶切:0.1~1 μg基因组DNA用限制性内切酶进行酶切,以得到适合的marker密度;
西北农林科技大学从美国引育的早熟新品种,为皇家嘎拉芽变。2006年通过了陕西省果树品种审定委员会审定。果实圆锥形,平均单果重180克;盖色浓红,着色有条纹;果皮光滑,果点大;果肉黄白色,风味酸甜适度;可溶性固形物含量14.6%,可滴定酸含量0.22%,果肉硬度11.3公斤/平方厘米;肉质硬脆,汁液多,有香气,为鲜食品种。果实发育期120天左右。丰产性好。红盖露树体适应性强,耐瘠薄,对早期落叶病、白粉病有一定的抗性。
2)加P1和P2接头:酶切后的片段两端加P1和P2 Adapter(可与酶切DNA缺口互补);
3)片段选择:PCR扩增两端分别含有P1和P2接头的tag序列,DNA片段pooling,电泳回收需要区间的DNA;
4)高通量测序:Cluster制备,上机测序。
5)文库构建完成后,使用Qubit 2.0进行初步定量,稀释文库至1 ng/μL,随后使用Agilent 2100对文库的insert size进行检测,insert size符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量(文库有效浓度>2 nM),以保证文库质量。
6)库检合格后,把不同文库按照有效浓度及目标下机数据量的需求pooling后进行Illumina Hiseq PE150测序。
1.4 基因组测序、比对和SNP检测 对Illumina HiSeq测序平台双端测序下机原始数据预处理,过滤掉含有接头序列的测序序列、低质量测序序列、N(未测出的碱基)含量超过该条测序序列长度10%的成对测序序列,最后得到高质量测序序列。以欧洲普通牛()为参考基因组(ftp://ftp.ensembl.org/pub/release-86/fasta/bos_taurus/),基因组大小为2 649 682 029碱基对(不含N),将所有样本作为一个群体与参考基因组比对,使用bwa软件(参数:mem-t4 -k32 -M)将双端高质量测序序列比对到参考基因组,使用samtools软件得到变异的vcf文件,共获得了3 601 237 个SNP位点,经过测序深度3x、Miss 0.2和次要等位基因频率(MAF)>0.01的条件过滤后,最后共获得441 548个SNP位点用于后续分析。
1.6 全基因组关联分析 以欧洲普通牛全基因组序列为参考基因组(ftp://ftp.ensembl.org/pub/release-86/fasta/bos_taurus/,Ensembl UMD3.1),使 用ANNOVAR软件对牛体尺性状相关的SNPs进行功能注释,过滤后的高质量SNP使用GEMMA软件混合线性模型进行关联分析。分析模型为:y=X+Z+W+e,其中,y为所要研究的表型性状;X为固定效应(Fixed Effect),影响y的其他因素,包括群体结构、性别、年龄等因素;Z,标记效应(Marker Effect);W,随机效应(Random Effect),一般指个体的亲缘关系;e,随机误差。采用Bonferoni correction方法来确定阈值。最后将关联到的结果用R软件绘制曼哈顿图和Q-Q图进行展示。
2 结果
2.1 样本表型测量结果 本实验测量了牛的体重(Body Weight,BW)、体高(Body High,BH)、体长(Body Length,BL)、胸宽(Chest Breadth,CB)、胸围(Chest Circumference,CC)、管围(Circumference of Cannon Bone,CCB)性状,这6个体尺性状的描述性统计见表1。
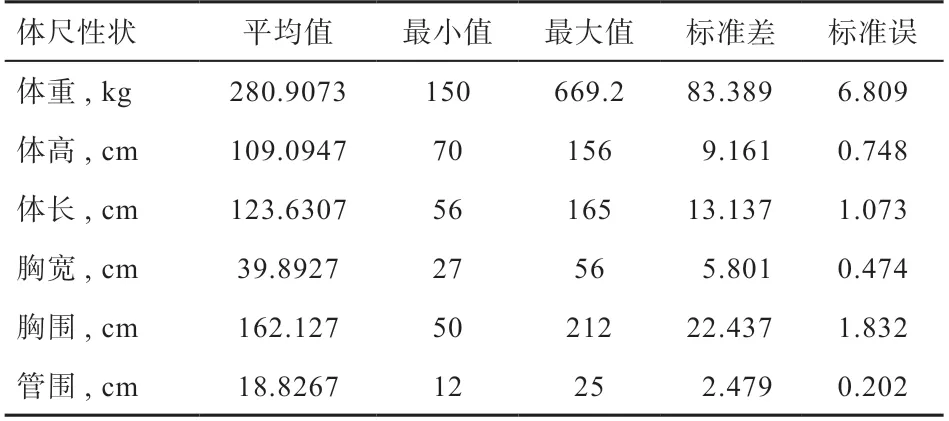
表1 样本体尺性状的描述性统计
2.2 GBS文库测序和SNP检测结果 本次选取了148头牛的样本数据,测序共产生98.396 G Raw data,平均每个样品664.835M Raw data,过滤后的Clean data共98.394G,平均每个样品664.823M。以欧洲普通牛基因组为参考序列,将每个样本测序得到的clean data和基因组进行比对,统计得到的平均比对率、平均测序深度和平均覆盖度见表2。

表2 测序质量
质控后数据与参考基因组比较,在思南黄牛群体中鉴定出SNP位点3 601 237个,平均SNP密度为100.627个/Mb。对获得的SNP质控后,获得高质量SNP位点441 548个,平均SNP密度为12.338个/Mb。
2.3 群体结构分析结果 主成分分析将牛群分为3个类群,样本牛群具有明显分层(图1A)。通过显著性检验纠正不同遗传因素和繁殖条件导致的种群分层。SNP位点构建的进化树在整体上聚为一个大分支和两个小分支,结果和主成分分析以及种群结构分析相吻合(图1B)。Structure软件分析种群结构,K值为3时,148个样本聚类为3簇,此时最低交叉验证值CV error(K=3)=0.464 84,与主成分分析结果相近(图1C)。
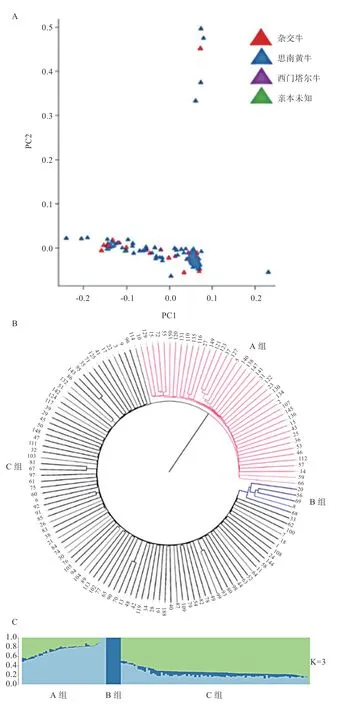
图1 基于SNPs的群体结构分析
2.4 连锁不平衡分析结果 连锁不平衡分析是全基因组关联分析的基础。本研究对148个牛外周血样本进行连锁不平衡分析,计算等位基因相关系数(),分析148个样本441 548个SNP位点在牛基因组的LD共线性。根据位点之间的物理距离,得到连锁不平衡度(LD度)(图2)。结果显示,LD度随距离增加迅速下降。经过注释发现,SNP在基因之间分布最多,其次是内含子,最后是外显子。
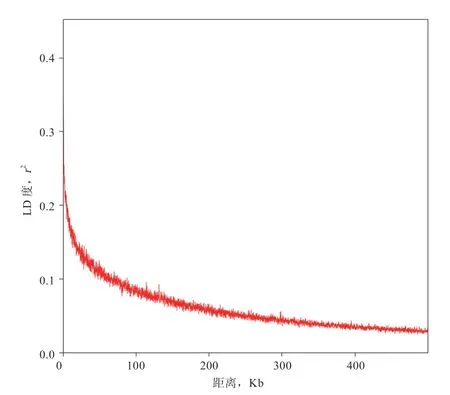
图2 连锁不平衡图
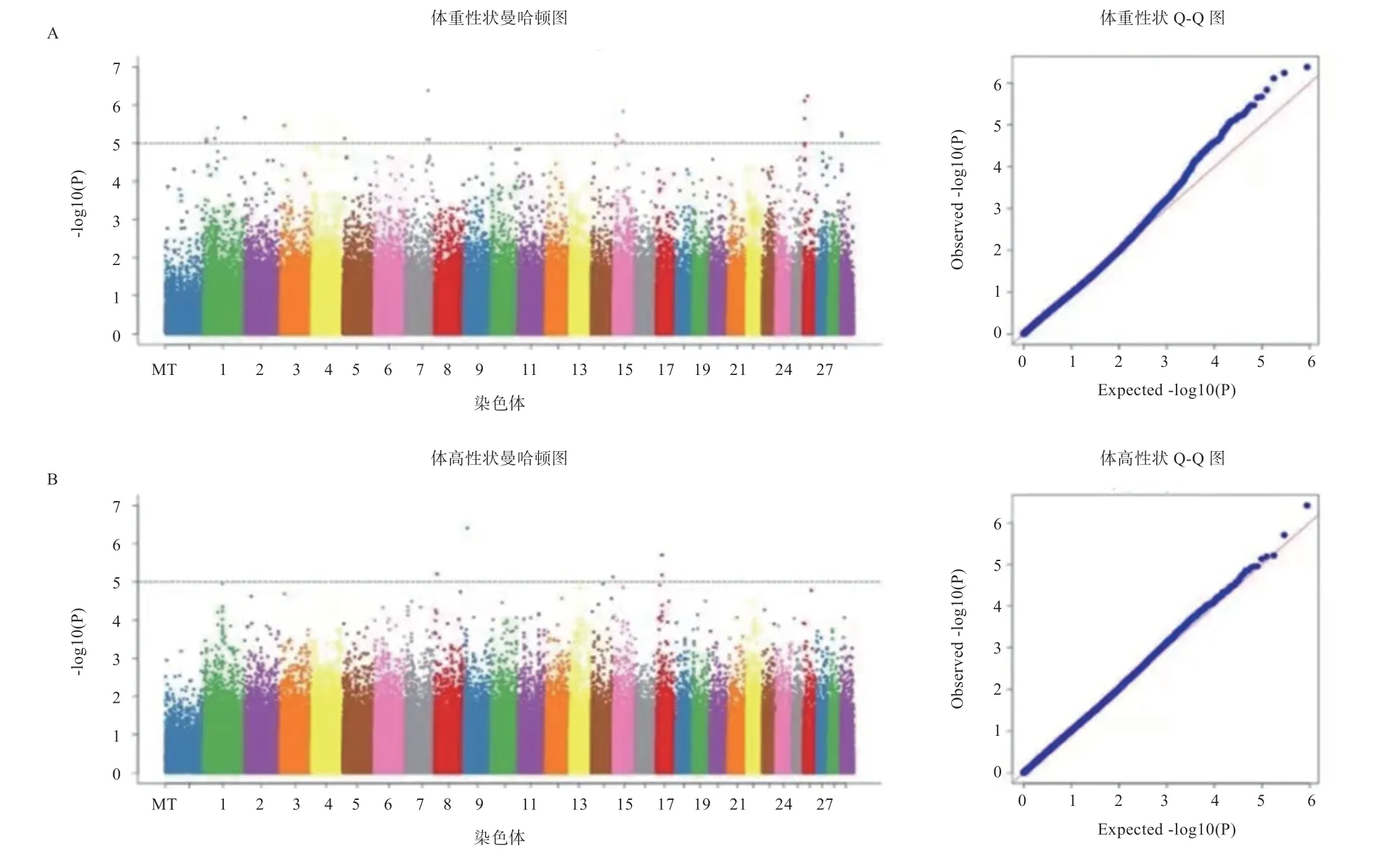
2.5 全基因组关联分析 主成分分析结果作为协变量,利用GEMMA软件混合线性模型对牛的6个体尺性状表现型进行全基因组关联分析(GWAS),得到牛体尺性状的GWAS正态化结果曼哈顿图和Q-Q图(图3)。图3左侧曼哈顿图显示了GWAS的优势位点,信号与原假设之间的偏差在右侧以Q-Q图的形式展示。通过Q-Q图观察发现,观测值与期望值基本在一条斜线上,每个性状Q-Q图结果仅在尾部区域稍微偏离值分布,表明混合线性模型不存在群体分层现象(λ接近1),SNP关联分析的假设与统计分布没有偏离,模型适合该资源群体。曼哈顿图中水平线是Bonferroni显著性阈值,本次关联分析共发现18个基因中的51个SNP与体重、体尺性状变化相关,筛选出体重、体高、体长、胸宽、胸围、管围等身体特征数据相关的基因分别有7、4、2、1、0个和4个(表3)。

表3 牛体重和体尺性状相关候选基因列表
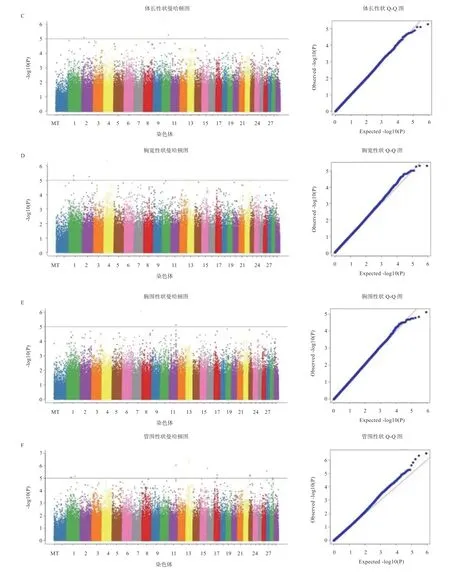
图3 混合线性模型对体重(A)、体高(B)、体长(C)、胸宽(D)、胸围(E)、管围(F)进行GWAS分析
3 讨 论
肉牛是全球主要肉类来源之一,体尺性状是衡量肉牛品种质量的指标。体尺性状和牛的健康、生产率、寿命息息相关。通过GWAS分析筛选牛体尺性状相关的候选基因,分别得到23、5、3、6、1、13个和体重、体高、体长、胸宽、胸围、管围等体重、体尺性状相关的SNP,这些SNP分别在体重的7个、体高的4个、体长的2个、胸宽的1个和管围的4个基因内或基因附近。
3.1 体重性状关联基因 牛的体重性状相关基因有()、、()、()、()、()和()。是牛肉品质的差异表达基因,影响牛的营养摄入,与牛体重性状相关。小鼠中IFT57蛋白的无效突变表现出纤毛病的特征,如上肢和下肢多指、上颌突缩短、神经管缺陷等。人的纯合突变会导致未分类的口面指综合征(Oral-facial-digital Syndrome,OFDS),表现为口腔、面部和四肢畸形,同时也会导致身材矮小。腺苷脱氨酶()基因可能通过脂类代谢影响牛皮下脂肪积累。基因与牛的性格发展有关,这影响了牛的活动频率、情绪控制和顺从性等性格,可能导致牛体重的改变。降钙素基因相关肽()能够影响哺乳动物骨密度,基因敲除小鼠的骨密度会增加,还导致小鼠去脂体重增加,说明信号参与小鼠骨骼分解代谢,同时可能与肠代谢有关。主要功能是促进消炎和免疫抑制性环境,多态性与人类炎症性肠病有关。此外,关于眼皮肤白化病2型基因()之前并无关于体重性状的报告,是一个新的体重性状关联候选基因。
3.2 管围性状关联基因 牛的管围性状关联基因有()、()、()和()。也被称为,与原因不明的肠道疾病克罗恩病有关,该基因可能与消化功能相关。RAB7B是一种GTPase,位于溶酶体、高尔基体和反式高尔基体网络,调节内体到高尔基体的运输,基因能够影响牛采食量与体重,与日本黑牛饲料利用和生长特性相关。在牛饮食受限时,该基因表达量也发生变化,对牛饮食限制及恢复,牛瘤胃上皮转录组分析显示基因出现差异表达。基因还与牛背最长肌的生长相关,也是小牛成骨不全症可能的候选基因。基因的SNP与猪的管围性状相关联,处于猪剩余饲料摄入量相关的重要候选QTL区域。
3.3 体长性状关联基因 牛的体长相关的基因有()和()。位于牛的腿型、蹄型关联区域,有报道附近存在脂肪酸性状相关的SNP位点,与牛的骨骼生长和脂类代谢相关,一定程度上能够解释其与体长性状的关联。基因存在人近端、远端结肠的差异甲基化位点,与消化系统的甲基化相关,由此可能会影响牛的体长性状。
3.4 身高性状关联基因 牛的身高性状关联基因有()、()、()和()。和都位于8号染色体上,都是牛的拷贝数变异基因。基因和基因都位于内罗尔牛背最长肌组织中锌含量有关QTL区域内。COL12A1是一种胶原蛋白,表达量随着牛背最长肌的发育而逐渐下降,该基因位于青海高原牛和金川牛胸椎骨长度选择压力区域,牛胸椎骨的节数和长度直接影响牛的骨架,从而和身高产生关联。的突变缺失会导致肌肉功能性和力量传导能力下降,此外,基因还是牛背最长肌组织中铁和铜含量重要的差异表达基因。SNBT1是一种外周膜蛋白,参与调控肌肉收缩过程,是快红肌和慢红肌的表达差异最大基因之一,同时处于牛背最长肌组织中的单不饱和脂肪酸相关区域,可能也在牛的脂质代谢中起到一定作用。
3.5 胸围性状关联基因 牛的胸围性状关联基因有()。位于影响鱼骨长的QTL区域,能够影响鱼头骨大小。还是猪体内共表达网络中肌肉肉质性状关联基因。
综上所述,在以上筛选的基因中:能够影响动物体型;和能够影响脂质代谢;影响牛的性格;影响动物骨骼生长发育;和影响动物消化系统;和影响动物的采食量;影响牛背最长肌组织中矿物质含量。这些基因共同作用,综合影响了牛的体重和体尺性状。
4 结 论
本研究通过对112头思南黄牛、17头西门塔尔牛以及19头本地杂交牛共计148个个体生产性状进行GWAS分析,筛选出体重、体尺性状相关的24个SNPs位于18个基因附近,发现和分别是体重、体高、体长、胸宽和管围相关性状最有相关性的候选基因,这些候选基因将有助于进一步探索思南黄牛的遗传改良性状,为后续思南黄牛遗传信息精细作图提供数据,为思南黄牛育种研究提供参考。