采用融合规则与BERT-FLAT模型对营养健康领域命名实体识别
郑丽敏,任乐乐
采用融合规则与BERT-FLAT模型对营养健康领域命名实体识别
郑丽敏1,任乐乐2
(1. 食品质量与安全北京实验室,北京 100083;2. 中国农业大学信息与电气工程学院,北京 100083)
人类营养健康命名实体识别旨在检测营养健康文本中的营养实体,是进一步挖掘营养健康信息的关键步骤。虽然深度学习模型广泛应用在人类营养健康命名实体识别中,但没有充分考虑到营养健康文本中含有大量的复杂实体而出现长距离依赖的特点,且未能充分考虑词汇信息和位置信息。针对人类营养健康文本的特点,该研究提出了融合规则与BERT-FLAT(Bidirectional Encoder Representations from Transfromers-Flat Lattice Transformer,转换器的双向编码器表征量-平格变压器)模型的营养健康文本命名实体识别方法,识别了营养健康领域中食物、营养物质、人群、部位、病症和功效作用6类实体。首先通BERT模型将字符信息和词汇信息进行嵌入以提高模型对实体类别的识别能力,再通过位置编码与词汇边界信息结合的Transformer模型进行编码以提高模型对实体边界的识别效果,利用CRF(Coditional Random Field,条件随机场)获取字符预测序列,最后通过规则对预测序列进行修正。试验结果表明,融合规则与BERT-FLAT模型的人类营养健康领域识别的准确率为95.00%,召回率为88.88%,F1分数为91.81%。研究表明,该方法是一种有效的人类营养健康领域实体识别方法,可以为农业、医疗、食品安全等其他领域复杂命名实体识别提供新思路。
营养;健康;食物;命名实体识别;自注意力机制;BERT模型;Transformer模型
0 引 言
随着中国居民生活水平不断提高,人们健康意识不断增强,人们的饮食观念从吃饱到吃好,再向实现营养均衡调理身体健康转变,从有病治病向无病预防转变[1]。通过良好的饮食习惯、注重食物的营养搭配以及食用具有健康功能的食物可以防止和降低疾病发生率[2]。但是目前人们无法在海量的互联网信息中快速定位准确的个性化健康信息,无法满足精准营养需求[3]。构建人类营养健康领域知识图谱,存储食物和病症数据,帮助用户提供符合营养标准的个性化饮食[4-5],对指导人们个性化健康饮食具有重要意义。营养健康实体识别是构建人类营养健康领域知识图谱的关键步骤,实体识别的效果会直接影响知识图谱的质量[6]。因此,需要研究有效的实体识别算法,准确识别营养健康领域实体,为营养健康知识图谱的构建提供数据支撑。
命名实体识别(Named Entity Recognition,NER)在很多领域进行了应用,并且随着深度学习的发展,模型能够融合多种特征,捕捉更深层次的语义关系,达到理想的识别效果[7-9]。杨培等[10]使用注意力机制对化学领域药物类别实体进行识别,解决了实体标签的全文非一致性问题。Li等[11]将BiLSTM(Bi-directional Long Short-Term Memory,双向长短时记忆网络)与CRF(Coditional Random Field,条件随机场)结合实现了中文电子病历领域NER,张晗等[12]通过引入对抗训练的方式结合注意力机制与BiLSTM-CRF实现了军事领域NER,董哲等[13]通过BERT与对抗网络训练结合实现了食品领域的命名实体识别。任媛等[6]提出了融合注意力机制与BERT+BILSTM+CRF模型的渔业标准定量指标识别方法,解决了渔业标准定量指标识别准确率不高的问题。以上方法对领域内实体识别效果较好,但是无法直接使用到营养健康领域识别复杂实体。
在营养健康领域,王璐[14]使用基于统计的特征模板和CRF模型对健康膳食知识进行命名实体识别,对于具有文本特征的数据取得了较好的识别效果,但是需依靠特征模板,对于多源数据,需编制不同的特征模板,无法全面概括文本特征。迟杨[15]采用了基于词典与统计机器学习相结合的方法,解决了在健康饮食领域非结构化数据中获取实体的问题,可以在一定程度上保证实体的质量,但是无法识别词典以外的实体以及复杂实体。以上垂直领域命名实体识别方法仅预测了实体边界和字符类别标签,但是在中文命名实体识别任务中,没有明显的词汇边界,导致序列模型难以识别出复杂实体[16],据研究发现,结合词汇信息的字-词格结构[17-19]对命名实体识别任务很有效,但是由于晶格结构复杂,不能实现并行计算,通常推理速度较慢。Li[20]等将格子结构转化为数个区间组成的平面结构,提出FLAT(Flat Lattice Transformer)模型,借助转换器充分利用格子信息,实现并行化处理,且识别性能达到最优。
在分析融合词汇信息方法的基础上,研究营养健康领域实体识别方法,对营养健康领域中食物、营养物质、人群、部位、病症和功效6类实体进行识别,提出了融合规则与BERT-FLAT模型的营养健康领域命名实体识别方法,以实现营养健康领域实体的精准识别,为农业、医疗、食品等领域实体识别提供新思路。
1 材料与方法
1.1 数据采集与数据预处理
1.1.1 语料采集
营养健康领域命名实体识别没有公开的语料数据集,本研究的数据来源主要通过Python语言构建爬虫框架,获取各网站(如生命时报网、中华养生网、食品科学网等)中关于饮食健康、食疗和养生文本语料,共获取2 135篇文本语料。
营养健康领域词典涉及到多个实体类型,且没有公共词典,本研究通过爬虫构建营养健康领域词典,词汇主要来源为《中国居民膳食营养素参考摄入量》2013修订版[21]、搜狗输入法细胞词库、百度输入法词库、清华大学自然语言处理(Natural Language Processing,NLP)词库以及现有医学数据库北京大学SymMap[22],共计170 000词汇。
1.1.2 语料处理
试验数据包含大量网页标签、链接、特殊字符等非文本数据结构,影响数据标注的质量。首先对数据进行初步清洗,删除包含特殊字符、无关营养健康等噪声文本。将初步清洗的文本语料进行分句,并清除噪声句子,最终得到113 747个句子。
1.1.3 词典处理
对营养健康领域词汇进行分类,包括食物、营养物质、人群、病症、功效和部位6类。其中营养物质包括《中国居民膳食营养素参考摄入量》中包含的33种膳食营养元素,而非膳食营养素目前研究较少没有明确的定义,但对人体的健康具有非常重要的作用,本研究将除膳食营养元素的物质均定义为非膳食营养元素。通过对词汇人工清洗,最终构建了6类词典,各类词典定义及词汇量如表1所示。
另外根据获取的词汇,定义了4类特征词典,其中烹饪词典表示菜肴的烹饪方式,颜色词典表示食材中常见的颜色,状态词汇和物质词汇分别表示食物和营养物质常见的词尾,4类特征词典词汇量与实例如表2所示。
1.1.4 语料标注
由于营养健康领域的标注数据库的匮乏,领域中没有一个广泛使用的标注规范[23]。结合营养健康领域知识特点,采用人工标注方式进行语料库标注,语料库包含实体共89 574个,其中食物40 582个、营养物质13 848个、人群2 528个、病症11 626个、功效13 797个和部位7 196个。不同类型实体标注符号与实例如表3所示。语料库标注示例如图1所示。
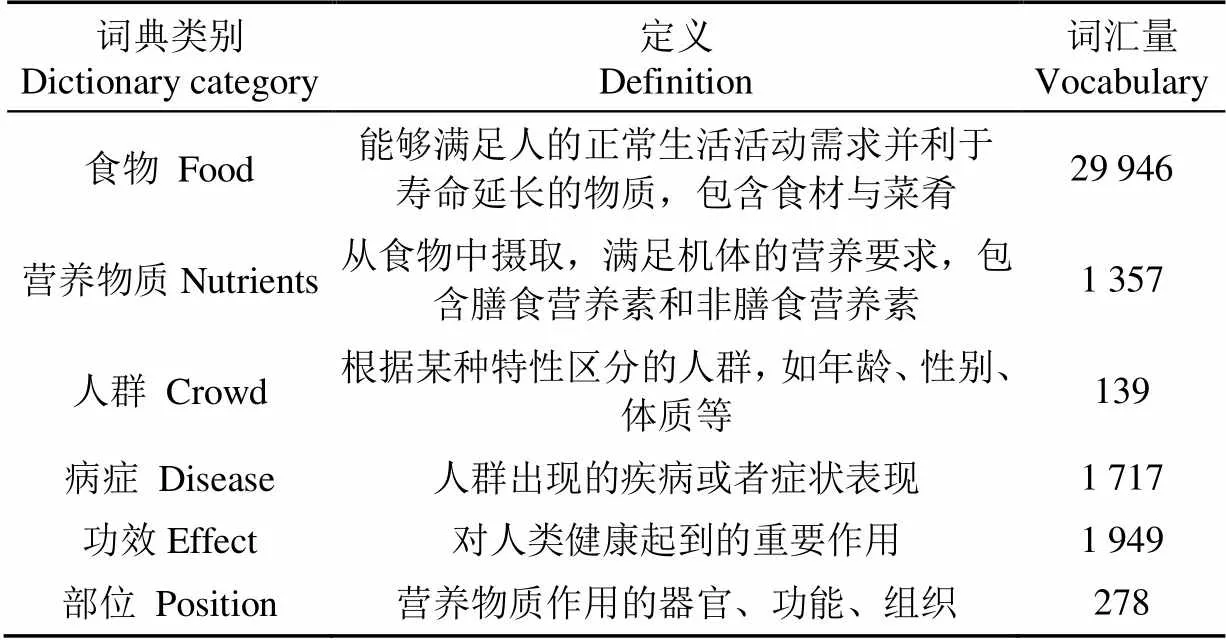
表1 领域词典类别、定义及词汇量
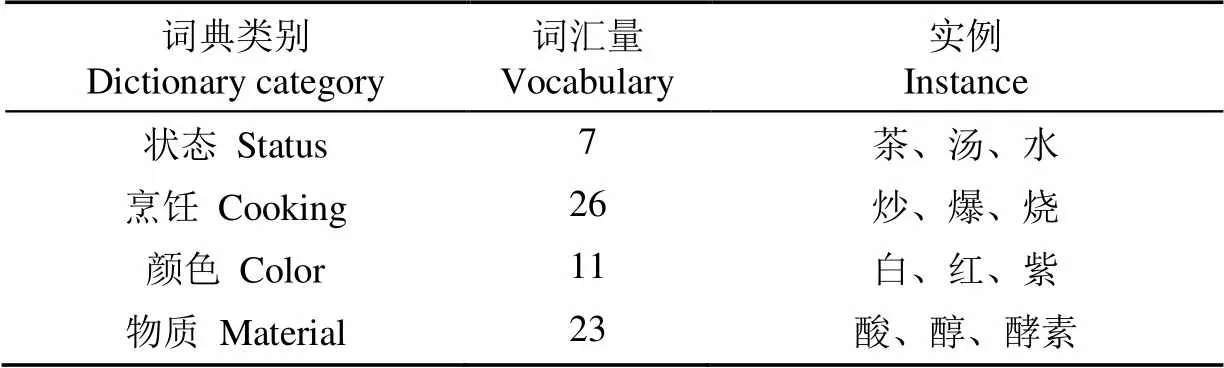
表2 4类特征词典词汇量与实例
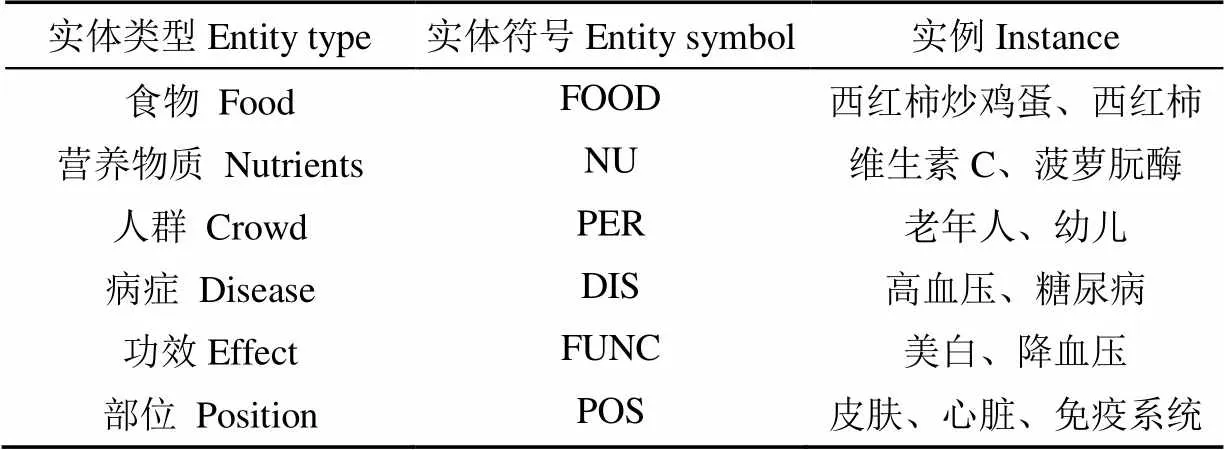
表3 营养健康实体标注符号与示例
1.2 融合规则与BERT-FLAT的营养健康领域命名实体识别模型
1.2.1 模型总体架构设计
在营养健康领域中存在大量的复杂实体和专有名词,如果使用基于字粒度的命名实体识别模型,会导致字在词汇中的语义表达缺失,并且丢失词边界信息,如果使用基于词粒度的命名实体识别模型会出现分词错误传播问题[17],词汇边界对实体边界通常起着至关重要的作用,因此提出将字符与词汇进行融合的实体识别方法。此外,由于部分营养健康领域的文本序列较长,若采用固定长度的上下文变量,会导致语料分片之间无法信息共享,丢失上下文信息,需要采用有效的方法解决长距离依赖问题。在设计营养健康领域实体识别模型时需要考虑字粒度和词粒度信息融合、词汇边界信息和长距离依赖等问题。
对常用的命名实体识别模型研究发现,BERT预训练模型可以用来提取包含上下文的文本信息,但是BERT在垂直领域命名实体识别任务中需要领域内的词汇信息加持;Transformer的自注意力机制能够捕捉句子之间的长距离依赖;CRF能够对预测标签进行解码;根据营养健康文本特点,制定文本规则,能够对解码序列进一步修正。因此,提出了一种融合规则与BERT-FLAT的命名实体识别方法。
融合规则与BERT-FLAT模型总体结构如图2所示。共分为3层网络模型,首先将字符信息和词汇信息进行拼接,输入到BERT模型中进行实体识别模型的预训练处理,然后为每个字符和词汇构建头位置编码和尾位置编码,表示字或词的开始位置和结束位置,将BERT层输出作为字符嵌入和构建的词嵌入输入到FLAT层进行编码,获取编码序列,为了解决梯度消失问题在自注意力机制和全连接层均加入残差连接与归一化,保证信息的无差传递。再将FLAT层输出的编码序列输入到CRF层进行解码,获取单词的预测标签,最后根据营养健康领域文本规则修正序列标签,得到最终预测结果。
1.2.2 嵌入层
嵌入层是将中文文本序列转换为字符或词汇的密集向量表示或分布式表示[24]。BERT模型是一个包含字符级、句子级特征的预训练语言模型[25],为捕捉上下文信息,BERT采用双向Transformer作为编码器,通过注意力机制对文本进行建模。BERT模型的输入将字嵌入、位置嵌入和句子嵌入进行拼接,输入堆叠的Transformer模型中进行特征提取,进而得到输出序列向量作为字符嵌入。BERT模型结构如图3所示。
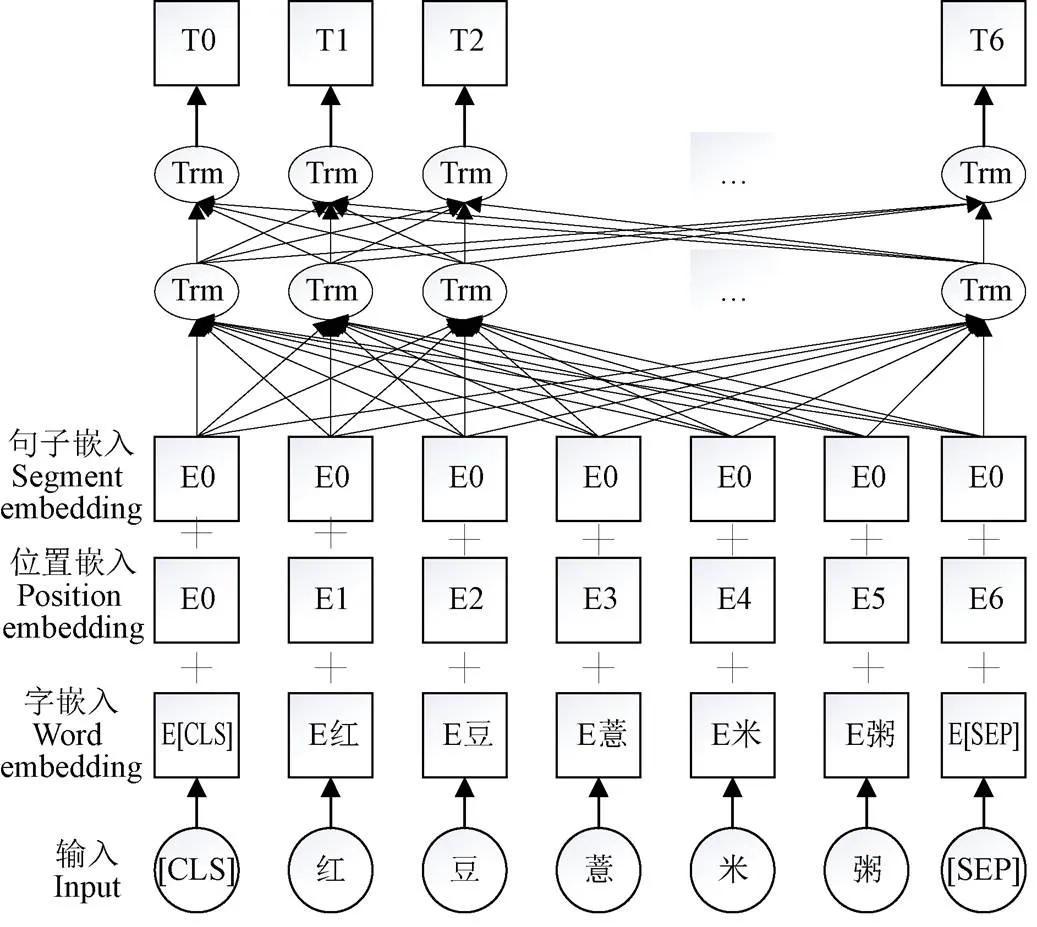
注:[CLS]和[SEP]表示BERT对序列的标记,[CLS]标识序列开始位置,[SEP]标识句子见分割;E表示每个字符的分布式表达;Trm表示BERT模型中堆叠的Transformer模型;T表示BERT模型输出的序列向量。
词嵌入即用向量表示词汇,主要分为两种:基于计数方法和基于推理方法。基于计数的方法是获取语料库的统计数据,一次性处理全部学习数据,但当词汇量很大时,会导致计算机难以处理。而基于推理的方法是通过神经网络在部分数据上学习,并反复更新权重[26]。Word2vec是基于推理方法实现的词嵌入学习模型,能够将神经网络中耗时的线性隐藏层去除,研究表明Word2vec模型的效果较好[27],因此本研究采取Word2vec训练词嵌入。
1.2.3 FLAT层
营养健康领域中存在大量的复杂实体,且实体中存在潜在实体,不同顺序的输入序列标识不同的实体,因此输入序列的位置信息对于NER任务非常重要。Transformer模型不仅可以对序列中的长距离依赖进行建模[20],同时还标记了序列的位置信息。本研究在Transformer编码器的基础上改进位置编码,为每个字符和词汇构建头位置与尾位置,分别表示字符和词汇的开始位置与结束位置,若头位置与尾位置相同表示当前表示为字符,反之为词汇。将BERT模型输出的字符嵌入与构建的词嵌入结合输入到FLAT层的Transformer编码器中,获取编码序列,字符嵌入和词汇嵌入如图4所示。
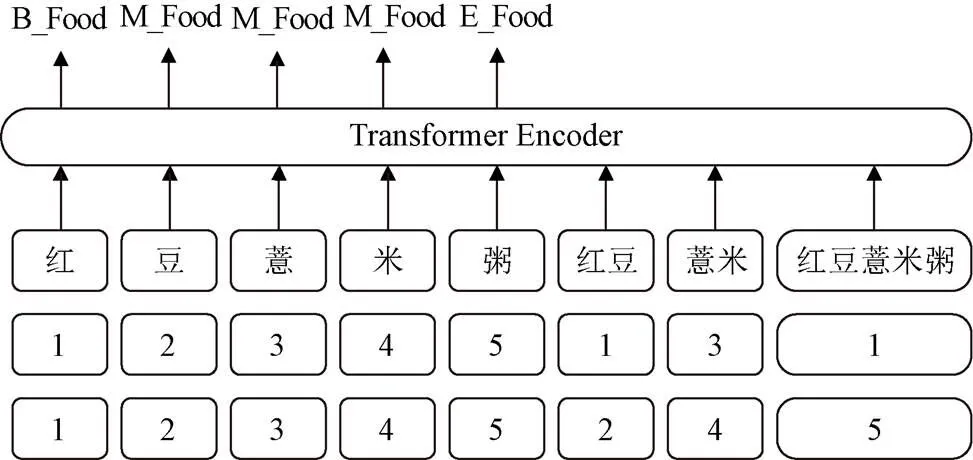
注:Transformer Encoder表示Transformer模型编码器;B_Food、M_Food、E_Food分别对应食物类型实体的开始位置、中间位置和结束位置。
Note: Transformer Encoder represents the encoder of the Transformer; B_Food, M_Food, E_Food represent the begin, middle, and end positions of the food type entity respectively.
图4 字符嵌入和词汇嵌入
Fig.4 Character embedding and vocabulary embedding
根据每个区间的头位置和尾位置,可将区间间的关系划分为包含、相交、相离。为表示3种区间关系,首先通过式(1)~式(4)构建区间的相对位置矩阵。

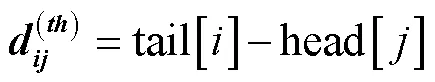
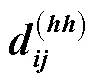
然后将4个相对位置矩阵拼接,计算区间x、x相对位置编码,如式(5)所示。

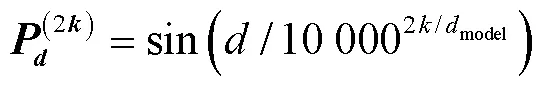
式中表示输入区间相对距离,表示位置编码的维度索引,model表示多头注意力机制中映射的向量维度。


1.2.4 解码层

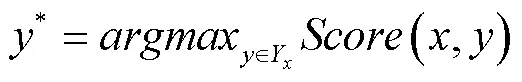

1.2.5 规则修正
由于营养健康领域中存在大量的复杂实体且实体规则性较强,本研究制定了相应的规则对BERT-FLAT模型的初步预测结果进行修正,具体的修正规则如下:
1)食物类型实体:部分食物名称存在实体重叠现象,设定规则当食物实体中前后紧邻食物词汇、烹饪词汇、表示颜色的词汇等,将其共同修正为食物实体;
2)营养物质类型实体:食物中含有的营养物质名称,如“类胡萝卜素”,模型将其预测为“O|B_FOOD| M_FOOD|E_FOOD|O”和“O|B_FOOD| M_FOOD| M_FOOD|E_FOOD”,没有对营养物质正确预测,因此设定规则将食物词汇周围出现的营养词汇,合并为营养物质实体;
3)病症类型实体:对于病症类型的实体,若紧邻词汇中出现了人群、器官等,则将其修正为病症类型实体;
4)器官类型实体:对于器官类型的实体,若紧邻词汇中出现了器官,则将其修正为器官类型的实体。
在规则修正过程中,定义滑动窗口大小为1,以关键词为中心,对上下文搜索1个实体,即若相邻预测标签为O,则判断是否为特征词汇,将特征词汇合并为相关实体,若相邻预测标签为实体词汇,则查找对应规则合并为相关实体,营养文本规则如表4所示。
1.3 试验设计
1.3.1 试验语料
语料标注的主要来源是从分句结果中选取与食物、营养物质、病症等相关的文本共计24 000句,如西红柿、红豆薏米粥、补血、膳食纤维、胡萝卜素等。将标注数据集按照8∶2的比例划分为训练集与测试集进行试验。
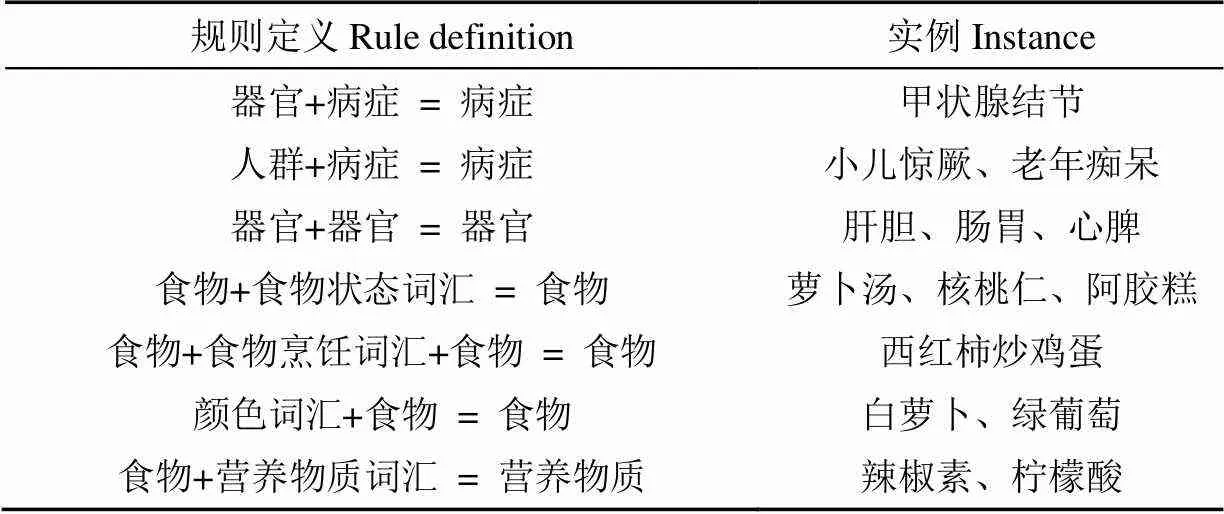
表4 营养文本规则
注:+表示连接相邻的实体类型;=表示连接后形成的新实体类型。
Note: + indicates the type of entities that are connected adjacent to each other; = indicates the type of new entities formed after the connection.
1.3.2 评价指标
在对比不同模型的能力时,需要采用相同的性能度量。命名实体识别任务中常使用准确率、召回率和1分数作为度量标准,并且1分数能够综合评价模型的识别性能[31]。本研究的目标是通过融合规则与BERT-FLAT模型自动识别文本中的实体,为构建营养健康领域知识图谱提供数据支撑,因此使用准确率、召回率和1分数衡量实体抽取效果,3种指标根据式(11)~式(13)计算。

式中表示精确率,表示召回率,1表示1分数。
1.3.3 试验参数
神经网络模型中超参数的设置对模型的性能有很大的影响,学习率用于控制权值的更新速度,加快模型训练并提升识别能力。优化器用来更新和计算影响模型训练和输出的网络参数,使其逼近或达到最优值[32],动量用以控制梯度下降的速度。权重衰减、丢失率、多头注意力机制3个参数会影响模型的收敛速度,需要设置合适的值以防模型出现过拟合。通过对数据集进行训练,融合规则与BERT-FLAT模型的参数设置如表5所示。

表5 融合规则与BERET-FLAT模型的参数设置
2 结果与分析
2.1 预试验
验证所使用的相对位置编码和外部词表引入的有效性,对BERT+BiLSTM+CRF模型和融入位置信息的BERT+BiLSTM+CRF模型进行了试验。融合位置信息的BERT+BiLSTM+CRF是对输入序列中单字标识为0,多字词开始位置标识为1,中间位置标识为2,结束位置标识为3,然后将输入文本序列的词汇相对位置编码信息与BERT模型输出进行拼接,试验结果如表6所示。从表6中可以看出在加入位置信息后,对营养健康领域命名实体识别的准确率、召回率和1分数分别为86.56%、91.01%和88.72%,与BERT+BiLSTM+CRF模型相比,准确率、召回率和1分数分别提升了1.55、0.20、0.32个百分点,说明位置信息的引入对营养健康领域命名实体识别任务有提升作用。
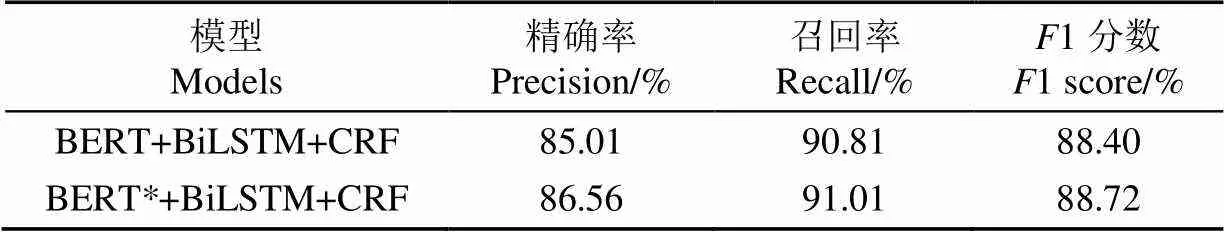
表6 预试验识别效果
注:BERT*+BiLSTM+CRF表示融合位置信息的BERT+BiLSTM+CRF模型。
Note: BERT*+BiLSTM+CRF represents the BERT+BiLSTM+CRF model fused with location information.
2.2 模型性能比较分析
使用精确率、召回率和F1分数三值作为评价指标,首先在BERT+BiLSTM+CRF[25]、迭代膨胀卷积神经网络[33](Iterated dilated convolutional neural networks,IDCNN)、BiLSTM+CRF[34]、FLAT等几个主流的命名实体识别模型进行了对比试验,试验结果如表7所示。

表7 不同模型的命名实体识别效果
注:IDCNN2和IDCNN的区别在于卷积层和全连接层的参数不同。
Note: The difference between IDCNN2 and IDCNN is the parameters of the convolutional layer and the fully connected layer.
从表8中可以看出,在IDCNN-CRF、IDCNN2-CRF、BiLSMT-CRF、BiLSTM-Attention-CRF、FLAT、BERT- FLAT中BERT-FLAT模型取得了最优结果,准确率、召回率、F1分数分别为85.89%、92.33%、88.99%,说明使用BERT预训练语言模型结合字词融合模型能够有效的对实体进行识别。BERT-FLAT模型性能提升的主要原因在于模型中引入的词汇、字符的相对位置关系有利于定位实体,其次引入词嵌入作为先验知识融入到模型中有利于实体的分类。在BERT-FLAT模型的基础上增加了营养领域文本规则修正解码序列,融合规则与BERT-FLAT模型具有很好的识别效果,虽然召回率低于BERT-FLAT模型,其主要原因在于部分正确识别的实体由于规则的引入调整为错误实体,导致召回率偏高,但是准确率和1分数分别提升了9.11和2.82个百分点,因此,通过试验证明了规则的加入在一定程度上对融合规则与BERT-FLAT模型的预测结果进行了优化,对部分错误标签实现了修正功能,且修正的数量大于干扰的数量。
2.3 识别详情分析
融合规则与BERT-FLAT模型在营养健康领域文本中各实体类别精确率、召回率、1分数如表8所示。从表中可以看出,无论在准确率、召回率还是1分数方面,食物类型实体相比于其他实体类型识别性能较低,主要包含了2个原因:1)食物实体类型中包含了大部分的嵌套实体,导致实体识别的不完全;2)由于规则的制定,导致词汇融合错误,干扰了模型的识别性能。除食物类型实体,其他实体类型的精确率、召回率和1分数大部分在90%以上,且人群实体准确率和1分数达到了97.68%和95.16%,说明本模型整体识别性能较好。
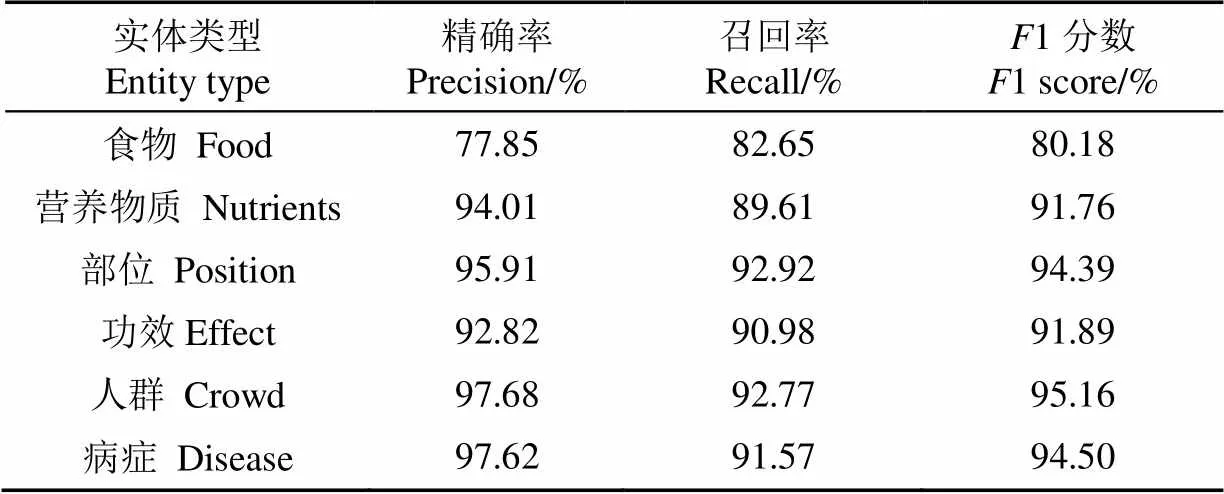
表8 融合规则与BERT-FLAT模型各类实体识别效果
3 结 论
1)本研究针对人类营养健康领域含有大量复杂实体且文本序列较长问题,提出了将字符信息与词汇信息融合的方法,结合外部此表为文本中的词汇构建位置向量,并通过预试验证明了词表及位置特征对于命名实体任务的有效性。
2)针对营养健康领域实体识别准确率不高的问题,根据营养健康文本特征,定义7种营养文本规则,并设计了融合规则与BERT-FLAT模型的营养健康命名实体识别方法,该方法通过BERT模型进行预训练,并借助位置向量定位实体位置,实现字符与词汇的交互,使用自注意力机制捕捉长距离依赖,通过规则后处理的方式,对解码序列进行修正。相比于BERT-FLAT模型,本研究提出的融合规则与BERT-FLAT模型的识别效果有了明显的提升,1分数为91.81%,解决了营养健康领域实体识别准确率不高的问题,是一种有效的人类营养健康领域实体识别方法,可以为农业、医疗、食品安全等其他领域复杂命名实体识别提供新思路。
虽然本研究提出的融合规则与BERT-FLAT模型证明在人类营养健康领域命名实体识别任务中有效,但是在提高食物类型实体识别准确度、复杂实体抽取、规则处理等方面仍有提升空间。
[1] 周旻. 中国特膳食品发展现状及建议[J]. 合作经济与科技,2021(14):52-54.
Zhou Min. Development status and suggestions of special food in China[J]. CO-Oerativeconomy & Science, 2021(14): 52-54. (in Chinese with English abstract)
[2] 任发政. 乳的营养与健康[J]. 中国食品学报,2020,20(7):1-9.
Ren Fazheng. Advances in milk nutrition and human health[J]. Journal of Chinese Institute of Food Science and Technology, 2020, 20(7): 1-9. (in Chinese with English abstract)
[3] 喻兵. 基于多维度特征的饮食干预方法研究[D]. 湘潭:湘潭大学,2019.
Yu Bing. Research on Dietary Intervention Bases on Multi-dimensional Characteristics[D]. Xiangtan: Xiangtan University, 2019. (in Chinese with English abstract)
[4] 董洪伟. 基于知识图谱的菜品推荐系统[D]. 北京:北京林业大学,2020.
Dong Hongwei. Dish Recommendation System Based on Knowledge Graph[D]. Beijing: Beijing Forestry University, 2020. (in Chinese with English abstract)
[5] 金碧漪. 基于多源UGC数据的健康领域知识图谱构建[D]. 上海:华东师范大学,2016.
Jin Biyi. Construction of Health Knowledge Graph Based on Multi-source UGC data[D]. Shanghai: East China Normal Unversity, 2016. (in Chinese with English abstract)
[6] 任媛,于红,杨鹤,等. 融合注意力机制与BERT+BiLSTM+CRF模型的渔业标准定量指标识别[J]. 农业工程学报,2021,37(10):135-141.
Ren Yuan, Yu Hong, Yang He, et al. Recognition of quantitative indicator of fishery standard using attention mechanism and the BERT+BiLSTM+CRF model[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2021, 37(10): 135-141. (in Chinese with English abstract)
[7] 檀稳. 基于深度学习的植物知识图谱的构建[D]. 北京:北京林业大学,2019.
Tan Wen. The Construction of Plant Knowledge Graph Based on Deep Learning[D]. Beijing: Beijing Forestry University, 2020. (in Chinese with English abstract)
[8] 王蓬辉,李明正,李思. 基于数据增强的中文医疗命名实体识别[J]. 北京邮电大学学报:2020,43(5):84-90.
Wang Penghui, Li Mingzheng, Li Si. Data augmentatoin for Chinese clinical named entity recognition[J]. Journal of Beijing University of Posts and Telecommunications, 2020, 43(5): 84-90. (in Chinese with English abstract)
[9] 吴赛赛,周爱莲,谢能付,等. 基于深度学习的作物病虫害可视化知识图谱构建[J]. 农业工程学报,2020,36(24):177-185.
Wu Saisai, Zhou Ailian, Xie Nengfu, et al. Construction of visualization domain-specific knowledge graph of crop diseases and pests based on deep learning[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2020, 36(24): 177-185. (in Chinese with English abstract)
[10] 杨培,杨志豪,罗凌,等. 基于注意机制的化学药物命名实体识别[J]. 计算机研究与发展,2018,55(7):1548-1556.
Yang Pei, Yang Zhihao, Luo Ling, et al. An attention-based approach for chemical compound and drug named entity recognition[J]. Journal of Computer Research and Development, 2018, 55(7): 1548-1556. (in Chinese with English abstract)
[11] Li L, Hou L. Named entity recognition in Chinese electronic medical records based on the model of bidirectional long short-term memory with a conditional random field Layer[J]. Studies in Health Technology and Informatics, 2019, 264: 1524-1525.
[12] 张晗,郭渊博,李涛. 结合GAN与BiLSTM-Attention-CRF的领域命名实体识别[J]. 计算机研究与发展,2019,56(9):1851-1858.
Zhang Han, Guo Yuanbo, Li Tao. Domain named entity recognition combining GAN and BiLSTM-Attention-CRF[J]. Journal of Computer Research and Development, 2019, 56(9): 1851-1858. (in Chinese with English abstract)
[13] 董哲,邵若琦,陈玉梁,等. 基于BERT和对抗训练的食品领域命名实体识别[J]. 计算机科学,2021,48(5):247-253.
Dong Zhe, Shao Ruoqi, Chen Yuliang, et al. Named entity recognition in food field based on BERT and adversarial training[J]. Computer Science, 2021, 48(5): 247-253. (in Chinese with English abstract)
[14] 王璐. 基于知识图谱的健康膳食知识智能问答系统[D]. 兰州:兰州大学,2020.
Wang Lu. A Healthy Diet Knowledge Q/A system Based on Knowledge Graph[D]. Lanzhou: Lanzhou University, 2020. (in Chinese with English abstract)
[15] 迟杨. 健康饮食领域知识图谱构建与应用研究[D]. 长春:吉林大学,2019.
Chi Yang. Knowledge Graph Construction and Application in Healthy Diet Domain[D]. Changchun: Jilin University, 2019. (in Chinese with English abstract)
[16] 武乐飞. 基于边界的嵌套命名实体识别方法研究[D]. 贵阳:贵州大学,2020.
Wu Lefei. Recreach on Recognition Method of Nested Named Entity Based on Boundary[D]. Guiyang: Guizhou University, 2020. (in Chinese with English abstract)
[17] Yue Z, Jie Y. Chinese NER using lattice LSTM[C]//The 56thAnnual Meeting of the Association for Computational Linguistics (ACL). Melbourne, AUS: Melbourne Convention and Exhibition Centre, 2018.
[18] Sui D, Chen Y, Liu K, et al. Leverage lexical knowledge for Chinese named entity recognition via collaborative graph network[C]//Proceedings of the 2019 Conference on Empirical Methods in Natural Language Processing and the 9th International Joint Conference on Natural Language Processing (EMNLP-IJCNLP). Hong Kong, China: World Expo, 2019.
[19] Gui T, Ma R, Zhang Q, et al. CNN-based Chinese NER with lexicon rethinking[C]//Twenty-Eighth International Joint Conference on Artificial Intelligence IJCAI-19. Macao, China: Venice Macao people Conference Center, 2019.
[20] Li X, Yan H, Qiu X, et al. FLAT: Chinese NER using flat-lattice transformer[C]//Proceedings of the 58thAnnual Meeting of the Association for Computational Linguistics. 2020.
[21] 中国营养学会. 中国居民膳食营养素参考摄入量速查手册:2013版[M]. 北京:中国标准出版社,2014.
[22] Wu Y, Zhang F, Yang K, et al. SymMap: An integrative database of traditional Chinese medicine enhanced by symptom mapping[J]. Nuclc Acids Research, 2018(D1): D1110-D1117.
[23] 钟友林. 营养学领域的知识抽取相关技术研究[D]. 哈尔滨:哈尔滨工业大学,2019.
Zhong Youlin. Research on Technologies of Knowledge Extraction in Nutrition[D]. Harbin: Harbin Institute of Technology, 2019. (in Chinese with English abstract)
[24] 郭旭超,唐詹,刁磊,等. 基于部首嵌入和注意力机制的病虫害命名实体识别[J]. 农业机械学报,2020,51(Supp2):335-343.
Guo Xuchao, Tang Zhan, Diao Lei, et al. Recognition of Chinese agricultural diseases and pests named entity with joint radical embedding and self-attention mechanism[J]. Transactions of the Chinese Society for Agricultural Machinery, 2020, 51(Supp2): 335-343. (in Chinese with English abstract)
[25] Devlin J, Chang M-W, Lee K, et al. BERT: Pre-training of deep bidirectional transformers for language understanding[J]// Melbourne, AUS: Melbourne Convention and Exhibition Centre, 2018.
[26] 斋藤康毅. 深度学习进阶:自然语言处理,陆宇杰译[M]. 北京:人民邮电出版社,2020. 10.
[27] Mikolov T, Sutskever I, Chen K, et al. Distributed representations of words and phrases and their compositionality[C]//Advances in neural information processing systems, Lake Tahoe, US: MIT Press, 2013.
[28] Dai Z, Yang Z, Yang Y, et al. Transformer-XL: Attentive Language Models beyond a Fixed-Length Context[C]//Proceedings of the 57thAnnual Meeting of the Association for Computational Linguistics. Florence, Italy, 2019.
[29] 王乃钰,叶育鑫,刘露,等. 基于深度学习的语言模型研究进展[J]. 软件学报,2021,32(4):1082-1115.
Wang Naiyu, Ye Yuxin, Liu Lu, et al. Language models based on deep learning: a review[J]. Journal of Software, 2021, 32(4): 1082-1115. (in Chinese with English abstract)
[30] 王海宁,周菊香,徐天伟. 融合深度学习与规则的民族工艺品领域命名实体识别[J]. 云南师范大学学报:自然科学版,2020,40(2):48-54.
Wang Haining, Zhou Juxiang, Xu Tianwei. Named entity recogniton in ethnic handicraft field with the deep learning and rules[J]. Journal of Yunnan Normal University: Natural Sciences Edition, 2020, 40(2): 48-54. (in Chinese with English abstract)
[31] 李明扬,孔芳. 融入自注意力机制的社交媒体命名实体识别[J]. 清华大学学报:自然科学版,2019,59(6):461-467.
Li Mingyang, Kong Fang. Combined self-attention mechanism for named entity recognition in social media[J]. Journal of Tsinghua University: Science and Technology, 2019, 59(6): 461-467. (in Chinese with English abstract)
[32] 刘全,翟建伟,章宗长,等. 深度强化学习综述[J]. 计算机学报,2018,41(1):1-27.
Liu Quan, Zhai Jianwei, Zhang Zongzhang, et al. A survey on deep reinforcement learning[J]. Chinese Journal of Computers, 2018, 41(1): 1-27. (in Chinese with English abstract)
[33] Strubell E, Verga P, Belanger D, et al. Fast and Accurate Entity Recognition with Iterated Dilated Convolutions[M]//Association for Computational Linguistics, Copenhagen, Denmark, 2017: 2670-2680.
[34] Huang Z, Wei X, Kai Y. Bidirectional LSTM-CRF Models for Sequence Tagging[J]. Computer Science, 2015, 20(2): 508-517.
Named entity recognition in human nutrition and health domain using rule and BERT-FLAT
Zheng Limin1, Ren Lele2
(1.,100083,; 2.,,100083,)
A nutritious and healthy diet can be widely expected to reduce the incidence of disease, while improving body health after the disease occurs. The nutritional diet knowledge can be acquired mostly through the Internet in recent years. However, reliable and integrated information is highly difficult to discern using time-consuming searching of the huge amount of Internet data. It is an urgent need to integrate the complicated data, and then construct the knowledge graph of nutrition and health, particularly with timely and accurate feedback. Among them, a key step is to accurately identify entities in nutritional health texts, providing effective location data support to the construction of knowledge graphs. In this study, a BRET+BiLSTM+CRF (Bidirectional Encoder Representations from Transformers + Bi-directional Long Short-Term Memory + Conditional Random Field) model was first used with location information. It was found that the precision of the model was 86.56%, the recall rate was 91.01%, and the F1 score was 88.72%, compared with the model without location information, indicating improved by 1.55, 0.20, and 0.32 percentage points. A named entity recognition was also proposed to accurately obtain six types of entities in text: food, nutrients, population, location, disease, and efficacy in the field of human nutritional health, combining rules with BERT-FLAT (Bidirectional Encoder Representations from Transformers-Flat Lattice Transformer) model. Firstly, the character and vocabulary information were stitched together and pre-trained in the BERT model to improve the recognition ability of the model to entity categories. Then, a position code was created for the head and tail position of each character and vocabulary, where the entity position was located with the help of a position vector, in order to improve the recognition of entity boundary. A long-distance dependency was also captured using the Transformer model. Specifically, the output of the BERT model was embedded into the Transformer as a character-embedding conjunction word, thus for the character-vocabulary fusion. Then the text prediction sequence was obtained from the CRF layer. Finally, seven rules were formulated, according to the text characteristics in the field of nutrition and health, where the prediction sequence was modified according to the rules. The experimental results showed that the F1 score of the BERT-FLAT model was 88.99%. The BERT model combined with the word fusion performed the best, compared with that without the Bert model, indicating an effective recognition performance. Correspondingly, the named entity recognition model in the field of nutrition and health using fusion rules and the BERT-FLAT model presented an accuracy rate of 95.00%, a recall rate of 88.88%, and an F1 score of 91.81%. The F1 score increased by 2.82 percentage points than before. The finding can provide an effective entity recognition in the field of human nutrition and health.
nutrition; health; food; named entity recognition; self-attention mechanism; BERT model; transformer model
郑丽敏,任乐乐. 采用融合规则与BERT-FLAT模型对营养健康领域命名实体识别[J]. 农业工程学报,2021,37(20):211-218.doi:10.11975/j.issn.1002-6819.2021.20.024 http://www.tcsae.org
Zheng Limin, Ren Lele. Named entity recognition in human nutrition and health domain using rule and BERT-FLAT[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2021, 37(20): 211-218. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2021.20.024 http://www.tcsae.org
2021-02-20
2021-08-10
现代农业产业技术体系北京市生猪产业创新团队项目(BAIC02-2021);国家重点研发计划(2017YFC1601803)
郑丽敏,教授,硕士生导师,研究方向为人工智能、知识图谱等。Email:zhenglimin@cau.edu.cn
10.11975/j.issn.1002-6819.2021.20.024
TP391.1
A
1002-6819(2021)-20-0211-08

