稻瘟病菌中小G蛋白Rho3的假定互作蛋白MoKin1的功能分析
蔡燕 林婕 何金 杨福长 张冬梅 张连虎 王宗华




摘 要:小G蛋白MoRho3在稻瘟病菌生命活动中具有重要的生物学功能。前期研究表明,MoRho3的缺失对于稻瘟病菌分生孢子的形态建设、附着胞萌发和侵入能力以及对宿主的致病性等表型都存在严重缺陷。为了进一步解析MoRho3在稻瘟病菌中的网络调控途径,通过生物信息学的方法预测了MoRho3的互作蛋白。在众多假定互作蛋白中,蛋白激酶MoKin1与酿酒酵母中KIN1和KIN2是同源蛋白。在酵母细胞中,KIN1和KIN2可以抑制△rho3的表型缺陷。为了验证在稻瘟病菌中MoKin1与MoRho3之间是否存在着相似的调控途径,本研究通过分析MoKin1与同源蛋白的进化关系,序列结构,定位状态以及相关的互作蛋白,揭示了MoKin1的功能特点,为后续病原菌中MoKin1与MoRho3互作机制的研究,以及病原菌致病机制的解析提供了新的思路和方向。
关键词:稻瘟病菌;MoRho3;MoKin1;互作分析
中图分类号:S432.4+4 文献标识码:A
Abstract: Small GTPase MoRho3 plays an important biological role in the rice blast fungus, Magnaporthe oryzae. The preamble research elucidated that the deletion of MoRho3 in this fungus caused serve phenotypic defects, such as ab-normal morphology of conidia, reduction of germination and penetration of appressoria and reduced pathogenicity. In order to understand the mechanism of the regulatory pathways of MoRho3, we adopted the bioinformatics to search the putative interacting protein of MoRho3. We found the homologus protein of Saccharomyces cerevisiae KIN1 and KIN2 in the proteins, named MoKin1. Because the over-expression of KIN1 and KIN2 could rescue some phenotypic defects of △rho3 in S. cerevisiae, in order to verify whether there was a similar regulatory pathway between Mokin1 and MoRho3 in M. oryzae, the study revealed the functional characteristics of Mokin1 by analyzing the evolutionary relationship, sequence structure, localization status and related interaction proteins, which would provide a new basis for the further study of the interaction between Mokin1 and MoRho3 and the pathogenic mechanism of M. oryzae.
Keywords: Magnaporthe oryzae; MoRho3; MoKin1; interaction analysis
DOI: 10.3969/j.issn.1000-2561.2021.11.026
稻瘟病菌(Magnaporthe oryzae)引起的稻瘟病(rice blast disease)是水稻生产中最重要的真菌病害之一。每年引起水稻产量的损失数以亿计,造成严重的粮食危害[1]。因此,稻瘟病的防控是水稻生产上最重要的任务。水稻与稻瘟病菌的互作机制是植物与病原物互作的经典模型之一,推动了植物病原真菌致病机制和寄主植物抗病机制的研究[2]。目前,主要从水稻的抗病和稻瘟病菌的致病两个方面进行研究,为水稻抗病品种的培育以及新型杀菌剂的研发提供理论方面的指导。
稻瘟病菌的分生孢子随着风雨的传播,散落在健康的水稻叶表皮。孢子分泌出粘液(spore tip mucilage)使其粘附在叶表皮[3]。在湿度适宜的情况下,孢子萌发,产生芽管。随后,芽管顶端膨大形成侵染性结构附着胞(appressorium)。附着胞内有甘油等,形成巨大的机械膨压[4]。经与水稻相互识别后,附着胞底部形成侵染栓(penetr¬ation peg),刺透水稻叶表皮层,进入水稻细胞,形成侵染菌丝(infection hypha),完成侵染[5]。稻瘟病菌的整个侵染过程受到一系列的信号调控,如cAMP途径和MAPK途径等[6]。此外,小G蛋白家族在稻瘟病菌分生孢子的產生和附着胞的侵入等过程中起着重要的作用[7]。
稻瘟病菌小G蛋白Rho家族有Rac1、Cdc42、Rho1、Rho2、Rho3和Rho4共6个成员,它们承担着重要的生物学功能。如Rac1分别与Chm1和Nox家族互作影响了病原菌的分生孢子形态建设以及致病性的产生[8];Cdc42的敲除也降低了病原菌的侵入能力和致病性[9];Rho2参与了cAMP途径的调控,能有效的影响附着胞的形成[10]。
前期研究表明Rho3的敲除使稻瘟病菌产生重要的表型缺陷:分生孢子畸形,附着胞侵染能力下降,致病性降低等。同时Rho3-DN(dominant negative,显性失活突变型)突变体和Rho3-CA (constitutively active,组成性激活突变型)突变体均表现出与野生型不同的表型[11],说明小G蛋白Rho3对稻瘟病菌致病机制的调控极其重要。解析Rho3的网络调控对于理解稻瘟病菌产孢和致病性形成具有重要的意义。同时,也将为寻求可能的药物靶标位点、生物农药的开发和病害的防控提供理论上的支撑。
Kin1/PAR-1/MARK家族属于丝氨酸/苏氨酸蛋白激酶[12]。从单细胞酿酒酵母到哺乳动物,Kin1/PAR-1/MARK激酶家族成员非常保守,具有相同的结构域,包括N端的丝氨酸/苏氨酸催化结构域(S_TKc)和C端的40个左右的氨基酸组成的调控结构域(KA1)[13]。某些成员在邻近催化结构域的位置有一个UBA结构域,其功能并不清楚,但是Rad23 UBA结构域能够结合泛素,参与蛋白酶体调控的蛋白水解[14]。Kin1/PAR-1/MARK激酶家族成员的功能具有多样性,包括细胞极性、微管稳定性、蛋白稳定性、胞外信号和细胞周期调控等[15]。
在秀丽隐杆线虫(Caenorhabditis elegans)中有6个编码PAR蛋白的基因:PAR1-6,其中PAR-1主要参与调控细胞极性以及细胞的不对称分裂[16]。酿酒酵母(Saccharomyces cerevisiae)中KIN1和KIN2是秀丽隐杆线虫中PAR-1的同源物[17]。研究表明,酿酒酵母中过表达KIN1和KIN2可以明显的恢复Rho3缺失突变体的表型缺陷,并且可以使rho3-V51突变体在14 ℃培养下正常存活[18]。同样的分泌型缺陷突变体cdc42-6也可恢复表型缺陷,说明蛋白激酶KIN1和KIN2在小G蛋白Rho3和Cdc42调控的下游途径中起作用[19]。因此我们在稻瘟病菌中寻找了酿酒酵母KIN1和KIN2的同源蛋白,命名为MoKin1,研究MoKin1是否可以功能互补MoRho3缺失造成的表型缺陷。
本研究对MoKin1的结构特点,细胞定位以及互作蛋白进行初步的分析,为后续开展MoKin1的功能鉴定提供一个基础,同时也为开展MoKin1和MoRho3的互作分析以及解析病原菌的致病机制提供一个方向。
1 材料与方法
1.1 材料
稻瘟病菌(M. oryzae)菌株Ku80在本实验室保存。
1.2 方法
1.2.1 稻瘟病菌MoRho3假定互作蛋白的预测 本研究利用酿酒酵母数据库(www.yeastgenome.org)中与Rho3之间具有physical interaction(物理相互作用)和genetic interaction(基因遗传互作)关系的蛋白序列在稻瘟病菌Ku80的基因组中进行序列比对,查找稻瘟病菌中的同源蛋白,得到蛋白序列。然后再根据NCBI(www.ncbi.nlm.nih.gov)中的注释进行整理,视为稻瘟病菌MoRho3的假定互作蛋白。
1.2.2 稻瘟病菌中MoRho3与MoKin1的关系 根据MoKin1的基因序列,构建MoKin1-AD载体,用于酵母双杂交实验。所用的载体是pGADT7,引物序列分别是:上游引物5'-TCCCCCGGGGATG TCCAGTAGACCGTCGTCAT-3'和下游引物5'-CG CGGATCCCTAGAGTTTAAGCTCCTTCAATA-3',酶切位点分别是SmaⅠ和BamHⅠ。同时我们构建了MoRho3-CA-BD和MoRho3-DN-BD载体。所用的载体是pGBKT7,引物序列是上游引物5'-CCG GGATCCGTATGCCTTTATCGCTTTGC-3和下游引物5-AACTGCAGCTACATGACGGTGCACTT- 3,酶切位点是BamHⅠ和PstⅠ。利用酵母双杂交的方法,验证稻瘟病菌中MoRho3-CA/ DN-BD与MoKin1-AD的互作关系。同时在3个突变体,MoRho3-KO,MoRho3-CA和MoRho3-DN,中检测MoKin1的表达量变化。首先提取野生型WT和3个突变体的RNA,反转录得到cDNA(反转录试剂盒使用的是TAKARA的PrimeScript RT reagent Kit with gDNA Eraser),然后使用TAKARA的荧光定量PCR试剂盒SYBR Premix ExTaq II完成qRT-PCR。根据得到的数值采用2–ΔΔCT的方法来计算MoKin1在野生型WT和3个突变体中的相对表达量变化。qRT-PCR中看家基因是β-tubulin(XP_368640),引物序列是5-GCTGTCC TCGTCGATCTCGA-3和5-CAGAGCAGGTCA GGTAACGA-3。检测MoKin1表达量的引物序列是5-GGAAGGAACAACCTGAG-3和5-GAAGC TCTTACGGATGG-3。
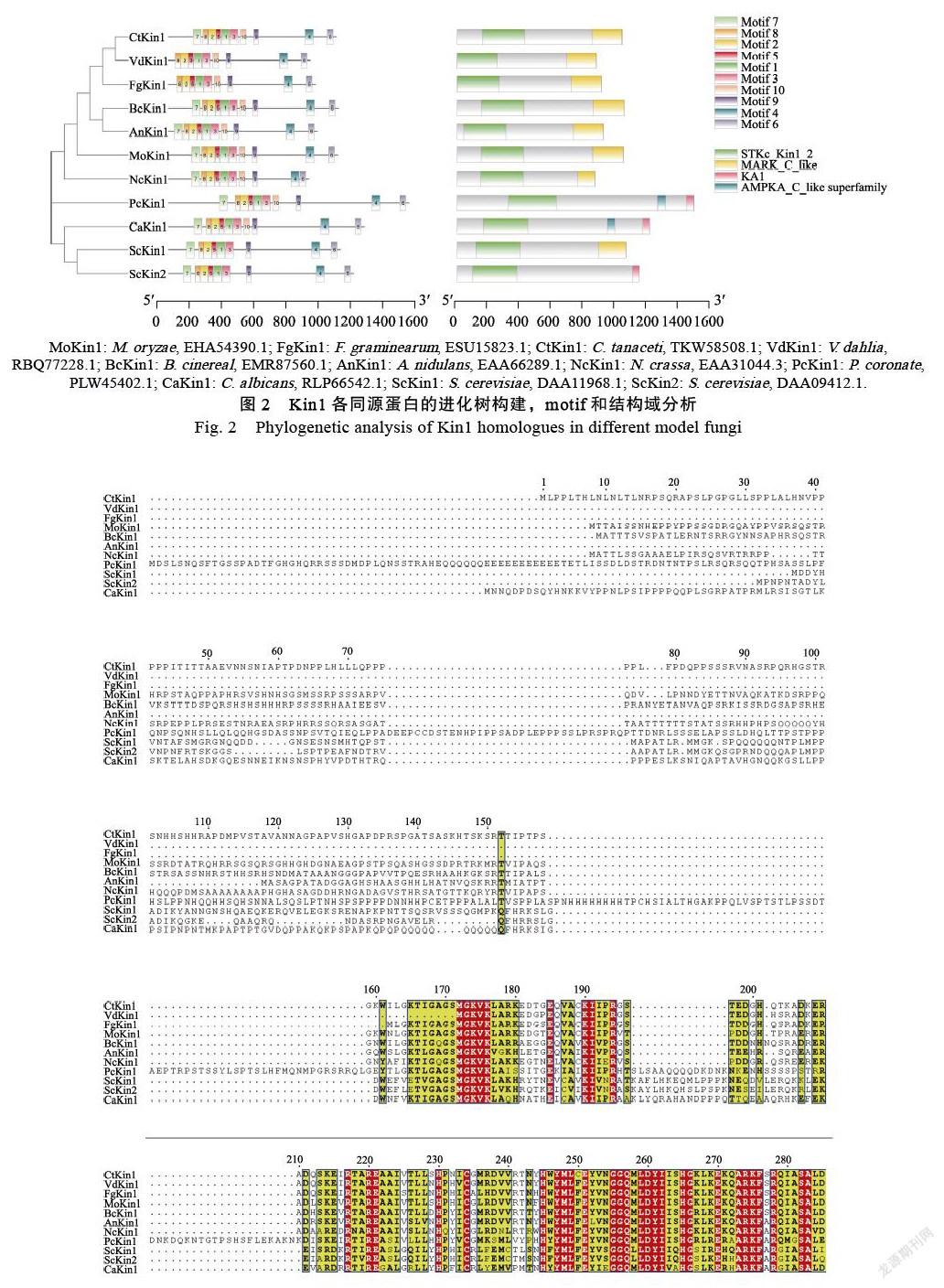
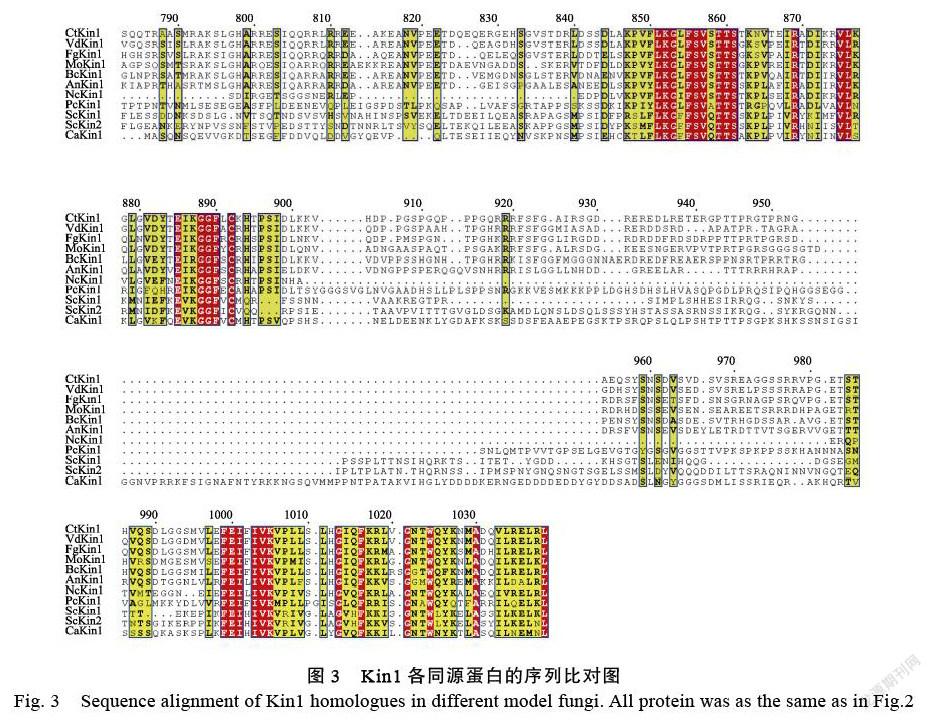
1.2.3 蛋白激酶MoKin1的结构特点及其进化分析 在NCBI(www.ncbi.nlm.nih.gov)进行BlastP搜索MoKin1在各种模式真菌中的同源蛋白,选取的模式真菌有禾谷鐮刀菌(Fusarium graminearum)、粗糙脉孢菌(Neurospora crassa)、灰葡萄孢(Botrytis cinerea)、酿酒酵母(S. cerevisiae)、构巢曲霉(Aspergillus nidulans)、炭疽菌(Colletotrichum tanaceti)、白色念珠菌(Candida albicans)、大丽轮枝菌(Verticillium dahliae)和禾柄锈菌(Puccinia coronata)。将上述得到的各模式真菌中Kin1同源蛋白的序列,在ESPript3.0(http://espript.ibcp.fr/ESPript/ESPript/index.php)在线软件中进行序列比对,所有参数设置成默认值,得到这些同源蛋白的序列比对图。同时利用MEGA7.0软件对Kin1的同源蛋白进行进化树分析,采用最大似然法(Maximum likelihood)构建系统发育树,并进行1000次的bootstrap的分析验证。
根据得到的各模式真菌中Kin1的同源蛋白,在MEME(https://meme-suite.org)在线软件中进行motif的分析;同时在NCBI(https://www. ncbi.nlm.nih.gov/ Structure/bwrpsb/bwrpsb.cgi)中进行蛋白结构域的预测分析。
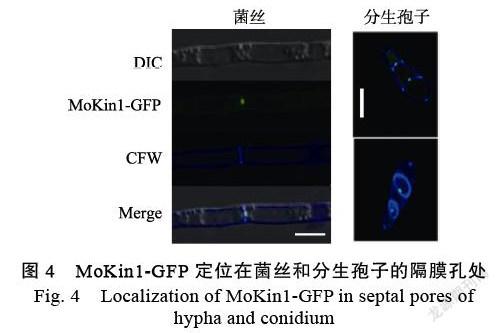
1.2.4 蛋白激酶MoKin1在稻瘟病菌中的定位分析 根据promoter 2.0(http://www.cbs.dtu.dk/services/ Promoter/)在线软件预测了稻瘟病菌MoKin1的启动子序列,然后设计引物构建MoKin1-GFP的定位载体Kin1G。使用的质粒是pKNTG,引物序列分别是:上游引物,5-TGGGCCCTTTAAGCA TTATG GCTTTAGG-3和下游引物,5-CCATC GATGAGTTTAAGCTCCTTCAATAT-3,所使用的酶切位点是ApaⅠ和ClaⅠ。将载体Kin1G转入到Ku80的原生质体中,使用G418作为筛选的抗生素,挑取阳性转化子,在激光共聚焦显微镜下观察MoKin1的定位情况。
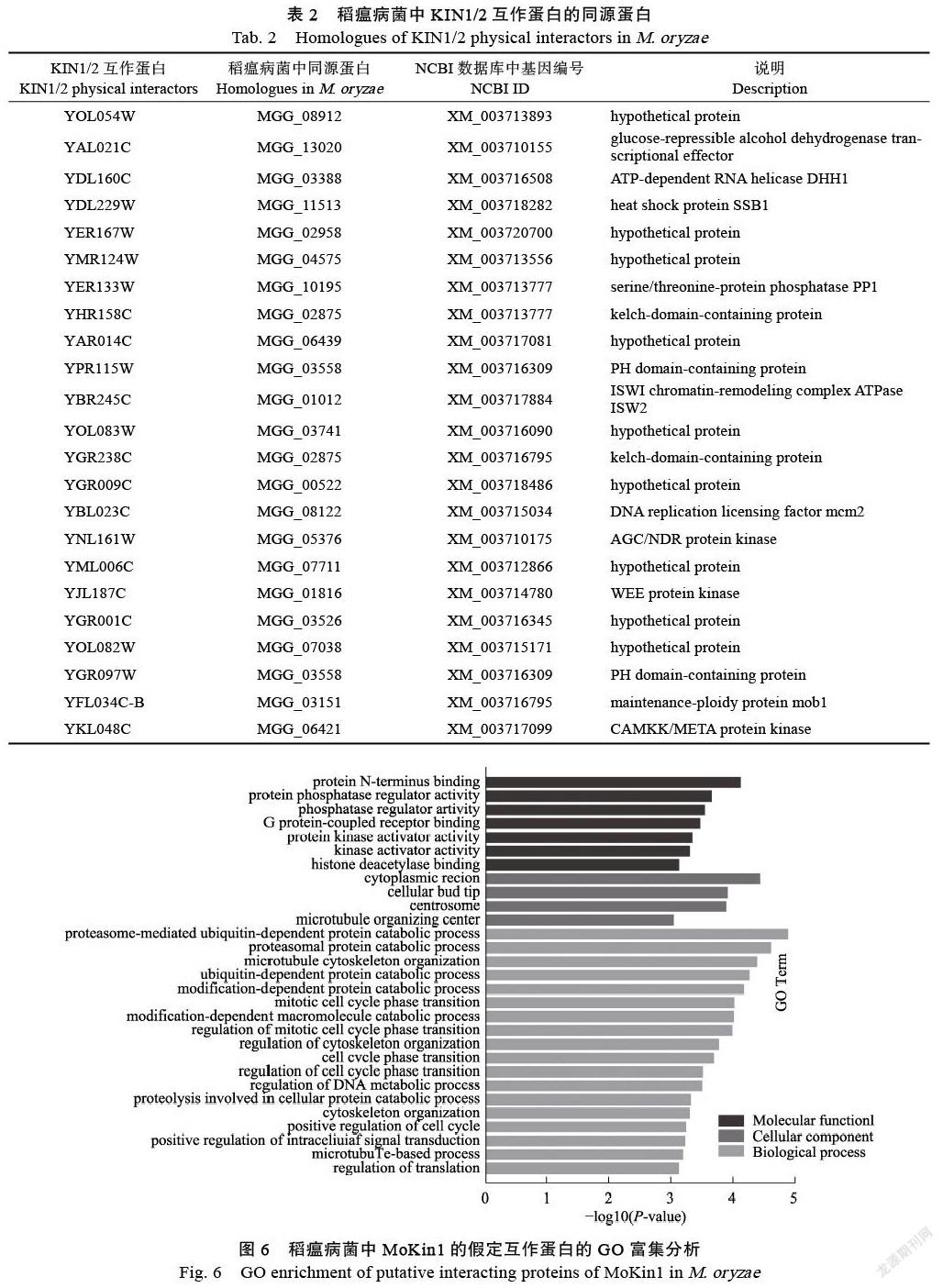
1.2.5 蛋白激酶MoKin1的互作蛋白分析 利用STRING(https://version11.string-db.org/)在线软件预测分析与稻瘟病菌MoKin1的互作蛋白。同时根据酵母数据库中KIN1和KIN2的physical和genetic互作蛋白,同源比对到稻瘟病菌中相应的蛋白序列,并进行简单的GO和KEGG分析,充分理解MoKin1的功能。
2 结果与分析
2.1 小G蛋白MoRho3的互作蛋白预测分析
利用酵母数据库中预测的Rho3的互作蛋白,在稻瘟病菌比对得到它们的同源蛋白。在酵母数据库(SGD)中,Rho3的互作蛋白分为physical interaction和genetic interaction。而且与Rho3具有genetic interaction的互作蛋白有224个(RHO3_genetic_interactors),这些蛋白参与了细胞中多种生命活动。此外,与Rho3具有physical interaction的蛋白有25个,与之相对应的稻瘟病菌中的同源蛋白有23个(表1)。通过分析这23个蛋白,最引起我们兴趣的是一系列的激酶,比如MGG_03696(CMGC/CK2 protein kinase),MGG_00446(casein kinase II subunit beta-1),MGG_04660(CMGC/CDK/CDK5 protein kinase),MGG_05199(protein kinase regulator Ste50)以及MGG_01279(CAMK/CAMKL/KIN1 protein kinase,命名为MoKin1)。Rho3作为开关分子,与细胞中激酶的互作丰富了稻瘟病菌中MoRho3调控的信号通路,对深入解析稻瘟病菌的致病机理提供了新的方向。
前期表明在酿酒酵母(S. cerevisiae)中,过表达Kin1/Kin2可以有效地抑制Rho3的缺失引起酵母细胞的表型缺陷[18]。因此在本研究将重点分析MoKin1的功能,为其后续的研究工作,如MoKin1和MoRho3的互作,MoKin1参与致病性产生等提供基础和方向。
2.2 稻瘟病菌中MoRho3与MoKin1的关系
为了明确MoKin1和MoRho3之间的关系,利用酵母双杂交的方法,检测MoKin1-AD和MoRho3-CA/DN-BD之间是否存在着直接互作关系,但是实验结果表明小G蛋白MoRho3和MoKin1之间没有明显的互作关系。为了推测二者之间是否存在遗传互作的关系。在突变体MoRho3- KO,MoRho3-CA和MoRho3-DN[11]中观察MoKin1的表达量变化。结果显示与野生型Ku80中相比,在3个突变体中,MoKin1的表达量均明显下降(图1)。说明MoRho3的不正常表达,影响了MoKin1正常生理功能,也说明了MoKin1参与了MoRho3的下游调控路径。
2.3 MoKin1与各个同源蛋白之间的进化关系以及蛋白结构分析
在NCBI中blast到MoKin1的同源蛋白,利用MEGA 7.0构建Kin1各同源蛋白间的ML进化树(图2),结合同源蛋白的序列比对图(图3),表明在各个丝状真菌中Kin1具有非常保守的区域。进一步分析Kin1同源蛋白的motif,表明各个Kin1同源蛋白具有相同的motif基序和顺序(VdKin1和FgKin1,缺少motif7),进一步的说明了Kin1的高度保守性。而且结构域显示丝状真菌的Kin1同源蛋白之间都包含有STKc_Kin1_2结构域和MARK_C_like结构域(图2),与序列比对图(图3)中序列保守的区域正好对应。
2.4 蛋白激酶MoKin1在稻瘟病菌中的定位分析
为了进一步的分析MoKin1的功能,将构建好的Kin1G载体转入到稻瘟病菌Ku80的原生质体中用于检测MoKin1在稻瘟病菌的定位。结果表明,MoKin1-GFP荧光主要集中在菌丝和分生孢子的隔膜孔处(图4)。
2.5 蛋白激酶MoKin1的假定互作蛋白分析
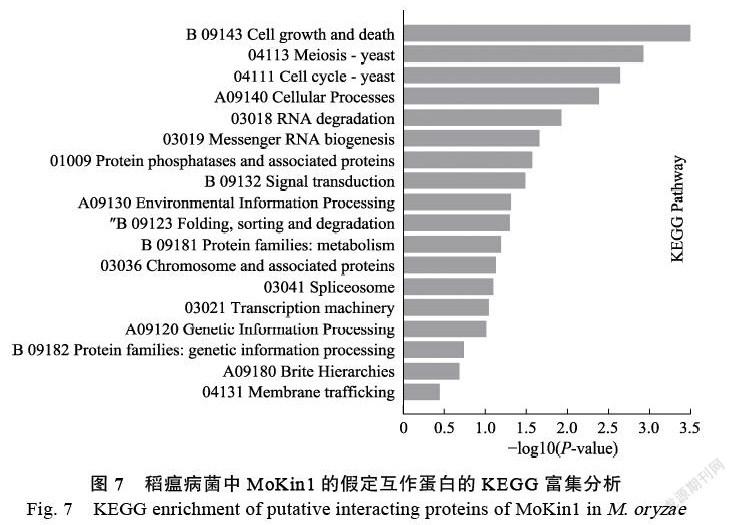
通过分析比对酵母数据库中KIN1/2的互作蛋白,在NCBI中blast稻瘟病菌的同源蛋白(图5)。酵母数据库中KIN1/2的互作蛋白分为physical interactors和genetic interactors。通过分析有23个蛋白与KIN1、KIN2均具有physical互作(图5, KIN1 physical interactors和KIN2 physical interactors共同含有的蛋白),表2所示相应的蛋白以及在稻瘟病菌中对应的22个同源蛋白。与此同时,这22个同源蛋白的GO富集(图6)和KEGG富集分析(图7)结果表明MoKin1互作蛋白主要參与了细胞生长发育、蛋白质磷酸化和信号转导等主要生化途径。
3 讨论
稻瘟病菌是水稻生产中最严重的病害,严重的影响了水稻的的产量和质量。对于防控病害的发生,通常采用种植抗病品种的方式。另一方面,研究致病病原的机理也对病害的防控起着理论指导的作用。前期的研究表明小G蛋白MoRho3病原菌的致病性起着重要的调控作用[11],因此理解MoRho3的精确功能有助于解析致病机理。本研究依据酵母数据库中Rho3的互作蛋白,预测了稻瘟病菌中MoRho3的一些互作蛋白。具有物理互作的同源蛋白有23个(表1),包含7个假定蛋白(蛋白编码基因是MGG_09923、MGG_ 03323、MGG_03048、MGG_06183、MGG_16213、MGG_05706、MGG_17697),4个细胞核内相关蛋白(蛋白编码基因是MGG_07120、MGG_ 01268、MGG_13020和MGG_06459),3个蛋白酶体相关蛋白(蛋白编码基因是MGG_07120、MGG_16706和MGG_ 04506),2个热激蛋白(编码基因是MGG_11513和MGG_06459),1个RNA-binding蛋白(编码基因MGG_14563),1个胞外分泌复合体的Exo70亚基(编码基因是MGG_01760)。除此之外,最引人兴趣的属于5个蛋白激酶,分别是构成MoCK2的2个亚基(MGG_03696和MGG_00446)[18],参与MAPK途径的MGG_05199[19],以及MGG_04660和MGG_01279(MoKin1)。通过分析这些互作蛋白,可以初步推断MoRho3参与很多的细胞生命活动途径,作为开关分子具有重要的调节功能。
相关文献研究表明在酿酒酵母中KIN1/KIN2作为一个抑制蛋白,可以恢复cdc42、Rho3的缺失所造成的表型缺陷[20-21],因此推测在稻瘟病菌中是否也存在着相似的网络调控路径?本文对KIN1/KIN2在稻瘟病菌中的同源蛋白MoKin1(MGG_01279)进行分析、预测和蛋白定位研究,为病原菌致病机制的解析提供新的方向和思路。
虽然酵母双杂交的实验表明MoRho3与MoKin之间没有明显的互作关系,但是qRT-PCR结果表明MoRho3与MoKin1之间具有明显的遗传互作关系。在MoRho3的各个突变体中,MoKin1的表达量明显降低,说明MoKin1受到MoRho3的影响,在MoRho3调控的下游途径中承担功能。这个结果也很好的佐证了酵母细胞中过表达的KIN1/2可以恢复Rho3的缺失造成的表型缺陷。
同时不同模式真菌中Kin1同源蛋白的进化树和蛋白序列的分析表明,Kin1蛋白在不同的真菌中具有非常高的保守性,且其蛋白结构包含有相同的motif基序和2个主要的结构域,分别是N端的STKc_Kin1_2结构域和C端的MARK_C_ like结构域,部分Kin1同源蛋白具有KAI结构域。其中S_TKc结构域(Catalytic domain of Kin1, Kin2, and similar Serine/Threonine Kinases)是激酶的主要活性中心,起着磷酸化作物底物的作用;而MARK_C_like结构域(C-terminal kinase associated domain 1 (KA1), a phospholipid binding domain, of microtubule affinity-regulating kinases, and similar domains)主要起调控作用,调节控制激酶的活性。
MoKin1蛋白的亚细胞定位分析表明,MoKin1- GFP的荧光主要集中在菌丝和分生孢子的隔膜孔处。MoKin1的定位与其他丝状真菌,如禾谷镰刀菌(F. graminearum),煙曲霉(Aspergillus fumigatus)中的定位相同,都定位在菌丝的隔膜孔处[22-23]。隔膜孔是稻瘟病菌菌丝细胞间物质交流和信息交流的主要通道,结合MoKin1定位正好在隔膜孔处,推测MoKin1可能具有关闭和打开隔膜孔的功能,影响着物质和信息的交流。在MoKin1的互作蛋白的GO富集分析中,蛋白富集在positive regulation of intracellular signal transduction,调控着细胞内的信号转导过程。
通过比对酵母细胞中KIN1/KIN2的互作蛋白,在稻瘟病菌中对MoKin1的假定互作蛋白做了初步的分析。韦恩图表明KIN1/KIN2的physical interactors有23个蛋白,表2中做了系统的汇总,这是后续试验重点分析的对象。通过GO和KEGG分析,表明MoKin1在稻瘟病菌的生长发育,蛋白磷酸化和信号转导中发挥着重要功能。这写功能与MoKin1的激酶特性以及隔膜孔定位具有一致性。
通过分析蛋白激酶MoKin1的功能,可以增加对小G蛋白调控途径的理解,同时进一步的分析MoKin1的互作蛋白的一些功能,可以丰富MoRho3-MoKin1的调控途径。在后续的研究中,将进一步的加深对该途径的研究,以期得到很好的生物农药靶标位点,为农业生产中稻瘟病的防控提供理论上的指导。
参考文献
[1] Fernandez J, Orth K. Rise of a cereal killer: the biology of Magnaporthe oryzae biotrophic growth[J]. Trends Microbiol, 2018, 26(7): 582-597.
[2] Dean R, Van Kan J A, Pretorius Z A, et al. The top 10 fungal pathogens in molecular plant pathology[J]. Molecular plant pathology, 2012, 13(4): 414-430.
[3] Hamer J E, Howard R J, Chumley F G, et al. A mechanism for surface attachment in spores of a plant pathogenic fun-gus[J]. Science, 1988, 239(4837): 288-290.
[4] Veneault-Fourrey C, Barooah M, Egan M, et al. Autophagic fungal cell death is necessary for infection by the rice blast fungus[J]. Science, 2006, 312(5773): 580-583.
[5] Dezwaan T M, Carroll A M., Valent B, et al. Magnaporthe grisea pth11p is a novel plasma membrane protein that me-diates appressorium differentiation in response to inductive substrate cues[J]. The Plant Cell, 1999, 11(10): 2013-2030.
[6] Xu J R, Hamer J E. MAP kinase and cAMP signaling regu-late infection structure formation and pathogenic growth in the rice blast fungus Magnaporthe grisea[J]. Genes and De-velopment, 1996, 10(21): 2696-2706.
[7] Zhou X, Zhao X, Xue C, et al. Bypassing both surface attachment and surface recognition requirements for appressorium formation by overactive ras signaling in Magnaporthe oryzae[J]. Molecular Plant-Microbe Interactions, 2014, 27(9): 996-1004.
[8] Chen J, Zheng W, Zheng S, et al. Rac1 is required for pa-thogenicity and Chm1-dependent conidiogenesis in rice fungal pathogen Magnaporthe grisea[J]. PLoS Pathogens, 2008, 4(11): e1000202.
[9] Zheng W, Zhao Z, Chen J, et al. A Cdc42 ortholog is re-quired for penetration and virulence of Magnaporthe gri-sea[J]. Fungal Genetics and Biology, 2009, 46(6-7): 450-460.
[10] Fu T, Kim J O, Han J H, et al. A small GTPase RHO2 plays an important role in pre-infection development in the rice blast pathogen Magnaporthe oryzae[J]. Plant Pathology Journal, 2018, 34(6): 470-479.
[11] Zheng W, Chen J, Liu W, et al. A Rho3 homolog is essential for appressorium development and pathogenicity of Magnaporthe grisea[J]. Eukaryotic Cell, 2007, 6(12): 2240-2250.
[12] Manning G, Whyte D B, Martinez R, et al. The protein ki-nase complement of the human genome[J]. Science, 2002, 298(5600): 1912-1934.
[13] Tassan J P, Le Goff. An overview of the KIN1/PAR- 1/MARK kinase family[J]. biology of the cell, 2004, 96(3): 193-199.
[14] Chen L, Shinde U, Ortolan T G, et al. Ubiquitin-associated (UBA) domains in Rad23 bind ubiquitin and promote inhibition of multi-ubiquitin chain assembly[J]. EMBO Reports, 2001, 2(10): 933-938.
[15] Wu Y, Griffin E E. Regulation of cell polarity by PAR-1/MARK kinase[J]. Current Topics in Developmen-tal Biology, 2017, 123: 365-397.
[16] Gomes J E, Bowerman B. Caenorhabditis elegans par genes[J]. Current Biology, 2002, 12(13): R444.
[17] Guo S, Kemphues K J. Par-1, a gene required for establish-ing polarity in C. elegans embryos, encodes a putative Ser/Thr kinase that is asymmetrically distributed[J]. Cell, 1995, 81(4): 611-620.
[18] Zhang L, Zhang D, Chen Y, et al. Magnaporthe oryzae CK2 accumulates in nuclei, nucleoli, at septal pores and forms a large ring structure in appressoria, and is involved in rice blast pathogenesis[J]. Frontiers in Cellular and Infection Microbiology, 2019, 9:113
[19] Zhao X, Kim Y, Park G, et,al. A mitogen-activated protein kinase cascade regulating infection-related morphogenesis in Magnaporthe grisea[J]. The Plant cell, 2005, 17(4): 1317-1329.
[20] Adamo J E, Rossi G, Brennwald P. The Rho GTPase Rho3 has a direct role in exocytosis that is distinct from its role in actin polarity[J]. Molecular and Cellular Biology, 1999, 10(12): 4121-4133.
[21] Adamo J E. Yeast Cdc42 functions at a late step in exocyto-sis, specifically during polarized growth of the emerging bud[J]. Journal of Molecular Cell Biology, 2001, 155(4): 581-592.
[22] Luo Y P, Zhang H C, Qi L L, et al. FgKin1 kinase localizes to the septal pore and plays a role in hyphal growth, ascospore germination, pathogenesis, and locali¬zation of Tub1 beta-tubulins in Fusarium graminearum[J]. New Phytologist, 2014, 204(4): 943-54.
[23] Praveen R. Juvvadi, D. Christopher Cole, Katie Falloon, et al. Kin1 kinase localizes at the hyphal septum and is dephosphorylated by calcineurin but is dispensable for septation and virulence in the human pathogen Aspergillus fumigatus[J]. Biochemical and Biophysical Research Communications, 2018, 505(3): 740-746.
責任编辑:沈德发

