P-糖蛋白的核酸适配体筛选及应用研究
李玉娟, 梁敏, 武广霞, 孙欣欣, 刘珊, 邓玉林
(北京理工大学 生命学院,北京 100081)
P-糖蛋白是最早由LING等[1]在中国仓鼠的卵巢细胞中发现的一种跨膜蛋白,分子量为170 kd,由人类基因mdr1编码[2],可通过消耗ATP的能量将胞内物质转运至胞外[3],也是迄今研究最多的三磷酸腺苷结合盒转运蛋白之一[4]. P-gp在人体中广泛分布在肝脏、肾脏[5]、胎盘、小肠及血脑屏障[6-7]等器官及组织中,可以防止有害物质进入,维持内环境的稳态,因而也会影响治疗药物穿越屏障到达靶点发挥疗效,如治疗一些神经系统疾病如癫痫、抑郁症、阿尔兹海默症等的药物[8]. 此外,P-gp在许多肿瘤组织中高表达,能将抗肿瘤药物如长春新碱、紫杉醇、阿霉素等排到肿瘤细胞外[9],严重影响抗肿瘤药物的治疗效果,由P-gp介导的肿瘤细胞多药耐药的机制,是抗肿瘤领域的研究热点. 研究学者多年来一直致力于开发新型P-gp抑制剂,但大多数P-gp抑制剂因其毒副作用高、特异性低等缺点,限制了其在临床的应用[10]. 因此,迫切需要开发新型安全、高效的P-gp抑制剂.
核酸适配体(Apt)是利用指数富集配体的系统进化(SELEX)技术,从随机寡核苷酸文库中体外筛选的,能与特定靶标结合的ssDNA或RNA,其靶标可以是小分子、蛋白或细胞[11-12],Apt以高亲和力、强特异性与靶标结合. Apt因为易于合成与修饰,生产成本低,已作为工具分子在生物分析检测领域广泛应用[13-15]. 此外,Apt也可作为治疗药物,如Macugen用于老年黄斑病变,还有很多抗肿瘤用的Apt已进入Ⅱ、Ⅲ期临床研究[16]. 目前尚未见P-gp的核酸适配体报道,Apt能否抑制P-gp外排功能也未有相关研究.
本文构建ssDNA文库,基于SELEX技术筛选P-gp的Apt,采用酶联免疫吸附测定法(ELISA)和表面等离子体共振法(SPR)两种方法表征Apt与P-gp的结合力大小,在人脑微管内皮细胞(hCMEC/D3)、人结直肠腺癌细胞(Caco-2)两种细胞系上考察Apt对P-gp外排功能的抑制效果,以期为P-gp相关研究提供潜在新型抑制剂.
1 实验材料
1.1 实验试剂
甲苯磺酰基磁珠(TMB)、链霉亲和素磁珠(SMB)(挪威Dynal公司);寡核苷酸文库、上下游引物及生物素修饰的上下游引物、核酸适配体、PCR纯化试剂盒(上海生工生物工程公司);P-gp(美国Cloud-Clone公司);辣根过氧化物酶标记的链霉亲和素蛋白(HRP-Strepavidin)(上海碧云天生物技术公司);氨基联试剂盒、再生试剂(甘氨酸-盐酸,pH 2.5)(美国GE Healthcare公司);RPMI1640培养基(美国Gibco公司);MEM培养基(美国GEHyclone公司);四季青胎牛血清(浙江天杭生物科技公司);牛血清白蛋白(BSA)、吐温-20、TE缓冲液、青链霉素混合液、DH5α大肠杆菌感受态细胞、Luria-Bertani(LB)培养基、TMB显色液、Triton X-100、罗丹明123(北京索莱宝科技公司);Na2HPO4·12H2O,NaH2PO4·H2O,KH2PO4·H2O(分析纯,西陇科学有限公司);NaCl,KCl(分析纯,北京化学试剂公司);SORFA胎牛血清(浙江硕华生命科学研究公司);环孢菌素A(上海梯希爱化成工业发展公司);pEASY-T1克隆载体(北京全式金生物技术公司).
1.2 仪器与耗材
磁力收集器(MPC,挪威Dynal公司);GE Biacore T200生物分子相互作用分析系统、Series S CM5芯片(美国GE Healthcare公司);多功能酶标仪(美国BioTek公司);凝胶成像分析系统ChampGel 3200(美国Bio Rad公司);R232 PCR仪(美国MJ Research公司);倒置显微镜(德国Leica公司);培养皿、离心管、96孔板(美国Corning公司).
1.3 细胞株
人脑微管内皮细胞(hCMEC/D3)购自上海赛百慷生物技术股份有限公司;人结直肠腺癌细胞(Caco-2)由首都医科大学馈赠.
2 实验方法
2.1 SELEX技术筛选Apt
2.1.1ssDNA初始文库的构建
设计两端18 bp固定序列,中间45 bp随机序列的81 bp寡核苷酸ssDNA文库,并用Primier Premier 5.0软件(加拿大Premier公司)设计上、下游引物及生物素修饰的上、下游引物. 引物序列如表1所示.
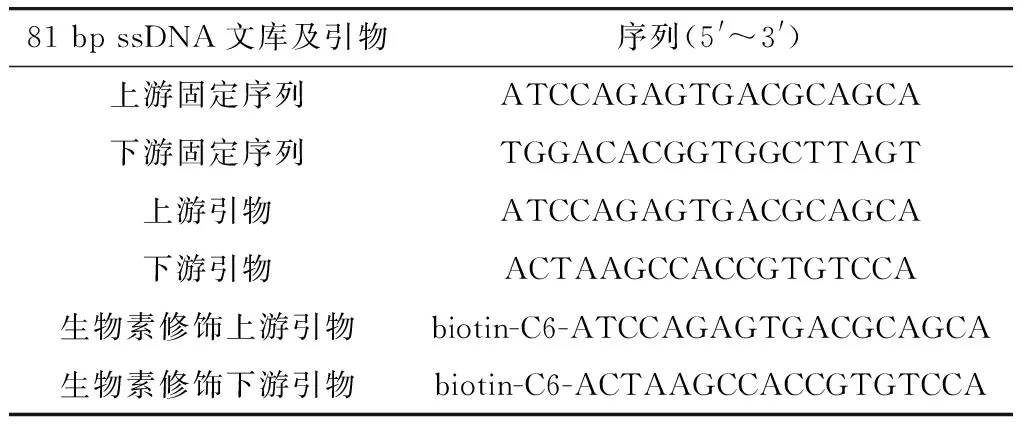
表1 81 bp寡核苷酸随机文库及引物Tab.1 81 bp oligonucleotide library and primers
2.1.2ssDNA初级文库的筛选
取10 μL TMB磁珠,与50 μL 0.1mg/mL的P-gp溶液(ddH2O配制)、100 μL缓冲液1、100 μL缓冲液2混合,4 ℃孵育过夜,后各用1 mL缓冲液3清洗1次,1 mL缓冲液4清洗2次,每次均在MPC上静置2 min,弃去上清,最后用12.5 μL的缓冲4重悬,得到TMB-P-gp复合物(筛选缓冲液的配方如表2所示). 将前述TMB-P-gp复合物与1 OD的ssDNA(100 μmol/L)混合,加入200 μL PBS,4 ℃孵育2 h,后用200 μL PBS清洗3次,得到TMB-P-gp-ssDNA复合物. 取40 μL的TE缓冲液加入到TMB-P-gp-ssDNA复合物中,95 ℃水浴15 min,将结合在TMB-P-gp复合物上的ssDNA洗脱,同时使用MPC清除TMB-P-gp复合物,可得到ssDNA溶液. 使用未生物素修饰的上游引物与生物素修饰的下游引物对得到的ssDNA进行PCR扩增,条件为:引物用量2.5 μL;94 ℃预变性5 min;95 ℃变性30 s;56.3 ℃退火30 s;72 ℃延伸30 s;循环数为15. 扩增结束后,使用4%琼脂糖凝胶电泳检测扩增产物.

表2 ssDNA筛选中使用的缓冲液及其组成Tab.2 The buffer and formula used to select ssDNA
2.1.3ssDNA次级文库的制备
将2.1.2中PCR扩增产物用柱式PCR产物纯化试剂盒纯化,使用酶标仪测定dsDNA的浓度. 根据磁珠推荐固载量(100 ng dsDNA每5 μL SMB)向dsDNA中加入适量的SMB,在37 ℃摇床孵育0.5 h,得到SMB-dsDNA复合物. 取200 μL 100 mmol/L的NaOH溶液加入到SMB-dsDNA复合物中,室温下碱变性15 min使未生物素化的ssDNA从SMB磁珠中洗脱下来. 取40 μL 1 mol/L的NaH2PO4(pH=3.2)中和ssDNA洗脱液,便可得到ssDNA次级文库.
2.1.4ssDNA次级文库的筛选
将2.1.3中的ssDNA次级文库,作为新的起始文库,重复上述实验步骤,本研究共进行了10轮重复筛选实验. 当筛选进行到第4轮时,进行一次反筛选,即用未固定P-gp的TMB磁珠与筛选的ssDNA文库孵育2 h,使用磁力分离器分离磁珠,得到不与TMB磁珠结合的ssDNA. 且每一轮筛选结束后,均使用ELISA法表征筛选的ssDNA与P-gp的结合能力,且每4轮进行一次反筛选实验. 实验中为得到特异性强的目标条带,每轮筛选的P-gp用量、PCR反应条件以及TMB-P-gp复合物与ssDNA的孵育时间略有不同. 其中,P-gp溶液用量的体积分别为:1~3轮5 μL,4~7轮4.8 μL,8~10轮4.6 μL. PCR反应中引物用量及循环数分别为:1~5轮2.5 μL,循环数15;第6轮5 μL,循环数20;7~8轮2.5 μL,循环数15;9~10轮5 μL,循环数20. TMB-P-gp复合物与ssDNA的孵育时间分别为:1~3轮120 min,4~6轮90 min,7~8轮70 min,9~10轮60 min.
2.1.5克隆与测序
第10轮筛选结束后,将得到的ssDNA用未生物素修饰的引物进行PCR扩增. 取上述扩增后产物0.5~4.0 μL与pEASY-T1克隆载体按1 μL载体:1.6 ng PCR产物的比例混合,补充ddH2O至5 μL,室温下反应10 min. 取50 μL的感受态细胞DH5α加入到上述混合物中,冰浴30 min,42 ℃热激30 s,然后置于冰上冷却2 min. 向混合物中加入250 μL的LB培养基,放置于37 ℃摇床中培养1 h. 将上述菌液4 000 r/min离心1 min后,保留100 μL涂布于平板中,37 ℃培养. 第二天选择生长状况良好的单个菌落,加入5 mL LB培养液培养12 h. 将上述含菌培养液送至上海生工生物工程股份有限公司进行DNA测序.
2.2 Apt与P-gp结合能力的表征
2.2.1ELISA方法表征结合能力
上述ssDNA测序后,即可得到目标Apt的核酸序列. 由上海生工生物工程股份有限公司按已知核酸序列合成Apt及进行生物素修饰. 将上述合成的Apt用双蒸水配制成0.1,0.4,0.8,1.0,2.0,5.0 μmol/L的系列溶液备用. 用100 μL 5 μg/mL的P-gp溶液包被96孔板(Nunc TM,丹麦),4 ℃过夜孵育. 每孔加入300 μL 0.1%的BSA封闭液(PBST配制,即含0.05%吐温-20的PBS溶液),在4 ℃封闭1 h. 微孔中加入100 μL相应浓度的Apt溶液,37 ℃孵育1 h后,向每孔中加入100 μL的HRP-Strepavidin,室温孵育1 h. 每步操作后均用300 μL的PBST清洗微孔3次. 最后向微孔中加入100 μL TMB显色液,避光反应直至蓝色褪去时,加入50 μL H2SO4终止反应,于酶标仪450 nm下测OD值. 以Apt浓度为横坐标,OD值为纵坐标进行非线性拟合可得到P-gp结合Apt的量随Apt浓度变化的关系,依据式(1)[17]求算复合物的解离常数
Y=BmaxX/(Kd+X)
(1)
式中:Y为OD值;Bmax为P-gp的最大吸附量;Kd为Apt-P-gp复合物的解离常数.
2.2.2SPR法表征结合能力
实验开始前需按照式(2)计算配体P-gp的偶联量,分析物为Apt,其中Rmax预设为100 RU,Sm为1,计算出RL后,P-gp的实际偶联量为1.5RL. 采用下述步骤将P-gp偶联到CM5芯片上,具体为:P-gp溶解在10 mmol/L、pH为4.5的醋酸钠缓冲液中,终浓度为20 μg/mL,使用氨基偶联试剂盒活化CM5芯片表面的羧基后,P-gp以脉冲方式进样,通过吸附方式偶联在芯片的表面上,并用乙醇胺封闭多余的羧基. 接下来选用pH 2.5、10 mmol/L的甘氨酸缓冲液为再生试剂,对浓度梯度为0,17,34,51,68,85 μmol/L的10条适配体依次进样,用GE Biacore T200生物分子互作分析系统收集系列浓度Apt的响应值,分析Apt与P-gp的结合力.
(2)
式中:RL为配体偶联水平;Sm为化学计量比;Ml为配体分子量;Ma为分析物分子量.
2.3 Apt对P-gp外排功能的抑制作用
2.3.1细胞培养
将hCMEC/D3复苏在含10%四季青胎牛血清,1%青链霉素的RPMI 1640培养基中;将Caco-2复苏在含10%SORFA胎牛血清,1%青链霉素的MEM培养基中. 上述两种细胞均培养在含5%CO2的37 ℃恒温培养箱中,每天置于倒置显微镜下观察细胞生长状况,待传代2~3次后,进行后续实验.
2.3.2P-gp底物罗丹明123的外排实验
将生长状态良好的细胞以2×105个/mL接种于96孔板中,每孔100 μL,复孔3个,放置于37 ℃恒温培养箱中培养12 h. 将96孔板中的培养基倒出,每孔加入100 μL含罗丹明123与Apt5的培养基,且罗丹明123的终浓度为5 μmol/L,Apt5的终浓度为0.5 μmol/L,同时使用环孢菌素A(Cyclo A)做阳性对照,终浓度为50 μmol/L;PBS做空白对照(Con). 将96孔板置于37 ℃恒温培养箱中孵育2 h后,将孔板中溶液倒出,并用PBS清洗3次,每孔加入100 μL 0.1%的Triton X-100,15 min后于酶标仪下检测每孔的荧光强度. 罗丹明123的激发波长为485 nm,发射波长为535 nm.
2.4 数据处理
使用GraphpadPrism v7.0(美国Graphpad软件公司)数据处理软件进行数据的统计分析,结果用平均值±标准差表示,使用单因素方差分析显著性,P<0.05时具统计学意义.
3 结果与讨论
3.1 P-gp核酸适配体的筛选结果
本文对核酸适配体筛选过程中的一些关键步骤,如寡核苷酸初始文库的大小、每轮结合时P-gp的用量、P-gp-TMB复合物与ssDNA文库的孵育时间、及每轮的PCR扩增条件进行了优化. 首先,构建了80 bp和81 bp两个不同大小的ssDNA文库,通过琼脂糖凝胶电泳,比较了80 bp和81 bp文库扩增后的PCR产物,发现81 bp文库得到的非特异性条带最少,因此将其作为筛选Apt的初始文库. 其次,在筛选过程中,发现每轮结合时P-gp的用量、及P-gp-TMB复合物与ssDNA文库的孵育时间是影响筛选产物特异性的重要因素,通过减少每轮的P-gp用量,缩短P-gp-TMB复合物与ssDNA文库的孵育时间,可以筛选得到与P-gp特异性结合能力更强的ssDNA. 此外,每轮PCR扩增条件也会显著影响筛选产物的特异性,其中,本文对影响PCR扩增的3个重要条件如退火温度、循环数及引物用量进行优化,结果表明56.3 ℃的退火温度适用于各轮筛选. 且在PCR的每轮进程中,要依据琼脂糖凝胶电泳的特异性条带结果,来适当调整每轮PCR扩增的循环数及引物用量,确保每一轮都得到特异性的扩增产物. 在整个筛选过程中,收集每轮筛选得到的ssDNA,取适量用ELISA法表征每轮筛选得到的ssDNA与P-gp的结合能力,当吸光度不再上升时,指示筛选循环结束,同时,采用其他蛋白如卵清蛋白为对照,以评价ssDNA与P-gp结合的特异性.
如图1所示,ssDNA与P-gp的亲和性随筛选进程增加,筛选至第10轮时,吸光度基本不再上升. 卵清蛋白与ssDNA亲和性不随筛选进程改变,且卵清蛋白与ssDNA的结合力远小于ssDNA与P-gp的结合力,这在一定程度上表明了Apt与P-gp的结合具有特异性. 本研究最终筛选得到10条长度为81 bp的P-gp特异性核酸适配体(依次标记为Apt1~ Apt10),其序列如表3所示. 使用Clustal X 2.1软件(European Bioinformatics Institute,)对Apt的一级结构进行比对分析时,发现10条Apt序列中同一碱基连续出现的频率较高(不含引物区),如GGG、TTT、AAA、CCC的碱基组合分别重复出现6次、5次、6次、5次;TTTT的碱基组合出现2次;GGGGG、CCCCC的碱基组合分别出现3次、2次;复杂的碱基组合TTTTCC也出现2次. 根据Apt与P-gp亲和力的表征结果,比对分析了与P-gp亲和力最强的三条核酸适配Apt5、Apt8与Apt1的序列,发现三者中GGG与AAA的碱基组合出现频率最高. 3条Apt序列的第2,4,6,8,30,34,39,40位具有相同的碱基. 以上结果或许说明特定的碱基顺序及其在序列中出现的频率决定了Apt与P-gp间的结合能力. 使用Mfold在线软件(http://unafold.rna.albany.edu/?q=mfold)分析了Apt的二级结构,发现10条Apt的二级结构显示均以茎环和口袋为主. 其中,Apt1,Apt5,Apt8的结构特点是小口袋大茎环(如图2所示),表现为序列从5′到3′端在23~46碱基形成单一口袋,在口袋的5′端、随机序列区或3′端形成2~3个较大的茎环,说明径环与口袋的结构特征或许是Apt能与P-gp特异性结合的结构基础.
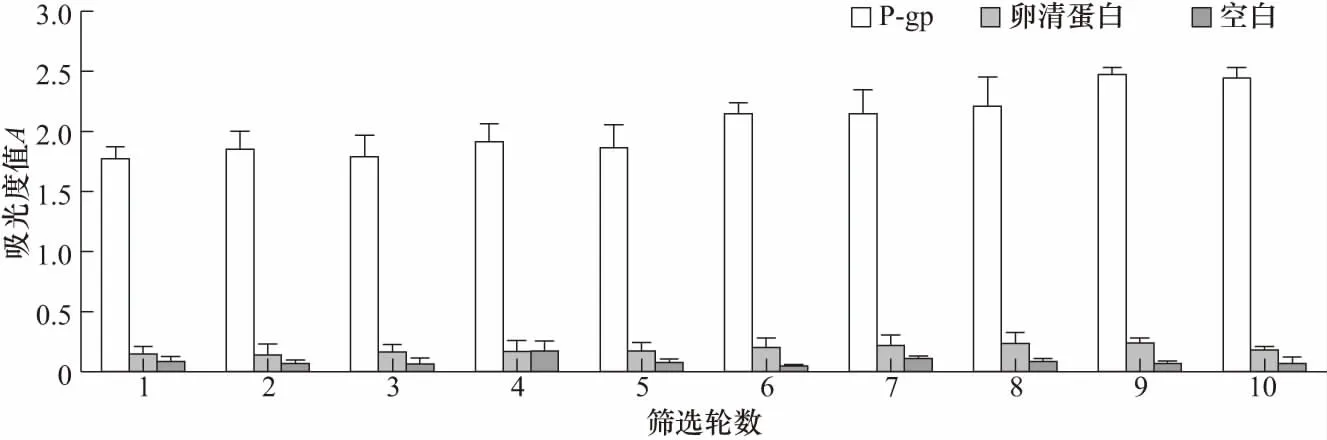
图1 ELISA法测得每轮筛选的ssDNA与P-gp、卵清蛋白的亲和性Fig.1 Affinity of selected ssDNA in every round binding with P-gp or ovalbumin by ELISA

表3 P-gp核酸适配体的核酸序列Tab.3 The nucleic acid sequence of P-gp aptamers
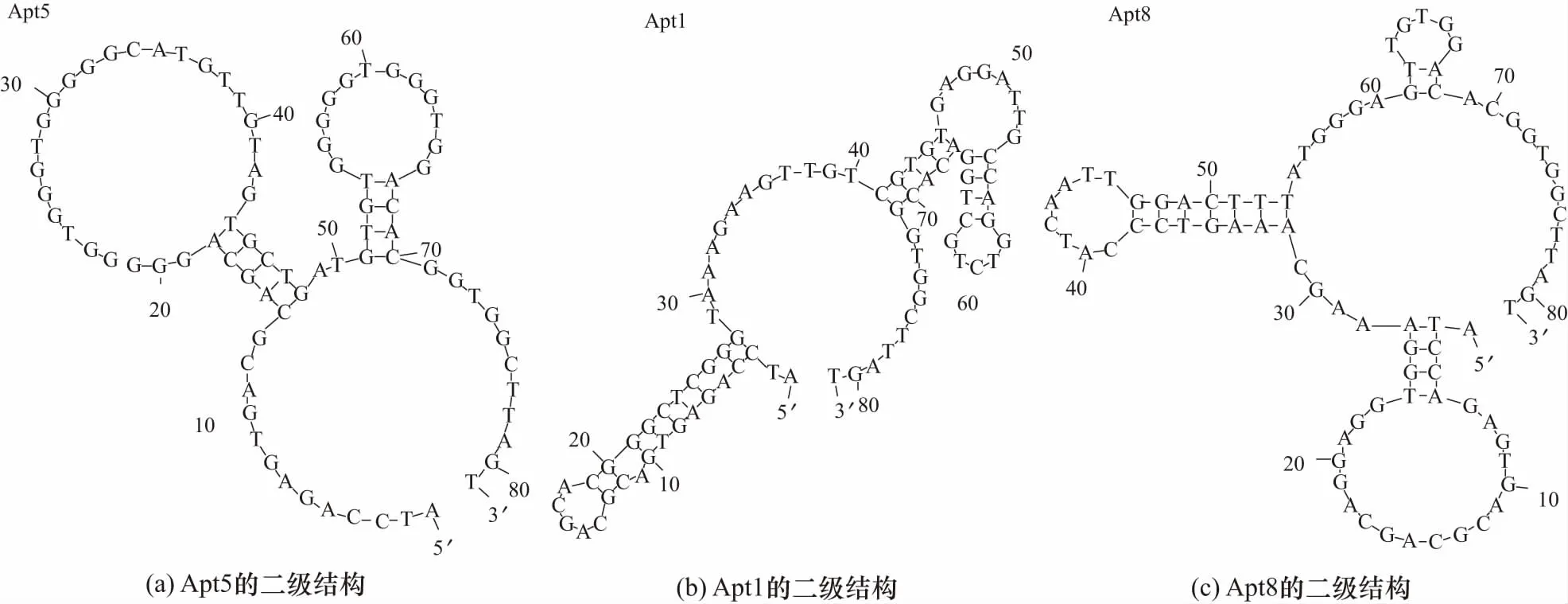
图2 Mfold软件预测Apt1, Apt5, Apt8的二级结构Fig.2 Predicted secondary structure of Apt1, Apt5 and Apt8 with Mfold software
3.2 ELISA法测定Apt与P-gp的结合能力
用ELISA法测定不同浓度的Apt与0.5 μg P-gp结合时的吸光度(如图3所示),根据式(1)可求得Apt与P-gp结合的解离常数,测定结果见表4. Apt与P-gp结合的解离常数大小可以反映二者结合能力的强弱,且解离常数越小,结合能力越强. ELISA法测定的Apt与P-gp结合的解离常数在10-8~10-7mol/L范围,表明Apt与P-gp具有较强的结合能力[18]. 其中,Apt5,Apt8和Apt1与P-gp结合的解离常数最小,依次为17.46,25.15和28.56 nmol/L,表明Apt5,Apt8和Apt1均与P-gp具有较高的结合能力. 此外,Apt5,Apt8和Apt1的核酸序列中GC含量丰富,分别为66.7%,50.6%和57.8%,说明Apt与P-gp结合能力的强弱可能与GC含量有关. ELISA法测得的10条Apt与P-gp结合能力的大小顺序为:Apt5>Apt8>Apt1>Apt4>Apt6>Apt10>Apt7>Apt2>Apt9>Apt3.
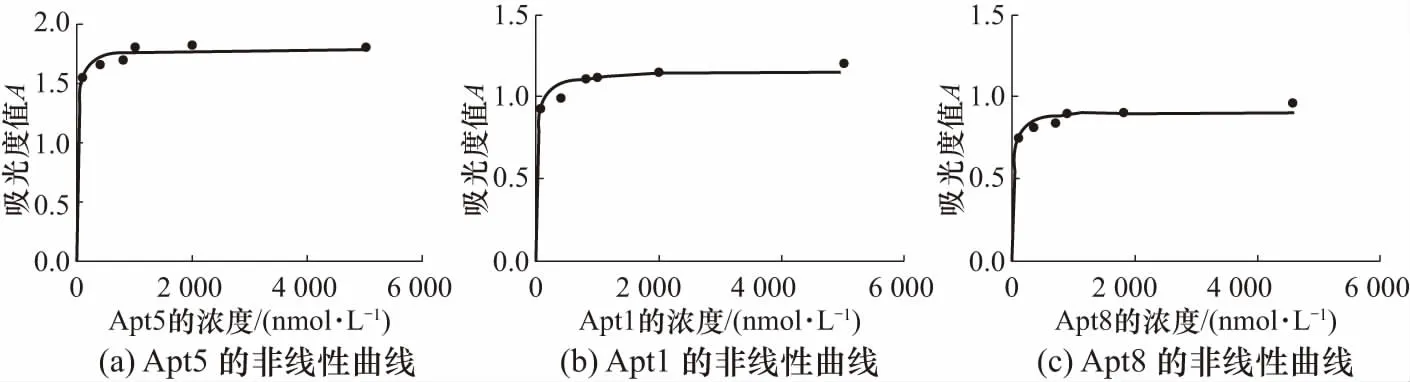
图3 ELISA法测得的Apt1,Apt5及Apt8吸光度与Apt浓度的非线性曲线Fig.3 Nonlinear curve between absorbance and concentration of Apt1, Apt5 and Apt8 by ELISA
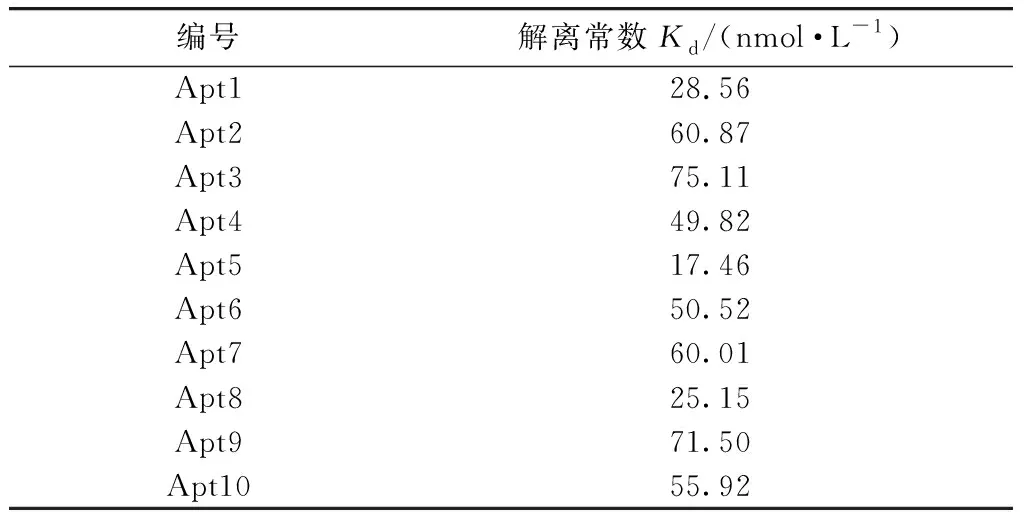
表4 ELISA法测得Apt与P-gp结合的解离常数
3.3 SPR法测定Apt与P-gp的结合能力
本研究也利用SPR法表征了Apt与P-gp的结合能力,仪器响应值随Apt浓度变化的曲线如图4所示. 在该实验中,为提高CM5芯片偶联P-gp的量,对溶解P-gp的缓冲液的PH值进行了优化. 通过比较不同PH值的醋酸钠缓冲液进样时芯片与P-gp的偶联量,最终选用PH为4.5的醋酸钠缓冲液,该pH条件下P-gp的荷电量增加(P-gp的等电点为8.95),富集在芯片上的量也增加,能达到较理想的偶联效果. 用SPR法测得的Apt与P-gp结合的解离常数如表5所示. 结果表明,Apt5,Apt1和Apt8的解离常数最小,依次为1.691,3.524和3.487 μmol/L,表明Apt5,Apt1和Apt8具有较强的结合P-gp的能力,该结果与用ELISA方法测得的结果基本一致.
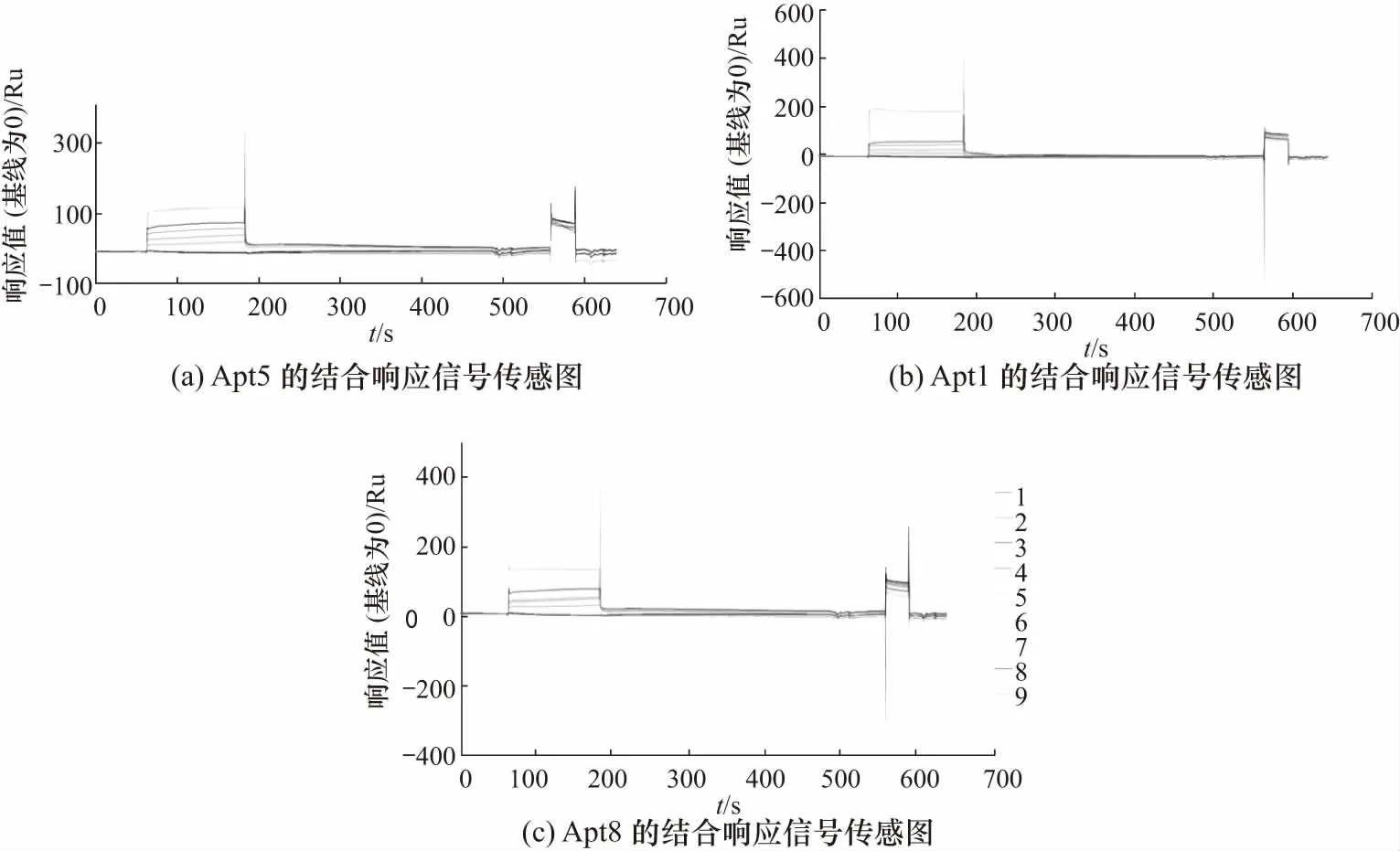
图4 Apt与P-gp的结合响应信号传感图Fig.4 Sensor-gram for the binding response signals of Apt with P-gp
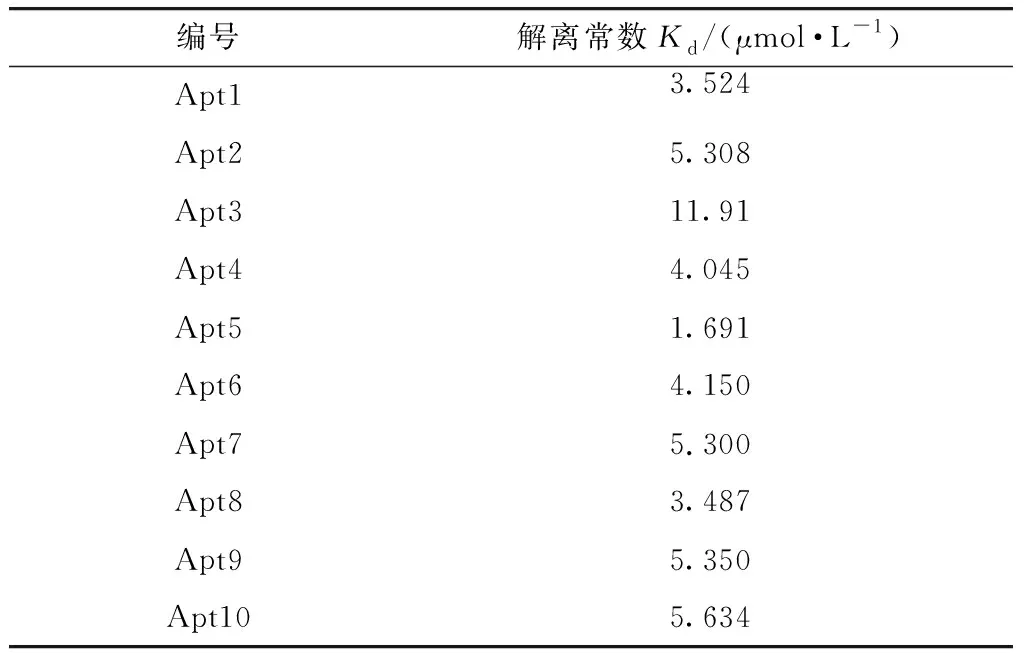
表5 SPR法测得Apt与P-gp结合的解离常数
ELISA和SPR法均是文献中用来测定蛋白及其配体间亲和力的常用方法,但二者的基本原理不同,产生误差的主要来源也不尽相同,因而测得的数值会存在一定的差异. 多个文献的研究结果表明,当采用不同方法测定同一组蛋白及其配体间的亲和力时,检测结果或会相差1~2个数量级. 研究者认为产生数值差异的原因与抗原包被的流程相关,虽然不同方法检出能力不同,但是相关结果仍具有参考性[19-20]. 也有研究者用SPR法测靶标蛋白及适配体间的解离常数,结果也在μmol/L范围级别,但并不能说明二者间相互作用弱[21],具体的原因或许与测定的方法、原理及误差等有关系,尚需进一步的研究. 此外,本实验经过多次重复,测定的结果也是稳定与重现的. 本实验中虽然SPR及ELISA法测定的数量级范围不同,但两种方法表征的Apt与P-gp亲和力大小的顺序基本一致. 本研究通过两种方法,均证明筛选得到的Apt与P-gp具有较强的亲和性. 其中,Apt5与P-gp的结合能力最强,因此选择Apt5进行后续实验.
3.4 Apt5对hCMEC/D3及Caco-2细胞内罗丹明123蓄积量的影响
Apt对P-gp外排功能的抑制作用通过P-gp的底物罗丹明123的荧光强度反映,且细胞内罗丹明123越多,荧光越强,表明P-gp的外排功能越受抑制[22]. 本研究在hCMEC/D3和Caco-2两种细胞系上考察了Apt5对P-gp外排功能的抑制作用,结果如图5所示. 在hCMEC/D3中,Apt5及阳性药环孢菌素A均显著地增加了胞内罗丹明123的蓄积量,分别为40.77%,44.60%,说明Apt5对hCMEC/D3中P-gp的外排功能有很强的抑制作用,且该抑制作用与阳性药的抑制作用相当. 在Caco-2中,Apt5及环孢菌素A也显著地增加了胞内罗丹明123的蓄积量,分别为32.39%,154.9%,说明Apt5对Caco-2中P-gp的外排功能也具有较强的抑制作用. 本实验在两种细胞系上均证明了Apt5对P-gp的外排转运功能有较强的抑制作用.
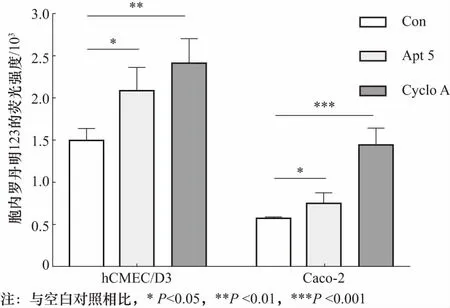
图5 环孢菌素A,Apt5存在时hCMEC/D3及Caco-2胞内罗丹明123的蓄积量Fig.5 Intracellular rhodamine 123 accumulation of hCMEC/D3 and Caco-2 treated with cyclosporine A or Apt5
4 结 论
本文基于SELEX技术首次筛选得到了P-gp的核酸适配体,并通过ELISA与SPR两种方法对Apt的结合能力进行表征,同时选择亲和性最强的Apt5考察其对P-gp功能的抑制作用. 结果筛选得到的10条Apt与P-gp均具有较高的结合能力. 经过Apt的序列分析与结构预测,发现Apt与P-gp特异性结合的能力与其特定的重复序列以及径环、口袋的结构特征相关. 且在对Apt结合能力表征的实验中发现Apt序列中的GC质量分数可能影响Apt与P-gp的结合能力,GC质量分数丰富的Apt对P-gp具有较高的结合能力. 此外,在底物外排实验中,Apt5能显著增加hCMEC/D3与Caco-2细胞内罗丹明123的蓄积量,对P-gp的外排功能表现出很强的抑制作用,或可成为潜在的P-gp抑制剂,这将为开发安全、高效的新型P-gp抑制剂提供新思路.
课题组前期曾对Apt体内抑制P-gp功能进行了初步研究,大鼠尾静脉注射给与Apt5和罗丹明123后,测定大鼠血浆和脑组织匀浆中罗丹明123的质量分数,来初步评价Apt5对鼠脑P-gp外排功能的抑制效果. 结果表明Apt5能显著增加鼠脑中罗丹明123质量分数,说明Apt5具有抑制P-gp外排功能的潜力,但后续还要进行更多的大量的体内实验来全面考察Apt的抑制能力. 未来对Apt仍需进行更多深入的研究,比如Apt5与P-gp的具体结合位点以及其抑制P-gp功能的机制等. 其次,对Apt的化学修饰也是一项重要的工作,比如通过硫代磷酸类似物取代DNA的磷酸二酯键,或用倒置胸腺嘧啶修饰Apt的3′端,都能增加Apt对体内核酸酶的抵抗力,延长半衰期,提高体内稳定性[23]. Apt作为分子工具,未来还具有很多潜在的应用价值,比如,可对Apt进行荧光标记,实现生物样本中P-gp质量分数分析,为P-gp的质量分数测定提供新方法;也可结合各种成像系统,实现基于Apt的P-gp在机体内的观察等.

