荧光RNA:点亮神秘的基因组暗物质
左方婷 杨弋
中心法则由弗朗西斯·克里克(F. Crick,1962年诺贝尔生理或医学奖获得者)于1958年提出,并于1970年丰富完善后在《自然》杂志(Nature)发表[1]。中心法则阐明了DNA、RNA以及蛋白质三者之间的关系:DNA可自行复制,并将遗传信息传递给RNA,再由RNA传递给蛋白质。无论是DNA还是蛋白质,在生命活动中都起到无可替代的作用,而起遗传信息传递的RNA则看起来平平无奇。其实这种过分简化的遗传信息传递流程忽略了RNA在其中扮演的角色。由于长期以来人们对RNA的了解仅浮于表面,在活细胞上也难以对RNA进行直接动态的观察,所以RNA,特别是种类繁多的非编码RNA也常被比喻成基因组中的暗物质。
研究RNA的意义
有关基因组学的深入研究表明:人类的大多数基因组被转录成RNA,但只有小部分被翻译成蛋白质。初代中心法则中的RNA即指这种可以被翻译成蛋白质的RNA,称为编码RNA。我们所熟知的信使RNA(messenger RNA, mRNA)就是编码RNA。通过对mRNA示踪,研究mRNA在细胞内的定位和转运,可以帮助科研人员获得更多关于mRNA的信息。改变RNA的定位可能会引起疾病的发生。例如,罹患脆性X染色体综合征的患者常表现出学习和记忆障碍。这是因为患者发生基因突变,缺失与mRNA转运相关的FMRP蛋白质,导致mRNA无法正确定位而引起疾病。因此,通过对RNA动态成像可以为了解该疾病或其他类似疾病的机理提供新的见解。
除mRNA起到传递遗传信息的功能外,RNA家族里还存在大量非编码RNA(non-coding RNA, ncRNA)。细胞中含量最高的核糖体RNA(ribosomal RNA, rRNA)和转运RNA(transfer RNA, tRNA)都属于非编码RNA,它们都能从基因组上转录出来,但是不翻译成蛋白质。此外,ncRNA还包括参与mRNA剪接的小核RNA(small nuclear RNA, snRNA)、参与RNA修饰的小核仁RNA(small nucleolar RNA, snoRNA),以及近年来发现的环状RNA(circular RNA, circRNA)、长链非编码RNA(long noncoding RNA, lncRNA)及microRNA等。大多数的ncRNA都是神秘的,与mRNA不同,ncRNA的序列仅能为它们潜在的功能提供很少的信息。因此,在细胞中对它们进行示踪显得尤为重要。其中,可以通过成像确定ncRNA的亚细胞器定位,从而对它们的功能进行预测。例如,细胞核中的ncRNA可能在调节基因转录中发挥功能,而细胞质中的ncRNA可能会影响该区域的其他过程。除此之外,对特定位置或结构的ncRNA的迁移进行动力学追踪,如细胞核中的卡哈尔体(Cajal body)或细胞质中的P小体(P-bodies),都可用于预测ncRNA的功能或调节通路。
研究RNA的生物学功能意义深远,它能帮助我们逐步揭开神秘的基因组面纱,更好地了解生命活动的本质。但是目前在研究RNA生物学功能上面临着巨大的挑战,缺乏能够在活细胞中高效工作的RNA可视化工具便是一个技术瓶颈。
荧光RNA的开发背景
提到RNA的可视化工具,则不得不提到蛋白质的可视化工具,其中遗传编码的荧光蛋白绝对占有重要的一席之地。2008年,绿色荧光蛋白(green fluorescent protein, GFP)的發现者下村修(O. Shimomura)、发现GFP具有荧光标签价值并将GFP的编码基因成功表达在原核模式生物的查尔菲(M. Chalfie),以及对GFP加以改造得到一系列不同荧光光谱的荧光蛋白并推动其广泛使用的钱永健,因为荧光蛋白技术的开发,共同获得该年度诺贝尔化学奖。
绿色荧光蛋白来自维多利亚多管发光水母,野生型绿色荧光蛋白由238个氨基酸构成。晶体学研究表明,绿色荧光蛋白是一个由11个β折叠环绕构成的桶状结构,生色团分子被α-螺旋固定在桶状结构的中心。该生色团由Ser65-Tyr66-Gly673个氨基酸(丝氨酸—酪氨酸—甘氨酸)残基通过自催化反应形成,系统将其命名为4-羟基亚苄基—咪唑啉酮(4-hydroxybenzlidene imidazolinone,简称HBI)[2]。化学合成的HBI没有荧光,这是HBI自身特性造成的:当HBI在水溶液中,分子将主要围绕结构中的乙炔桥进行类似“呼啦圈旋转”的运动,使得激发光能量以非辐射衰减的方式消散,宏观表现为受激发时不会发出荧光。而当HBI固定时(正确折叠的GFP中HBI被蛋白质结构固定),分子内运动被阻止,导致其无法通过分子运动而只能以辐射的方式耗散能量,宏观表现为受激发时发出荧光。
荧光蛋白可以说是大自然赠予人类的礼物,它的出现将“死物学”变成了真正的“生物学”。它发出的荧光犹如明灯,照亮生命体在细胞、分子层面的诸多黑暗角落,正如钱永健教授所言,荧光蛋白将生命活动这部晦涩的小说改编成了一部生动的电影。
自然界中存在天然的荧光蛋白,但尚未发现天然的荧光RNA。所以在RNA早期研究中,科研人员另辟蹊径,开发出一系列基于荧光蛋白的RNA示踪工具,即RNA结合蛋白—荧光蛋白系统(RNA binding protein-fluorescent protein, RBP-FP)。其中,基于噬菌体得到的MCP-FP系统最早被开发出来且应用最为广泛。该系统来源于噬菌体,MS2蛋白是一种噬菌体衣壳蛋白,它也是一种天然存在的RNA结合蛋白,可以特异性识别一段由19个碱基形成的茎环结构RNA,即MS2结合位点,并与之相互作用。在使用时,通常在靶标基因上融合一组MS2结合位点,同时将MS2衣壳蛋白与荧光蛋白融合,将两者在同一个细胞中共表达。由于MS2结合位点与MS2蛋白具有较高的亲和力,与靶标RNA融合的MS2结合位点会拉近融合有荧光蛋白的MS2衣壳蛋白,从而使靶标基因被“照亮”,实现对细胞内靶标RNA定位和动态变化的实时监测。在这之后,科研人员不断丰富与优化,相继开发了Cys4、PP7、λN22、Cas等诸多基于RNA结合蛋白的RNA成像系统[3]。
上述方法其实都还在借助编码荧光蛋白的这一报告基因,但荧光蛋白自身存在的劣势,如荧光团成熟需要时间且需要氧气参与,以及系统不可避免的游离荧光蛋白造成的背景信号,都会对示踪效果产生不利的影响。尚未发现天然存在的荧光RNA,那我们是否可以根据荧光蛋白发光原理人工“制造”荧光RNA,从而实现对靶标RNA的直接示踪呢?
答案是肯定的。钱永健教授除了在荧光蛋白领域造诣颇高,在荧光RNA领域也是开山鼻祖。他在2003年报道了首个在结合RNA后可以发出荧光的荧光团,该荧光团是一种三苯甲烷染料——孔雀绿(malachite green, MG)[4]。然而,有研究报道,MG染料在荧光辐照后会产生活性氧自由基(reactive oxygen species, ROS)而对细胞产生明显的毒性。虽然MG染料并不能应用于活细胞内的RNA成像,但这也为荧光RNA的出现提供了新的想法。科研人员可以合成类似于荧光蛋白生色团的荧光团,再结合20世纪90年代发展起来的指数富集的配体系统进化(systematic evolution of ligands by exponential enrichment,SELEX)技术,经体外人工筛选得到可以与荧光团结合的RNA适配体。RNA适配体是一段RNA序列,經A-U、C-G、G-U碱基配对形成较为复杂的空间结构。当荧光团分子嵌入RNA适配体形成的结合口袋时,分子内运动被阻碍,从而实现人造“荧光RNA”。这个想法虽然听起来不难,但是实际操作起来还是会面临许多意想不到的问题。所以时隔8年后,第一个可以应用于哺乳动物细胞的荧光RNA才被《科学》杂志(Science)正式报道。
荧光RNA的发展历程
2011年,来自美国康奈尔大学的一个课题组首先报道了一个名叫Spinach-DFHBI的复合物[5]。因其受激发后发出绿光,发射波长位于505 纳米,因而该RNA适配体取名“Spinach”,中文译为“菠菜”。生色团DFHBI是在绿色荧光蛋白生色团HBI的基础上加以修饰而得到,因此它也被称为拟荧光蛋白RNA(RNA mimics of fluorescent protein,RMFP)。Spinach虽然实现了在哺乳动物细胞内表达,但由于其是二聚体,在细胞内折叠效率低且荧光弱等,应用十分受限。
但科研人员们并没有放弃,而是继续对Spinach进行优化,于2012年报道了性质得到明显提升的二代菠菜,Spinach2[6]。脆性X震颤/共济失调综合征是一种神经退行性疾病,它的发生常与“有毒RNA”CGG错误碱基重复序列相关,即在转录过程中,出现长度不等的CGGCGGCGGCGG重复序列,严重干扰正常生理功能。利用Spinach2-DFHBI复合物,科研人员可以实现对“有毒RNA”CGG碱基的标记,并且观察到药物治疗该疾病后CGG碱基重复序列消散的动力学过程。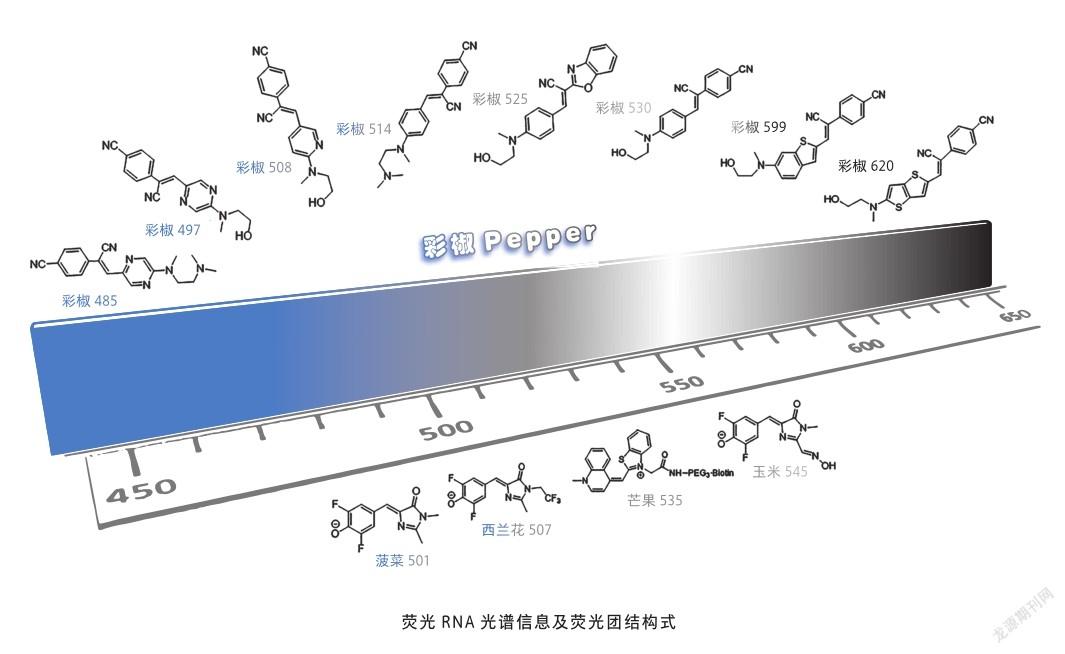
没有最好,只有更好,科研之路没有绝对的顶峰。2014年,在Spinach2的基础上,科研人员在大肠杆菌中表达突变文库,结合流式细胞术筛选得到性质更为出色的RNA适配体Broccoli,中文译为“西兰花” [7],并进一步得到优化后的生色团DFHBI-1T。由于以上拟荧光蛋白RNA的荧光团都是在GFP生色团基础上做的化学修饰,因而它们的光谱并没有发生明显变化,无论是“菠菜”还是“西兰花”,都还属于“绿色蔬菜”。
波长红移一直是科研人员不断拓展的方向,更长的波长不仅穿透力更强,可以对深层组织内的靶标RNA进行示踪,同时可以在一定程度上避免光毒性及细胞自发荧光造成的背景噪声。2017年,依旧是康奈尔大学的那个课题组,他们对红色荧光蛋白DsRFP生色团进行修饰,得到荧光团DFHO。同时利用SELEX技术筛选得到可以与之结合的RNA适配体,最终得到发射波长位于545 纳米的黄绿色拟荧光蛋白Corn-DFHO,“Corn”中文译为“玉米” [8]。Corn-DFHO拥有较好的光稳定性,可用于对活细胞中转录效率的定量分析。
以上拟荧光蛋白RNA生色团的设计思路全部来自荧光蛋白。而与此不同的是,2014年,来自加拿大西蒙弗雷泽大学的课题组利用已知能结合双链核酸的噻唑橙(thiazole Orange, TO1)的优势,对TO1进行生物素(biotin)修饰,并利用微流控技术相结合的SELEX技术,筛选得到了可以结合TO1-biotin的RNA 适配体,开发了基于花青素染料的荧光RNA,Mango-TO1-biotin[9]。该荧光RNA发射波长位于535 纳米,发出黄绿色荧光,所以取名“Mango”,中文译为“芒果”。近年来,他们对Mango进行突变优化,得到二代、三代、四代芒果(MangoⅡ、MangoⅢ、MangoⅣ),性能进一步提升。
无论是“菠菜”“西兰花”“玉米”还是“芒果”,RNA 适配体本身都有一个缺陷,即天生自带G-四链体。而细胞中存在天然识别G-四链体的核酸降解酶,这些荧光RNA在细胞中表达后,会很快被细胞识别并降解,这给它们发挥靶标RNA示踪功能带来了非常不利的影响。
2019年,笔者团队和朱麟勇教授合作开发了全新的荧光RNA,Pepper-HBC家族[10]。全新设计的荧光团分子HBC,经修饰后可得到HBC系列衍生物,与RNA 适配体Pepper结合后发出不同颜色的荧光(从485 纳米的青色荧光、530 纳米的绿色荧光、599 纳米的橙色荧光,到620 纳米的红色荧光),因而该RNA适配体起名“Pepper”,中文译为“辣椒”或“彩椒”。全新理性设计的HBC更易于被锁定在RNA适配体中,且背景荧光低、无细胞毒性。同时Pepper本身是单体参与结合,不含有G-四链体,与HBC的结合力也非常强,可达到纳摩尔级。此外,Pepper系列荧光RNA亮度高且稳定,无论是在体外还是活细胞内均具有出色的表现,这使得Pepper可实现对不同丰度功能性RNA的示踪研究,同时不影响它们的空间分布和动态变化。
研究人员联用Pepper和荧光蛋白,通过流式细胞术,同时检测Pepper标记的mRNA及其翻译的融合荧光蛋白的蛋白质水平,首次实现了大规模的活体单细胞mRNA的翻译调控分析。利用Pepper得到的翻译调控分析数据显示,人结肠癌细胞HCT-116、人胃癌细胞MKN-45、人神经胶质瘤细胞U-87、人食管癌细胞TE-1和人前列腺癌细胞PC-3等癌细胞系的R2数值(通过米氏方程拟合得出,表示相关性强度)均低于正常细胞系(小鼠胚胎成纤维细胞NIH/3T3和人胚肾细胞293T/17)的R2数值,这意味着在癌细胞中翻译调控存在失衡。此外,研究者们还利用Pepper实现了活细胞中RNA的动态追踪。结果显示,肿瘤细胞中蛋白质与其mRNA的含量存在着很弱的相关性,换而言之,肿瘤细胞中的翻译过程是严重失调的。这为肿瘤的诊疗提供了一种全新的思路和方法。
联用Pepper和CRISPR基因标记系统,可实现对不同基因位点的多色且可快速切换荧光的成像标记及分析。此外,Pepper620发射波长达到红色波段620 纳米,荧光亮度高、光稳定性强,可用于结构光照明超分辨成像(structure illumination microscopy,SIM),这也是首次实现荧光RNA对光学衍射极限的突破,为功能性天然RNA的超分辨成像提供新的强有力工具。因此,这些出色的性能使Pepper-HBC成为目前可以在哺乳动物细胞中对靶标RNA进行示踪的最佳熒光RNA。
荧光RNA的未来发展
荧光蛋白的发现源于人类对自然的观察与好奇,而荧光RNA的发展过程则表现出人类的理性思考,以及对生命本质的不断探索。从第一个可以在哺乳动物细胞中实现标记的荧光RNA算起,荧光RNA的发展还不足10年。这得益于其站在巨人的肩膀上,赶在生命科学蓬勃发展的黄金时代。Pepper作为目前表现效果最好的荧光RNA,除可用于活细胞内靶标RNA的示踪,进行基础的科学研究外,未来还可用于发展任意标靶的检测探针,为活细胞与活体生物传感、即时诊断,甚至实时诊断技术的发展提供新的机遇。
实际上荧光RNA的发展仍处在起步阶段,未来的荧光RNA需朝着远红、甚至近红外波段发展,提高亮度与稳定性,以实现单分子、超分辨成像,实现活体动物的靶标RNA实时监测,直至最终应用于临床。荧光RNA归根结底是一门生物技术,一个工具,如何用好这个工具推动生物学向前发展,解决人类在疾病诊疗中遇到的问题,真正造福于全人类,这需要各领域科研人员的共同努力。本文介绍的都是十分经典且成功的荧光RNA代表,其实还有数以亿计的候选荧光RNA被淘汰和淹没。科学之路从来都不会一帆风顺,也许真正值得铭记的是那些已被遗忘的无数次的失败与尝试,真正伟大的是那些在黑暗中仍旧不断求索的人。
[本文得到国家重点研发计划(2017YFA050400、2019YFA0904800),国家自然科学基金(91857202、21937004、31971349、31600688)和上海市科学技术基金(18JC1411900)的资助。]
[1]Crick F. Central dogma of molecular biology. Nature, 1970, 227(5258): 561-3.
[2]Cody C W, Prasher D C, Westler W M, et al. Chemical structure of the hexapeptide chromophore of the Aequorea green fluorescent protein. Biochemistry, 1993, 32(5): 1212-1218.
[3]Evelina T, Nathan M L, Robert H S, et al. Imaging mRNA in vivo, from birth to death. Annual review of biophysics, 2018, 47: 85-106.
[4]Jeremy R B, Stephen R A, Roger Y T. Aptamers switch on fluorescence of triphenylmethane dyes. Journal of the American Chemical Society, 2003, 125 (48): 14716-14717.
[5]Jeremy S P, Karen Y W, Samie R J. RNA mimics of green fluorescent protein. Science, 2011, 333 (6042): 642-646.
[6]Rita L S, Matthew D D, Samie R J. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods, 2013, 10 (12): 1219-1224.
[7]Grigory S F, Jared D M, Nina S, et al. Broccoli: rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society, 2014, 136 (46): 16299-16308.
[8]Wen J S, Grigory S F, Hyaeyeong K, et al. Imaging RNA polymerase III transcription using a photostable RNA-fluorophore complex. Nature Chemical Biology, 2017, 13 (11): 1187-1194.
[9]Elena V D, Sunny C Y, Shanker S S, et al. RNA mango aptamerfluorophore: a bright, high-affinity complex for RNA labeling and tracking. ACS Chemical Biology, 2014, 9 (10): 2412-2420.
[10]Xiaojun C, Dasheng Z, Ni S, et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology, 2019, 37:1287-1293.
关键词:荧光RNA 荧光蛋白 活细胞成像 ■

