基于特有基因VY93_RS02885为靶标的滑液支原体qPCR检测方法建立
徐彬 张敬峰 卢凤英 孙华伟 赵莎 张晓曦 刘青涛 张小飞

摘要:利用CD-HIT-EST快速聚类分析和BLASTN同源性比对共鉴定出17个滑液支原体(Mycoplasma synoviae)特有基因,并从中选取保守基因VY93_RS02885作为分子诊断的靶标基因。根据该基因核苷酸序列设计了1对引物,建立了滑液支原体的SYBR green qPCR检测方法。结果表明,该方法对不同浓度的模板和对应的CT值具有较好的线性关系,R2值为0.998 8;该方法的特异性较好,BLASTN比对,检测引物对仅能匹配滑液支原体的特有基因VY93_RS02885,结果显示,该方法能特异性地检测滑液支原体,与其他常见的引起禽关节炎的细菌/支原体性病原无交叉反应;该方法的敏感性较好,100%阳性检测的靶标基因最小拷贝数为10;该方法的重复性较好,对3个不同浓度的模板进行组间和组内重复检测,变异系数≤1.172 7%。此外,该方法对临床样品的检出率与传统检测方法相符。因此,本研究建立的针对滑液支原体特有基因的SYBR green qPCR检测方法具有用时短、特异性高、准确性高、敏感性高、可重复性强的优点,可为滑液支原体的快速诊断和流行病学调查提供技术手段。
关键词:滑液支原體;特有基因;qPCR;检测方法
中图分类号:S852.62 文献标志码: A文章编号:1002-1302(2021)19-0183-07
滑液支原体(Mycoplasma synoviae)是一种重要的禽病病原,是OIE必通报疫病病原。该病原所致疾病的致死率不高,但能导致鸡关节炎、气囊炎、生长迟缓、产蛋量下降、产畸形蛋并增加鸡对其他病原的易感性和疾病程度等,疾病发展缓慢,病程长。该病原在鸡群中感染后很难根除,对家禽养殖业造成严重的经济损失[1-3]。
对滑液支原体的快速诊断对该病原所致疾病的防控非常重要[4],荧光定量PCR(qPCR)相对于常规PCR具有反应时间短、更高的敏感性、结果不用跑胶直接获得等优点。现阶段应用最广泛的qPCR方法分别是荧光探针法qPCR(如TaqMan qPCR)和核酸染料法qPCR(如SYBR green qPCR)。对于单基因检测,SYBR green qPCR相对于TaqMan qPCR等荧光探针法省去了探针的合成,经济上更实惠。许多报道也表明SYBR green qPCR具有不输于TaqMan qPCR的准确性和敏感性[5-6]。因此,本研究着眼于建立滑液支原体SYBR green qPCR的诊断方法。
到目前为止,针对滑液支原体的SYBR green qPCR检测方法仅有Jarquin等建立的以16S rRNA的基因序列为靶标的方法。多项研究表明,仅针对单一基因位点进行病原检测,可能出现由于靶标病原基因引物区的突变、其他物种同源性较高基因的非特异性扩增等诸多情况导致结果不可靠[7]。如16S rRNA属于所有物种的共有基因,在不同物种特别是近源物种之间具有不低甚至较高的同源性。Mekkes等建立的针对鸡毒支原体(Mycoplasma gallisepticum)16S rRNA的检测方法能非特异地检测出模仿支原体(Mycoplasma imitans)[8]。因此,建立针对其他靶点特别是特异性更高靶点的SYBR green qPCR具有研究价值。本研究选择以滑液支原体的特有基因作为诊断靶标建立SYBR green qPCR快速诊断方法,为滑液支原体的临床诊断和流行病学调查提供有效手段。
1材料与方法
1.1菌种和病料
试验用滑液支原体标准株ATCC 25204、20株滑液支原体临床分离株、3株鸡毒支原体、6株金黄色葡萄球菌(Staphylococcus aureus)、4株禽致病性大肠杆菌(Avian pathogenic Escherichia coli)、2株鸡白痢沙门氏菌(Salmonella pullorum)、3株鸡伤寒沙门氏菌(Salmonella gullinarum)、3株鼠伤寒沙门氏菌(Salmonella typhimurium)、48份滑液支原体阳性鸡跗关节组织、10份SPF健康鸡跗关节组织均由江苏省农业科学院兽医研究所保存。试验时间自2019年底至2021年初,试验地点在江苏省农业科学院兽医研究所。
1.2主要仪器和试剂
北京格瑞德曼公司GT200组织细胞样品均质仪;美国ABI公司StepOnePlus荧光定量PCR仪;德国Eppendorf公司Mastercycler nexus gradient 多用途PCR仪和BioPhotometer D30核酸蛋白测定仪。2×Taq Plus Master Mix Ⅱ(Dye Plus),AceQ?qPCR SYBR Green Master Mix (High ROX Premixed)购自Vazyme公司;pMD18-T Vector Cloning Kit,MiniBEST Bacteria Genomic DNA Extraction Kit Ver.3.0购自TaKaRa公司;Mycoplasma gDNA Miniprep Kit购自BIOMIGA公司;Gel Extraction kit和Plasmid Mini Kit Ⅰ购自OMEGA公司。
1.3生物信息学分析筛选和鉴定滑液支原体核苷酸水平特有基因
从NCBI GenBank中收集包括6株滑液支原体在内的46株来自25种/亚种动物致病性支原体的全基因组所有基因的核苷酸序列集。46株支原体分别是:鸭支原体(Mycoplasma anatis)NCTC10156株;关节炎支原体(Mycoplasma arthritidis)158L3-1株和NCTC10162株;牛生殖支原体(Mycoplasma bovigenitalium)HAZ596株和NCTC10122株;牛鼻支原体(Mycoplasma bovirhinis) GS01株和HAZ141_2株;牛支原体(Mycoplasma bovis) NADC58株和Ningxia-1株;加州支原体(Mycoplasma californicum) HAZ106_1株和ST-6株;犬支原体(Mycoplasma canis)LV株和PG 14株;山羊支原体山羊亚种(Mycoplasma capricolum subsp. capricolum)ATCC 27343株;山羊支原体山羊肺炎亚种(Mycoplasma capricolum subsp. capripneumoniae)04012株和87001株;鸽口支原体(Mycoplasma columborale)NCTC10179株;犬支原体(Mycoplasma cynos)C142株;猫支原体(Mycoplasma felis)Myco-2株;禽支原体(Mycoplasma gallinaceum)NCTC10183株和Peacock20181011株;生殖支原体(Mycoplasma genitalium)G37株和M6282株;嗜肝糖支原体(Mycoplasma glycophilum)NCTC10194株;猪肺炎支原体(Mycoplasma hyopneumoniae)168株和7448株;猪鼻支原体(Mycoplasma hyorhinis)DBS 1050株和HUB-1;猪滑液支原体(Mycoplasma hyosynoviae)M60株;衣阿华支原体(Mycoplasma iowae)695株和NCTC10185株;火鸡支原体(Mycoplasma meleagridis)NCTC10153株;丝状支原体丝状亚种(Mycoplasma mycoides subsp. mycoides)Ben1株和T1/44株;肺炎支原体(Mycoplasma pneumoniae)M29株和M2192株;雏支原体(Mycoplasma pullorum)B359_6株;鸡毒支原体mx-4株、NCTC10115株、str. Rlow株;滑液支原体53株、86079/7NS株、ATCC 25204株、HN01株、MS-H株、NCTC10124株。
将核苷酸同源性的阈值设置为99%、核苷酸序列长度的差异阈值设置为70%,其他参数均为默认参数,利用CD-HIT-EST快速聚类方法(v 4.8.1)[9]获取上面所提46株支原体的特有基因和共有基因。选取6株滑液支原体共有且相对于其他支原体特有的基因集进行BLASTN分析,鉴定滑液支原体相对于已知基因序列的其他物种的核苷酸水平特有基因。
1.4支原体、细菌、跗关节组织基因组DNA的提取
支原体的基因组DNA利用Mycoplasma gDNA Miniprep Kit并按用户手册提取,细菌的基因组DNA利用MiniBEST Bacteria Genomic DNA Extraction Kit Ver.3.0并按用户手册提取。
取“1.1”节中病料和健康组织,经反复冻融2~3次、剪碎、按质量体积比1 ∶3与PBS(pH值7.4)混合后,利用均質仪进行组织的充分均质。均质物经6 000 r/s离心5 min,取上清液用于基因组DNA的提取。利用Mycoplasma gDNA Miniprep Kit并按用户手册提取各样品基因组DNA。
使用核酸蛋白测定仪测定各样品中DNA的含量。获得的样品基因组DNA立即使用,或置于 -40 ℃ 冰箱中保存,避免反复冻融。
1.5引物设计与合成
利用Primer Premier 5软件对引物进行设计。qPCR检测的上游引物命名为qMS-2885F;下游引物命名为qMS-2885R。设计好的引物经BLASTN分析是否能非特异性地结合除滑液支原体外其他物种的DNA。同时设计用于常规PCR扩增VY93_RS02885基因片段的引物对MS-2885F和MS-2885R。引物序列信息见表1,交由南京擎科生物科技有限公司进行引物合成。
1.6阳性重组质粒的构建
以滑液支原体标准株ATCC 25204的基因组DNA为模板,以MS-2885F和MS-2885R为引物,利用2×Taq Plus Master Mix Ⅱ(Dye Plus)并按用户手册设置PCR反应体系和反应程序,扩增基因片段,基因片段命名为MS-2885。
将MS-2885利用pMD18-T Vector Cloning Kit并按用户手册连接至pMD-18T。将连接产物转化至DH5α。经PCR检测含有阳性重组质粒pMD-18T::MS-2885的DH5α送至南京擎科生物科技有限公司测序。测序结果显示没有碱基突变的pMD-18T::MS-2885作为后续使用的阳性重组质粒。
1.7SYBR green qPCR反应体系和反应程序
按照AceQ?qPCR SYBR Green Master Mix(High ROX Premixed)的用户手册设置qPCR反应体系和程序。具体如下:
qPCR反应体系(20 μL):AceQ?qPCR SYBR Green Master Mix(High ROX Premixed)(2X)10 μL,10 μmol/L的qMS-2885F和qMS-2885R各 0.4 μL,样品DNA 1 μL(浓度<100 ng/μL),其余用灭菌双蒸水补齐。
qPCR反应程序:预变性,95 ℃ 5 min;PCR反应,95 ℃ 10 s、60 ℃ 30s,40个循环;按仪器默认设置进行解离步骤。
1.8标准曲线的建立和溶解曲线分析
将“1.6”节中所得阳性重组质粒pMD-18T::MS-2885进行DNA浓度测定和调整及10倍梯度稀释。以1×109到10 拷贝/μL的质粒为模板,参照“1.7”节反应体系和反应程序进行SYBR green qPCR检测,并绘制标准曲线和进行熔解曲线分析。
1.9敏感性分析
以1 000、100、10、5、2.5、1 拷贝/反应的阳性重组质粒为模板,分别进行10次“1.7”节所述qPCR反应,统计每个拷贝能获得CT值的百分率。以100%能阳性扩增获得CT值的最小靶标基因拷贝数作为检测极限。
1.10重复性分析
以1×106、1×105、1×104拷贝/反应的阳性重组质粒为模板,分别进行4次(组间)“1.7”节所述qPCR反应,每次反应相同条件设置4个重复(组内)。计算组内、组间CT值的平均值、标准差和变异系数。
1.11特异性分析
取鸡毒支原体、禽致病性大肠杆菌、金黄色葡萄球菌、鸡白痢沙门氏菌、鸡伤寒沙门氏菌、鼠伤寒沙门氏菌和滑液支原体标准株和临床分离株的基因组DNA。测定各基因组DNA的浓度并将浓度调整到1 ng/μL。以各菌/支原体基因组DNA为模板进行“1.7”节所述qPCR反应。以灭菌双蒸水为阴性对照。每组qPCR反应进行3次独立试验。
1.12滑液支原体阳性鸡组织和SPF健康鸡组织的检测
取实验室保存的48份基于病原分离培养、基因组DNA提取、常规PCR鉴定获得的滑液支原体阳性鸡跗关节组织和10份SPF健康鸡跗关节组织,提取这些鸡跗关节组织的基因组DNA,并测定和调整DNA浓度到1 ng/μL。以这些基因组DNA为模板进行“1.7”节所述qPCR反应。以灭菌双蒸水为阴性对照,每组qPCR反应进行3次独立试验。
2结果与分析
2.1滑液支原体核苷酸水平特有基因的筛选和后续分子诊断靶标的确定
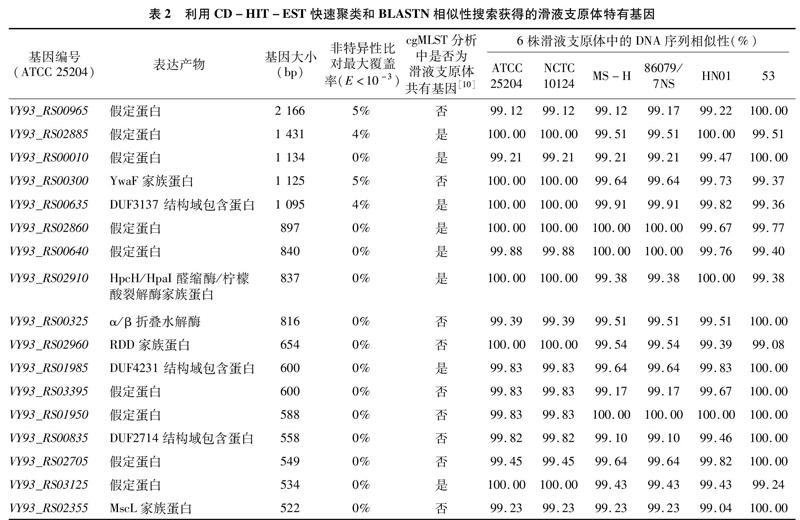
CD-HIT-EST快速聚类显示46株支原体中总共有22 044个泛基因集。进一步筛查,总共有433个基因集是6株滑液支原体所共有且相对于其他40株支原体特有。以往的研究报道将BLASTN比对下,在E值<103的情况下,匹配的非本物种基因覆盖度不大于40%的基因作为该物种的特有基因。将这433个基因集进行逐一的BLASTN比对,符合这一条件的特有基因集有96个。其中,在E值<103的情况下,匹配的非滑液支原体基因覆盖度不大于5%的基因集总共有27个。由表2可知,为方便后续用于分子诊断靶标特有基因的确定,仅选取>500 bp的17个特有基因集用作后续分析;这17个基因集包含的基因均为各滑液支原体中的单拷贝基因。参照先前研究报道对滑液支原体共有基因的鉴定[10],最终选择编码假定蛋白的特有基因VY93_RS02885作为后续分子诊断的靶标。
2.2引物的设计和特异性分析
利用Primer Premier 5在VY93_RS02885的开放阅读框中设计SYBR green qPCR诊断引物,预测的上游引物和下游引物的Tm值分别为59.4 ℃和596 ℃。引物扩增的片段大小为122 bp(表1)。经过BLASTN比对,该引物对仅能匹配滑液支原体的基因VY93_RS02885,说明这对引物的特异性良好。
2.3标准曲线的建立和溶解曲线分析
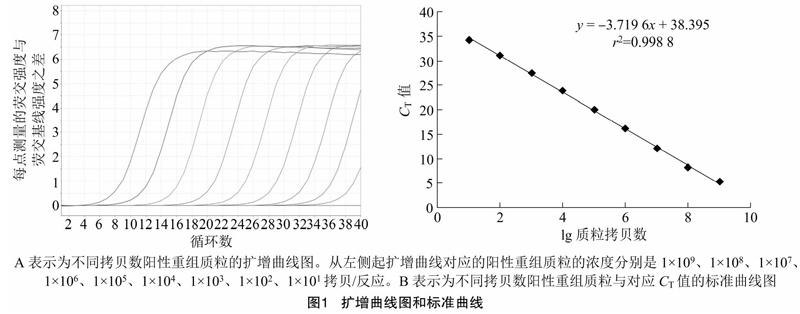
以10倍稀释所构建的阳性重组质粒,用上述引物对进行SYBR green qPCR检测。由图1可知,不同浓度的模板均能扩增出典型的“S”形扩增曲线,采用模板浓度从1×109~1×10拷贝/反应及其对应的扩增CT值作的标准曲线呈现良好的线性关系。以模板浓度的lg值为横坐标,以扩增CT值为纵坐标,所作标准曲线的方程式为y=-3.719 6x+38395,r2为0.998 8。由图2可知,熔解温度(Tm)在75.54 ℃左右出现狭窄、单一的特异性峰,没有引物二聚体及非特异性扩增的峰,说明所构建的方法特异性较好。特异性扩增峰的Tm值相对较低,可能与滑液支原体基因的GC比在28%左右有关[11]。
2.4敏感性分析
将阳性重组质粒倍比稀释到1 000、100、10、5、2.5、1拷贝/反应并作为模板,以本研究建立的qPCR方法分别扩增10次,结果(图3)表明,当模板在1 000、100、10拷贝/反应时,均能100%扩增出CT值;当模板在5.0、2.5、1.0拷贝/反应时,分别有90%、90%、80%的概率扩增出CT值。这说明本研究构建的SYBR green qPCR检测方法的敏感性为10拷贝/反应。
2.5重复性分析
由表3可知,将3种不同浓度的阳性重组质粒模板分别组内和组间重复4次,组内标准差在0031 0~0.155 1间,变异系数在0.153 5%到 0.651 5% 之间;组间标准差在0.178 4到0.235 6之间,变异系数在0.747 1%~1.165 8%间,说明本研究建立的SYBR green qPCR检测方法的可重复性较好。表3重复性分析结果
项目1×106拷贝CT值1×105拷贝CT值1×104拷贝CT值平均值标准差变异系数(%)平均值标准差变异系数(%)平均值标准差变异系数(%)第一次(组内)15.923 80.043 50.273 120.200 30.031 00.153 523.811 50.155 10.651 5第二次(组内)15.897 50.073 10.460 019.818 80.092 40.466 424.042 00.078 30.325 6第三次(组内)16.174 00.104 30.645 020.387 30.055 60.272 723.697 30.083 40.351 9第四次(组内)16.296 00.082 20.504 319.945 80.070 90.355 323.985 50.146 80.611 9组间16.072 80.187 41.165 820.088 00.235 61.172 723.884 10.178 40.747 1
2.6特异性分析
利用本研究建立的SYBR green qPCR诊断方法检测滑液支原体标准株ATCC 25204和20株临床分离株。由图4可知,结果均能扩增出现典型的“S”形扩增曲线,且CT值在13.595~17.377间;检测其他6种临床上常见的引起鸡关节炎的细菌/支原体性病原和灭菌双蒸水均检测不到CT值,结果说明本研究建立的SYBR green qPCR检测方法具有较高的特异性。
2.7鸡组织中病原检测
用本研究建立的SYBR green qPCR诊断方法检测实验室保存的48份滑液支原体阳性鸡跗关节组织样品的基因组DNA,结果显示均能扩增出CT值,最大的CT值为29.155;检测实验室保存的10份SPF健康鸡的跗关节组织基因组DNA和灭菌双蒸水均不能扩增出CT值。说明本研究建立的诊断方法准确性较高。
3讨论
滑液支原体的生长缓慢,且没有针对滑液支原体的选择培养基,从宿主分离出来的滑液支原体,前2代液体培养往往需要1周甚至更长的时间。因此,病原分离不适用于滑液支原体感染的及时诊断,且病原分离后也需要进行例如常规PCR等方法鉴定。滑液支原体所致疾病发展缓慢,往往需要 1~3 周的时间才能刺激宿主产生抗体[12]。因此,ELISA和血凝抑制试验不适用于滑液支原体感染的早期诊断。多种多样的PCR检测方法成为了滑液支原体感染的快速诊断方法。
国外研究学者建立了以滑液支原体16S rRNA、23S rRNA及其周圍基因间序、vlhA保守区为靶标的常规PCR检测方法[13-15]。此外,针对以上3个靶点的TaqMan qPCR检测方法和针对16S rRNA的SYBR green qPCR检测方法也已建立[16-17]。鉴于临床上区分野毒和弱毒活疫苗的需要,逐步建立了以obg基因为靶标的SYBR green qPCR检测方法和以obg基因或者oppF基因为靶标的巢式PCR与高分辨率熔解曲线分析相结合的方法来鉴别MS-H疫苗株与野毒株[18-20]。以及建立了以HIT家族蛋白编码基因的单碱基突变为靶标的错配放大突变分析方法区分疫苗株MS1和野毒株[21]。2017年开始我国开放进口弱毒苗MS-H[(2017)外兽药证字33号],因此针对MS-H与野毒的鉴别诊断在我国也具有了临床意义。我国研究学者也以vlhA保守区为靶标建立了TaqMan qPCR诊断方法[22]。鉴于如前言所述单基因靶标的潜在问题、qPCR相对于常规PCR的检测敏感度、SYBR green qPCR的经济实惠等因素,本研究重点建立针对其他靶点的滑液支原体SYBR green qPCR检测方法。
PCR檢测的特异性是临床诊断的重要方面,是准确诊断何种病原所致感染的关键。本研究利用生物信息学手段鉴定出多个核苷酸水平上的滑液支原体特有基因,并结合国外报道的滑液支原体共有核心基因来选择qPCR诊断的靶标,以从靶标选择上确保PCR检测的特异性。本研究鉴定的特有基因也为其他研究学者建立相关的分子诊断、病原基因分型方法提供备选靶标。
qPCR的敏感性是该分子诊断方法的另一个重要方面。想要做到及时快速诊断,就要省略病原分离步骤,从发病甚至疑似发病的组织或者气管拭子、鼻拭子等直接提取基因组DNA用于诊断。从宿主组织中提取的DNA会掺杂很高比例的宿主基因组DNA。从拭子中提取的DNA又往往浓度不高。在这些情况下,就需要qPCR的检测敏感性足够高。本研究建立的qPCR诊断方法的敏感性在10拷贝/反应,高于国外针对16S rRNA所建立的SYBR green qPCR检测方法1倍以上[12]。这对待检样品中病原含量较低时的快速准确诊断具有积极意义。
本研究建立的以特有基因为靶标的滑液支原体SYBR green qPCR检测方法具有用时短、特异性高、敏感性高、可重复性强、经济实惠等优点,能满足大批量样品的快速准确鉴定,有利于滑液支原体感染的早期诊断、及时治疗和预防病原的进一步扩散,有利于减少滑液支原体感染所致养禽业的经济损失,促进养禽业的健康发展。
参考文献:
[1]Dufour-Gesbert F,Dheilly A,Marois C,et al. Epidemiological study on Mycoplasma synoviae infection in layers[J]. Veterinary Microbiology,2006,114(1/2):148-154.
[2]Kursa O,Pakua A,Tomczyk G,et al. Eggshell apex abnormalities caused by two different Mycoplasma synoviae genotypes and evaluation of eggshell anomalies by full-field optical coherence tomography[J]. BMC Veterinary Research,2019,15(1):1.
[3]Oh K,Lee S,Seo J,et al. Rapid serodiagnosis with the use of surface plasmon resonance imaging for the detection of antibodies against major surface protein A of Mycoplasma synoviae in chickens[J]. Canadian Journal of Veterinary Research-Revue Canadienne de Recherche Veterinaire,2010,74(1):71-74.
[4]Kleven S H. Control of avian mycoplasma infections in commercial poultry[J]. Avian Diseases,2008,52(3):367-374.
[5]Tajadini M,Panjehpour M,Javanmard S H. Comparison of SYBR green and TaqMan methods in quantitative real-time polymerase chain reaction analysis of four adenosine receptor subtypes[J]. Advanced Biomedical Research,2014,3:85.
[6]Gomes C M,Cesetti M V,de Paula N A,et al. Field validation of SYBR green- and TaqMan-Based Real-Time PCR using biopsy and swab samples to diagnose American tegumentary leishmaniasis in an area where Leishmania (Viannia) braziliensis is endemic[J]. Journal of Clinical Microbiology,2017,55(2):526-534.
[7]Diene S M,Bertelli C,Pillonel T,et al. Comparative genomics of Neisseria meningitidis strains:new targets for molecular diagnostics[J]. Clinical Microbiology and Infection,2016,22(6):1-7.
[8]Mekkes D R,Feberwee A. Real-time polymerase chain reaction for the qualitative and quantitative detection of Mycoplasma gallisepticum[J]. Avian Pathology,2005,34(4):348-354.
[9]Huang Y,Niu B,Gao Y,et al. CD-HIT suite:a web server for clustering and comparing biological sequences[J]. Bioinformatics,2010,26(5):680-682.
[10]Ghanem M,El-Gazzar M. Development of Mycoplasma synoviae (MS) core genome multilocus sequence typing (cgMLST) scheme[J]. Veterinary Microbiology,2018,218:84-89.
[11]Xu B,Liu R,Tao L,et al. Complete genome sequencing of Mycoplasma synoviae strain HN01,isolated from chicken in Henan province,China[J]. Microbiology Resource Announcements,2020,9(6):e01419-e01480.
[12]Jarquin R,Schultz J,Hanning I,et al. Development of a real-time polymerase chain reaction assay for the simultaneous detection of Mycoplasma gallisepticum and Mycoplasma synoviae under industry conditions[J]. Avian Diseases,2009,53(1):73-77.
[13]Lauerman L H,Hoerr F J,Sharpton A R,et al. Development and application of a polymerase chain reaction assay for Mycoplasma synoviae[J]. Avian Diseases,1993,37(3):829-834.
[14]García M,Jackwood M W,Head M,et al. Use of species-specific oligonucleotide probes to detect Mycoplasma gallisepticum,M. synoviae,and M. iowae PCR amplification products[J]. Journal of Veterinary Diagnostic Investigation,1996,8(1):56-63.
[15]Ramírez A S,Naylor C J,Hammond P P,et al. Development and evaluation of a diagnostic PCR for Mycoplasma synoviae using primers located in the intergenic spacer region and the 23S rRNA gene[J]. Veterinary Microbiology,2006,118(1/2):76-82.
[16]Fraga A P,de Vargas T,Ikuta N,et al. A multiplex real-time PCR for detection of Mycoplasma gallisepticum and Mycoplasma synoviae in clinical samples from Brazilian commercial poultry flocks[J]. Brazilian Journal of Microbiology,2013,44(2):505-510.
[17]Sprygin A V,Andreychuk D B,Kolotilov A N,et al. Development of a duplex real-time TaqMan PCR assay with an internal control for the detection of Mycoplasma gallisepticum and Mycoplasma synoviae in clinical samples from commercial and backyard poultry[J]. Avian Pathology,2010,39(2):99-109.
[18]Shahid M,Markham P F,Marenda M S,et al. High-resolution melting-curve analysis of obg gene to differentiate the temperature-sensitive Mycoplasma synoviae vaccine strain MS-H from non-temperature-sensitive strains[J]. PLoS One,2014,9(3):e92215.
[19]Zhu L,Konsak B M,Olaogun O M,et al. Identification of a new genetic marker in Mycoplasma synoviae vaccine strain MS-H and development of a strategy using polymerase chain reaction and high-resolution melting curve analysis for differentiating MS-H from field strains[J]. Veterinary Microbiology,2017,210:49-55.
[20]Dijkman R,Feberwee A,Landman W. Development,validation and field evaluation of a quantitative real-time PCR able to differentiate between field Mycoplasma synoviae and the MS-H-live vaccine strain[J]. Avian Pathology :2017,46(4):403-415.
[21]Kreizinger Z,Sulyok K M,Grózner D,et al. Development of mismatch amplification mutation assays for the differentiation of MS1 vaccine strain from wild-type Mycoplasma synoviae and MS-H vaccine strains[J]. PLoS One,2017,12(4):e0175969.
[22]黃莉,谢芝勋,王盛,等. 鸡滑液囊支原体和禽呼肠病毒的实时荧光定量PCR快速检测[J]. 动物医学进展,2018,39(2):1-6
基金项目:国家重点研发计划(编号:2017YFD0500706);国家自然科学基金(编号:32002291)
作者简介:徐彬(1987—),男,河北廊坊人,博士,助理研究员,主要从事动物传染病防治研究。E-mail:xubin9999@foxmail.com。
通信作者:张小飞,博士,研究员,主要从事动物传染病防治研究。E-mail:xiaofei0804@sina.com。

