基于ESI的全球作物生物育种领域研究前沿分析
齐世杰 赵静娟 郑怀国

摘要:全球作物生物育种已进入至关重要的、以抢占技术制高点与经济增长点为目标的战略机遇期,基础研究是科技创新的重要途经,明晰全球生物育种领域的研究前沿和发展方向,对我国作物生物育种发展来说具有迫切性和必要性。基于ESI数据平台,经专家咨询对全球生物育种领域研究前沿和核心论文进行遴选与解读,借助DDA分析工具,采用计量分析和数据挖掘方法,从期刊、国家、机构及合作、研究方向等角度对核心论文进行全方位的深入剖析。Plant Biotechnology Journal是作物生物育种领域前沿的代表期刊;美国和中国是全球重点研究国家;我国代表性研究机构优势显著,但影响力有待提升,可根据机构特色研究方向,有针对性地积极开展跨国合作;基因编辑技术、碱基编辑等生物育种技术及应用是重点关注的研究前沿。
關键词:作物生物育种;研究前沿;高被引论文;文献计量;DDA;基因编辑技术;基因组学;高通量表型平台
中图分类号:S336 文献标志码: A文章编号:1002-1302(2021)19-0009-10
农作物种业是国家战略性、基础性核心产业,事关粮食安全、民族兴衰。种业发展可以分为4个阶段:第1阶段:农家育种时代;第2阶段:杂交育种时代;第3阶段:分子育种时代;第4阶段:“生物技术+人工智能+大数据信息技术”育种时代[1]。发达国家已经进入第2阶段至第3阶段之间。2021年,生物育种作为前瞻性、战略性国家重大科技项目之一,被写入国家“十四五”规划纲要,是种子产业变革的重大举措。育种基础科学创新能力的提升从源头上支撑了我国现代种业发展[2]。因此,把握全球作物生物育种研究热点,追踪作物生物育种技术前沿,对于管理决策者和研究者都具有十分重要的意义。
生物育种是利用遗传学、细胞生物学、现代生物工程技术等方法原理培育生物新品种的过程,主要包括转基因育种、分子辅助标记、分子设计育种、基因组学育种,以及基因编辑育种。近年来,随着高通量测序平台、计算机技术的发展,作物生物育种研究获得了重大的突破。通过跟踪全球的研究前沿与核心论文,从期刊、资助基金、学科领域、国家、机构进行多维度分析,有助于对作物生物育种的研究前沿进行全方位的了解,进而揭示领域热门研究和技术前沿的主要贡献者和潜在合作者。
研究前沿能提供一个独特的视角来揭示作物生物育种的研究脉络,可以简明扼要地概括该领域的最新进展和前沿技术,为未来研究方向的把握拓展奠定基础。
1材料与方法
1.1数据来源及范围
基本科学指标(Essential Science Indicators,简称ESI)数据库是科睿唯安在汇集和分析Web of Science核心合集(SCIE、SSCI)所收录的学术文献及其所引用的参考文献的基础上建立起来的分析型数据库。研究前沿(Research Frontier)是基于该数据库中近5年内的高被引论文的引证关系,采用共被引分析计算得出,这1组高被引论文称为“核心论文”,每2个月更新1次[3]。这些论文及相关研究受到了同行高度认可和关注,代表着该领域最具影响力和发展前景的研究方向。研究前沿的遴选工作基于ESI数据库中的研究前沿以及前沿所对应的核心论文,数据截至2020年6月。
1.2研究前沿遴选
利用ESI数据库,经专家研讨和文献分析,为保证查全率,首先筛选出与“作物生物育种”相关的研究前沿137个,作为研究前沿遴选的基础数据;然后建立情报专家和农业领域专家团队,对137个前沿进行2轮反复研讨,剔除不相干前沿36个,以研究前沿对应的核心论文超过5篇为筛选目标,经过剔除和合并,最终确定了5个研究前沿,并获得研究前沿与前沿对应的核心论文103篇。
2研究前沿计量分析
2.1核心论文概况
对作物生物育种领域ESI研究前沿核心论文103篇进行分析,主要分布在2014—2019年,发文量分别是21、12、20、24、11、15篇。学科方向主要涉及植物科学62篇,科学、技术等多学科科学20篇,生物化学与分子生物学16篇、生物技术应用微生物学16篇、遗传学13篇。103篇核心文献中,基金资助论文94篇,基金资助率达到92%,主要资助机构为中国国家自然科学基金(NSFC)、英国生物技术与生物科学研究委员会(BBSRC)、美国国家科学基金会(NSF)、中国科学院基金项目以及澳大利亚研究委员会。
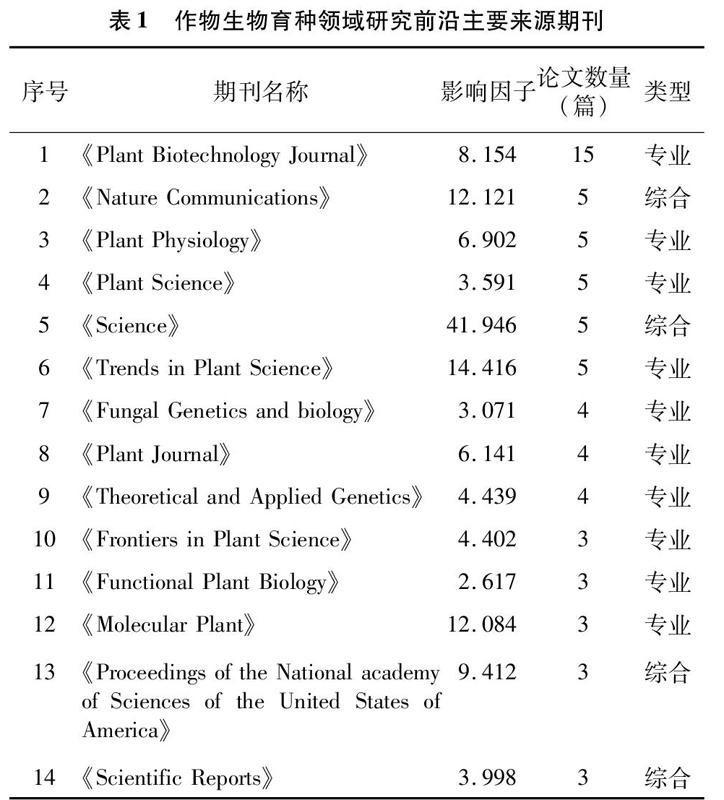
2.2主要来源出版物
研究前沿论文的来源期刊共有41本,其中既包括《Nature Communications》《Science》等综合性顶级期刊,也包括生物、农业、遗传学等学科领域的专业期刊,如《Plant Science》(植物科学)、《Fungal Genetics and biology》(遗传学)等。其中发表3篇及以上的期刊如表1所示,《Plant Biotechnology Journal》期刊刊载的论文数量最多,为15篇,反映了该刊在作物生物育种领域具有较强的专业性和前瞻性,刊载的论文对该领域的发展方向具有一定的引领作用,是作物生物育种领域研究前沿的重要来源期刊。其次是《Plant Physiology》《Plant Science》《Trends in Plant Science》3本专业性期刊,以及《Nature Communications》和《Science》2本综合性期刊,载文量均为5篇,也是作物生物育种领域研究前沿的代表性期刊。
2.3重点代表国家
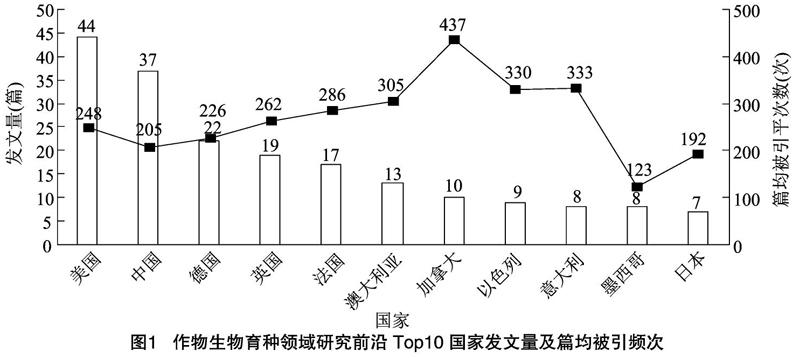
从统计数据来看,作物生物育种领域的热点前沿论文共涉及39个国家/地区,利用数理统计和 TF-IDF 算法对排名前10的11个国家的发文量、篇均被引频次和主要关键词进行计算,从数量、影响力和研究内容3个方面,对该领域研究前沿的主要国家进行深入分析(图1、表2)。
从文章数量来看,美国是本领域研究前沿核心论文的主要来源国家,共发表论文44篇,中国37篇排名第2。 以上2个国家位于第1梯队, 遥遥领先
于其他国家,是作物生物育种领域研究前沿的突出代表国。德国、英国、法国和澳大利亚位于第2梯队,发文量占10%以上,是该领域欧洲和大洋洲的主要代表国家。加拿大、以色列、意大利等5个国家位于第3梯队,以亚洲、欧洲和北美国家为主。
从篇均被引频次(图1折线)和研究主题(表2)来看,加拿大排名第1,篇均被引频次超出排名第2的意大利(333次)100余次,一定程度上反映出加拿大在生物育种领域的基础研究具有较高质量和影响力,特别是在多倍体(小麦)基因组、基因组学育种方面。意大利、以色列和澳大利亚位于第2梯队,篇均被引频次在300~400次之间,竞争优势显著,且具有“少而精”的发文特点,具有较大的发展潜力和合作空间,研究主题集中在利用QTL技术对番茄和水稻进行作物改良方面。法国、英国、美国、德国和中国位处第3梯队,篇均被引频次在200~300次之间,具有发文活跃、研究方向多元化、影响力一般的发文特点,整体发展较好,特别是中国、美国2个农业大国,已经率先在CRISPR/Cas9 技术、基因编辑等生物育种技术方面进行探索,也开展了复杂农艺性状的相关研究,相对而言,影响力有待进一步提升。日本、墨西哥位处第5梯队,处于追赶的状态,在提升优良小麦群体产量、植物表型鉴定方面表现突出
2.4重要研究机构及合作
作物生物育种领域的研究前沿共有280个研究机构主导或参与,利用DDA分析工具,采用TF-IDF算法对前10个机构的研究主题词进行提取,并绘制出10个机构的合作情况(表3、图2)。
全球作物生物育种领域研究前沿前10个机构中包括6所高等院校和4个科研院所,发展较为均衡。从发文数量(表3)来看,前10个研究机构具有明显的阶梯特征,中国农业科学院发表的前沿核心论文最多,研究集中在复杂农艺性状、甘蓝型油菜、作物驯化、全基因组关联方面,产出能力较强,处于世界领先地位。美国农业部农业研究院排名第2,在遗传多样性、基因分型、多倍体小麦、单核苷酸多态性方面表现突出,以上2个机构位于第1梯队,是该领域基础研究的主要产出机构。中国科学院和华中农业大学位于第2梯队,科研氛围较为活跃,特别是在QTL、CRISPR/Cas9、基因编辑技术、基因组工程、异源四倍体等育种技术方面,研究对象包括小麦、水稻、番茄、棉花,是我国作物生物育种领域的代表性研究机构。其他机构位于第3梯队,发文量相差不大,但研究方向各有不同,法国国家农业科学研究院侧重在QTL和全基因组关联分析[4],以及作物生长模型和基因组预测[5]方面;加州大学戴维斯分校在多倍体小麦的基因分型、基因编辑技术在香蕉抗病性育种[6]、提升油茶品质[7]中的应用有所突破;约翰·英纳斯研究中心在下一代测序技术、六倍体小麦的基因分型[8-9]阵列进行相关探究;国际玉米和小麦改良中心在利用遥感、无人机等智能育种技术方面有所进展[10-12];堪萨斯州立大学在基因组育种方法[13-14]上进行了探究。
结合篇均被引频次分析,法国国家农业科学研究院(351次)排名第1,在本领域具有较强的影响力和行业关注度。加州大学戴维斯分校和堪萨斯州立大学,属于“少而精”的研究机构,竞争优势显著,能够引起同领域科研人员的高度关注。其次是美国农业部农业研究院和华中农业大学,能够兼顾论文的数量与质量,整体发展相对较好。中国农业科学院、中国科学院2个机构,相对于发文量而言,其篇均被引频次表现一般,仍有一定的发展潜力和上升空间。但整体而言,排名前5位的研究机构中我国机构占据3个,证明我国研究机构的力量十分雄厚。约翰英纳斯研究中心和国际玉米和小麦改良中心处于追赶阶段。
作物生物育种领域研究前沿Top10机构的合作情况见图2,图2中节点代表研究机构,节点大小代表发文量的多少,连线的实虚代表合作的强弱。从图2可以看出,重要机构间合作呈现出以中、美2国的国内合作为主,跨国合作逐步形成的特点,包括2个较为明显的国内合作团体:(1)美国农业部农业研究局、堪萨斯州立大学和加州大学戴维斯分校之间较强的合作网络;(2)以中国农业科学院为主导的,联合中国科学院、华中农业大学之间的合作关系。此外,康奈尔大学的国际合作表现突出,在全球各国间起到重要的链接作用。约翰英纳斯研究中心的合作关系较弱,偏向于机构内部团队间的合作。
3研究前沿重点解析
对全球生物育种领域的五大前沿及前沿对应的核心论文信息进行整理。从表4可以看出, 全球
生物育种领域的五大前沿分别是:(1)基因组学分析在作物育种中的应用;(2)高效基因编辑技术及其在作物育种中的应用;(3)高通量表型平台与植物育种;(4)雄性不育与作物杂种优势利用;(5)野生亲缘关系在作物育种中的研究。其中前沿(1)“基因组学分析在作物育种中的应用”核心论文最多,是重点研究前沿。前沿(5)“野生亲缘关系在作物育种中的研究”相对较新,核心论文平均出版年2017年,是新兴研究前沿。
3.1基因组学分析在作物育种中的应用
基因组测序技术的快速发展使作物基因组研究取得了突破性进展。继2002年第一个水稻基因組测序完成之后,世界各国已完成或接近完成64种作物的基因组测序。我国是世界上较早启动作物基因组学研究的国家,已完成了世界上70%~80%重要作物的基因组测序。此外,还开发了基于高通量基因组测序的基因型鉴定方法,成功开展了水稻、玉米重要农艺性状的基因组关联分析和功能研究。
分子标记辅助育种(MAS)是作物改良的有效手段。研究前沿中首次挖掘出小麦抗赤霉病的TaHRC-R基因[15]和甘蓝型油菜产油量的新的QTL位点[16],为新品种的培育提供了靶位点和筛选手段。利用高通量SNP芯片技术、基因分型测序(GBS)以及重测序技术,结合表型组、代谢组、蛋白组等组学技术,全基因组关联分析(GWAS)获得了与性状直接关联的SNP标记,提供了分子辅助育种候选位点。小麦中,研究人员设计了35、90[17]、660 K[18]芯片,获得了产量相关因素、耐旱性相关QTLs,提供了检测基因渗入的SNP位点。番茄中,进行了番茄属的属间和种间多样性研究,挖掘出控制番茄风味的化学物质以及QTLs,发现了番茄驯化和改良的过程为2组QTLs,以及大果驯化过程中丢失了的控制风味基因,全面揭示了育种是如何全面改变风味物质的。油菜中,研究人员梳理了甘蓝型油菜进化历史,通过60 K芯片的GWAS分析,获得植株高度和分支数的8个QTLs,挖掘到控制产油量的50个QTLs[16]。玉米中,Wen等结合代谢组,定位到1 459个QTLs[19]。
基因组学的发展催生了基因组选择(GS)育种技术,它是一种新型的、针对数量性状由微效多基因控制的育种难题的更高效的分子辅助育种方法。研究前沿集中在GS育种方法、GS实施中的准则、模型,以及预测的准确性方面。通过选择训练群体,可使水稻开花时间的预测准确率达到63%[20]。基因组选择育种技术在作物育种中具有广阔的应用前景。
3.2高效基因编辑技术及其在作物育种中的应用
基因编辑是对生物体基因组特定目标基因进行精确修饰的一种基因工程技术。目前的基因编辑技术包括:巨型核酸酶、锌指核酸酶(ZFNs)、转录激活样效应因子核酸酶(TALEN)和成簇规律间隔短回文重复(CRISPR/Cas)系统。其中CRISPR/Cas因其简单、高效和多功能性迅速成为基因编辑的首选技术,广泛应用于研究和生产中。植物CRISPR/Cas基因编辑的首次成功应用报道于2013年,随后,更多植物的CRISPR/Cas基因编辑体系成功建立,为作物品质改良提供了全新途径。
该前沿主要集中在对技术本身问题的探讨以及在作物育种应用两大方面。就技术本身而言,研究热点主要集中在作物基因编辑的效率及遗传稳定性、转化方式的优化、基因编辑工具的完善以及CRISPR/Cas9基因编辑监管问题;作物基因编辑育种则主要体现在增强作物抗病性、改善作物品质等方面。
3.2.1CRISPR/Cas9植物基因组编辑方法、编辑效率及遗传方式朱建康等检测了CRISPR/Cas在拟南芥的12个不同靶点针对7个基因的编辑情况,结果证明,CRISPR/Cas系统在编辑拟南芥基因方面是高效和特异的,编辑后的目的基因可以稳定地遗传。同时他们还测试了2个水稻亚种11个靶基因对CRISPR/Cas9诱导的编辑情况,证明CRISPR/Cas9系统在水稻中也是高效特异且稳定遗传的[21]。美国研究人员报道了水稻Cas9/sgRNA诱导的大染色体片段缺失,Cas9/sgRNAs在水稻原生质体中产生涉及3个不同基因簇的大染色体缺失(115~245 kb),并在再生的T0代植株中验证了2个基因簇的缺失[22]。美国杜邦公司利用Cas9和Guide RNA在玉米和大豆基因组中成功实现定向突变、精确基因编辑和位点特异性基因的插入[23-24]。
3.2.2避免转基因中间体产生的编辑技术方面CRISPR/Cas9编辑植物基因组通常涉及转基因中间体,引发监管方面的担忧。相关报道介绍了2种简单而有效的基因组编辑方法,一种是从以DNA或RNA形式引入的瞬时表达CRISPR/Cas9的愈伤组织细胞中再生植株[25];另一种是使用CRISPR/Cas9核糖核蛋白(RNPs)对小麦进行基因组编辑[26]。这2种方式获得的突变体是完全无转基因的,因此可广泛应用于基因组编辑作物的生产,具有很好的商品化前景。此外,美国杜邦公司也采用预先组装的Cas9-gRNA核糖核蛋白导入玉米胚细胞,并再生了具有突变和编辑等位基因的玉米植物[27]。
3.2.3碱基编辑技术方面基于CRISPR/Cas系统发展起来的新型靶基因修饰技术碱基编辑技术,为创造作物碱基突变提供了新的手段,在改变作物基因功能方面具有显著优势。包括胞嘧啶碱基编辑器(cytosine base editor,简称CBE)和腺嘌呤碱基编辑器(adenine base editor,简称ABE)2种。有研究者利用碱基编辑系统成功在水稻、小麦及玉米中实现精准碱基编辑[28]。日本研究人员在番茄和水稻中利用CBE碱基编辑系统实现C-T (G-A)碱基替换。研究人员分别将人源胞嘧啶脱氨酶替换了大鼠胞嘧啶脱氨酶,大大提高了CBE碱基编辑效率[29]。后续的研究中,研究团队发现CBE碱基编辑器在水稻产生大规模脱靶,还需要进一步优化。2018年来自中国农业科学院的研究团队,几乎同时分别实现ABE碱基编辑器的成功应用。CBE碱基编辑器的编辑效率显著高于ABE碱基编辑器。张倩倩等利用CBE碱基编辑器成功实验西瓜ALS基因的碱基突变,获得世界首例抗除草剂西瓜[30]。常规CRISPR/Cas9编辑技术使用化脓性链球菌Cas9(SpCas9),需要NGG作为邻接基序(PAM),大大限制了碱基编辑器的使用范围。中国科学院的研究团队使用带有SpCas9和金黄色葡萄球菌Cas9(SaCas9)变体的新ABES和CBE来绕过这一限制,从而大大增加了水稻基因组中碱基编辑的目标范围。日本研究人员使用SpCas9(SpCas9-NGv1)变体,有效地诱变水稻和拟南芥基因组中的NG PAM的内源靶位点且拓展了CBE的碱基识别窗口[31]。
3.2.4基因编辑技术监管、安全性评估方面基因编辑技术飞速发展,但与之对应的政策法规的设立存在一定的滞后性。尽管基因编辑技术不引入转基因,并且基因编辑作物在多个国家已被认定为非转基因产品,但对这些新技术的监管、安全性评估标准、大众接受度及相应的立法尚存在很大争议,各个国家的评判标准也不统一。通过基因编辑进行植物育种的快速进展要求为新的生物技术建立新的全球政策,同时填补基于过程和基于产品的转基因法规之间的空白。德国、日本和美国[32-33]等国家对上述问题开展了一些调研和探讨,建议通过基因编辑或其他未来技术修改的植物应根据新的性状和由此产生的最终产品进行评估,而不是根据创造新植物品种所使用的技术进行评估。
3.2.5作物基因编辑育种在增强作物抗病性、改善作物品质方面CRISPR/Cas基因编辑育种可以实现优质育种资源从无到有的突破,Macovei等利用CRISPR/Cas9系统在水稻东格鲁病敏感品种IR64中产生eIF4G突变其中导致SVLFPNLAGKS残基(主要是NL)与YVV残基相邻的框内突变的等位基因,使其获得东格鲁病抗性,为培育更多样化的RTSV抗性品种提供了有价值的材料[34]。Ortigosa等鉴定了AtJAZ2的番茄同源基因,并通過CRISPR/Cas9介导的SlJAZ2编辑成功地在商品品种MoneyMaker中获得了抗细菌性斑点病的番茄[35]。Tripathi等利用CRISPR/Cas9技术敲除香蕉B基因组中香蕉内源条纹病毒激活序列。在水分胁迫条件下,与未编辑的对照植物相比,75%的编辑植株保持无症状[6]。
在园艺作物单性结实的农艺性状方面,Ueta等利用CRISPR/CAS9系统突变单性结实的关键基因SlIAA9,获得单性结实番茄纯合突变体[36]。利用CRISPR/Cas9敲除SlAGL6,获得的番茄突变体能够在严重阻碍受精坐果的热胁迫条件下产生果实且无核果实质量和形状正常,花粉生活力正常并保持有性繁殖能力[37]。
在大米和油茶作物中,Tang等利用CRISPR/CAS9系统敲除金属转运蛋白基因OsNramp5,培育出低Cd积累量、无转基因的籼稻新品系[38]。利用CRISPR/Cas9在2个广泛栽培的优良粳稻品种的Waxy基因中引入功能缺失突变,实现了在不影响其他理想农艺性状的情况下将水稻转化为糯性水稻。Jiang等利用CRISPR/Cas9靶向新兴油料植物山茶中FAD2基因,成功地获得了油酸含量从脂肪酸组成的16%提高到50%以上的山茶种子[7]。Morineau等通过CRISPR/Cas9基因编辑,实现了3个δ-12-脱饱和酶(FAD2)基因的选择性、针对性突变,导致多不饱和脂肪酸水平降低,油酸积累增加[39]。3个FAD2基因座的不同等位基因的组合提供了具有不同脂肪谱的茶树品系的多样性,油酸在油中的积累量从10%到62%不等。不同的等位基因组合可以对这个六倍体物种的基因剂量和功能进行公正的分析,也为植物育种提供了一个独特的遗传变异来源。
3.3高通量表型平台与植物育种
高通量表型平台的出现及发展可以对植物的表型组学数据进行充分挖掘与分析,最终构建与丰富基因组数据相匹配的植物表型测量技术体系。近5年来,高通量作物表型平台获得飞速发展,出现了卫星、有人驾驶固定翼飞机、无人机、地面移动机器人、室外移动式龙门架、室内流水线及微型CT机等各类平台。当前热点集中在自动化机器人田间表型平台、无人机表型平台、表型信息获取传感及分析技术等方向。
田间高通量表型测量平台(HTPP)中,Li等建立了具有专用传感器阵列的全自动机器人田间表型测量平台,所用传感器包括高分辨率可见光成像仪、叶绿素荧光和热红外成像仪、高光谱成像仪和3D激光扫描仪[40]。另有1种悬挂式表型测量平台主要对温室内植物进行监测,该平台在距离地面 2~5 m范围内运行,并可在恶劣天气条件下完成高分辨率成像[41]。低成本的无人机系统(UAS)在近距离快速测量作物表型方面具有巨大潜力。基于无人机的平台可在30~100 m的高度获得高分辨率热成像和多光谱成像,既能为少量小区提供高分辨率的测量,也可对成千上万的小区进行快速评估。
利用高通量表型平台对作物生育期内的冠层发育进行测量研究,发现冠层高度的广义遗传力最高[42]。Zhang等开发了一套评估温室入射光空间分布的方法对数百株玉米进行监测,利用平台测量了玉米重组自交系群体(n=167)在16个发育时期的106个机器识别性状[43]。结合高密度遗传连锁图谱(包括2 496个重组区)共鉴定到包括已知的3个QTL热点区间在内的988个QTL。此外,研究发现UAS平台获得的植被指数VI与地面实际测量值之间具有较好的相关性,并发现VI具有很高的广义遗传性[10]。该高空作业平台能够有效评估低氮胁迫下的农作物表现,光谱成像和地面测量的归一化植被指数(NDVI)数据均表明农作物衰老指数和谷物产量之间具有很强的相关性。
在作物表型预测中,为了评估高通量表型数据的效用,科研人员等提出了候选G2P模型用于整合大量基因组和新表型数据,并基于基因与环境进行建模[44]。
3.4雄性不育與作物杂种优势利用
植物雄性不育通常是指雄性器官不能产生正常功能的雄配子(花粉)的现象,在杂种优势利用及杂交种生产等方面具有重要价值。根据遗传机制不同,可将雄性不育分为受核基因控制的核不育类型(GMS)和受细胞质基因与核基因共同控制的质核互作不育类型(CMS)。
目前,在雄性不育与作物杂交育种研究主要集中在雄性不育的遗传资源、分子机制及其在杂种优势中的应用等领域。其中,在雄性不育的分子机制研究领域已发现导致小孢子减数分裂异常、绒毡层发育异常、花粉壁发育异常等与细胞核不育相关的功能基因,及在细胞叶绿体和线粒体中发现导致细胞质不育的相关功能基因。
本研究前沿主要集中在3个方面:作物杂交育种中雄性不育的分子调控机制研究、雄性不育基因在杂交育种中的应用及作物雄性不育与育性恢复。其中“Male sterility and fertility restoration in crops”是本前沿论文中被引频次(210次)最高的论文。文章综述了近年来作物细胞质雄性不育系和细胞核雄性不育系的研究进展,总结了雄性不育和育性恢复的几种模型及进化意义,具有重要价值[45]。
7篇核心论文中我国主导或参与贡献了6篇,表明我国在这一热点研究方面处于世界领先水平,特别是我国杂交稻生产技术:基于核质互作雄性不育“三系法”和光温敏核不育“两系法”。我国植物遗传学家在阐明杂交水稻育性调控的分子遗传基础领域作出了重要贡献。在三系杂交稻育种应用最广泛的CMS系统是水稻野败型细胞质雄性不育系统,其不育基因为线粒体不育基因WA352,WA352通过结合核编码蛋白Cox11抑制其降解ROS,引起花粉绒毡层提前程序性死亡,是导致花粉败育的分子机制。将核不育恢复基因与来自于玉米的α-淀粉酶基因、1种来自于珊瑚的红色荧光蛋白基因串联,可用于批量生产获得非转基因的雄性不育系[45]。
我国科学家在玉米核不育恢复基因ZmMs7[46]、ZmMs30[47]等的克隆和功能研究也取得了重大突破,为玉米雄花发育的分子机制研究提供了重要线索;同时,提出了作物多控不育技术系统的概念,利用该技术体系生产的玉米不育系和杂交种都不含任何转基因成分,可规避转基因对人类食品安全的困扰。通过对多种植物核不育基因的比较基因组学和生物信息学分析,预测到62个玉米核不育候选新基因,据此构建了玉米花药/花粉发育的遗传与生化调控网络,为玉米和其他作物不育基因的理论研究和育种应用提供了重要参考和技术指导。
3.5野生亲缘关系在作物育种中的研究
相对于驯化作物来说,野生亲缘关系(crop wild relatives,简称CWR)作物在进化过程中具备很强的生物或非生物抗性,从而拥有更高的遗传多样性。利用野生近缘种提高作物的抗病性、产量等性状是热点研究。该热点前沿集中在CWR保护、基因组水平上对作物野生种遗传多样性的认知以及CWR在作物育种的应用现状与展望3个方向。
在CWR保护方面,世界上部分国家已发出倡议并设立了资助项目,用于保护世界范围内的CWR,作物种类包括苹果、花生、香蕉、大麦、胡萝卜、鹰嘴豆、豇豆、茄子、蚕豆、小米、豌豆、扁豆、燕麦、珍珠小米、木豆、马铃薯、大米、黑麦、高粱、向日葵、甘薯、野豌豆和小麦等。工作内容则包括:确定那些现有基因库中缺失的、最有可能包含适应农业适应气候变化的价值多样性以及最濒危的CWR;从野生环境中收集新的和受威胁的CWR多样性,并将其提供给基因库进行保护;与全世界研究人员和其他用户共享这些物种;评估新收集的CWR和其他已收集的CWR的有用特性,并准备用于作物改良;广泛提供信息,以便研究人员、收集者、育种者和植物遗传资源的其他用户能够访问信息和信息系统,以改进CWR的保护和使用。其次是利用现有数据结合科学方法提出CWR保护的量化指标,既可清晰了解全球CWR保护的现状,也可确定那些需要优先保护的野生种类型。有研究利用已有信息模拟了与81种作物相关的 1 076 个分类群的全球分布,并将这些分布所包含的潜在地理和生态多样性与目前可在基因库中获得的多样性信息进行比较,以此来评估遗传多样性保护的全面性[48]。研究结果表明,CWR的多样性在基因库中代表性很差。有63种作物相关的313个类群(占总数的291%)没有种质资源,另外257个(23.9%)只有不到10个。超过70%的分类群被认为需要进一步收集,以提高它们在基因库中的代表性,而超过95%的分类群在其自然分布中的地理和生态变异方面的代表性不足。研究提出一个差距分析指标方法,用来评估迁徙地和原生地野生植物遗传多样性保护工作[49],不仅能够确定物种的优先次序,还可以量化区域范围内植物保护的进展情况。对近7 000 个分类群评估,发现每100个分类群中仅有不到3个被评估为充分保护,并提出建议。
在基因组水平上对CWR基因库多样性的认知研究中,基因组测序数据表明,CWR的确是具有实用价值的植物育种基因库,野生种能够扩大作物的遗传多样性。此外,DNA测序技术的进步使得育种家认识到需要结合重测序来有效地探索CWR中有益的遗传变异。对CWR核基因组、转录组和母体(叶绿体和线粒体)基因组的分析有助于它们在作物改良中的应用[50]。以高优先级CWR为靶点进行测序将使CWR基因组测序的贡献最大化。
CWR在作物改良应用研究中,从作物类型来看,野生种利用研究最多的作物依次为向日葵、小麦、马铃薯、花生、苹果、水稻、燕麦、木豆、梨和番茄;而从改良性状类型来看,多为生物抗性、非生物抗性、农艺性状、育性、数量性状、表型性状;从应用年代来看,自1980年以来,野生种利用受到关注,在2000年以后发生了跳跃性的提升直到最近依然保持这种状态,通过一些实例来表达野生种遗传多样性对作物遗传改良的重要推动。在非生物胁迫抗性方面,通过杂交育种将野生种单粒小麦(Triticum monococcum)中的TmHKT1:5-A基因转入硬粒小麦中去从而使其在盐环境下的产量增加25%。在生物胁迫抗性方面,将玉米野生种墨西哥玉米(Tripsacum dactyloides L.)的抗枯萎病等位基因转移后,至少使美国1978年的玉米损失减少50%。在作物产量性状上,将野生番茄(Solanum hirsutum)与小果泛起杂交后形成的番茄品种产量和可溶物含量增加20%。如何将目标性状或基因从野生种渗透至作物基因组中,涉及到诸多技术,比如胚抢救技术、体细胞融合杂交技术、多倍体化技术等。即使这样,也仍有部分野生种的基因未能成功转移进入作物,需要持续研究。进一步研究发现,当野生种基因渗入到作物基因组中以后,需要利用分子标记辅助选择技术、QTL定位技术、全基因组关联分析技术、单核苷酸变异芯片技术、基因组测序技术、功能基因组学、代谢组学以及转基因技术和最新的基因编辑技术等来进一步获得并利用野生种的基因及其控制性状。尽管如此,仍有一些因素阻碍了CWR的利用进程,包括野生种表型和基因型数据的缺乏、杂交亲和性障碍、野生种基本信息的缺失、连锁累赘、野生种的劣势认知缺乏、对复杂性状的遗传基础缺乏了解、对迁徙地野生种管理和维护的不善、缺乏机构支持和资金限制等问题,仍需要进一步解决。
4结论与讨論
作物育种的前沿始终围绕生物育种技术与应用,也是全球重点农业组织与国家基金的重点资助方向。《Plant Biotechnology Journal》是领域前沿的重要期刊,美国和中国仍是该研究领域的突出贡献者和领军者,处于全球领先地位。总体而言,全球研究机构和大学发展较为均衡,合作以中、美两国的国内合作为主,康奈尔大学是国际合作的典范。重要研究机构中,我国的中国科学院、中国农业科学院和华中农业大学是突出代表机构,在全球机构中具有显著的竞争优势,但影响力有待进一步提升,未来应在兼顾产出的基础上,更加重视论文的质量,提升国际影响力。在合作方面,应由重点机构带头,基于全球重要机构的不同研究方向,有针对性地开展国际学术交流,积极开展跨国合作模式,增进我国的优势技术,补足我国作物育种中的短板。
生物育种技术的发展引领了作物生物育种领域的前进方向,随着计算机信息技术的发展,高通量表型平台也有了较大的突破,能够在技术应用过程中发挥重要作用。面对粮食供给现状,雄性不育与作物杂种优势这一传统方向是关系到世界粮食供给和农业产业发展的重要内容,是基础性和必要性的研究方向,野生亲缘关系及在作物育种中的研究是较新的研究方向。我国正值“十四五”的开局之年,须在掌握国际发展动态和前沿技术的基础上,基于我国作物育种产业发展的国情,结合大力发展生物种业的相关政策,顺势而为,利用我国育种优势,加强育种技术的创新,突破生物育种的“卡脖子”技术,尽早跨越种业第2阶段至第3阶段的过渡时期,向种业第4阶段迈进。
參考文献:
[1]育种:实现跨越发展,打好种业翻身仗[EB/OL]. (2021-03-10)[2021-04-20]. https://baijiahao.baidu.com/s?id=1693809839546724507&wfr=spider&for=pc.
[2]李新海,谷晓峰,马有志,等. 农作物基因设计育种发展现状与展望[J]. 中国农业科技导报,2020,22(8):1-4.
[3]Incites Essential Science Indicators [EB/OL]. (2019-03-10)[2021-04-20]. http://help.incites.clarivate.com/incitesLiveESI/ESIGroup/indicatorsGroup/indicatorsESI/researchFro nts.html.
[4]Sauvage C,Segura V,Bauchet G,et al. Genome-wide association in tomato reveals 44 candidate loci for fruit metabolic traits[J]. Plant Physiology,2014,165(3):1120-1132.
[5]van Eeuwijk F A,Bustos-Korts D,Millet E J,et al. Modelling strategies for assessing and increasing the effectiveness of new phenotyping techniques in plant breeding[J]. Plant Science,2019,282:23-39.
[6]Tripathi J N,Ntui V O,Ron M,et al. CRISPR/Cas9 editing of endogenous banana streak virus in the B genome of Musa spp.overcomes a major challenge in banana breeding[J]. Communications Biology,2019,2:46.
[7]Jiang W Z,Henry I M,Lynagh P G,et al. Significant enhancement of fatty acid composition in seeds of the allohexaploid,Camelina sativa,using CRISPR/Cas9 gene editing[J]. Plant Biotechnology Journal,2017,15(5):648-657.
[8]Winfield M O,Allen A M,Burridge A J,et al. High-density SNP genotyping array for hexaploid wheat and its secondary and tertiary gene pool[J]. Plant Biotechnology Journal,2016,14(5):1195-1206.
[9]Allen A M,Winfield M O,Burridge A J,et al. Characterization of a Wheat Breeders Array suitable for high-throughput SNP genotyping of global accessions of hexaploid bread wheat (Triticum aestivum)[J]. Plant Biotechnology Journal,2017,15(3):390-401.
[10]Haghighattalab A,Pérez L G,Mondal S,et al. Application of unmanned aerial systems for high throughput phenotyping of large wheat breeding nurseries[J]. Plant Methods,2016,12:35.
[11]Zaman-Allah M,Vergara O,Araus J L,et al. Unmanned aerial platform-based multi-spectral imaging for field phenotyping of maize[J]. Plant Methods,2015,11:35.
[12]Tattaris M,Reynolds M P,Chapman S C.A direct comparison of remote sensing approaches for high-throughput phenotyping in plant breeding[J]. Frontiers in Plant Science,2016,7:1131.
[13]Isidro J,Jannink J L,Akdemir D,et al. Training set optimization under population structure in genomic selection[J]. Theoretical and Applied Genetics,2015,128(1):145-158.
[14]Appels R,Eversole K,Feuillet C. et al. Shifting the limits in wheat research and breeding using a fully annotated reference genome.[J]. Science,2018,361(6403):1-13.
[15]Su Z Q,Bernardo A,Tian B,et al. A deletion mutation in TaHRC confers Fhb1 resistance to Fusarium head blight in wheat[J]. Nature Genetics,2019,51(7):1099-1105.
[16]Liu S,Fan C C,Li J N,et al. A genome-wide association study reveals novel elite allelic variations in seed oil content of Brassica napus[J]. Theoretical and Applied Genetics,2016,129(6):1203-1215.
[17]Sukumaran S,Dreisigacker S,Lopes M,et al. Genome-wide association study for grain yield and related traits in an elite spring wheat population grown in temperate irrigated environments[J]. Theoretical and Applied Genetics,2015,128(2):353-363.
[18]Cui F,Zhang N,Fan X L,et al. Utilization of a Wheat660K SNP array-derived high-density genetic map for high-resolution mapping of a major QTL for kernel number[J]. Scientific Reports,2017,7:3788.
[19]Wen W W,Li D,Li X A,et al. Metabolome-based genome-wide association study of maize kernel leads to novel biochemical insights[J]. Nature Communications,2014,5:3438.
[20]Spindel J,Begum H,Akdemir D,et al. Correction:genomic selection and association mapping in rice (Oryza sativa):effect of trait genetic architecture,training population composition,marker number and statistical model on accuracy of rice genomic selection in elite,tropical rice breeding lines[J]. PLoS Genetics,2015,11(6):e1005350.
[21]Feng Z Y,Mao Y F,Xu N F,et al. Multigeneration analysis reveals the inheritance,specificity,and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America,2014,111(12):4632-4637.
[22]Zhou H B,Liu B,Weeks D P,et al. Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice[J]. Nucleic Acids Research,2014,42(17):10903-10914.
[23]Svitashev S,Young J K,Schwartz C,et al. Targeted mutagenesis,precise gene editing,and site-specific gene insertion in maize using Cas9 and guide RNA[J]. Plant Physiology,2015,169(2):931-945.
[24]Li Z S,Liu Z B,Xing A Q,et al. Cas9-guide RNA directed genome editing in soybean[J]. Plant Physiology,2015,169(2):960-970.
[25]Zhang Y,Liang Z,Zong Y A,et al. Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA[J]. Nature Communications,2016,7:12617.
[26]Liang Z,Chen K L,Li T D,et al. Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes[J]. Nature Communications,2017,8:14261.
[27]Svitashev S,Schwartz C,Lenderts B,et al. Genome editing in maize directed by CRISPR-Cas9 ribonucleoprotein complexes[J]. Nature Communications,2016,7:13274.
[28]Zong Y A,Wang Y P,Li C,et al. Precise base editing in rice,wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nature Biotechnology,2017,35(5):438-440.
[29]Zong Y A,Song Q N,Li C,et al. Efficient C-to-T base editing in plants using a fusion of nCas9 and human APOBEC3A[J]. Nature Biotechnology,2018,36(10):950-953.
[30]張倩倩,许勇,田守蔚,等. 利用CRISPR/Cas9介导的单碱基编辑技术创制抗除草剂西瓜新种质[J]. 中国瓜菜,2019,32(8):219-220.
[31]Endo M,Mikami M,Endo A,et al. Genome editing in plants by engineered CRISPR-Cas9 recognizing NG PAM[J]. Nature Plants,2019,5(1):14-17.
[32]Araki M,Ishii T.Towards social acceptance of plant breeding by genome editing[J]. Trends in Plant Science,2015,20(3):145-149.
[33]Hartung F,Schiemann J.Precise plant breeding using new genome editing techniques:opportunities,safety and regulation in the EU[J]. The Plant Journal,2014,78(5):742-752.
[34]Macovei A,Sevilla N R,Cantos C,et al. Novel alleles of rice eIF4G generated by CRISPR/Cas9-targeted mutagenesis confer resistance to Rice tungro spherical virus[J]. Plant Biotechnology Journal,2018,16(11):1918-1927.
[35]Ortigosa A,Gimenez-Ibanez S,Leonhardt N,et al. Design of a bacterial speck resistant tomato by CRISPR/Cas9-mediated editing of SlJAZ2[J]. Plant Biotechnology Journal,2019,17(3):665-673.
[36]Ueta R,Abe C,Watanabe T,et al. Rapid breeding of parthenocarpic tomato plants using CRISPR/Cas9[J]. Scientific Reports,2017,7:507.
[37]Klap C,Yeshayahou E,Bolger A M,et al. Tomato facultative parthenocarpy results from SlAGAMOUS-LIKE 6 loss of function[J]. Plant Biotechnology Journal,2017,15(5):634-647.
[38]Tang L,Mao B G,Li Y K,et al. Knockout of OsNramp5 using the CRISPR/Cas9 system produces low Cd-accumulating indica rice without compromising yield[J]. Scientific Reports,2017,7:14438.
[39]Morineau C,Bellec Y,Tellier F,et al. Selective gene dosage by CRISPR-Cas9 genome editing in hexaploid Camelina sativa[J]. Plant Biotechnology Journal,2017,15(6):729-739.
[40]Li L,Zhang Q,Huang D F.A review of imaging techniques for plant phenotyping[J]. Sensors,2014,14(11):20078-20111.
[41]Kirchgessner N,Liebisch F,Yu K,et al. The ETH field phenotyping platform FIP:a cable-suspended multi-sensor system[J]. Functional Plant Biology,2017,44(1):154.
[42]Andrade-Sanchez P,Gore M A,Heun J T,et al. Development and evaluation of a field-based high-throughput phenotyping platform[J]. Functional Plant Biology,2013,41(1):68-79.

