胶红酵母Rhodotorula mucilaginosa CM-1菌株的鉴定及胞外多糖的分离纯化
马文锦,李梅林,王博,周彦兵,张永显,于长青,彭涛
(甘肃省轻工研究院有限责任公司,甘肃 兰州,730000)
酵母菌胞外多糖(exo-polysaccharide,EPS)是由酵母细胞产生的,分布于细胞壁之外的同多糖或异多糖,可以荚膜的形式附到细胞壁上或以黏液的形式分泌于胞外环境,由于其易与菌体细胞分离,因此可通过液态发酵实现产业化生产。与植物多糖、动物多糖等其他来源的多糖相比,EPS最大的优势在于易于分离纯化、可连续化发酵,不受地域、季节等环境的影响,它不但在微生物领域具有重要应用,而且在生物、医药和食品等行业上也有广泛应用。因此,EPS具有广阔的发展前景。
胶红酵母(Rhodotorulamucilaginosa)属子囊菌门隐形酵母科红酵母属,是单细胞真核生物[1-2]。研究发现,胶红酵母富含虾青素、β-胡萝卜素和β-葡聚糖,可将糖蜜、三乙醇胺、二乙醇胺和一些简单的芳香族化合物作为满足自身生产的氮源[3]。目前研究较多的为海洋胶红酵母,杨铿等[4]测出海洋胶红酵母粗蛋白质含量为49.2%、粗脂肪1.5%、总糖22.3%、灰分7.9%。含有16种常见的氨基酸,包括7种人体必需氨基酸和6种脂肪酸,并且含有丰富的β-胡萝卜素、维生素E、核苷酸和虾青素等微量营养素。本文通过形态学、碳源/氮源同化实验及18S rDNA生物学方法,对菌株CM-1进行鉴定,经细胞液体培养累积胞外多糖,进一步分离纯化制备出组分单一、纯度较高的胶红酵母EPS组分,为后续胶红酵母EPS的结构解析与活性分析提供理论和数据支撑。
1 材料与方法
1.1 实验材料
1.1.1 材料与试剂
胶红酵母(RhodotorulamucilaginosaCM-1)菌株的获得见筛选方法(1.2.1.1小节)。浓H2SO4,国药集团化学试剂有限公司;苯酚、抗坏血酸,天津市天力化学试剂有限公司;考马斯亮蓝(G250),上海麦克林生化科技有限公司;麦芽糖,合肥博美生物科技有限责任公司;蔗糖、乳糖,天津开发区乐泰化工有限公司;蛋白胨,北京双旋微生物培养基制品厂;Ezup柱式真菌基因组DNA抽提试剂盒,上海易利生物科技有限公司;DreamTaqTMDNA Polymerase,上海浩洋生物科技有限公司;SanPrep柱式质粒DNA小量抽提试剂盒,上海生工生物工程股份有限公司。
1.1.2 仪器与设备
LDZX-30KBS高压蒸汽灭菌锅,上海申安医疗器械厂;UB103i生物显微镜,重庆澳浦光电技术有限公司;HPS-160恒温生化培养箱,哈尔滨东联电子技术有限公司;Universal Hood Ⅱ凝胶电泳呈像仪、7370XL测序仪、2720 thermal cycler PCR仪,美国伯乐BIO-RAD公司;EM-30 Plus扫描电镜,北京天耀科技有限公司;DYCP-31DNDNA电泳槽,北京六一仪器厂;Vector 33 FT-IR分光光度计,德国Bruker。
1.2 实验方法
1.2.1R.mucilaginosaCM-1菌种的鉴定
1.2.1.1 胶红酵母菌株筛选方法
酵母菌接入酵母浸出粉胨葡萄糖(yeast peptone detrose,YPD)液体培养基中培养后,用保护液对酵母菌培养液进行稀释。取菌体稀释液,制备菌膜。将制备的菌膜分别置于离子注入机小真空靶室的无菌靶台上,采用不同能量、不同剂量的N+在真空状态下,以5 s脉冲方式注入酵母菌菌膜。以真空条件下未注入N+的酵母菌膜为对照。离子注入结束后,立即用发菜基因组DNA的Tris-EDTA缓冲液浸泡酵母菌膜,30 ℃静置温育2 h。未注入N+的酵母菌膜也按相同方法处理,并以2 mL无菌水代替发菜基因组DNA TE缓冲液,与注入后的菌膜互为对照。温育完毕后加入无菌水洗脱菌膜。取洗脱液涂于平板培养基中培养。以不注入N+的等量菌膜作为对照。利用构建的多重PCR筛选体系对重组酵母进行筛选得到。
1.2.1.2 形态学观察
菌种在YPD培养基平板上划线接种,28 ℃培养2 d进行菌落形态观察,并使用扫描电子显微镜对细胞形态进行观察[5]。
1.2.1.3 同化实验
参考文献[9-13]的方法并稍作改动。
(1)R.mucilaginosaCM-1菌株基因组DNA的提取
取酵母菌菌液,常温6 000 r/min离心2 min,吸去上清液;加入300 μL裂解液、2 μL β-巯基乙醇和20 μL蛋白酶K溶液,振荡;将离心管置于恒温水浴中至细胞完全裂解;取出冷却至常温,加入100 μL Buffer PE,涡旋振荡后冰浴,加入20 μL的核糖核酸酶A,放置2~5 min;室温12 000 r/min离心5 min,吸取上清液,加入200 μL Buffer BD,200 μL无水乙醇,充分混匀;放入吸附柱,加入溶液和半透明纤维状悬浮液,静止2 min,离心后依次用500 μL PW溶液、500 μL洗液清洗,除去残留,吸附柱放入1.5 mL离心管,加入50 μL TE缓冲液静置3 min后离心,收集DNA溶液用于后续实验。
(2)PCR扩增
用NS序列通用引物(P1:5′-GTAGTCATATGCT TGTCTC-3′,P2:5′-GCATCACAGACCTGTTATTGCCTC-3′)进行基因扩增并测序。PCR反应体系见表1。PCR循环条件见表2。

表2 PCR循环条件Table 2 The PCR cycling conditions
(3)凝胶电泳
1%琼脂糖电泳,150 V、100 mA、20 min电泳观察。
(4)纯化回收
PCR产物电泳条带切割所需DNA目的条带,PCR产物用PCR引物直接测序。
(5)序列比对
将测序结果登录NCBI网站(http://www.ncbi.nlm.nih.gov/)BLAST程序(http://www.ncbi.nlm.nih.gov/BLAST)进行序列比对,并提交GenBank数据库申请登录号ClustalX(1.83)软件进行多序列比对(multiple alignments),用软件 PHYLIP3.57进行分析获得系统进化树。
1.2.2R.mucilaginosaCM-1胞外多糖的制备
1.2.2.1R.mucilaginosaCM-1细胞固体培养基培养
配制YPD固体培养基,将培养基、培养皿、锥形瓶等物品高压蒸汽灭菌,待培养液冷却至40 ℃,倒平板备用;将R.mucilaginosaCM-1从-80 ℃冰箱中取出,室温下接种于平板上,于28 ℃培养2~3 d活化;用稀释涂布法将活化好的胶红酵母菌进一步分离纯化;挑选单个菌落,按照上述的方法再次培养菌种,得到胶红酵母菌种的纯培养菌落;挑取单菌落于在载玻片上,滴加无菌水,放显微镜下观察;使菌悬液在载玻片上形成均匀薄膜。待玻片自然干燥后,滴加少量美蓝染液,静置3 min,镜检。
1.2.2.2R.mucilaginosaCM-1细胞液体培养基培养
挑取活化好的R.mucilaginosaCM-1菌落接种于种子液体培养基(酵母膏1%、葡萄糖2%、蛋白胨2%,pH 6)扩大培养,配制4 000 mL的无机培养液,种子液按5%的接种量接入。于28 ℃、80~100 r/min条件下培养6 d,分离上清液与细胞,提取EPS。
1.2.2.3R.mucilaginosaCM-1胞外粗多糖的分离
将液体培养液于9 000 r/min、4 ℃、6 min条件下离心分离酵母菌与培养液,取上清液,旋蒸浓缩。
1.2.3R.mucilaginosaCM-1胞外粗多糖分离纯化
1.2.3.1R.mucilaginosaCM-1胞外粗多糖制备
为得到单一、纯度高的EPS,先将浓缩好的上清液在乙醇溶液中醇沉24 h,乙醇终体积分数为70%。将醇沉后的EPS在4 ℃、5 000 r/min、10 min的条件下离心,得白色沉淀,即为EPS粗品。收集粗品置于通风橱干燥,挥发乙醇。蒸馏水复溶后冷冻干燥,保存备用。
1.2.3.2 三氯乙酸沉淀蛋白
称取EPS样品,蒸馏水溶解,缓慢滴加质量分数为10%的三氯乙酸。置于4 ℃的冰箱内放置24 h,于5 000 r/min、4 ℃、10 min条件下去除白色沉淀。将多糖溶液醇沉后进行冻干处理,保存后备用。
1.2.3.3 葡萄糖标准曲线的绘制
准确称取烘干到恒重的葡萄糖0.025 g,蒸馏水溶解,使葡萄糖质量浓度达到0.5 mg/mL。将葡萄糖溶液分置于6个具塞试管中,具体操作方法按照表3执行。

表3 葡萄糖标准曲线的测定 单位:mL
向各管内滴入配制好的苯酚溶液1.0 mL,滴加5.0 mL的浓H2SO4振荡,静置5 min,100 ℃的水浴锅中水浴25 min,冷却,在484 nm处测量吸光值,以0号管为空白,横坐标为标准葡萄糖体积,纵坐标为吸光值A,得到标准曲线。
1.2.3.4 DEAE-52纤维素层析柱纯化
在文献[14-15]的方法基础上稍作修改。将处理好的树脂置于缓冲液内进行填料,保证填料柱子完整致密,无小泡。剩余树脂浸泡在体积分数75%的乙醇溶液中保留备用。直立固定层析柱,加缓冲溶液,将DEAE-Cellulose 52处理后的浑浊液缓慢的注入到玻璃柱内,自然沉降到柱高的1/3时,打开层析柱下方的排液口,使液体均匀的流出,确保树脂上表面平整且不能暴露在缓冲液外,观察填料柱无小气泡产生。将粗多糖RmEPS溶于蒸馏水中,离心去除未溶解的沉淀与杂质,将上清液添加到DEAE-Cellulose 52柱上,用蒸馏水洗脱填料柱以除去未结合的EPS。将粗多糖依次用蒸馏水,0.3,0.6,0.9和1.2 mol/L NaCl以0.5 mL/min的流速洗涤,采用苯酚-硫酸法测定每管收集洗脱液的吸光值,绘制洗脱曲线,收集各峰值部分备用。
1.2.3.5 Sephadex G-100凝胶柱层析
用蒸馏水将主要组分进一步洗脱[16],浓缩,透析并冻干后备用。通过苯酚-硫酸法测定RmEPS-11的产量。
1.2.4 RmEPS-11多糖组分的初级结构解析
1.2.4.1 分子质量分布
通过高效凝胶渗透色谱(high performance gel permeation chromatography,HPGPC)分析RmEPS-11的平均分子质量。测试条件:取10 mg多糖样品,加1 mL 0.1 mol/L NaNO3,60 ℃孵育过夜,稀释后过0.22 μm滤膜,上样。仪器:凝胶色谱-示差-多角度激光光散射;检测器:RI,MALS;流动相:0.1 mol/L NaNO3;流速:0.8 mL/min;柱温:45 ℃,分析柱型号:Ohpak SB-804 HQ,Ohpak SB-805 HQ;上样量:100 μL。
1.2.4.2 傅里叶变换红外光谱(Fourier transform infrared spectroscopy,FT-IR)分析
RmEPS-11的FT-IR分析通过KBr法实施[17]。将样品RmEPS-11与KBr粉末充分混合,研磨并压制成1 mm颗粒。使用FT-IR分光光度计以4~4 00 cm-1的分辨率记录2 cm-1的FT-IR谱图。
1.2.4.3 单糖组成分析
采用离子色谱法(ion chromatography,ICS)对RmEPS-11的单糖组成进行测定。将RmEPS-11溶解于三氟乙酸,在121 ℃下保存2 h,采用N2气流对其风干。风干后的样品用甲醇进一步萃取、洗涤、风干,循环3~4次。得到的干样品用蒸馏水溶解,取25 μL样液采用ICS进行分析,采用PAD软件做出相应的峰图。设定条件如下:流动相为NaOH,流速梯度为0~40 min,5 mmol;40~45 min,100 mmol;45~50 min,200 mmol;50.2~55 min,5 mmol;其中40~45 min内同时加入100 mmol的醋酸钠,流速设定为1 mL/min,上样量25 μL,柱温30 ℃,柱型为CarboPac-PA10。
1.2.4.4 甲基化分析
将甲基化的样品RmEPS-11在100 ℃下用90.0%甲酸处理3 h,用甲醇萃取去除甲酸。产物进一步用2.0 mol/L三氟乙酸在121 ℃水解2 h,用NaBH4还原,然后乙酰化。通过GC-MS分析所得的部分O-甲基化糖醇乙酸酯,进行GC-MS测定。温度设定为140 ℃、2 min,然后以2 ℃/min的速度升温至250 ℃保持20 min。使用氦气作为载气,流速为1.24 mL/min。
2 结果与分析
2.1 R.mucilaginosa CM-1菌株的鉴定
2.1.1R.mucilaginosaCM-1菌株形态学观察
如图1-a所示,菌株R.mucilaginosaCM-1在琼脂平板上的菌落形态为中等大小、圆形、隆起、湿润,边缘整齐,菌落颜色均一,为橙色、深橙色或腥红色。通过扫描电镜(图1-b)观察,菌株R.mucilaginosaCM-1细胞形态呈球形、椭圆形、卵圆形或长形,大小约3~4 μm,生殖方式多为芽殖。
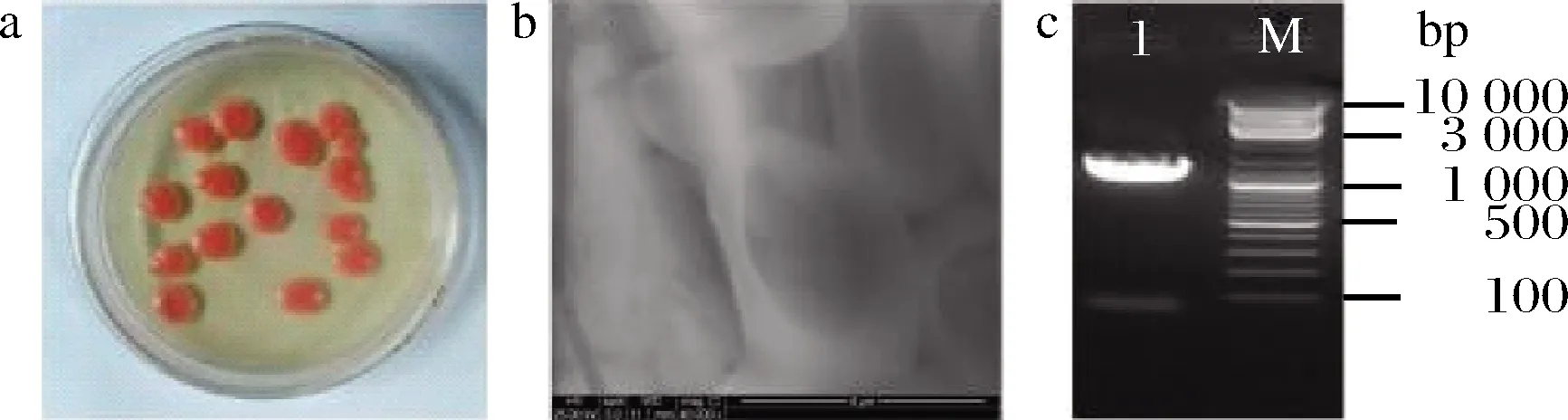
图1 R.milailaginosa CM-1的菌落(a)、 细胞形态(b)与电泳条带图(c)Fig.1 Colony(a), cell morphology(b) and electrophoretic band(c) of R.milailaginosa CM-1
2.1.2R.mucilaginosaCM-1的生理生化鉴定
在28 ℃培养3~7 d的R.mucilaginosaCM-1细胞能够同化葡萄糖、麦芽糖、蔗糖、棉子糖、海藻糖和D-阿拉伯糖,但不能同化半乳糖、乳糖、蜜二糖、肌醇、甲醇、KNO3、放线菌酮(1%)和乙胺。根据酵母菌的特征与鉴定手册[18],菌株CM-1初步确定为酵母菌。
2.1.3 18S rDNA分子生物学鉴定
R.mucilaginosaCM-1菌株的18S rDNA全长约1 323 bp。选择的引物(NS1和NS6)能够清晰地扩增泳道1中18S rDNA的靶标(图1-c)。测定R.mucilaginosaCM-1的NS区和18S rDNA基因以及相关物种的菌株的保守序列。NS区比5.8S rDNA和26S rDNA更快的替换率对于检查物种内的遗传变异或紧密相关物种间的系统发育是至关重要的[19]。与GenBank中的BLAST基因序列进行比对,结果表明:R.mucilaginosaCM-1的18S rDNA序列与R.mucilaginosaKM222281,R.mucilaginosa5H3-M3,R.mucilaginosa4H1-P3-P7-1,R.mucilaginosaRM-1,R.mucilaginosaL10-2的基因高度同源,分别为99%、99%、99%、99%、99%。经鉴定,菌株CM-1为红酵母属(Rhodotorulasp.)。
2.2 R.mucilaginosa CM-1胞外多糖分离与纯化
通过浓缩、醇沉、脱蛋白、脱色和透析等方法得到R.mucilaginosaCM-1胞外多糖RmEPS,经苯酚-硫酸法测定多糖含量为7.35 g/L。通过DEAE-52纤维素阴离子交换层析分离获得RmEPS-1、RmEPS-2、RmEPS-3、RmEPS-4、RmEPS-5和RmEPS-6六个多糖组分(图2),占主要部分的中性多糖RmEPS-1用蒸馏水洗脱,通过凝胶渗透在Sephadex G-100柱中分离纯化,分别获得了2个组分RmEPS-11、RmEPS-12组分(图3)。收集RmEPS-11组分(RmEPS-11占RmEPS-1的73.4%),透析、冷冻干燥,用于后续的结构解析及活性分析。
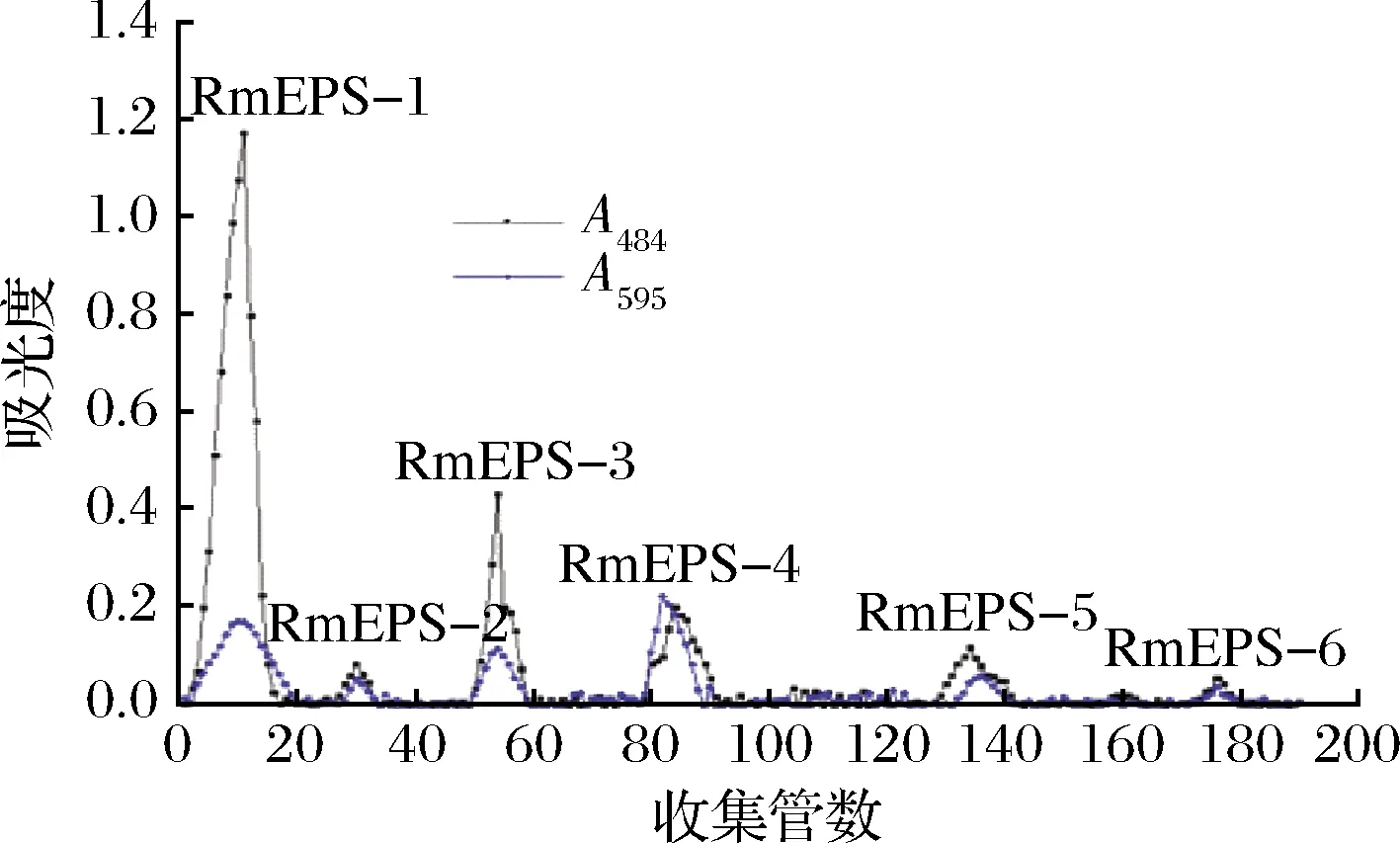
图2 DEAE-52纤维素柱层析洗脱曲线Fig.2 Elution curve of DEAE-52 cellulose column chromatography

图3 SephadexG-100凝胶柱层析洗脱曲线Fig.3 Elution curve of SephadexG-100 gel column chromatography
2.3 RmEPS-11多糖组分的初级结构解析
2.3.1 RmEPS-11多糖组分的均一性和分子质量分布
HPGPC图显示多糖RmEPS-11的色谱图为单一对称洗脱峰(图4),表明多糖为相对均一的多糖组分,RmEPS-11的平均分子质量为2.3×105Da。
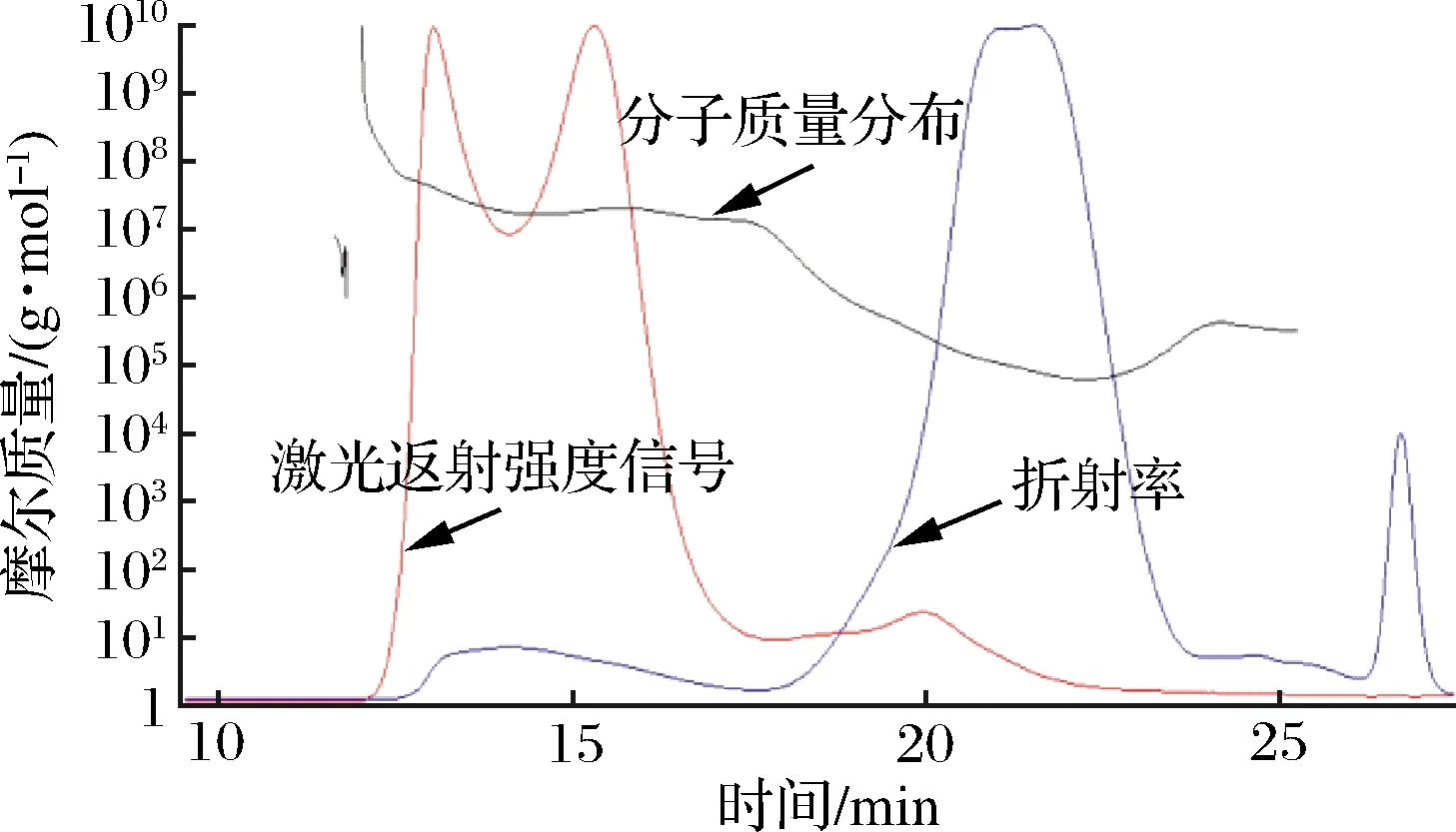
图4 多糖RmEPS-11分子质量分布图Fig.4 The molecular weight distributions of RmEPS-11
2.3.2 多糖RmEPS-11红外光谱分析


图5 多糖RmEPS-11红外光谱图Fig.5 FT-IR spectrum of the RmEPS-11 fraction
2.3.3 多糖RmEPS-11单糖组成分析
通过离子色谱测定多糖RmEPS-11的单糖组成,不仅可以分析多糖的构成,而且准确测定各单糖在多糖中所占的比例,单糖含量之和可以用来衡量对比多糖的质量。试验中以阿拉伯糖、核糖、木糖、葡萄糖、果糖、甘露糖、D-半乳糖为标准糖,测定每个标准单糖的保留时间,通过相应的峰面积和响应因子对RmEPS-11的单糖组成进行测算。基于标准单糖的ICS分析(图6-a),RmEPS-11主要由Ara,Gal,Glc和Man四种单糖构成(图6-b),摩尔比分别为1.3∶4.4∶7.8∶86.4,其中甘露糖含量最高。
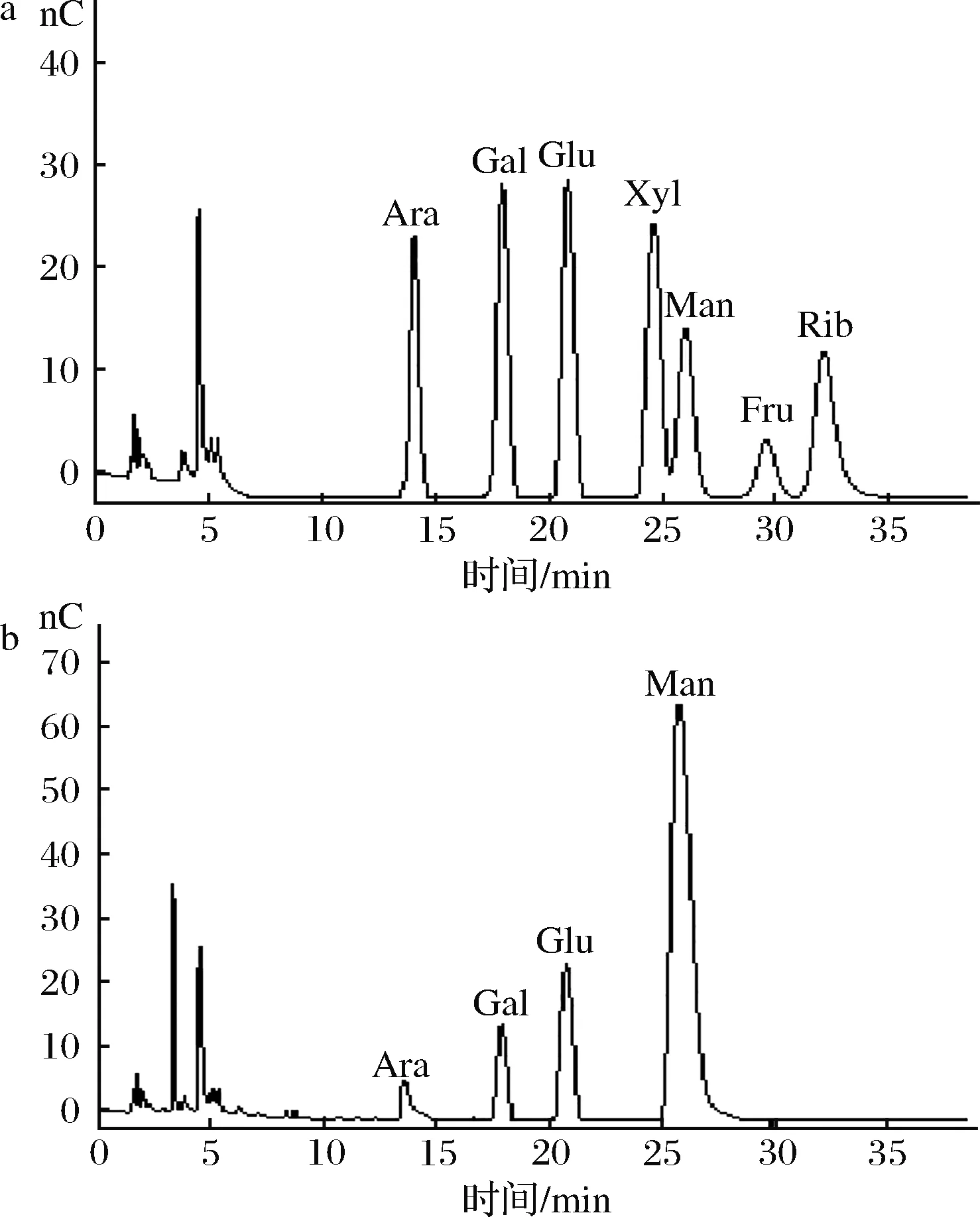
a-单糖标准品;b-RmEPS-11的单糖组成图6 单糖标准品和RmEPS-11的ICS图谱Fig.6 ICS profiles of monosaccharide standards and RmEPS-11注:Ara,Rib,Xyl,Glc,Fru,Man和Gal分别代表阿拉伯糖, 核糖,木糖,葡萄糖,果糖,甘露糖和半乳糖。
2.3.4 RmEPS-11的甲基化分析
如图7所示,根据保留时间和质谱图分析RmEPS-11的碎片峰。表4分析结果表明,RmEPS-11的非还原末端含有甘露糖,分支位于半乳糖基和一些阿拉伯糖残基上。可以得出以下结论:(1)甘露糖残基以末端和1,3-连接的Manp残基存在;(2)葡萄糖残基作为1,2-连接的Glcp残基存在;(3)半乳糖残基以1,2,6-连接的Galp残基存在;(4)阿拉伯糖残基以1,2,3,4连接的Arap残基存在。除了末端Manp残基之外,上述这些残基的数量占总甲基化糖的64.5%,这表明RmEPS-11的骨架由1,3-连接的Manp和1,2-连接的Glcp残基组成,分支上是1,2,6连接的Galp和1,2,3,4连接的Arap。

1-2,3,4,6-Me3-Man;2-3,4,6-Me3-Glc;3-2,4,6-Me3-Glc; 4-3,4-Me3-Gal;5-阿拉伯糖醇图7 RmEPS-11甲基化总离子流图Fig.7 Total ion chromatogram of partially methylated alditol acetates of RmEPS-11

表4 RmEPS-11甲基化分析数据表Table 4 Methylation analysis of RmEPS-11
3 讨论
微生物多糖是发酵工业的新产品,尤其是胞外多糖,相比动物多糖与植物多糖,微生物多糖具有许多优点,不仅可以方便地提取,而且成本低、无毒、环保,不受季节和地理条件及病虫害的影响,生产周期短,产量及质量都很稳定,性价比高,具有动植物多糖不具备的特殊功能,因此很大程度上满足了人们对天然绿色产品的需求。本文通过形态学、碳源/氮源同化实验及18S rDNA生物学方法,鉴定菌株CM-1为胶红酵母菌(R.mucilaginosa)。从R.mucilaginosaCM-1无机培养基中分离得到水溶性胞外多糖RmEPS。采用苯酚-硫酸法测定其产量为7.35 g/L。多糖RmEPS经纤维素交换柱、凝胶渗透等纯化方法得到单一组分多糖RmEPS-11,并对其分子质量分布、红外光谱、单糖组成及甲基化进行了分析,为后续胶红酵母EPS的结构解析与活性分析研究提供了理论和数据支撑。

