苦荞黄酮含量与SSR标记的关联分析
石桃雄 黎瑞源 潘凡 黄娟 朱丽伟 汪燕 梁成刚


摘要:【目的】開展苦养籽粒总黄酮含量与SSR标记的关联分析,挖掘与黄酮含量相关的分子标记,为高黄酮含量苦荞品种的遗传改良提供依据。【方法】以193份苦荞种质为供试材料,基于62对SSR引物分析了供试种质的遗传多样性,对总黄酮含量和SSR标记进行了关联分析。【结果】193个种质籽粒总黄酮含量的变幅为1.04%- 2.99%,变异系数为27.93%。62对引物在193个种质中共扩增出267个等位基因,每个SSR位点平均检测到245个有效等位基因,基因多样性为0 503,Shannon信息指数为0 942,观测杂合度为0.513,引物多态信息量为0.74。群体结构分析将193份种质划分为3个亚群。基于广义线性模型( GIM)共检测到5个与籽粒总黄酮含量显著关联的SSR标记,表型贡献率为6.3%- 12.9%,其中标记TatG0124( 12.9%)和S6853( 8.5%)的表型贡献率较高。【结论】供试苦荞种质遗传多样性丰富,可用于苦荞重要农艺和品质性状的关联分析。TatG0124和SSR6853可能是控制籽粒黄酮含量的重要位点,对改良苦荞籽粒黄酮含量具有重要意义。关键词:苦荞;SSR标记;黄酮含量;遗传多样性;群体结构;关联分析
中图分类号:S 517
文献标志码:A
文章编号:1008-0384( 2021) 08088408
Correlation Between Flavonoids Content and
SSR Markers of Tartary Buckwheat
SHI Taoxiong. LI Ruiyuan. PAN Fan, HUANG Juan. ZHU Liwei. WANG Yan. LIANG Chenggang*
( Buckwheat Industry Technology Research Center. Guizhou Normal Universily, Guiyang, Guizhou 55000I . China )
Abstract: 【Objective】 Correlation between the flavonoids content and associated markers was analyzed for geneticimprovement studies on Tartary buckwheat. 【Method】Sixty-two SSR primer pairs were used to assess the genetic diversityof 193 Tartary buckwheat germplasms. The SSR markers closely related to the total flavonoids content were selected byassociation analysis. 【Result】The total flavonoids content of 193 Tartary buckwheat germplasms varied from l.04% to2.gg% with a coefficient variation of 27.93%. Sixty-two pairs of SSR primers amplified 267 polymorphic sites with an averagenumber of effective alleles of 2.45. The mean values of gene diversity, Shannon index. observed heterozygosity andpolymorphism information content of SSR markers were 0.503. 0.942. 0.5 13 and 0.74. respectively. The population structureanalysis divided the 193 germplasms int0 3 subgroups. Five SSR markers were determined to be significantly associated withflavonoids content by the general linear model (GLM). The phenotypic variations ranged from 6.3% t0 12.9% with TatG0124at 12.9% and S6853 at 8.5% being the higher markers. 【Conclusion】The germplasms used in this study were diverse ingenetic variation and feasible for related genome-wide mapping on important traits of Tartary buckwheat. The loci. TatG0124and S6853, appeared to closely associate with the flavonoids content of Tartary buckwheat and were considered to bepotentially useful for breeding new varieties with high content of the functional components.
Key words: Tartary buckwheat: SSR marker; flavonoids content; genetic diversity; population structure; correlation analysis
0 引言
【研究意义】苦荞(Fagopyrum tataricum),又名鞑靼荞麦( Tartary buckwheat),属蓼科(Polygonaceae)荞麦属(Fagopyrum Mill)雙子叶植物,是荞麦的两大栽培品种之一[1],广泛种植于云、贵、川、藏等省区的高原和高寒地带[2]。苦荞是我国重要的杂粮作物之一,其籽粒含有丰富的多酚类化合物、抗性淀粉、可溶性纤维、蛋白质、氨基酸、维生素及微量元素等,可有效地预防和改善肥胖、胆结石和心血管等疾病[3-4],在食品、保健品、医药等领域具有广泛的应用前景。黄酮类化合物属多酚类物质,苦荞黄酮类化合物含量和种类丰富,生物活性独特,是苦荞保健功能最突出的一类生物活性物质,对肿瘤、“三高”(高血脂、高血压,高血糖)和炎症等都有一定的辅助治疗作用[3-4]。因此,研究苦荞籽粒黄酮含量的遗传机制,挖掘与之紧密连锁的分子标记,对苦荞高黄酮含量专用型品种的选育具有重要意义。【前人研究进展】目前国内外学者对荞麦黄酮类化合物的研究主要集中在其含量和组分的遗传变异评价[5-9]、生物合成调控机理揭示及代谢途径上一些关键基因的挖掘与鉴定[10]等方面。苦荞种质资源中黄酮含量变异丰富,表现出数量性状遗传的特点‘69],所以传统育种方法很难短期改良苦荞品种的黄酮含量。目前苦荞数量性状基因座( QTL)定位还处于遗传群体和图谱的构建阶段[ll],还没有和黄酮含量关联的QTL或分子标记的报道。关联分析是解析数量性状、挖掘有利等位基因、开发与目标性状紧密连锁分子标记的有效手段,在作物遗传育种中发挥着重要作用。近年来,随着苦荞参考基因组的公布[12],Zhang等[13]1对510份苦荞种质黄酮含量和重要农艺性状进行了全基因组关联分析,推测FtUFGT3和FtAP2 YT1可能分别是调控籽粒黄酮含量积累和粒重的关键基因。【本研究切入点】SSR标记是关联分析常用的分子标记之一。随着测序技术的发展和测序成本的降低,荞麦属植物SSR标记已被大规模开发[14-18],主要用于苦荞种质资源遗传多样性和群体结构的研究[19-24]。但目前SSR标记与苦荞黄酮含量关联分析的研究鲜有报道。【拟解决的关键问题】本研究调查了来自我国12个省区的193个苦荞审定品种、地方品种和育种品系的籽粒总黄酮含量,利用SSR标记关联作图法寻找可能与籽粒黄酮含量紧密连锁的标记,以期为分子标记辅助选育高黄酮含量苦荞品种提供依据。
1材料与方法
1.1供试材料
搜集来自全国不同省区的审定品种、育种品系和地方品种等193个苦荞种质作为试验材料,其中贵州139个、四川16个、云南13个、陕西7个、山西6个、江西2个,甘肃和湖南各3个,辽宁、内蒙古、宁夏和西藏各1个。193个苦荞资源由36个审定品种、75个地方品种和82个育种品系组成。
1.2田间种植
供试种质于2016年种植于贵州师范大学柏杨试验基地。每个种质种植四行区,行长2.0 m,行间距为0.33 m,常规田间管理。籽粒成熟后,按小区收获,脱粒、风干至恒重。
1.3籽粒总黄酮含量的测定
称取风干后的饱满籽粒30 9充分研磨,称取0.1000 g粉末置于10 mL离心管中。采用75%的甲醇60℃恒温水浴浸提,0.1 mol·L-l AIC13-1.0 mol·L-lKAc显色,在波长420 nm处测定吸光度(新世纪T6紫外可见分光光度计,北京普析通用仪器有限责任公司),芦丁标准曲线的绘制和黄酮含量的具体测定过程参照吕丹等[8]的方法和步骤。
1.4 SSR基因型检测和遗传多样性分析
在每份种质种植小区中混合采集幼嫩叶片,采用植物基因组DNA提取试剂盒(北京天根)提取DNA。对本课题组前期合成的350对SSR引物进行筛选,包含了150对Genic-SSR引物和200对Genomic-SSR引物,分别以“S”和“TatG”开头命名,引物信息详见黎瑞源等[25]的报道。PCR反应体系为10 uL,包括:DNA模板( 50ng·uL-l) 2.0 uL,2×Taq PCR MasterMix 5 uL,上下游引物( 100 ng·uL-I)各0.5 uL,ddH20 2.0 uL。
PCR扩增产物在2000 V,80 W,100 mA/板的电泳条件下,采用6%非变性聚丙烯酰胺凝胶垂直板电泳后,胶板用0.15% AgN03染色15min,1.5% NaOH显影,具体步骤参考杨美[26]的试验方法。胶板晾干后读带,将可重复的、清晰的条带记为1,同一位置无带或不易分辨的弱带计为0,建立原始数据矩阵。利用PowerMarker V3.25软件计算每对引物扩增的等位基因数( Na)、有效等位基因数(Ne)、Shannon信息指数(I)、观察杂合度(Ho)和基因多样性(He)及多态性信息量( PIC)。
1.5群体结构分析
利用STRUCTURE 2.2软件对供试种质SSR数据矩阵进行基于数学模型的亚群划分。群体结构K值设定为1~9,每个设定的K值进行10次独立运算,Burn-in与MCMC周期均设定为100000次。将STRUCTURE 2.2软件的运算结果先导入在线T具STRUCTURE HARVESTER( http://taylorO.biology.ucla.edu/structureHarvester/)计算统计值AK,确定最有可能的亚群数;再导入在线工具CLUMPAK( http://clu-mpak.tau.ac.i1/)计算群体结构。最后将计算结果导入到EXCEL 2019完成结果图的绘制。
1.6黄酮含量与SSR分子标记的关联分析
使用软件TASSEL 3.0计算、绘制标记连锁不平衡( LD)配对检测的矩阵图,观测LD水平,Permutation设置1000次。使用该软件的广义线性模型( GIM),将各个材料相应的Q值作为协变量进行群体矫正,将黄酮含量与62个SSR标记基因型数据进行回归分析,标记与表型性状的显著性水平设定为p=0.05。TASSEL 3.0软件中表型变异解释率的计算公式为:R 2-SSM/SST,其中SSM为标记基因型间平方和,SST为总平方和。
2 结果与分析
2.1苦荞种质SSR标记的遗传多样性分析
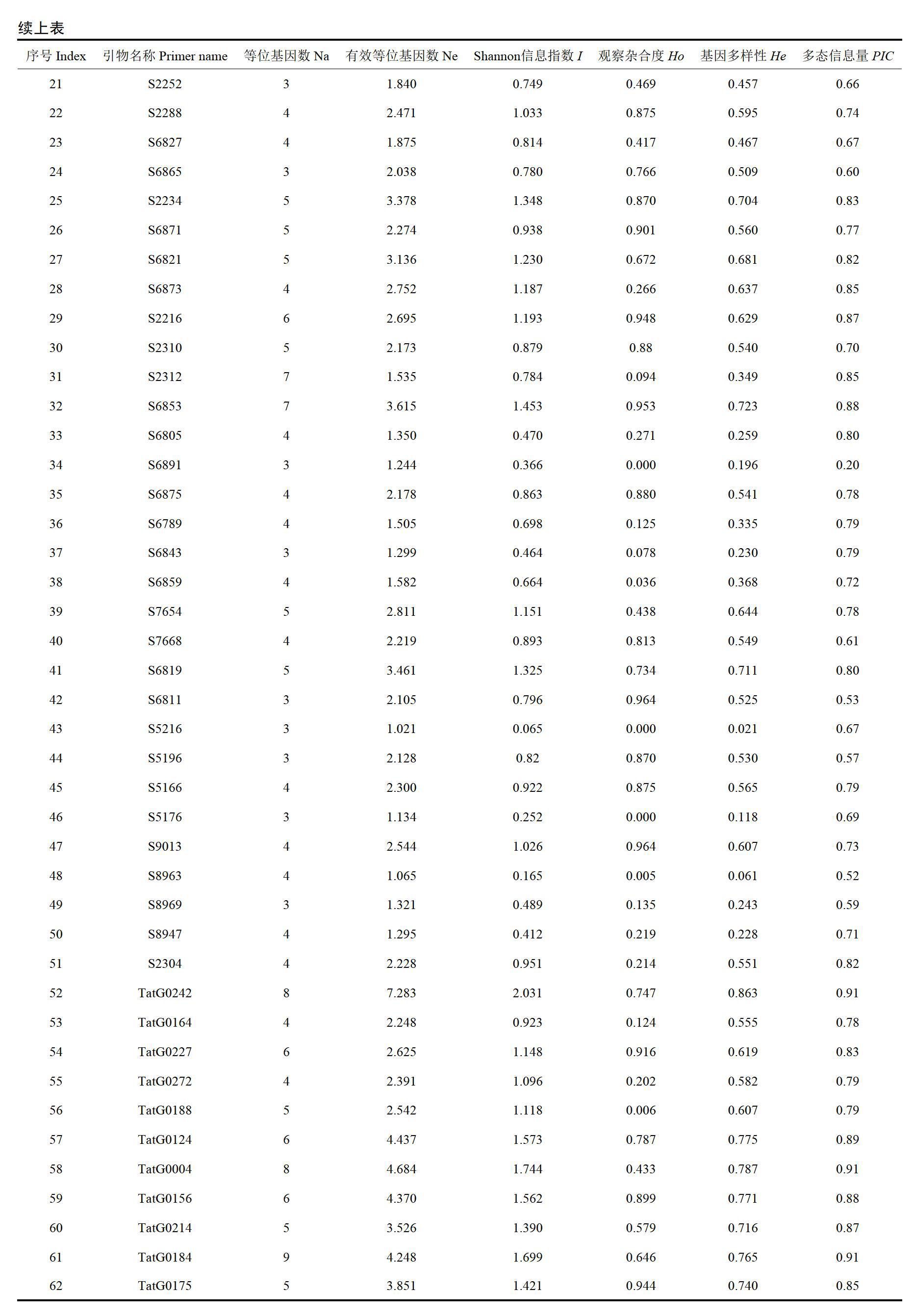
利用从350对SSR引物中筛选到的多态性较好的62对引物,对193个苦荞种质进行扩增(图1),共检测H1 267个等位变异,每个SSR标记平均检测到4.31个等位基因( Na),2.45个有效等位基因(Ne),范围分别为3~9和1.02~7.28。观测杂合度(Ho)平均为0.503,范围为0~0.964。基因多样性(He)平均为0 513,范围为0.021~0.863。Shannon信息指数(I)平均为0.942,范围为0.065~2.031。引物多态信息量( PIC)平均为0.74,范围为0.20—0.91。在62个SSR标记中,有35.5%的标记至少能检测到5个以上等位基因,98.3%的标记PIC> 0.5,64. 5%的标记He> 0.5(表1),表明供试的193个苦荞种质具有丰富的遗传多样性。
2.2苦荞种质群体结构的分析
利用62个SSR标记对供试种质进行群体结构分析,在K=3时,AK值最大(图2),因此将193个苦荞种质划分为3个亚群,命名为POPI、POP2和POP3(图3),分别包含88、90和15个种质。对各亚群的遗传多样性参数的统计可以看出,POPI是遗传多样性最丰富的亚群,平均每个SSR标记检测出4.21个Na,He为0.513,,为0.943。其次是POP2,平均每个SSR标记检测出3.74個Na,He为0.484,,为0.870(表2)。亚群POP2与POP3之间的Nei's遗传距离最小,为0.026;POPI与POP3之间的Nei's遗传距离最大,为0.076。3个亚群之间的成对Fst值平均为0.027;亚群POP2和POP3之间的成对固定系数Fst最小,为0.014;POPI和POP3之间的成对Fst值最大,为0.037(表3),表明划分的3个亚群间遗传分化程度低。
2.3连锁不平衡分析
对62个SSR标记形成的1891个成对组合进行连锁不平衡( LD)分析,整体共有644个组合存在显著(P<0.05)的LD,占总组合数的34.1%,而三个亚群POP1、POP2和POP3中,存在显著的LD成对标记组合数分别只有90(4.8%)、206( lO.9%)和97(5.1%)个(表4),远少于在全体种质中检测到的数目,表明群体结构会影响连锁不平衡的分析。
2.4黄酮含量的变异和关联分析
193个苦荞种质籽粒黄酮含量平均为2.11%,变幅为1.04%~2.99%,变异系数为27.93%。偏度和峰度分别为0.29和1.17,说明籽粒黄酮含量呈近似的正态分布。以上结果表明,供试种质籽粒黄酮含量的变异丰富,可以进行关联分析。
对黄酮含量与62个SSR标记进行基于群体结构的关联分析,检测到5个与黄酮含量显著关联的SSR标记(表5),共解释43.30%的表型变异率。其中TatG0124解释了12.g%的表型变异率,是5个位点中贡献率最大的。其次是S6853,解释了8.5%的表型变异率。
3讨论与结论
群体具有丰富的遗传和表型变异,且内部没有明显的群体结构是关联分析成功的关键条件[27]。群体的遗传多样性与群体的大小、来源、标记数量和引物的类型等因素有关。有效等位基因数目( Ne)、期望杂合度/基因多样性(He)、Shannon信息指数(I)和多态信息量(PIC)等参数能有效地度量和比较不同群体的遗传多样性。一般认为He> 0.5,群体没有高强度的选择,拥有丰富的多态性,He< 0.5的群体遗传多样性低;PIC> 0.5的位点为高度多态座位[28]。本文利用62对SSR引物分析了193个苦荞种质遗传多样性,其,和PIC平均分别为0.942和0.74,高于山西地方品种(He和PIC的均值分别为0.4475和0. 3869)[29]、云南地方品种(He和PIC的均值分别为0.508和0.44)[20]、我国审定品种(PIC均值为0.44)[19]和国内外苦荞种质(PIC均值为0.40)[16] SSR标记的遗传多样性,说明本研究中的193个苦荞种质的遗传多样性丰富。62对SSR引物中11对来自苦荞基因组序列,其Ne、I、He和PIC均值分别为3.837、1.427、0.707和0.855;51对来自苦荞转录组序列,其Ne、I、He和PIC分别为2.154、0.837、0.471和0.719。可见,基因组SSR位点的多态性程度要高于转录组SSR位点。62个引物中,有35.5%的引物至少能检测到5个以上等位基因,98.3%的引物PIC>0.5,64.5%的引物He> 0.5,这些等位基因丰富、高度多态性的引物可有效地用于今后苦荞种质遗传多样性的研究。
群体结构是指一个群体内存在亚群的情况,群体结构会增加染色体间的LD,造成目标性状和基因位点的伪关联[27]。本研究利用群体结构分析将193个供试苦荞种质划分为3个亚群,亚群间的成对固定系数Fst为0.014~0.037,表明3个亚群间遗传分化程度低。62个SSR标记之间的LD分析显示,三个亚群POPI、POP2和POP3间存在显著的LD的标记组合数分别有90、206和97个,数目远少于在全体材料中检测到的644个,这表明通过群体结构可对供试种质进行有效的划分,降低目标性状与标记关联的假阳性,提高关联分析的准确度。以上结果为利用该群体进行苦荞重要性状的关联分析提供了重要参考。
本研究中193份苦荞种质籽粒总黄酮含量变幅为1 .04%~2.99%,变异系数为27.93%,黄酮含量的变异程度大于Gupta等[30]报道的印度喜马拉雅地带的195份苦荞种质和刘三才等[6]调查的来自我国四川、云南、贵州、宁夏、湖南和山西等地的76份苦荞种质籽粒总黄酮含量的变异程度,也大于吕丹等[8]和郑冉等[9]报道的苦荞遗传群体籽粒总黄酮含量的变异程度。本研究对供试验种质籽粒黄酮含量与62个SSR标记进行了基于群体结构的关联分析,定位到了5个与籽粒黄酮含量显著关联的SSR标记,共解释43.3%的表型变异率。其中TatG124( 12.9%)和SSR6853( 8.5%)解释的表型变异率较大,由此推测SSR0124和SSR6853是控制籽粒黄酮含量的重要位点,可进一步验证开发为实用性标记,将为分子标记辅助选育高籽粒黄酮含量苦荞品种提供依据。
参考文献:
[1]陈庆富荞麦属植物科学[M]北京:科学出版社,2012: 10 - ll
[2]黄凯丰,李振宙,王炎,等我国荞麦高产栽培生理研究进展[J]贵州师范大学学报(自然科学版),2019,37(1):115-120
HUANG K F.LI Z Z.WANG Y.et al.Research progress onphysiology of buckwheat under high-yield cultivation[J].Journal ofGuiZhou Normal University (Natural Sciences),2019,37(1):115-120 (in Chinese)
[3]吴韬,肖丽,李伟丽苦荞的营养与功能成分研究进展[J].西华大学学报(自然科学版),2021,40(2):91-96. 109
WU T XIAO L, LI W L Research progress of chemicals in Tartarvbuckwheat[J].Journal ofXihua University (Natural Science Editiion),2021. 40(2): 91-96. 109.(1n Chinese)
[4] LUTHAR z.GOLOB A.GERM M et al Tartary buckwheat inhuman nutrition [J].Plants 202l,10 (4):700
[5]王璐瑗,荣玉萍,黄娟,等21l份金荞麦收集系根茎黄酮含量的分析评价[J]贵州师范大学学报(自然科学版),2019,37(4):25 30.48
WANG L Y,RONG Y P,HUANG J,et al Analysis and evaluatlon ofthe navonoid coment of rhizomes of 211 different Golden buckwheataccesslons(Fagopyrum cymosum comples)[J].Journal of Guizhou Normal University (Natural Scienes),2019,37(4):25 30,48(inChinese)
[6]刘二才,李为喜,刘方,等苦荞麦种质资源总黄酮利蛋白质含量的测定与评价[J]植物遗传资源学报,2007,8(3):317-320
LIU S C.LI W X LIU F et al Identificalton and evaluatlon of totalflavones and protein coment ln Tartary buckwhea genIlplasm [J]Journal ofPlant Genetic Resources,2007,8(3):317-320(inChinese)
[7]吕丹,黎瑞源,郑冉,等苦荞种质资源籽粒黄酮含量及籽粒性状的变异分析[J]分子植物育种,2020,18(14):4762 4774
LYU D,LI R Y,ZHENG R,et al.V al'1atlon analysis of navonoldsconteIlt m seeds and seed tralts of Tan8ry buckwheat gemlplasmresources[J].Molecular Plant Breeding,2020,18(14) 4762-4774(in Chinese)
[8]呂丹,黎瑞源,郑冉,等苦荞“翅米荞×野苦荞”重组自交系群体籽粒黄酮含量及籽粒性状的分析[J]贵州师范大学学报(自然科学版),2019,37(6):40-46
LYU D,LI R Y,ZHENG R,et al Analysls of favonoids content ingrams and grain traits on'Chimiqiao'and'Yekuqlao'recormbinantinbled lines populatlon of Tartary buckwheat[J].Journal of GuizhouNormal University(Natural Sciences,2019,37(6):40-46(inChinese)
[9]郑冉,黎瑞源,吕丹,等苦荞重组自交系群体籽粒黄酮含量与产量性状分析[J]广西植物,202l,4l(2):216-224
ZHENG R,LI R Y,LYU D,et al_Vallatlon analysls of navonoidcontems ln seeds and yleld tralts on recomblnant lnbred linepopulation of Tartary buckwheat[J] Guihaia,202l,4l(2):216-224(in Chinese)
[10]MATsuI K,wALKER A R Biosyhthesis and r.gulatlon ofnavon01ds in buckwheat[J]. Breeding Science,2020,70(1)74-84
[11]sHI T x,LI R Y,zHENG R,et al_Mappmg QTLs for looo—grainweight and genes conn'01ling huU type using SNP malker in Tartarybuckwheat(Fagopyrum tataricum)[J] BMC Genomics,2021,22142
[12]zHANG L J,LI x x,MA B,et al The Tart8ry buckwheat genomeprovldes insights ino rutin biosynthesis aIld ablotic stlesstolelance[J].Moleccular Plant,2017,10(9):1224-1237
[13]ZHANG K x,HE M,FAN Y,et al Res.quencing of global Tartarybuckwheat accesslons leveals mltiple domestlcatlon events and k.yloci assoclated with agronomic tralts[J].Gemome Biology,202l,22(1):23
[14]馬名川,刘龙龙,刘璋,等苦荞全基因组ssR位点特征分析与分子标记开发[J]作物杂志,202l(1):38 46
MA M C,LIU L L,LIU Z,et al Analysls of SSR locl m wholegeIlome and development of molecular malkers in Tallarybuckwheat[J].Grops,2021(1):38-46(in Chinese)
[15]贺润丽,尹桂芳,李春花,等苦荞种皮转录组ssR位点信息分析及其分子标记的开发[J]分子植物育种、2020、l8(18):60856092
HE R L,YIN G F,LI C H,et al DevelopmeIlt of molecular markersand SSR locl mfbnnatlon analysls of n'anscrlpt01ue m Tal18rybuckwheat seed coat[J].Molecular Plant Breeding,2020,l8(18):6085-6092(in Chinese)
[16]杜伟,王东航,侯思宇,等基于苦荞全长转录组测序开发ssR标记及遗传多样性分析[J]植物生理学报,2020,56(7):1432 1444
DU W,WANG D H,HOU S Y,et al Development of SSR malkersbased on full-length traIlscriptome sequencing and its application forgenetic diversity analysls in Fagopyrum tataricum[J].Plant,Physiology Journal,2020,56(7):1432-1444(in Chinese)
[17]sHI T x,LI R Y,cHEN Q J,et al De novo squencing of seedtranscriptome and development of genlc—SSR malkers m cmmonbuckwheat(Fagopyrum esculentum)[J].Molecular Breeding,2017,37(12):1-15
[18]黎瑞源,潘凡,陈庆富,等苦荞转录组EsT—ssR发掘及多态性分析[J]中国农业科技导报,2015,17(4):42-52
LI R Y,PAN F,CHEN Q F,et al.Excavatlon and polyluorphismanalysis of EST—SSR flom transc.1ptome of Tal18ry buckwheat[J]Journal of Agricultural Science and Technology 2015.17(4):42—52(in Chinese)
[19]黎瑞源,石桃雄,陈其皎,等中国35个苦荞审定品种EsT—ssR指纹图谱构建与遗传多样性分析[J]植物科学学报,2017,35(2):
267-275
LI R Y,SHI T X.CHEN Q J,et al_Constmctlon of EST—SSRfingerinting and analysls ofgenetlc diversity ofthirty—five registeredTa11ary buckwheat cuhlvars(Fagopyrum tataricum)in China [J]Plant Science Journal 2017.35(2):267-275(in Chinese)
[20]李春花,陈蕤坤,王艳青,等利用ssR标记构建云南苦荞种质资源分子身份证[J]分子植物育种,2019,17(5):1575-1582
LI C H,CHEN R K,WANG Y Q,et al EstabllsluueIlt of themolecular ID for Yuman Tart8ry buckwheat genuplasm resourcesbased on SSR marker[J].Molecular Plant Breeding,2019,l7(5):1575-1582(in Chinese)
[21]屈洋,周瑜,王钊,等苦荞产区种质资源遗传多样性和遗传结构分析[J]中围农业科学,2016,49(11):2049—2062
QU Y,ZHOU Y,WANG Z,et al.Analysis of genetlc diversity andstrucnlre of Tartary buckwheat resoulces flom production regions[J]Scientia Agricultura Sinica,2016,49(ll)2049-2062(inChinese)
[22]徐笑宇,方正武,杨璞,等苦荞遗传多样性分析与核心种质筛选[J]干旱地区农业研究,2叭5,33(1):268 277
XU X Y,FANG Z W,YANG P,et al Genetic diversity analysls ofTallary buckwheat and selection of core coUections[J].Agricultural,Research in the Arid Areas.2015 33(1):268-277(in Chinese)
[23]杨学文,丁素荣,胡陶,等l04份苦荞种质的遗传多样性分析[J]作物杂志、2013 (6):13-18
YANG X W. DING S R,HU T, et al Genetic diversity of 104 Tartarybuckwheat accessions [J].Crops. 2013(6):13-18.( in Chinese)
[24]HOU s Y. SUN z X. BIN L H. et al Genetic diversity of buckwheatcultivars (Fagopyrum tartaricum Gaertn.) assessed with SSR markersdeveloped froin genome survey sequences [J]. Plant MolecularBiology Reporter. 2016. 34(1):233-241
[25]黎瑞源,粱龙兵,石桃雄,等苦荞重组自交系群体Fs代SSR遗传图谱的构建[J]贵州师范大学学报(自然科学版),2017.35(4):31-45
LI R Y. LIANG L B SHI T X. et al Consturction of a microsatellite-based genetic map of Tartary buckwheat using F5 recombinant inbredlines EJl Journal of Guizhou Normal University (Natural Sciences).2017. 35 (4): 31-45. (in Chinese)
[26]杨美甘蓝型油菜根系形态对低磷胁迫的反应及其QTL分析[D]武汉:华中农业大学,2010
YANG M Response of root morphology to low phosphorus stress andQTL analysis in Brassica napus[D] Wuhan: Huazhong AgriculturalUniversity. 2010 (in Chinese)
[27]赵宇慧,李秀秀,陈倬,等生物信息学分析方法I:全基因组关联分析概述[J]植物学报、2020,55(6):715 732
ZHAO Y H,LI X X,CHEN Z,et al.An overvlew of genome—wldeassoclation smdies m plants[J] chinese Bulletin of Botany,2020,55(6):715 732(in Chinese)
[28]徐刚标植物群体遗传学[M]北京:科学出版社,2012:193 200
[29]马名川,张丽君,刘璋,等基于SSR标记的山西省小同地区苦荞遗传多样性分析[J]山西农业大学学报(自然科学版),2021,41(3):25 3l
MA M C,ZHANG L J,LIU Z,et al Analysls of genetlc dlverslty ofTal18ry buckwheat flom dlfkrem r.glons of ShaIlXl Provmce based onSSR marker[J].Journal of Shanxi Agricultral University(Natural,Science Edition.2021 4l(3):25-31(in Chinese)
[30]GuPTA N,sHARMA s K,RANA J c,et al.AFLP fingerprinting ofTartary buckwheat accesslons(Fagopyrum tataricum)displaying rutincontemvarlatlon[J].Fitoterapia,2012,83(6):ll31-ll37
(責任编辑:于洪杰)
收稿日期:2021-0425初稿;2021-0629修改稿
作者简介:石桃雄( 1980-),女,博士,副教授,研究方向:荞麦种质资源保育及创新(E-mail: shitaoxiong@126.com):共同第作者:黎瑞源
( 1980-),男,博士,副教授,研究方向:生物信息学(E-inail: ruiyuan li@126.coin)
*通信作者:粱成刚( 1985-),男,博士,副教授,研究方向:荞麦遗传育种(E-mail: jesselcg@163.com)基金项目:围家重点研发计划项日(2019YFDl001300. 2019YFDl001301):国家自然科学基金项日(31960125):贵州省科技支撑计划项日(黔科合ZC[2019]2298);贵州省荞麦种质资源保育及创新重点实验室建设基金项日(黔教合KY[2017]002);贵州师范大学博士启动基金项日(11904/0514027)

