兰科虾脊兰属HSP70基因家族及其适应性进化
朱雅婷 俞霞 李笑平 刘仲健 吴沙沙 兰思仁 翟俊文

摘 要:生物體受环境刺激,尤其是高温刺激时会迅速产生热激蛋白(Heat shock proteins,HSPs),帮助受逆境胁迫导致的变性蛋白恢复正常到折叠状态,以维持其正常生长。兰科(Orchidaceae)虾脊兰属(Calanthe R. Br.)植物海拔分布范围可从海平面至3 500 m,是研究HSP基因家族的理想材料。本研究通过虾脊兰属7种植物叶片转录组数据进行HSP70基因的筛选,再对各基因的理化参数、家族标签、保守结构域和选择压力等参数进行分析。结果显示,7种植物中共鉴定出47个HSP70基因,且发现随着物种海拔的降低基因数量依次增多,其中大黄花虾脊兰(C. striata)和三棱虾脊兰(C. tricarinata)分别为4个,三褶虾脊兰(C. triplicata)5个,香花虾脊兰(C. odora)7个,长距虾脊兰(C. sylvatica)8个,银带虾脊兰(C. argenteo-striata)9个,中华虾脊兰(C. sinica)10个,其基因数量可能与物种对温度的适应进化相关;HSP70s基因有典型的3个结构域和标签,且N-末端motif数量最多;选择压力Ka/Ks< 基因家族较保守,表明其受到了纯化选择;系统发育分析显示,HSP70s基因聚为6组,相同亚细胞的基因聚为一组,说明同一细胞器的HSP70s可能具有相似的功能。该研究结果为虾脊兰属植物HSP基因家族的功能和对环境的适应性进化提供了新视角,为其适应性育种提供参考。
关键词:虾脊兰属;适应性进化;热激蛋白
中图分类号:S682.31 文献标识码:A
Analysis of HSP70 Gene Family and Adaptive Evolution in Calanthe (Orchidaceae)
ZHU Yating, YU Xia, LI Xiaoping, LIU Zhongjian, WU Shasha, LAN Siren, ZHAI Junwen*
Key Laboratory of National Forestry and Grassland Administration for Orchid Conservation and Utilization, College of Landscape Architecture, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
Abstract: Heat shock proteins (HSPs) emerge quickly when organisms are stimulated, especially by high temperature, which help the denatured protein return to its normal folding state so as to maintain its normal growth. Calanthe R. Br. (Orchidaceae) is an ideal group of plant to study the HSP gene family as they present an altitude distribution range from 0 m up to 3 500 m. In this study, HSP70s genes of seven Calanthe plants were identified through transcriptome data; consequently physical and chemical parameters, gene family tags, conserved domains and selection pressure of the each gene were analyzed. A total of 47 HSP70 gene sequences were confirmed in the seven plants, and the gene numbers increased in descending order along with the altitude. Among them, there were four sequences in C. striata and C. tricarinata, five in C. triplicata, seven in C. odora, eight in C. sylvatica, nine in C. argenteo–striata and ten in C. sinica. HSP70 genes had three typical domains and tags, and the number of N–terminal motifs was the largest. The selection pressure Ka/Ks<1, which revealed the gene family was conservative, subject to purification selection. Phylogenetic analysis showed that HSP70s were clustered into 6 groups. The genes from the same subcells were clustered into one group, which inferred that the HSP70 from the same organelle had similar functions. The results of this study would provide a new perspective for the study of the function and adaptive evolution of HSP family genes in the genus Calanthe, and provide suggestions for the adaptive breeding.
Keywords: Calanthe R. Br.; adaptive evolution; heat shock protein
DOI: 10.3969/j.issn.1000-2561. 2021.09.009
热激蛋白(Heat shock proteins,HSPs)几乎存在所有生物体中,按分子量的大小,可分为HSP100、HSP90、HSP70、HSP60及HSP20等5个大家族,其中HSP70家族是生物体中分布最广泛、最保守的基因家族,研究也最为深入[1]。该基因的一级结构的氨基酸序列由2个部分组成:N-末端44 kD的ATP酶结构域(NBD),约由450个氨基酸残基组成的高度保守区;C-末端25 kD的底物结合域(SBD),约由200个氨基酸组成,又可以分为一个15 kD左右相对保守的多肽的结合部位,另一个10 kD可变区域结合域[2]。
HSP70基因家族的生物学功能包括2大类,一类作为分子伴侣,辅助蛋白质的正确折叠、组装和转运且维持构象的稳定[3-4]。如新生肽链从核糖体释放后,由于细胞质内高浓度的溶质环境,易使肽链包裹一些疏水性残基,造成错误的卷曲折叠,HSP70s便可以保护新生的肽链不聚集,防止非天然构象形成[5]。另一类参与一些应激反应,包括植物体内的干旱、低温、高温、盐度和重金属等多个代谢调控。如铁皮石斛(Dendrobium officinale)受低温胁迫后HSP70开始表达且能维持数小时,表明参与了低温的逆境胁迫[6];参与植物病毒组分及金属胁迫的互作机制,相同物种在冶炼厂污染区和未污染生境条件下,前者体内HSP70表达水平明显更高[7-9]。及可被热激诱导并提高耐热性,拟南芥(Arabidopsis thaliana)经热处理后HSP70表达量激增,且过表达可以提高植株的耐热或耐旱能力[10-11]。同时部分研究显示物种的耐热程度与HSP70基因的表达相关,如低海拔蝴蝶的HSP70表达比高海拔蝴蝶高[12]。
中国分布的虾脊兰属(Calanthe R. Br.)植物有60余种,属内物种多样性较高,超过20种为中国特有。该属植物在气候温和的中新世中期分化,后因为青藏高原的抬升运动导致气候变冷却,使得高山起源的虾脊兰属植物为应对气候变化逐渐向低海拔区域扩散,形成从海平面至3500 m以上的垂直分布格局[13]。虾脊兰属植物在应对温度变化时产生了新的适应性,使得不同种演化出不同的海拔分布格局,是研究适应性较理想的材料。热激蛋白是适应环境变化的一个重要媒介分子,在生物长期适应环境温度中起着重要的作用。本研究中以分布于不同海拔范围的虾脊兰属7种植物作为研究对象,从生物信息学角度分析虾脊兰属植物的HSP70基因家族特性,对研究该属的适应性进化有一定的参考价值。
1 材料与方法
1.1 材料
实验材料中华虾脊兰(C. sinica)、银带虾脊兰(C. argenteo-striata)、长距虾脊兰(C. sylvatica)和三褶虾脊兰(C. triplicata)已种植于海拔35 m的福建农林大学兰苑1年,2016年6月取样。香花虾脊兰(C. odora)来源于云南昆明海拔810 m原生境,大黄花虾脊兰(C. sieboldii)来源于湖南阳明山海拔1200 m原生境,三棱虾脊兰(C. tricarinata)来源于四川黄龙风景区海拔1700 m原生境,这3种虾脊兰属植物于2017年6月取样。中华虾脊兰、银带虾脊兰、香花虾脊兰、长距虾脊兰和三褶虾脊兰是低海拔耐热物种,海拔分布范围分别为0~1100、500~1200、750~1300、800~2000、0~3000 m,三褶虾脊兰分布较广,总体上低海拔分布较多。大黄花虾脊兰和三棱虾脊兰是高海拔耐寒物种,海拔分布范围为1200~1500 m和300~3500 m[13]。外类群用深圳拟兰(Apostasia shenzhenica)和铁皮石斛(Dendrobium catenatum)已测序的基因组数据。
1.2 方法
1.2.1 样品的采集与测序 采集虾脊兰属植物新鲜幼嫩的叶片作为转录组样品,采样前用70%的酒精和超纯水对植株叶片表面进行喷洒清洗,样品于液氮中冷冻并转移至–80 ℃超低温冰箱保存。提取总RNA并构建文库,在Illumina Hiseq 2000平台上对文库进行测序。深圳拟兰、铁皮石斛基因组数据信息在NCBI下载所得(https:// www.ncbi.nlm.nih.gov/nuccore)。
1.2.2 HSP70基因家族成员的鉴定 使用蝴蝶兰‘满天红的PhHSP70 cDNA为查询序列(GenBank登录号为 MG214259),BLAST(比对阈值设置为1e-5)鉴定虾脊兰属植物、深圳拟兰和铁皮石斛中HSP70基因家族成员。设置默认E值的HMMER 3.0程序,用HSP70结构域(PF00012)来搜索鉴定候选基因[14]。将候选基因的蛋白序列分别提交至CDD(https://www. ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)、Pfam(https://pfam.xfam.org/)和SMART(http://smart. embl-heidelberg.de/)以确认HSP70的保守域[15-16]。最后用软件cdhit-4.6.2去除冗余序列[17]。
1.2.3 虾脊兰属HSP70基因家族物理特性与保守基序的获取 所有高可信度HSP70s序列均提交至ExPASy(http://web.expasy.org/protparam/),以计算氨基酸数量、分子量和理论等电点[18]。使用MEME程序(版本4.11.2,http://alternate.meme- suite.org/tools/meme)[19]來识别HSP70序列中的保守基序,其参数如下:任意重复次数最多10个,最佳基序宽度6~200个氨基酸残基。
1.2.4 亚细胞定位与多重序列比对 将HSP70蛋白序列分别提交至BIOCOMP(http:// busca.biocomp.unibo.it)和WoLF PSORT II(http:// www.genscript.com/wolf-psort.html),以分析HSP70s的亚细胞定位[20]。最后确认的HSP70基因用Jalview软件可视化比对序列[21]。
1.2.5 系统演化关系构建 同源基因的选择压力采用tbtool(simple Ka/Ks calculator)工具分析。将检索到的深圳拟兰、铁皮石斛和7种虾脊兰属植物HSP70基因家族成员用MEGA比对[22],再用CIPRES Science Gateway网站(https://www. phylo.org/)构建ML系统发育树,参数使用默认值[23]。
2 结果与分析
2.1 HSP70基因家族同源序列鉴定
本研究基于虾脊兰属7个植物的转录组数据共获得47个HSP70基因,其中大黄花虾脊兰所含的4个被命名为CSIEHSP7001~CSIEHSP7004,三棱虾脊兰的4个被命名为CTRAHSP7001~ CTRAHSP7004,三褶虾脊兰的5个被命名为CTRIHSP7001~CTRIHSP7005,香花虾脊兰的7个被命名为CODOHSP7001~CODOHSP7007,长距虾脊兰的8个被命名为CSYLHSP7001~ CSYLHSP7008,银带虾脊兰的9个被命名为CARGHSP7001~CARGHSP7009,中华虾脊兰的10个被命名为CSINHSP7001~CSINHSP7010,其等电点、分子量和亚细胞定位信息见表1。此外,本研究从深圳拟兰基因组数据中筛共选出17个HSP70候选基因(如Ash012061等),铁皮石斛基因组数据中筛选出23个HSP70候选基因(如XP_020702966.1等)。
ExPASy计算的结果如表1所示,虾脊兰属植物的47个HSP70蛋白序列差异较大,三棱虾脊兰、大黄花虾脊兰、三褶虾脊兰、香花虾脊兰、长距虾脊兰、银带虾脊兰和中华虾脊兰的氨基酸长度分子量分别为663~704、652~709、131~751、583~716、115~706、137~704、和101~706;等电点分别为5.09~6.08、4.99~6.01、4.65~8.12、4.87~ 6.63、4.55~7.31、4.72~8.86和4.30~9.05;分子量分别为73 008.97~75 104.05、71 400.99~76 285.95、73 457.43~82 452.76、63 570.05~76 428.61、12 825.61~ 76 046.77、14 961.48~75 176.07、10 940.19~ 75 985.74 kDa。
使用BIOCOMP和WoLF PSORT进行亚细胞定位分析,检测到虾脊兰属植物HSP70成员分别位于叶绿体、线粒体、细胞质和内质网中。细胞质中数量变化最大,三棱虾脊兰和大黄虾脊兰各只含有1个,中华虾脊兰和长距虾脊兰各含有5个。
2.2 HSP70基因序列特征和結构分析
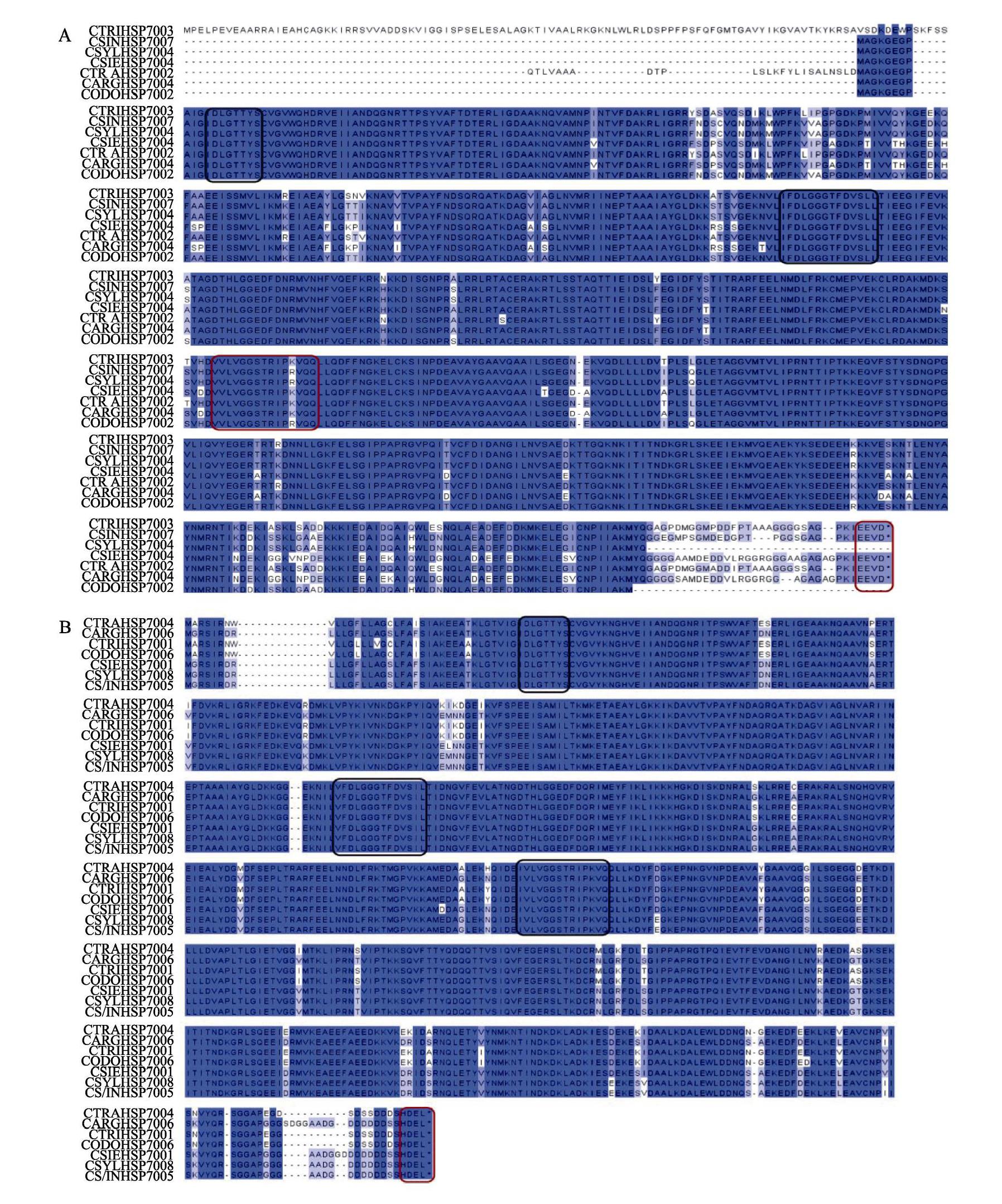
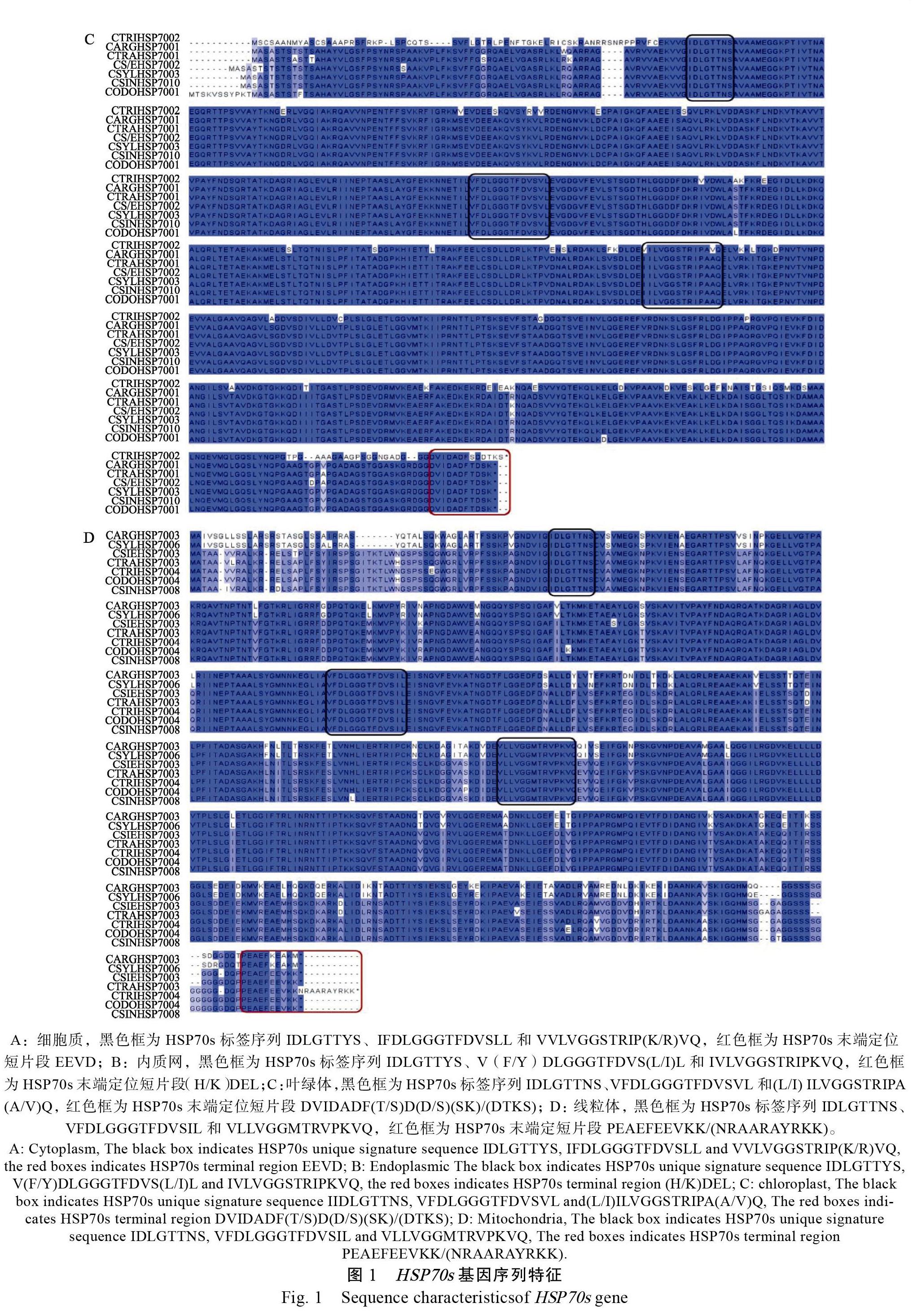
虾脊兰属植物HSP70含有3个典型的家族标签:IDLGTT(Y/N)S、(I/V)(F/Y)DLGGGTFD(I/V) S(L/I/V)L和(L/I/V)LVGG(S/M)TRIP(K/R/A)(V/ A)Q。HSP70s基因C-末端含有特定的短片段序列,这些有特征的短序列可将HSP70引向不同亚细胞内。虾脊兰属植物叶绿体HSP70的定位序列含有DVIDADF(T/S)D(D/S)(SK)/(DTKS)基序,线粒体的定位序列含有PEAEFEEVKK/ (NRAARAYRKK),内质网的定位序列含有(H/K) DEL基序,细胞质的定位序列EEVD与其他生物一样(图1A~图1D)。
同时该属植物HSP70的氨基酸序列含有3个保守结构域,N端ATPase结构域,中间铰链区和C端区域,其中N-端存有的ATP结合位点为A(S/E)(K/S/A)(F/Y)L(N/G)(D/K/S/T)(K/S/N/T/P)。
2.3 进化压力计算
为了确认HSP70在演化过程中的选择压力,对虾脊兰属内HSP70同源基因编码序列进行Ka/Ks计算。系统进化树直系同源基因对的Ka/Ks值除CSYLHSP7005-CSINHSP7004较大,为0.56,其余均小于0.5(图2)。
2.4 虾脊兰属HSP70保守基序分析
使用MEME工具分析了HSP70的保守基序,结果显示虾脊兰属7个物种存在10个保守基序。利用CDD进一步分析10个保守基序,再物理定位10个motif在基因中的位置。N端motifs数量多于C端,其中motif 1、2、5、6、8、9、10为HSP70的N端核苷酸结合结构域(nucleotidebinding domain,NBD)的结合位点(图3)。此外motif 8被鉴定为生物素化钙调蛋白(CaM)结合位点(RAL(R\S\G)(R\K)LR(T\R)(A\E) (C\A)ERAKR(A\T)LS(S\N)(T\Q)(A\H)),见图4。
2.5 虾脊兰属植物HSP70基因家族的系统进化分析
以铁皮石斛和深圳拟兰为外类群,对虾脊兰属植物47条HSP70s蛋白序列进行比对,构建ML系统发育树,结果见图5,不同细胞器的蛋白质序列聚为6组,不同物种中同一细胞器的HSP70聚集在一起。第Ⅰ组为内质网中的HSP70s基因。第Ⅱ组主要为铁皮石斛和深圳拟兰细胞质中的HSP70s基因,虾脊兰属植物细胞质中的HSP70s基因主要位于第Ⅲ组。第Ⅳ组中只包含铁皮石斛和深圳拟兰HSP70s基因,位于叶绿体中。第Ⅴ组为虾脊兰属植物叶绿体HSP70s基因。第Ⅵ组为线粒体中的HSP70s基因。系统发育树鉴定虾脊兰属中11对直系同源基因,其中海拔相近的物种的HSP70聚为一对(CODOHSP7004和CTRIHSP7004)。
3 讨论
3.1 HSP70基因数量与适应性进化
高等植物HSP基因家族的数量高于低等植物,可能是高等植物需要更多的基因来维持生物过程,大豆和烟草中各含有61个HSP70基因[1,24],水稻中含有32个HSP70基因[25],辣椒中含有21个HSP70基因[26],拟南芥中含有18个HSP70基因[27],海洋真核微藻Ostreococcus lucimarinus中含有8個HSP70基因及Ostreococcus tauri中含有7个HSP70基因[28]。也有研究表明高等植物中HSP70基因家族进化存在多样性,这种多样性可能与其对环境适应性相关,如兰科中铁皮石斛的HSP70基因家族成员比小兰屿蝴蝶兰多2倍,推测铁皮石斛比小兰屿蝴蝶的适应性更强,分布更加广泛[29]。
本研究中所用虾脊兰属7种植物共鉴定47个HSP70基因家族成员,不同植物基因家族数量不一,中华虾脊兰10个、银带虾脊兰9个、长距虾脊兰8个、香花虾脊兰7个、三褶虾脊兰5个、大黄花虾脊兰和三棱虾脊兰各4个。结合前人研究,虾脊兰属在冷气候的选择压力下,从高海拔逐渐扩散到低海拔。推测随着海拔的降低,虾脊兰属内HSP70基因数量逐渐增多,以适应低海拔高温气候。另外银带虾脊兰、香花虾脊兰、中华虾脊兰、长距虾脊兰和三褶虾脊兰从中新世开始逐渐积累和形成[13],在长期面对古气候胁迫时形成的环境功能基因HSP70多,有利于适应气候变换。
3.2 虾脊兰属植物HSP70s基因结构特征
虾脊兰属植物HSP70s有3个保守结构域,N端一个较保守的ATPase结构域,中间铰链区和C端可变区,C末端的变化可极大地改变螺旋的位置,对蛋白质功能上产生影响,极可能决定了单个HSP70的功能特异性[30-32]。同时鉴定到7种虾脊兰属植物HSP70基因的N-端ATP结合位点为A(S/E)(K/S/A)(F/Y)L(N/G)(D/K/S/T)(K/S/N/T/P)。N端ATP基序的结合是重要调制器,直接影响HSP70表达[33]。ATP水解是HSP70家族蛋白质执行分子伴侣功能所必须的条件,能诱导ATPase结构域构象变化,ATP结合到HSP70上才能使底物从结合结构域上释放出来[2]。
虾脊兰属植物HSP70s与其他植物相比同样具有该家族高度保守的序列特征,含有3个典型的家族标签:IDLGTT(Y/N)S、(I/V)(F/Y) DLGGGTFD(I/V)S(L/I/V)L和(I/V)VLVGG (S/M) TRIP(K/R/A)(V/A)(Q/S)。在真核生物中HSP70基因C末端序列,是将HSP70向特定细胞器进行转移的靶标[31,32,34-35]。在不同细胞器上,虾脊兰属HSP70基因家族的末端序列有各自的特点,且在序列上有高度保守和变化的位点。例如,叶绿体HSP70s末端定位片段为DVIDADF(T/S)D (D/S) (SK)/(DTKS),线粒体HSP70s末端定片段为PEAEFEEVKK/(NRAARAYRKK),内质网HSP70s末端定位片段为(H/K)DEL,而细胞质HSP70s末端为EEVD,该特征在动物和植物中具有高度的保守性,可能具有特定的功能。有些真核生物细胞质HSP70的EEVD参与HSP70功能的分子内调控和与分子间相互作用,可与一个保守的肽重复(TPR)结构域相互作用(如Hsp40、CHIP、HIP等具有TRP结构域),促进HSP70复合物的组装,如HIP与HSP70的结合可诱导HSP70的ATP酶结构域发生构象变化,从而抑制HSP70的ATP酶活性[36-37]。若删除或突变EEVD发现会影响HSP70的ATP酶活性以及与底物结合活性[38]。
3.3 不同物种GC含量差异的原因及基因选择压力
在选择压力分析中,Ka/Ks表示非义替换(Ka)和同义替换(Ks)之间的比例,这个比例可以判断是否有选择压力作用于这个蛋白质编码基因。当Ka/Ks> 基因受正选择(positive selection)即非纯化选择(diversifying selection),表明基因在该进化时间点可能产生一些新功能,这往往可能导致新基因或者物种新品种的产生;如果Ka/Ks= 则基因受中性选择;如果Ka/Ks< 则基因受负选择(negative selection)即纯化选择(purifyingd selection),当基因处于强烈负选择时,则意味着该物种的蛋白质保持原有的重要功能且趋于稳定[39-40]。虾脊兰属植物HSP70直系同源基因Ka/Ks<0.5,说明了该基因在虾脊兰属中演化较慢且高度保守,在结构甚至是功能上已经具有一定的稳定性。
3.4 虾脊兰属植物HSP70s基序与保守性
虾脊兰属植物中HSP70 N端的结合位点多于C端,且存在生物素化钙调蛋白(CaM)结合位点(RAL(R\S\G)(R\K)LR(T\R)(A\E)(C\A)ERAKR (A\T)LS(S\N)(T\Q)(A\H)),即motif 8。研究显示,拟南芥和玉米(Zea mays)幼苗都存在CaM位点[41-42],用CaCl2溶液预处理玉米种子可显著增强其耐热性[43]。虾脊兰属植物中该位点的作用能否增加耐热性,还需要进一步验证。
结合虾脊兰属系统发育树分析发现,距离较近的HSP70具有相似的motif组成,motif组成越相似,进化方式和基因功能则可能越相近,这与玉米[35]、拟南芥[10, 27]、大豆[1]、水稻[25, 44]等模式植物的研究结果较为相似。虾脊兰属植物motif除了细胞质中HSP70存在缺失、移位和置换,其他HSP70家族成员之间几乎没有缺失,证明了HSP70s家族在虾脊兰属内保守性非常高。
3.5 HSP70s在兰科中的分化及功能
通过牛耳草[45]、大豆[1]、煙草[24]等HSP70s基因家族的研究,发现该基因在双子叶植物/单子叶植物分化之前产生,从单个共同的祖先HSP70基因经过远古复制事件产生进化而来,在不同物种内基因数量、结构等有自身的特异性扩展,从而增加了HSP70s在物种内的多样性,这种特异性进化体现了物种在适应环境过程有自身的选择性。对虾脊兰属植物HSP70基因家族系统发育分析发现,所有不同物种HSP70s在6组中均有分布,不存在完全单独的分枝;虾脊兰属直系同源基因11对,深圳拟兰和铁皮石斛有4对。由于直系同源片段起源于物种分化,因此可以判断,起源于共同祖先的直系同源HSP70基因在兰科植物分离前就存在。
系统发育分析中,相同细胞器中HSP70s聚为一枝,类似现象出现在玉米[35]、拟南芥[10,27]、水稻[25, 44]、烟草[24]等植物HSP70s基因家族中。此外还发现,细胞质HSP70更倾向于结合富含亮氨酸的肽基序(即富含脂肪族残基),线粒体同源HSPA5更倾向于芳香残基序[46]。说明虾脊兰属不同植物的相同细胞器中HSP70s比同一物种不同细胞器中HSP70s的同源性要高,相同细胞器的HSP70s基因具有相似的功能,且有9对直系同源基因序列来源于海拔分布范围较接近的物种序列,意味着直系同源基因功能可能更加相似。即在面对胁迫时,海拔分布范围较近虾脊兰属植物面对温度胁迫反应机制可能相似。
虾脊兰属植物HSP70s基因数量相差主要是细胞质中HSP70的数量多于其他亚细胞器,在虾脊兰属植物中HSP70的基因数量差异最大的是细胞质中的数量,三褶虾脊兰、香花虾脊兰、银带虾脊兰、中华虾脊兰和长距虾脊兰主要是细胞质数量多,高海拔分布的三棱虾脊兰和大黄花虾脊兰仅含有1条。推测其原因:(1)细胞质是生物体新陈代谢的主要场所[47],蛋白质是在细胞质中合成,组成型的HSC70需要向各个亚细胞器运送蛋白质,且也需要作为分子伴侣参与新生肽的折叠和错误蛋白的降解等多种细胞活动[48];(2)提高抗性的遗传基础大部分是细胞质HSP70s的因素,通过杂交获得的遗传变异可提高物种的耐热性[49]。因此自然选择可能导致了虾脊兰属物种之间HSP70基因的差异,这种差异很可能造成了热适应性的强弱,也使不同虾脊兰属植物获得不同耐热性以形成各自的海拔和纬度分布区。
4 结论
虾脊兰属植物在应对环境变化方面具有多样化的适应机制,且物种分化的过程中产生了新的适应性。但截至目前,该属植物对环境适应相关的功能基因挖掘,及其生态适应性的分子机制研究尚未见报道。
本研究对虾脊兰属HSP70基因家族进行生物信息学分析,探索虾脊兰属植物适应性演化。获得的主要结论和发现如下:
(1)虾脊兰属7种植物共鉴定47个HSP70基因家族,其中低海拔虾脊兰比高海拔虾脊兰的基因数量多,且推测细胞质HSP基因数量的差异可能造成了虾脊兰属植物之间的耐热性的差异,以此适应不同海拔区域环境温度。
(2)虾脊兰属的HSP70基因结构与其他植物该基因家族特征一致,有3个保守结构域,N端ATPase结构域,中间铰链区和C末端区域序列。且含有HSP70基因家族3个典型的家族标签:IDLGTT(Y/N)S、(I/V)(F/Y)DLGGGTFD(I/V)S(L/I/V)L和(I/V)VLVGG(S/M)TRIP(K/R/A)(V/A) (Q/S)。Motif组成显示,HSP70基因N端有较多的转录结合位点,且motif 8被鉴定为CaM结合位点。
(3)HSP70基因家族纯化选择显示,该基因家族在虾脊兰属中较保守,说明该基因在功能上已经具有一定的稳定性。
(4)HSP70s基因系统发育树聚为6组,相同细胞器HSP70s基因比同一物种不同细胞器HSPs的同源性要高,说明同一细胞基因功能相似。
参考文献
[1] Zhang L, Zhao H K, Dong Q L, et al. Genome-wide analysis and expression profiling under heat and drought treatments of HSP70 gene family in soybean (Glycine max L.)[J]. Frontiers in Plant Science, 2015, 6: 773.
[2] 于晓丽. 三角涡虫热激蛋白70cDNA的克隆及生物信息学分析[D]. 陕西: 陕西师范大学, 2009.
[3] WANG W X, Vinocur B, Shoseyov O, et al. Role of plant heat-shock proteins and molecular chaperones in the abiotic stress response[J]. Trends in Plant Science, 2004, 9(5): 244-252.
[4] 刘 娜, 曲延英, 陈全家, 等. 棉花HSP70基因的克隆与原核表达[J]. 西北植物学报, 2015, 35(4): 688-693.
[5] Abrams J L. Functional analysis of cytosolic Hsp70 nucleotide exchange factor networks in Yeast[D]. State of Texas: The University of Texas MD Anderson Cancer Center, 2014.
[6] 李东宾, 高燕会, 斯金平, 等. 铁皮石斛HSP70基因的克隆及冷胁迫表达分析[J]. 中国中药杂志, 2013, 38(20): 3446-3452.
[7] K?hler H R, Triebskorn R, St?cker W, et al. The 70 kD heat shock protein (hsp 70) in soil invertebrates: a possible tool for monitoring environmental toxicants[J]. Archives of Environmental Contamination and Toxicology, 1992, 22(3): 334-338.
[8] Mathioudakis M M, Veiga R, Ghita M, et al. Pepino mosaic virus capsid protein interacts with a tomato heat shock protein cognate 70[J]. Virus Research, 2012, 163(1): 28-39.
[9] 王 勇, 任亞峰, 李冬雪, 等. 热休克蛋白与植物病毒组分的互作机制研究进展[J]. 应用与环境生物学报, 2018, 24(2): 415-424.
[10] Sung D Y, Vierling E, Guy C L. Comprehensive expression profile analysis of the Arabidopsis Hsp70 gene family[J]. Plant Physiology, 2001, 126(2): 789-800.
[11] Su P H, Li H M. Arabidopsis stromal 70-kD heat shock proteins are essential for plant development and important for thermotolerance of germinating seeds[J]. Plant Physiology, 2008, 146(3): 1231-1241.
[12] Karl I, S?rensen J G, Loeschcke V, et al. HSP70 expression in the copper butterfly Lycaena tityrus across altitudes and temperatures[J]. Journal of Evolutionary Biology, 2009, 22(1): 172-178.
[13] 陈燕琼. 广义虾脊兰属系统基因组学、生物地理学及网状进化[D] 福建: 福建农林大学, 2020.
[14] Finn R D, Tate J, Mistry J, et al. The Pfam protein families database[J]. Nucleic Acids Research, 2007, 36(suppl 1): D281-D288.
[15] Marchler-Bauer A, Anderson J B, Chitsaz F, et al. CDD: specific functional annotation with the Conserved Domain Database[J]. Nucleic Acids Research, 2009, 37(suppl 1): D205-D210.
[16] Letunic I, Copley R R, Schmidt S, et al. SMART 4.0: towards genomic data integration[J]. Nucleic Acids Research, 2004, 32: D142-D144.
[17] Huang Y, Niu B F, Gao Y, et al. CD-HIT Suite: a web server for clustering and comparing biological sequences[J]. Bioinformatics, 2010, 26(5): 680-682.
[18] Gasteiger E, Gattiker A, Hoogland C, et al. ExPASy: the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research, 2003, 31(13): 3784-3788.
[19] Bailey T L, Williams N, Misleh C, et al. MEME: discovering and analyzing DNA and protein sequence motifs[J]. Nucleic Acids Research, 2006, 34(suppl 2): W369-W373.
[20] Horton P, Park K J, Obayashi T, et al. WoLF PSORT: protein localization predictor[J]. Nucleic Acids Research, 2007, 35(suppl 2): W585-W587.
[21] Waterhouse A M, Procter J B, Martin D M A, et al. Jalview Version 2: a multiple sequence alignment editor and analysis workbench[J]. Bioinformatics, 2009, 25(9): 1189-1191.
[22] Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870- 1874.
[23] Miller M A, Pfeiffer W, Schwartz T. Creating the CIPRES Science Gateway for inference of large phylogenetic trees[C]//2010 Gateway Computing Environments Workshop (GCE). New Orleans, LA, USA. IEEE, 2010: 1-8.
[24] Song Z P, Pan F L, Lou X P, et al. Genome-wide identification and characterization of Hsp70 gene family in Nicotiana tabacum[J]. Molecular Biology Reports, 2019, 46(2): 1941- 1954.
[25] Sarkar N K, Kundnani P, Grover A. Functional analysis of Hsp70 superfamily proteins of rice (Oryza sativa)[J]. Cell Stress and Chaperones, 2013, 18(4): 427-437.
[26] Guo M, Liu J H, Ma X, et al. Genome-wide analysis of the Hsp70 family genes in pepper (Capsicum annuum L.) and functional identification of CaHsp70-2 involvement in heat stress[J]. Plant Science, 2016, 252: 246-256.
[27] Lin B L, Wang J S, Liu H C, et al. Genomic analysis of the Hsp70 superfamily in Arabidopsis thaliana[J]. Cell Stress and Chaperones, 2001, 6(3):201-208.
[28] 張毓婷. 雷蒙德氏棉HSP70基因家族进化与GhHSP70-20的功能解析[D]. 杭州: 浙江农林大学, 2014.
[29] Zhang G Q, Xu Q, Bian C, et al. The Dendrobium catenatum Lindl. genome sequence provides insights into polysaccharide synthase, floral development and adaptive evolution[J]. Scientific Reports, 2016, 6(1): 1-10.
[30] Fuertes M A, Jos M P, Soto M, et al. Thermodynamic stability of the C-terminal domain of the human inducible heat shock protein 70[J]. Biochimica et Biophysica Acta, 2004, 1699(1-2):45-56.
[31] Park H, Ahn I Y, Lee H E. Expression of heat shock protein 70 in the thermally stressed Antarctic clam Laternula elliptica[J]. Cell Stress and Chaperones, 2007, 12(3): 275-282.
[32] Wang Z L, Wu Z H, Jian J C, et al. Cloning and expression of heat shock protein 70 gene in the haemocytes of pearl oyster (Pinctada fucata, Gould 1850) responding to bacterial challenge[J]. Fish & Shellfish Immunology, 2009, 26(4): 639-645.
[33] Henics T, Nagy E, Oh H J, et al. Mammalian Hsp70 and Hsp110 proteins bind to RNA motifs involved in mRNA stability[J]. Journal of Biological Chemistry, 1999, 274(24): 17318-17324.
[34] 顾颖慧. 龙须菜热激蛋白70(HSP70)基因克隆及热激下的表达模式分析[D]. 青岛: 中国海洋大学, 2011.
[35] 宋晋辉, 马海莲, 瓮巧云, 等. 玉米HSP70基因家族的全基因组鉴定与分析[J]. 核农学报, 2017, 31(7): 1245-1254.
[36] 高娟玉. Hsp70调控蛋白HBP21在人视网膜色素上皮细胞及PVR增生膜中的表达[D]. 南京: 南京医科大学, 2008.
[37] Smock R G, Blackburn M E, Gierasch L M. Conserved, disordered C Terminus of DnaK enhances cellular survival upon stress and DnaK in vitro chaperone activity[J]. Journal of Biological Chemistry, 2011, 286(36): 31821-31829.
[38] Freeman B C, Myers M P, Schumacher R, et al. Identification of a regulatory motif in Hsp70 that affects ATPase activity, substrate binding and interaction with HDJ-1[J]. Embo Journal, 1995, 14(10): 2281-2292.
[39] Hurst L D. The Ka/Ks ratio: diagnosing the form of sequence evolution[J]. Trends in Genetics, 2002, 18(9): 486-487.
[40] 王 捷, 徐 羽, 石 瑛, 等. 念珠藻屬植物nifH基因的适应性进化分析[J]. 西北植物学报, 2019, 39(11): 1961-1969.
[41] Sun X T, Li B, Zhou G M, et al. Binding of the maize cytosolic Hsp70 to calmodulin, and identification of calmodulin- binding site in Hsp70[J]. Plant and Cell Physiology, 2000, 41(6): 804-810.
[42] Cha J Y, Su'udi M, Kim W Y, et al. Functional characterization of orchardgrass cytosolic Hsp70 (DgHsp70) and the negative regulation by Ca2+/AtCaM2 binding[J]. Plant Physiology and Biochemistry, 2012, 58: 29-36.
[43] Gong M, Li Y J, Dai X, et al. Involvement of calcium and calmodulin in the acquisition of heat-shock induced thermotolerance in maize seedlings[J]. Journal of Plant Physiology, 1997, 150(5): 615-621.
[44] Jung K H, Gho H J, Nguyen M X, et al. Genome-wide expression analysis of HSP70 family genes in rice and identification of a cytosolic HSP70 gene highly induced under heat stress[J]. Functional and Integrative Genomics, 2013, 13(3): 391-402.
[45] 张振南. 复苏植物牛耳草HSP基因家族分析及BhHSP70-1基因功能研究[D]. 大庆: 黑龙江八一农垦大学, 2013.
[46] Rosenzweig R, Nillegoda N B, Mayer M P, et al. The Hsp70 chaperone network[J]. Nature Reviews Molecular Cell Biology, 2019, 20(11): 665-680.
[47] 马炜梁, 王幼芳, 李宏庆. 植物学 [M]. 北京: 北京高等教育出版社, 2007: 12-38.
[48] 祁茂冬. 玉米HSP70家族成员的鉴定及抗旱基因的筛选和功能分析[D]. 保定: 河北农业大学, 2018.
[49] 崔旭红. B型烟粉虱和温室粉虱热胁迫适应性及其分子生态机制[D]. 北京: 中国农业科学院, 2007.
责任编辑:黄东杰

