基于信息熵的肿瘤样本纯度估算研究∗
王丽华
(中国石油大学(华东)计算机科学与技术学院 青岛 266580)
1 引言
随着高通量技术的发展和各基因组学和表观遗传学数据的出现,为研究肿瘤致病模式及解释肿瘤发病机制提供了可能。肿瘤研究中一个重要问题是临床获得的肿瘤样本总是混有正常细胞[1],我们称为“肿瘤纯度”,即肿瘤样本中肿瘤细胞所占比例。准确评估肿瘤纯度有利于肿瘤样本的准确鉴别分析,降低肿瘤基因分型、复发风险及疗效预测的分析偏差[2~4]。传统的肿瘤纯度估算方法基本上是由病理研究者通过图像分析获得,以及后来出现基于细胞分类的技术,这些方法耗人力且成本高,不适合用来大规模推广。巧合的是,肿瘤细胞和正常细胞之间存在着显著的遗传和表观遗传差异,因此利用现有的高通量数据来估计肿瘤纯度是可行的。
目前,已有很多方法利用基因表达、拷贝数变异和单核苷酸多态性作为预测因子来估计肿瘤纯度[5~12],但很少是基于DNA甲基化。异常的DNA甲基化模式和肿瘤的发生密切相关,几乎在所有的癌症中都存在,并且发生在癌症的早期,有望成为癌症早期诊断的理想标志物。ABSOLUTE[5]利用拷贝数变异数据结合最大似然估计方法直接计算肿瘤样本的纯度;ESTIMATE[13]利用基质、免疫细胞的基因表达谱结合经验累计分布函数来估计肿瘤纯度;MethylPurify[6]利用DNA甲基化测序数据识别差异位点结合EM算法来评估肿瘤纯度;Infinium⁃Purify[8,12]利用秩和检验识别DNA甲基化差异位点并结合高斯核密度函数计算肿瘤纯度。不难发现,目前利用甲基化数据评估肿瘤纯度的方法多是基于信息位点的选择。选择信息位点是指在肿瘤样本和正常样本中甲基化程度出现差异的CpG位点,差异越显著越有可能被识别为信息位点。尽管目前根据肿瘤和正常组织甲基化水平差异确定差异甲基化位点的方法已经得到了很好的研究,但不同的信息位点选择方法对肿瘤纯度的估计结果不尽相同,选择与肿瘤相关的差异甲基化位点作为信息位点显得尤为重要。与此同时,DNA甲基化数据相对于测序数据[14~15]来说是稳定且容易获得的,测序数据昂贵且应用范围有限,而突变数据则有样本不稳定的风险。近年来,利用DNA甲基化数据估计肿瘤纯度的方法开始出现,但仍然很少。
肿瘤纯度估算方法侧重于肿瘤间异质性[16],同一种肿瘤类型的样本识别一组差异基因或CpG位点,忽视了肿瘤生长空间的异质性,即样本特异性。本文基于DNA甲基化数据,利用样本位点的“信息熵”识别具有样本特异性的差异甲基化位点,并进行样本的肿瘤纯度评估工作。
2 数据来源与处理
本文使用了来自UCSC数据库[17~18]的肝癌LI⁃HC(Liver Hepatocellular Carcinoma)的DNA甲基化样本数据进行实验。
为了提高分类预测的准确性、有效性和可伸缩性,需要对下载到的数据进行预处理:数据清理和数据过滤。选择DNA甲基化数据中的具有癌旁样本的肿瘤样本数据;为了消除和减小数据噪声,我们对其中的缺失值进行了删除或填补处理。去除质量较差的CpG位点,过滤掉X、Y染色体上、SNP相关的CpG位点。
3 识别特异性信息位点
为了确定甲基化位点的显著差异,基于位点的“信息熵”来识别肿瘤样本的特异性信息位点。对于CpG位点i来说,其正常样本信息熵INi定义如下:
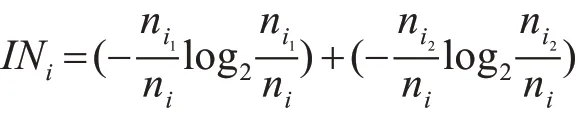
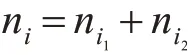
“超甲基化”定义如下:
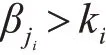
式中,βji表示正常样本j的CpG位点i的β-νalue,ki为用户定义的阈值。同理,“低甲基化”定义为
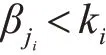
本文基于信息熵识别肿瘤样本的信息位点,IEi越小,说明对于CpG位点i在正常样本中信息熵越小,甲基化程度表现越稳定。对于CpG位点i,其肿瘤信息熵ITi定义如下:

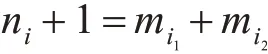
加入肿瘤样本后带来的信息量可以表示为
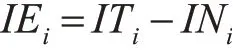
选择信息量增幅大的前s个CpG位点作为信息位点。为了评估选出的信息位点的显著差异性,即识别出的信息位点是个小概率事件。换句话说,信息位点的差异性不是随机的。当然,每个信息位点的p值可以根据公式进行计算,以保证信息位点选择的概率极小。根据王等[19]在全局零假设的前提下,信息位点的具体p值计算公式可以表示为

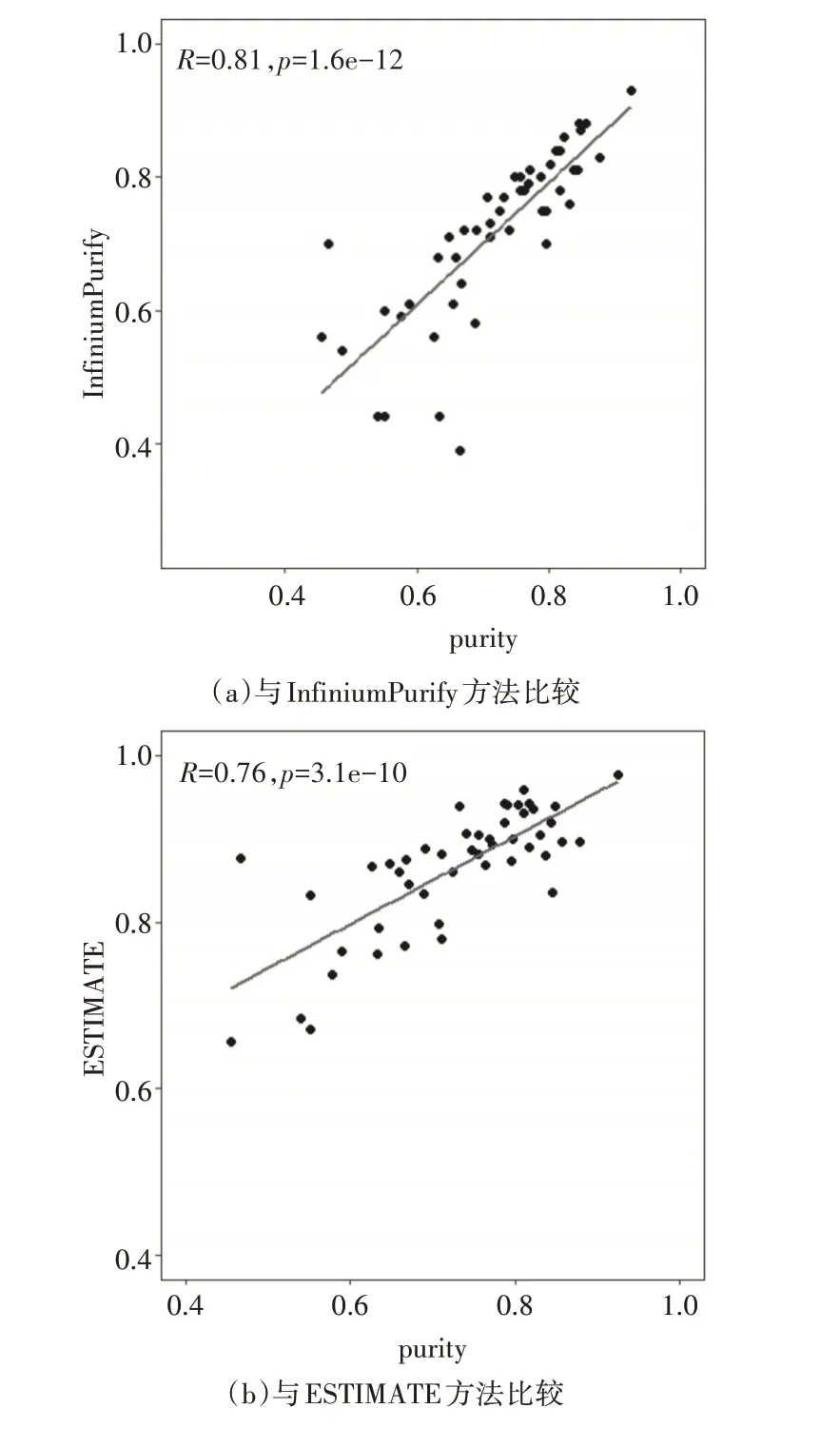
式中,D f为每次选择的信息位点的数量,m为置换检验重复的次数,Di为信息位点i的原始排名,~Di为信息位点i在置换检验中的排名,函数rank是用来计算信息位点的原始排名比置换检验中排名靠前的次数。当信息位点的pi 根据信息位点的β值估计肿瘤纯度,测定方法源于郑等人[12]。首先,确定肿瘤样本信息位点的甲基化程度,判定依据是正常样本每个CpG位点的平均β值。如果肿瘤样本中信息CpG位点的β值高于正常样本中相应的β值,则该CpG位点为高甲基化;如果β值低于正常样本,则该CpG位点为低甲基化。其次,转换肿瘤样本信息位点的β值。转换的规则是,如果CpG位点是超甲基化,则β值保持不变;如果该CpG位点是低甲基化,则β值转换为1-β。最后,利用高斯核密度估计方法对转换后的信息位点的β值进行估计。 本文采用皮尔森相关系数R(Pearson correla⁃tion coefficient)来度量不同数量信息差异甲基化位点的选择对肿瘤纯度的估算结果的影响。同时为了研究信息位点的显著差异性,将肿瘤纯度结果与数据集中随机选择的多组“信息位点”估算的结果进行比较。 图1表示的是选择不同数量的信息位点和随机位点的情况下,纯度估计值的相关系数R,这里计算的相关系数是与InfiniumPurify方法相比的。图1的横坐标表示选择不同数量的CpG位点,纵坐标表示本文方法估算出的结果与InfiniumPurify的相关性。图1中虚线表示的是利用本文方法选出的信息位点进行估计的,实线表示利用随机选择位点进行估计的相关性。从图1中可以看出,利用本文方法选出的信息位点估算出的肿瘤纯度结果与InfiniumPurify方法的相关性更高,这也表明本文方法选出的信息位点更具有显著差异性。同时,利用信息位点估算肿瘤纯度的曲线趋势,在信息位点数目未达到1000之前,相关性不断增加,选择的信息位点数目达到1000后相关性基本不再增加,后续基本保持稳定,因此我们后续实验过程中,信息位点的数目选择为1000。与此同时,利用随机位点估算肿瘤纯度的相关性随着位点选择数目的增加呈现出增长的趋势,但仍旧低于利用信息位点估计的相关性。我们后续随机选择20000个CpG位点,相关性会呈现出略微下降的趋势,这是由于选择的位点数目越多,冗余信息也越多。 图1 选择不同数量CpG位点的相关性 本文得到的肿瘤纯度估算结果与InfiniumPuri⁃fy、ESTIMATE、CPE方法比较的散点图分别如图2(a)、(b)、(c)所示。图中的横坐标表示的都为本文方法估算出的肿瘤纯度值,纵坐标分别表示Infini⁃umPurify、ESTIMATE、CPE方法估计出的肿瘤纯度值。图中的斜线表示相关性近似程度的趋势线。图中的R标识两种方法皮尔森相关系数,p是指p值,表示的是显著性水平。 图2 与InfiniumPurify、ESTIMATE、CPE方法肿瘤纯度比较散点图 通过图2可以看出,本文方法估算出的肿瘤纯度值与现有的其他方法具有较高的一致性。图2(a)中与InfiniumPurify方法相关性最高,为0.81,最低为与CPE方法的相关性,为0.54。图2(c)中的CPE方法的结果是取ABSOLUTE、ESTIMATE、HE染色和LUMP方法肿瘤纯度结果的中值获得的,而ABSOLUTE方法没有对应的肿瘤样本纯度值。与CPE方法的相关性略低一些,这不排除是因为我们实验所用的样本数略少的原因,肿瘤纯度的差异容易影响相关性的高低。将方法应用于更多的肿瘤样本,这也是后续要继续研究的方面。 本文使用UCSC数据库中肝癌的DNA甲基化数据,筛选出其中的疾病样本及其配对的正常样本数据,基于CpG位点的“信息熵”识别出肿瘤样本的特异性信息位点,根据高斯核密度估计方法,利用甲基化信息位点的显著差异性估算肿瘤样本的纯度。实验结果表明本文能够准确地估算出肿瘤纯度,与现有的其他方法具有高度一致性,且估算出的肿瘤纯度结果考虑了样本特异性,更具生物学意义,且DNA甲基化数据相较于突变数据、拷贝数变异数据更具有稳定性,为研究肿瘤样本提供了不同方面的解释。4 最佳信息位点数目选取

5 实验结果及分析

6 结语

