基于生物信息学分析的卵巢癌中miR- 4324调控机制的研究
眭怡群,张永胜
(苏州大学附属第二医院 病理科,江苏 苏州 215004)
卵巢癌是女性生殖系统常见的恶性肿瘤之一,其发病隐匿,缺乏有效的治疗手段,致死率高,因此了解卵巢癌发生发展的分子机制并制定新的治疗策略十分重要。微小RNA(microRNA,miRNA)是一类长度19~25个核苷酸的非编码单链小分子RNA,通过与靶mRNA的3′端非编码区特异性结合,从而在转录后水平上对目的基因表达进行负调控[1]。miRNA异常表达与肿瘤的关联是目前的研究热点。近来的研究表明miR- 4324的异常表达参与了多种肿瘤的生物学过程。Li等[2]研究表明,miR- 4324通过靶向HOXB2基因,从而抑制结直肠癌的发生。Inamoto等[3]报道miR- 4324的表达与膀胱尿路上皮癌患者的预后有关。陈军莹等[4]发现,与正常宫颈组织相比,宫颈鳞状细胞癌组织中miR- 4324表达下调。目前miR- 4324在卵巢癌中的研究尚未见报道,作者旨在利用生物信息学研究miR- 4324在卵巢癌中的表达,并进一步分析miR- 4324在卵巢癌发生、发展中的作用。
1 材料与方法
1.1 提取卵巢癌中miRNA表达数据
从GEO数据库(Gene Expression Omnibus,http:∥www.ncbi.nlm.nih. gov/ GEO/)中筛选并下载卵巢癌miRNA表达的数据集GSE83693和GSE119055,评估卵巢癌中miR- 4324表达情况。数据集GSE83693包括8例原发性卵巢癌组织、8例复发性卵巢癌组织和4例正常卵巢组织。数据集GSE119055包括 6例卵巢癌组织和3例正常卵巢组织。
1.2 分析卵巢癌中miR- 4324的表达水平
用GEO2R在线工具(https:∥www.ncbi.nlm.nih.gov/geo/GEO2R/)基于R软件“LIMMA”包筛选卵巢癌组织和正常卵巢组织之间差异表达的miRNAs(DE- miRNAs),分为4个不同的组,其中数据集GSE83693分成3个组,分别为4例正常卵巢组织与8例原发性卵巢癌组织(83693NP组)、4例正常卵巢组织与8例复发性卵巢癌组织(83693NR组)、4例正常卵巢组织与16例(原发+复发)卵巢癌组织(83693NC组),最后1个组是数据集GSE119055中3例正常卵巢组织与6例卵巢癌组织(119055组)。筛选阈值为| log2FC |>1和adjP<0.05,4组分别得到各自差异表达的miR- 4324。
1.3 预测miR- 4324的下游靶基因(TG- miR4324)
利用TargetScan数据库(www.targetscan.org/vert_72)预测miR- 4324的TG- miR4324。
1.4 提取卵巢癌中mRNA表达数据
从GEO数据库(Gene Expression Omnibus,http:∥www.ncbi.nlm.nih. gov/GEO/)中下载卵巢癌mRNA表达的数据集GSE66957和GSE14407,筛选并识别卵巢癌中差异表达基因(DEGs)。数据集GSE66957包括57 例卵巢癌标本和12例正常卵巢标本,数据集GSE14407包括12例卵巢癌标本和12例正常卵巢标本。
1.5 DEGs的筛选
利用GEO2R筛选卵巢癌中的DEGs,筛选标准为adjP<0.05和| log2FC |>1。
1.6 TG- miR4324与DEGs的交集
应用在线工具绘制文氏图,取TG- miR4324与数据集GSE66957 DEGs、GSE14407 DEGs的交集,筛选出验证后的miR- 4324相关性差异靶基因,并作进一步研究。
1.7 GO注释和KEGG路径富集分析
利用DAVID 数据库(https:∥david.ncifcrf.gov/)对miR- 4324相关性差异靶基因进行GO功能注释和KEGG途径富集分析。P<0.05表示差异具有统计学意义。
1.8 PPI网络构建与模块选择
利用检索相互作用基因的搜索工具(STRING,11.0版,https:∥STRING- db.org/)数据库[5],基于miR- 4324相关性差异靶基因构建蛋白质- 蛋白质相互作用(PPI)网络,然后由Cytoscape软件[6](3.7.2版)可视化,以置信度C≥0.4为截止标准。用cytoHubba插件筛选hub基因。然后用MCODE插件筛选功能模块,筛选度阈值=2,节点数阈值=0.2,最大深度=100,K核=2的PPI网络模块。
1.9 生存分析
利用在线生存分析工具Kaplan- Meier Plotter(http:∥kmplot.com/ analysis)评估hub基因对卵巢癌患者总生存期(OS)的影响。
2 结 果
2.1 miR- 4324在卵巢癌中表达下调
miR- 4324的表达是基于GEO数据库在一系列卵巢癌和正常卵巢组织中评估的。共收集了两个GEO数据集(GSE83693和GSE119055)。分为83693NP、83693NR、83693NC、119055组共4组。GEO2R分析显示,与正常卵巢组织相比,各组卵巢癌中miR- 4324表达均下调(表1)。
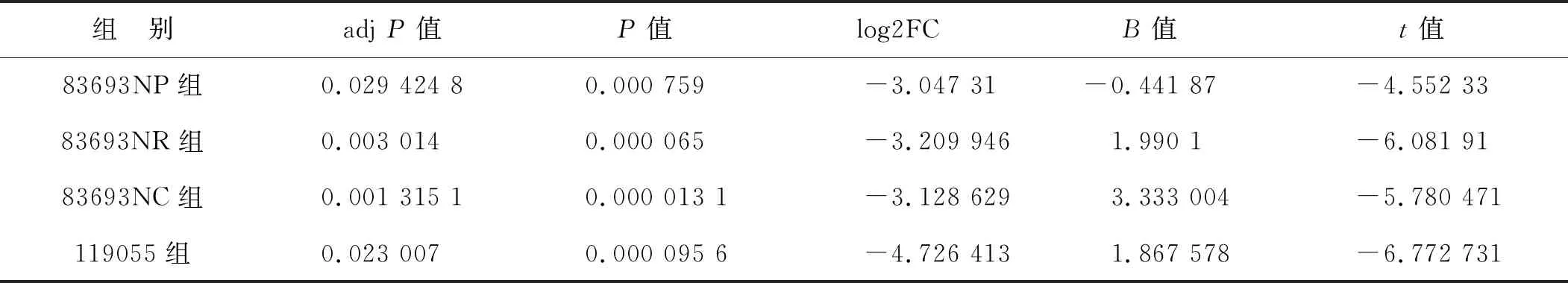
表1 miR- 4324在不同组别中的表达
2.2 TG- miR4324的预测
利用TargetScan数据库预测了TG- miR4324,共计5 292个靶基因。
2.3 miR- 4324相关性差异靶基因的筛选与验证
从GSE66957和GSE14407数据集中分别筛选出5 775 和5 665个DEGs,其中上调的DEGs分别为4 487 和3 574个,然后上调的DEGs与TG- miR4324取交集,以此验证TG- miR4324,共得到290个miR- 4324相关性差异靶基因(图1)。
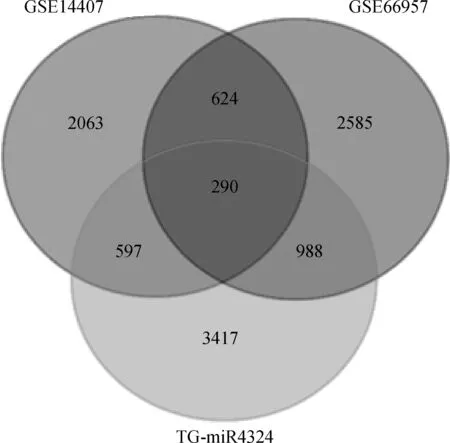
图1 GSE66957上调的DEGs及GSE14407上调的DEGs与TG- miR4324的交集
2.4 290个miR- 4324相关性差异靶基因在卵巢癌中的功能分析
使用DAVID 数据库对290 个差异靶基因进行GO 和KEGG富集分析。GO 分析(表2)显示,细胞学过程(biological processes,BP) 变化主要是血管再生、血管内皮细胞增殖、细胞外基质、细胞黏附、迁移、胞内循环、蛋白糖基化、蛋白类泛素化修饰、硫酸角质素生物合成过程、RNA聚合酶Ⅱ启动子的转录调控、ARF蛋白信号转导的调控、对肽类激素的反应、细胞命运锁定等104个功能簇;分子功能(molecular function,MF) 变化主要是染色体结合、ATP结合、泛素蛋白转移酶活性、细胞外基质组织、转录因子活性、序列特异性DNA结合、赖氨酰氧化酶活性等93个功能簇;细胞成分(cell component,CC) 变化主要是细胞外基质、细胞质、细胞基膜、蛋白质性细胞外基质、高尔基体、初级内含体、细胞骨架、基底膜等143个功能簇。KEGG 通路分析(表3)显示,基因富集于细胞外基质受体的相互作用、糖胺聚糖生物合成(硫酸角质素)和癌症相关通路共3条信号通路。

表2 卵巢癌中miR- 4324相关性差异靶基因GO(前10)富集分析
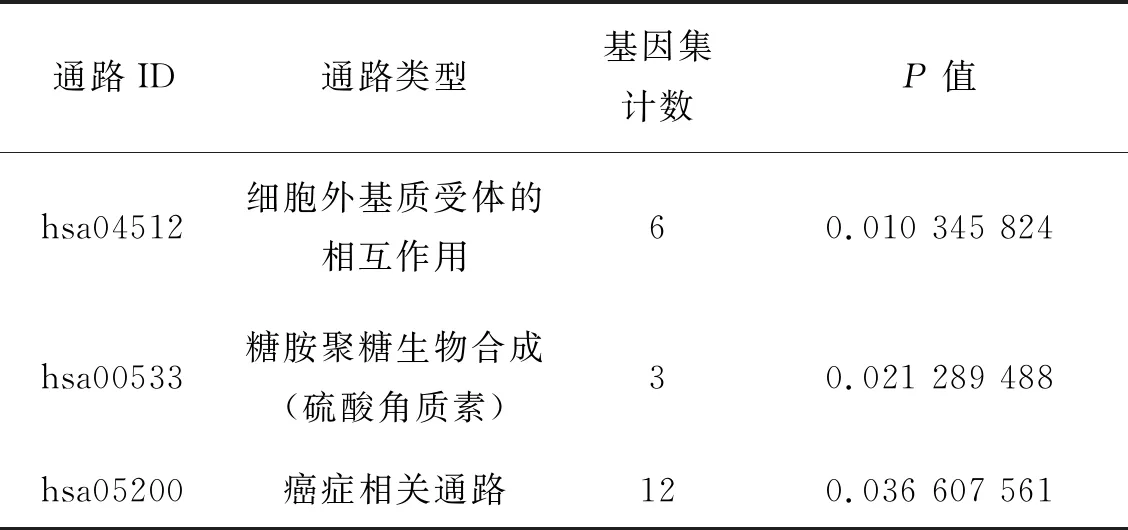
表3 卵巢癌中miR- 4324相关性差异靶基因KEGG通路分析
2.5 PPI网络构建与模块选择
290个miR- 4324相关性差异靶基因的PPI网络由198个节点和330个边缘组成(图2)。以度≥10为截止标准[7],得到8个hub基因(图3)。使用MCODE从PPI网络中获得一个重要模块,包括10个节点和45个边缘(图4)。
图2 miR- 4324相关性差异靶基因的PPI网络
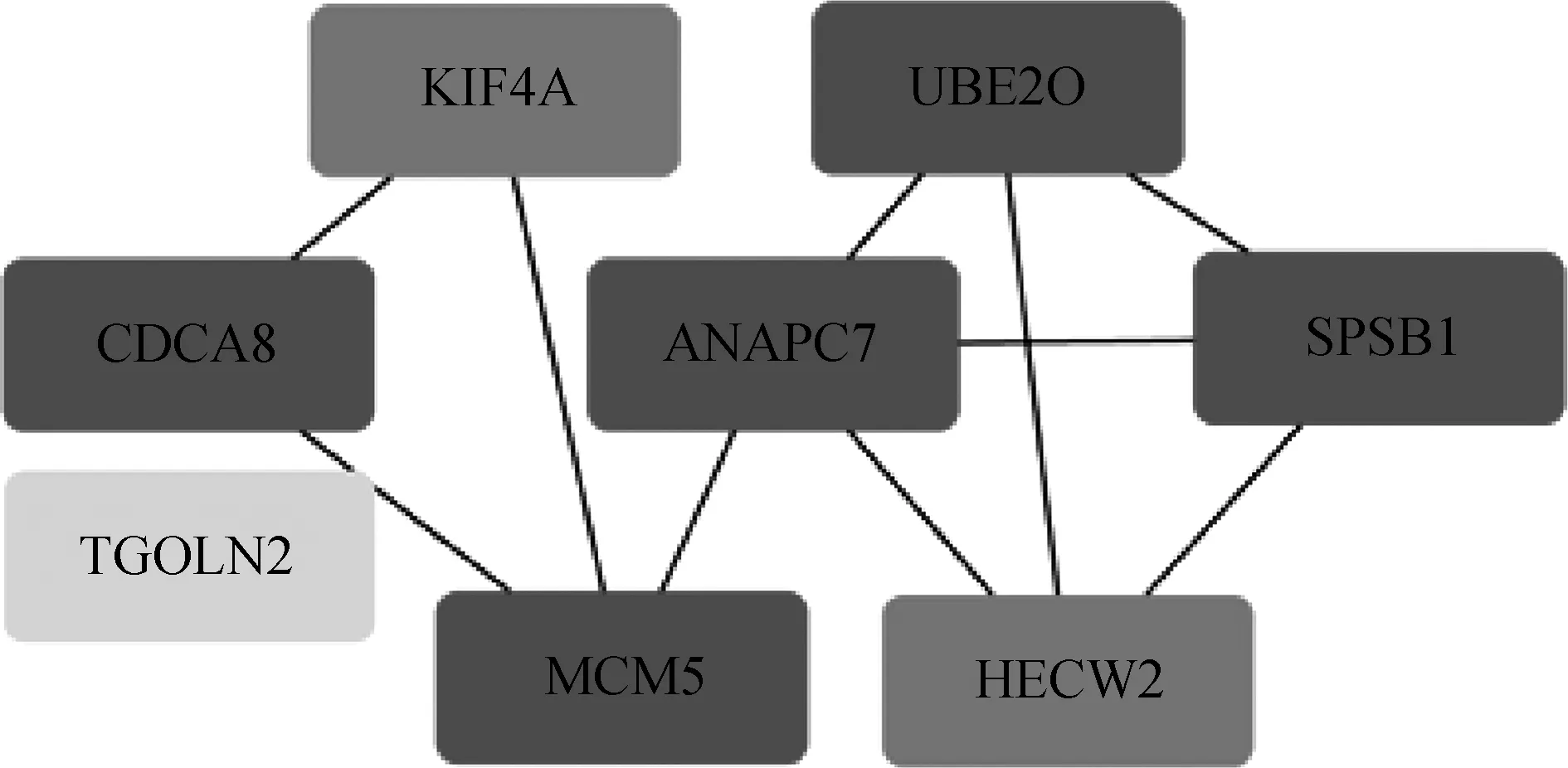
图3 PPI网络中筛选出的hub基因
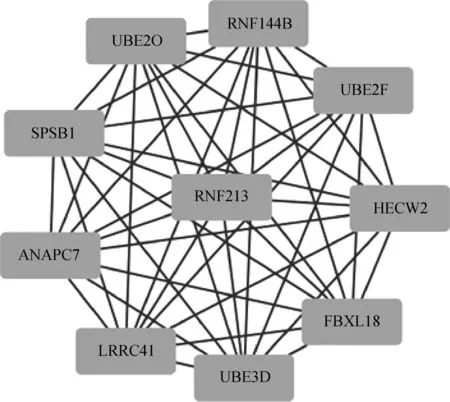
图4 PPI网络中筛选出的重要功能模块
2.6 生存分析
根据hub基因的高表达和低表达分析卵巢癌患者的OS。结果发现卵巢癌患者中SPSB1、ANAPC7、HECW2、TGOLN2基因高表达者及UBE2O、MCM5基因低表达者,其OS明显缩短(图5)。
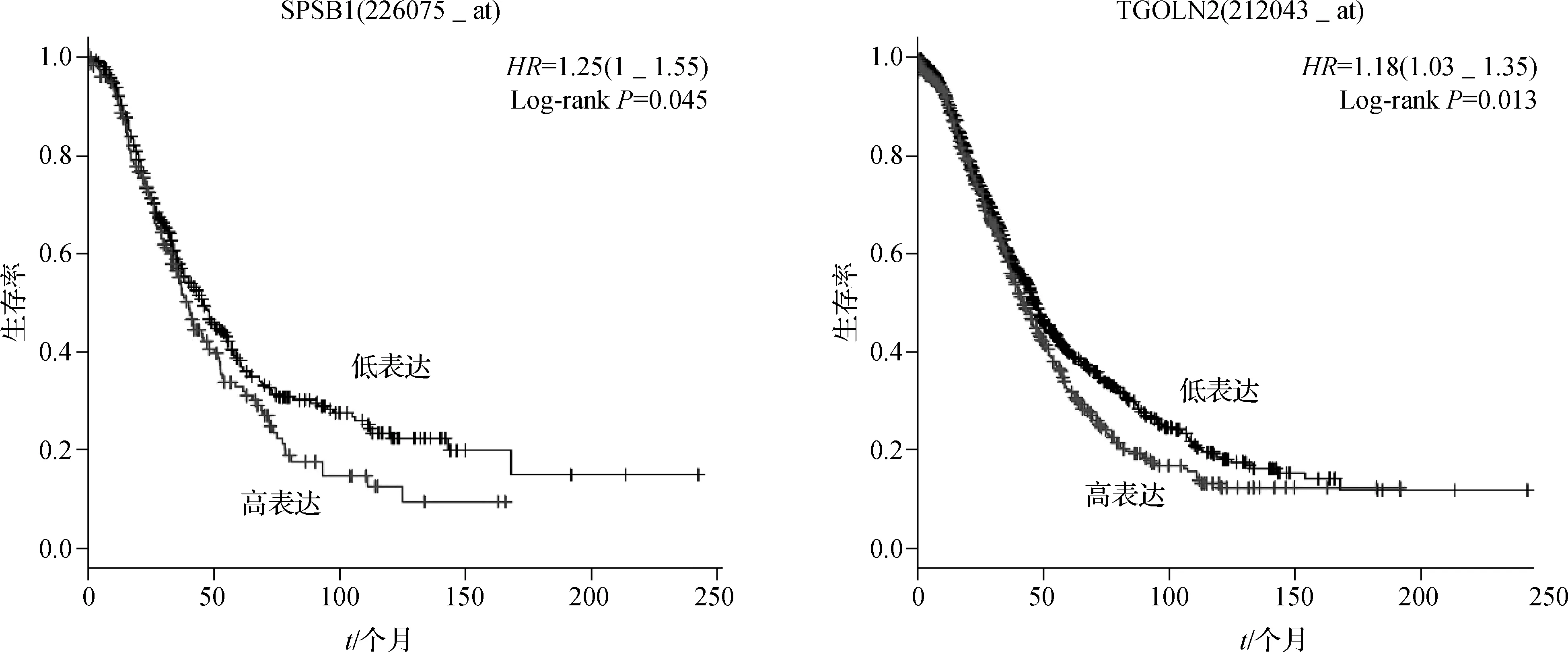
图5 hub基因表达与卵巢癌患者OS的K- M生存曲线
3 讨 论
目前miR- 4324表达与肿瘤关系的研究逐渐成为热点。本研究通过比较卵巢癌组织和正常卵巢组织中的miRNA表达谱,发现卵巢癌中miR- 4324异常表达。数据分析表明,与正常卵巢组织相比,卵巢癌组织中miR- 4324的表达下调,这与大多数肿瘤中miR- 4324的表达情况[3- 4,8- 10]一致。此外,我们还通过靶基因预测、筛选验证、GO分析、KEGG途径富集、PPI网络构建和Kaplan- Meier Plotter分析,研究miR- 4324参与卵巢癌关键生物学过程中可能的新型标志物和潜在治疗靶点。GO分析表明,在卵巢癌中,miR- 4324相关性差异靶基因主要富集于细胞黏附、细胞迁移、血管再生、细胞外基质、内吞循环、泛素化修饰等功能簇。KEGG途径注释表明,miR- 4324相关性差异靶基因主要富集于细胞外基质受体的相互作用、糖胺聚糖生物合成(硫酸角质素)和癌症相关通路共3条信号通路。PPI网络的构建及hub基因的筛选表明CDCA8、SPSB1、UBE2O、ANAPC7、MCM5、KIF4A、HECW2、TGOLN2是miR- 4324参与卵巢癌生物学过程的关键基因。且预后分析表明,卵巢癌患者中,SPSB1、ANAPC7、HECW2、TGOLN2基因高表达者及UBE2O、MCM5基因低表达者,OS明显缩短。
SPSB1基因高表达和低表达患者的中位OS分别为39.87个月和45.77个月(P<0.05)。SPSB1基因(splA/ryanodine receptor domain and SOCS box containing 1 gene)位于1号染色体p36.22处,属于细胞因子抑制物(suppressor of cytokine signaling,SOCS)家族中的Sps6亚家族成员之一。越来越多的研究[11- 14]表明,SPSB1基因参与肿瘤的生物学过程。本研究表明,miR- 4324相关性差异靶基因SPSB1作为hub基因,参与主要功能模块的构建,在卵巢癌的发生发展中起重要作用,且其高表达与卵巢癌患者的不良预后有关。
TGOLN2基因高表达和低表达患者的中位OS分别为42个月和46.6个月(P<0.05)。TGOLN2基因编码位于反面高尔基体网状蛋白上的Ⅰ型跨膜整合蛋白,这个蛋白在胞外囊泡的形成中起重要作用。目前的研究表明包括反面高尔基体网在内的高尔基复合体在自噬过程中起重要作用[15- 16],而自噬与卵巢癌一直是研究热点[17- 18]。许多学者认为,卵巢癌细胞中自噬活性的增强可促进其耐药的产生,而抑制自噬的产生或降低自噬活性,或许可以提高化疗药物敏感性。本研究表明,miR- 4324相关性差异靶基因TGOLN2,可能通过调节自噬活性,从而参与卵巢癌的发生发展和转归,其基因高表达与卵巢癌患者的不良预后有关。
通过生物信息学分析和研究我们发现,miR- 4324在卵巢癌中表达下调,miR- 4324可能通过调控SPSB1、TGOLN2等miR- 4324相关性差异靶基因的表达,从而介导卵巢癌的发生、发展和转归。

