基于TCGA和GEO数据库构建胃癌ceRNA调控网络
冯伟,宗炜,鞠少卿
(南通大学附属医院医学检验科,江苏南通 226001)
胃癌(gastric cancer, GC)是起源于胃粘膜上皮的恶性肿瘤,死亡率位于全球恶性肿瘤的第三位[1]。GC的发生、发展是一个多阶段、多因素的病理过程[2]。临床诊断主要依靠影像学检查、病理活检以及血清标志物检测,但早期胃癌诊断的阳性率较低,易造成漏诊、误诊。近年来研究表明,包括GC在内的许多肿瘤组织中存在大量的非编码RNA,其中关于LncRNA、miRNA和mRNA研究最为丰富[3]。ceRNA假说最早是由Salmena等[4]提出,其认为任何含有miRNA反应元件(miRNA response elements,MREs)的基因都可以通过MRES与miRNA结合,从而影响其转录表达。LncRNAs广泛分布于真核细胞中,具有多种MREs,能够竞争性结合miRNAs。因此,深入挖掘胃癌中“LncRNA-miRNA-mRNA”的ceRNA调控网络,为胃癌诊治提供新的依据和靶点,具有巨大的临床应用价值和广泛的前景。
本研究拟联合TCGA和GEO数据库进行基因筛选,挖掘胃癌中差异表达的LncRNAs、miRNAs以及mRNAs,构建ceRNA调控网络;利用DAVID数据库对ceRNA调控网络中的差异mRNAs进行GO分析以及KEGG分析,探究差异基因在胃癌发生、发展中的潜在调节机制;利用荧光定量PCR对部分差异基因进行临床标本验证。
1 资料与方法
1.1数据下载 利用TCGA数据库(https://portal.gdc.cancer.gov/)下载人类胃癌的RNA-Seq和miRNA-Seq的Count数据,其中包含343例胃癌组织样本和30例胃正常组织样本;利用GEO数据库(https://www.ncbi.nih.gov/geo/)下载GSE93415数据集的miRNA芯片数据,其中包含20对胃癌组织样本和癌旁组织样本。
1.2差异基因筛选 利用edgeR函数包对TCGA数据整理分析,筛选并确定差异表达的LncRNAs、mRNAs和miRNAs;利用limma函数包对GEO数据整理分析,筛选并确定差异表达的miRNAs。差异基因筛选条件:(1)对LncRNAs和mRNAs差异表达倍数取对数:|logFC|>2,P<0.05;(2)对miRNAs差异表达倍数取对数:|logFC|>1,P<0.05。错误发现率(FDR)用于校正所有P值的统计学意义;鉴于GEO数据库胃癌组织样本量偏少,|logFC|>2,P<0.05的DEmiRNAs数量较少,筛选|logFC|>1,P<0.05的DEmiRNAs进行后续研究。
1.3ceRNA网络构建 基于ceRNA理论,利用miRcode数据库预测与miRNAs存在调控关系的LncRNAs;利用starBase数据库预测与miRNAs存在调控关系的mRNAs;将预测结果分别与数据库筛选出的差异表达的LncRNAs和mRNAs取交集,将TCGA数据库与GEO数据库中差异表达的miRNAs取交集;利用Cytoscape软件将差异表达的LncRNAs、miRNAs和mRNAs三者的调控网络可视化。
1.4富集分析 利用DAVID数据库对25个mRNAs进行功能富集分析和通路富集分析,根据筛选条件(P<0.05)对分析结果进行汇总,结果以气泡图呈现。
1.5表达量验证 利用血清RNA提取试剂盒(北京百泰克生物技术公司)提取总RNA,使用逆转录试剂盒(美国Thermo Fisher Scientific公司)和特异引物合成互补DNA(cDNA)。采用LightCycler 480实时荧光定量仪(Roche公司)检测DELncRNAs的相对表达量,LncRNAs引物序列见表1。选择18S内参对LncRNAs的表达量进行标准化,采用2-ΔΔCt法计算相对表达量。
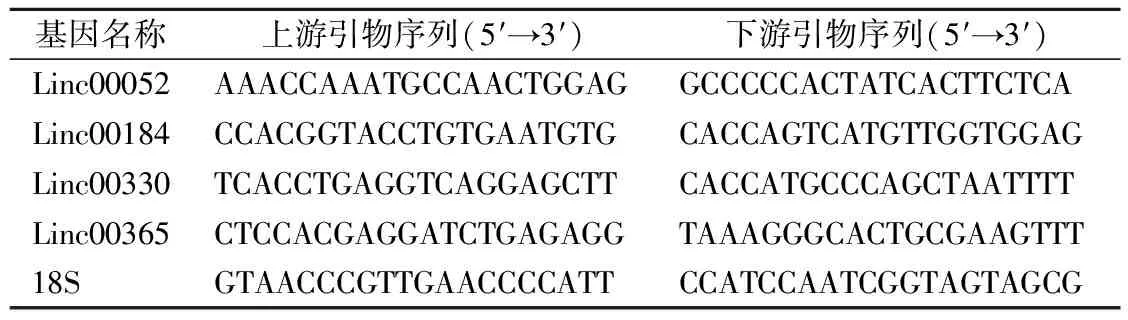
表1 LncRNAs引物序列
2 结果
2.1差异基因筛选 根据设定的筛选条件,分别从TCGA数据库中获得高表达的LncRNAs 895个,低表达的LncRNAs 207个,高表达的mRNAs 937个,低表达的mRNAs 740个, 高表达的miRNAs 180个,低表达的miRNAs 60个;从GEO数据库中获得高表达的miRNAs 49个,低表达的miRNAs 23个。对TCGA数据库和GEO数据筛选的差异miRNAs取交集,得到18个共同的miRNAs,其中上调的miRNAs 12个,下调的miRNAs 6个。
2.2ceRNA网络构建 将miRcode数据库、TCGA数据库和GEO数据库三者联合筛选差异LncRNAs,得到52个LncRNAs。利用starBase数据库和TCGA数据库筛选差异mRNAs,得到25个mRNAs。最终,将52个LncRNAs、18个miRNAs以及25个mRNAs进行ceRNA网络的构建(图1),ceRNA调控网络见表2。

注:菱形、圆形和矩形分别表示LncRNAs、mRNAs和miRNAs;红色表示高表达,蓝色表示低表达。

表2 ceRNA调控网络
2.3基因功能和通路富集分析 利用DAVID数据库进行GO分析,结果发现存在6个生物学过程,包括受体配体激活、信号受体激活、丝氨酸型肽酶激活、丝氨酸水解酶激活、化学排斥物激活以及跨膜受体蛋白络氨酸激酶激活等(P<0.05,图2A);KEEG通路分析结果涉及癌症中的转录调节失调、癌症中的miRNAs(P<0.05,图2B)。

注:A,DEmRNAs的GO分析气泡图;B,DEmRNAs的KEGG分析气泡图。
2.4血清验证差异LncRNAs 利用荧光定量PCR检测胃癌患者血清中差异LncRNAs的相对表达水平,结果显示,Linc00052、Linc00365和Linc00330在胃癌患者和健康人血清样本中存在明显差异(P<0.05),但Linc00184在胃癌患者和健康人血清样本中的差异无统计学意义(图3)。
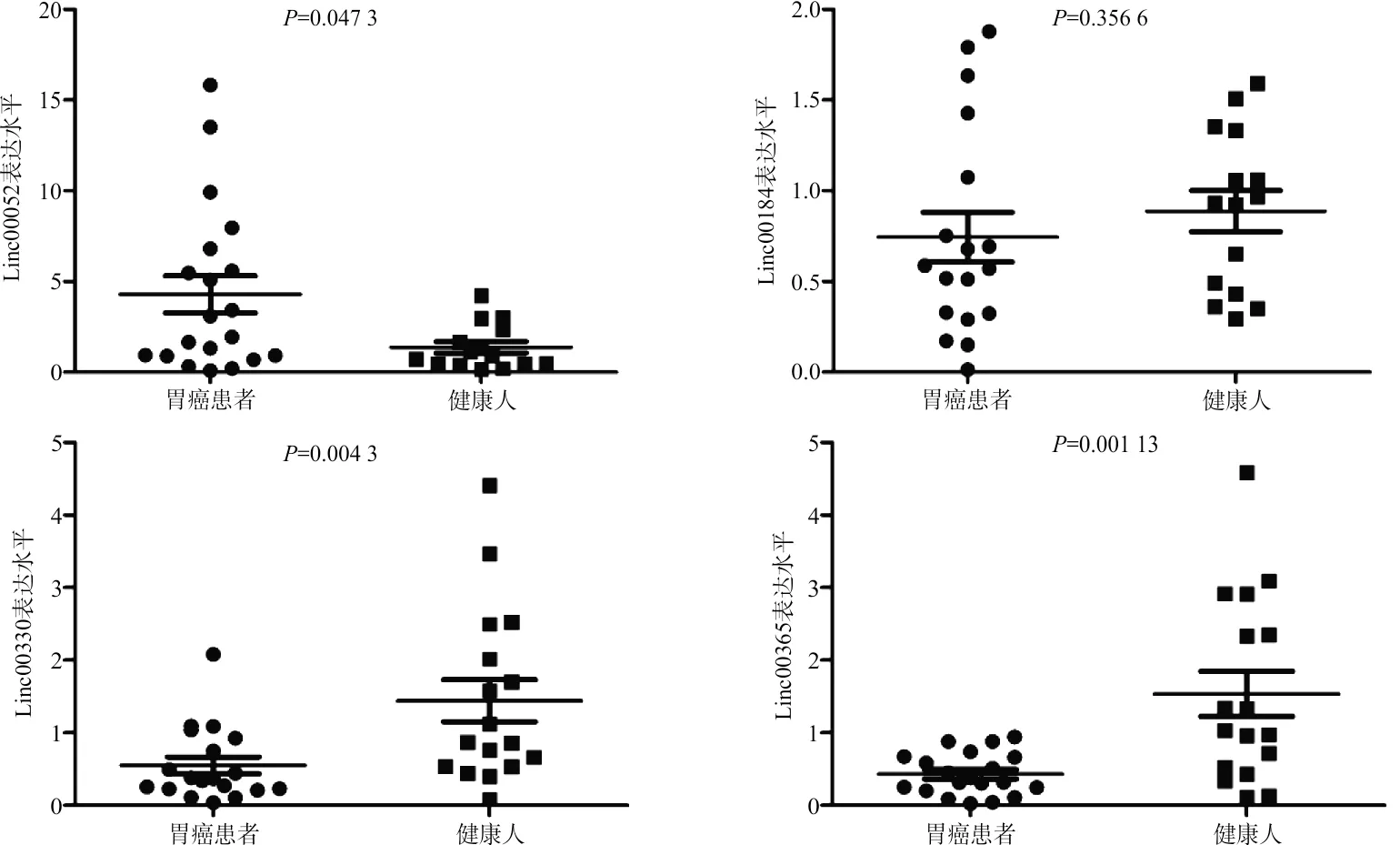
图3 胃癌患者血清中DELncRNAs的表达水平
3 讨论
胃癌的发生、发展是一个多基因参与的、多水平调控的复杂过程。绝大多数胃癌患者早期无明显症状,易与胃炎、胃溃疡等胃良性疾病混淆。胃癌诊断主要依靠影像学检查以及实验室检测,但传统实验室检查不能满足临床需求[5]。近年来,国内外研究表明,异常表达的ncRNAs不仅可以作为疾病的早期筛查指标,还能作为患者预后监测及治疗的靶点,为癌症患者带来了新的希望[6-7]。
本研究通过对现有胃癌样本基因表达谱进行筛选,结合样本的临床病理数据,分析各基因差异表达水平以及患者生存预后,根据“高表达LncRNA-低表达miRNA-高表达mRNA”和“低表达LncRNA-高表达miRNA-低表达mRNA”这两种调控轴构建ceRNA网络,笔者筛选出潜在的ceRNA调控网络,并发现部分ceRNA调控网络已有相关研究发表。Teng 等[8]研究发现,LncRNA NKX2-1-AS1能够竞争性结合miR-145-5p,从而促进SERPINE1的表达,激活VEGFR-2信号通路,促进胃癌的恶性进展。Piao等[9]研究发现,Linc00184同样能够竞争性结合miR-145,促进ANGPT2表达,促进胃癌细胞的生长和上皮-间质转化(EMT)。Yan等[10]研究发现,Linc00365在胃癌中的表达降低,而低表达的Linc00365能够抑制胃癌细胞增殖,其进一步通过机制研究发现,Linc00365能够抑制NF-κB信号通路,从而抑制胃癌的恶性进展。以上研究均表明,生物信息学预测能够对癌症的基础研究有所启发,同时还能为癌症新的诊治靶点和思路提供理论支撑,使得后续的基础验证和临床研究更具有针对性,能够避免盲目的实验探究并减少科研资源的浪费。
虽然生物信息学对科学研究有很大的帮助,但其仍有一定的局限性,例如,芯片分析结果的质量存在一定差异,以及易与实际实验验证存在一定误差等。因此,生物信息学分析的结果最终需要大量的临床及基础实验进行验证。笔者针对部分差异表达的LncRNAs设计了特异的引物(表2),并利用荧光定量PCR在胃癌患者血清样本中进行验证,结果显示,Linc00052、Linc00330、Linc00365在胃癌患者血清样本中的表达水平与芯片结果一致,但Linc00184的表达水平差异无统计学意义,且表达水平与芯片结果并不一致,可能由于样本量较少的原因,也可能由于芯片结果主要来源于胃癌组织,其表达水平可能与血清样本存在一定的差异。后期,我们将针对构建的ceRNA调控网络进行基础实验验证,特别是针对目前在胃癌中暂无相关研究的Linc00330,进一步扩大样本量和样本类型,收集胃良性病变的血清和组织样本,同时在不同胃癌细胞系进行体内外功能验证,或许能够为胃癌的诊断和治疗提供新的依据。

