基于ITS序列对河南省山区部分野生食药用菌种质资源鉴定分析
崔筱 付晓雨 胡素娟 宋志波 刘芹 孔维威 康源春 袁瑞奇 张玉亭 孔维丽


摘 要:为了对收集的河南省野生食药用菌资源开展分类鉴定,应用ITS(Internal transcribed spacer)序列分析方法对收集于不同地区的51株野生食用菌菌株进行鉴定,基于NJ(Neighbour-Joining)法构建rDNA ITS1、ITS4序列的分子进化系统树,同时对相同属间各菌株ITS序列进行比对分析。 NCBI数据库比对结果表明,51个菌株的核酸序列相似性在92.9%~100%,隶属于2个亚纲5目14科14属28种;遗传进化树分为3个分支,侧耳属(Pleurotus)的盖囊侧耳(Pleurotus cystidiosus)单独聚为一支,肺形侧耳(Pleurotus pulmonarius)、哥伦比亚侧耳(Pleurotus columbines)、糙皮侧耳(Pleurotus ostreatus)和美味侧耳(Pleurotus sapidus)聚为第二支,丝膜菌属(Cortinarius)、马勃属(Lycoperdon)、口蘑属(Tricholoma)、乳牛肝菌属(Suillus)、灵芝属(Ganoderma)、乳菇属(Lactarius)等聚为第三支,种间各自聚为小支,表现出良好的单系性;侧耳属的25个菌株为6个差异菌株,丝膜菌属、马勃属、口蘑属、乳牛肝菌属、灵芝属、乳菇属等的18株菌株为14个差异菌株。本研究为深入了解河南省野生资源分布奠定基础。
关键词:野生食药用菌;鉴定分析;ITS
中图分类号:S646 文献标识码:A DOI 编码:10.3969/j.issn.1006-6500.2021.07.001
Abstract: To carry out classification and identification of the collected wild edible and medicinal fungi resources in Henan Province,the internal transcribe spacer sequence analysis was used to identify 51 strains of wild edible fungi collected from different regions, a phylogenetic tree of rDNA ITS1/ITS4 sequences was constructed by NJ(Neighbor-joining) method, and the ITS sequence of each strain in the same genus was analyzed.The NCBI blast results showed that the sequence similarity of nucleic acid sequences of 51 strains on NCBI was between 92.9% and 100%, which were belonging to 2 subclasses 5 orders 14 families 14 genera 28 species.The genetic evolution trees showed that the 51 strains were divided into three branches, of which Pleurotus cystidiosus was isolated into one branch, Pleurotus pulmonarius, Pleurotus columbines, Pleurotus ostreatus, and Pleurotus sapidus clustered into the second branch, and Cortinarius, Lycoperdon, Tricholoma, Suillus, Ganoderma, Lactarius etc.clustered into the third branch, and each species aggregated into small branches showing a good monophyletic property.Analysis of the ITS sequences of each strain of the same genus showed that 25 strains of the Pleurotus genus were identified as 6 different strains, and 18 strains of the Cortinarius, Lycoperdon, Tricholoma, Suillus, Ganoderma, Lactarius etc. were identified as 14 different strains. This study could lay a foundation for further understanding of the wild resources distribution in Henan Province.
Key words: wild edible and medicinal fungi; identification analysis; ITS
河南省地域廣阔,位于南暖温带与北亚热带的过渡地带,气候温暖,雨量较为充沛,山林茂密,植被类型复杂,适于各种菌类繁殖生长,野生资源相当丰富。随着人们生活水平的不断提高,食用菌营养价值的关注度得到提高,许多国家和地区对食用真菌资源状况进行了调查研究。Pera等[1]采集西班牙北部天然林和人工林中海岸松下的98株菌根真菌,共有35种,隶属于16属,均为大型食用真菌,可形成外生菌根,证明了真菌和海岸松之间共生关系;Rincón等[2]从加泰罗尼亚松林混合林中收集60个菌株,发现仅有17个菌株,隶属于大型真菌的8个属,可形成外生菌根,其中有几个是新发现的物种,进一步证明了真菌和海岸松之间共生关系;李碧琼等[3]连续3年多次对莆田老鹰尖自然保护区进行野外定点调查,初步发现该区拥有22科、95种野生食用菌资源;徐凌川等[4]通过对山东省各地大型真菌调查研究,以拍照和采集子实体的方法,借助放大镜、体视显微镜、生物显微镜和扫描电镜进行性状和显微观察,根据Ainsworth分类系统,发现山东省具有食用和药用价值的真菌共有255种,隶属于6纲14目45科113属;陈光富等[5]通过对玉龙雪山云冷杉针叶林内野生食用菌资源组成进行调查,发现有食用菌资源17科21属37种,22种可食用,15种药食同性;张根伟等[6]对承德坝上及其邻近地区松林下野生食用菌的资源状况进行了调查研究,共鉴定标本148号,计16科31属102种(含变种、变型)。上述研究均基于形态学对大型食用真菌的鉴定研究,然而,关于河南省野生食用菌资源的研究,仅申进文等[7]、杜敏华等[8]对伏牛山区开展了部分调查研究分类,尚没有进行全面系统的调查研究。
对大型真菌分类的常规鉴定主要是依据子实体的形态、颜色、气味以及孢子特征等,这些形态特征经常是易变、模糊且易混淆的,因而依据传统的形态特征进行分类鉴定总是带有一定的局限性和主观性[9],同时有些种内食用菌外观特征差异较小,仅凭传统的形态特征进行分类不能鉴定出其遗传学多态性。真菌核糖体基因转录间隔区(internal transcribed spacer ITS),又叫内转录间隔区,是位于真菌核糖体DNA(rDNA)上18S和28S基因之间的区域片段,主要包括内转录间隔区1(ITS1)、5.8S rDNA、内转录间隔区2(ITS2),其两侧分别是18S RNA基因和28S RNA基因[10]。因其序列种间变异度高,在物种和亚种水平上的识别信息最为丰富[11]且在测序错误及假基因出现的情况下保持高准确度[12],因此可作为物种新的可识别特征之一,为分类和精确识别奠定基础[13-14]。因此,本研究采用ITS法對近几年河南省豫西南、豫北、豫南等各地采集分离的部分野生食用菌进行鉴定,以期对野生食用菌种质资源的发掘和利用提供科学依据和参考。
1 材料和方法
1.1 试验材料
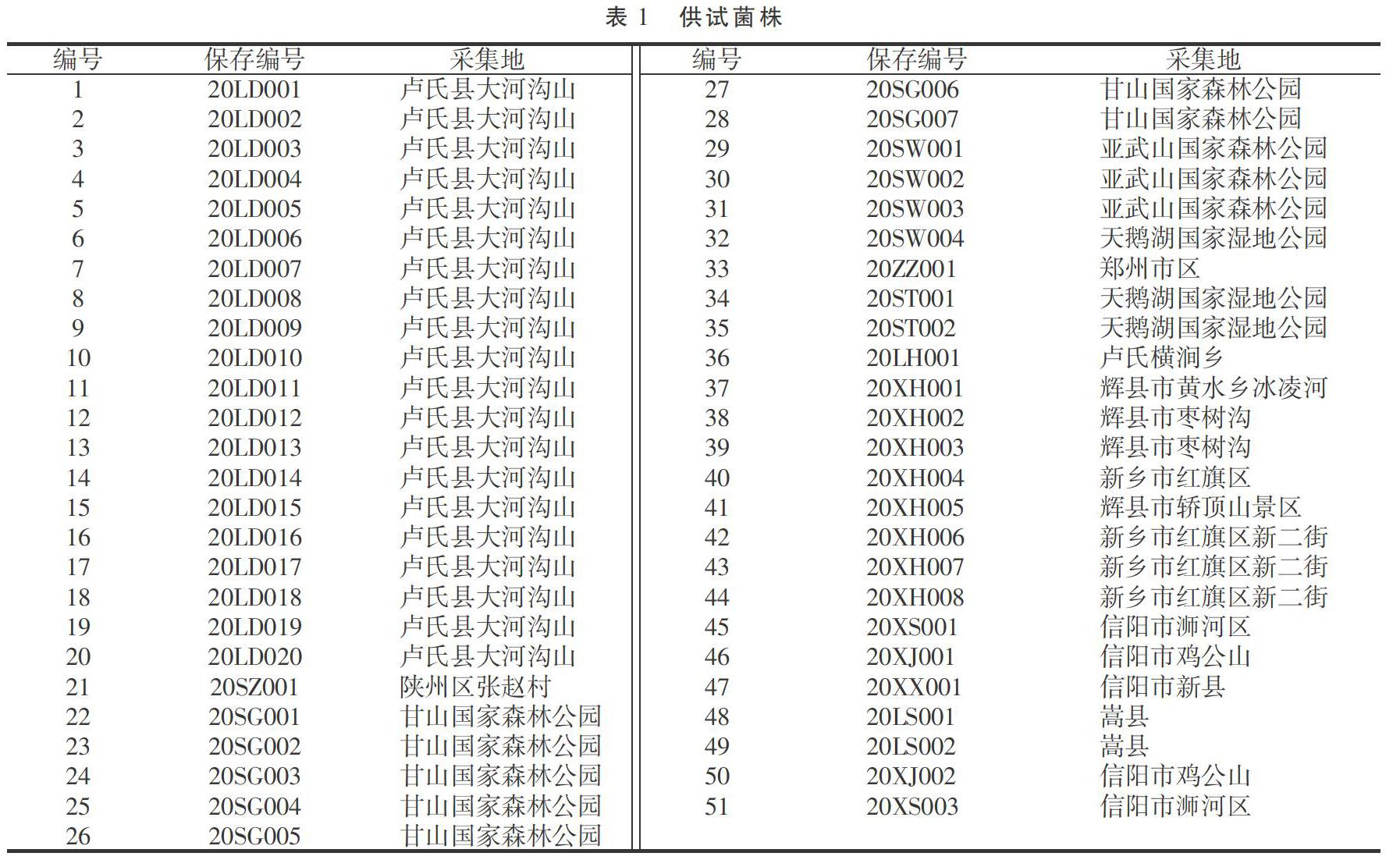
1.1.1 供试菌株 收集保藏的51株野生食用菌菌株(表1)样品采集于三门峡、新乡、信阳等地,现保存于“河南省食用菌种质资源库”。
1.1.2 主要试剂及引物 Taq PCR Master Mix聚合酶、DNA纯化试剂盒、DNA凝胶回收试剂盒、PCR 扩增试剂及4sGelRed染液均购自上海生工有限公司;pMD-18T载体、大肠杆菌DH5α感受态细胞购自宝生物工程(大连)有限公司;rDNA ITS 分析所用的ITS 通用引物ITS1(5′-TCCGTAGGTGAACCTGCGG-3′)和ITS4(5′-TCCTCCGCTTATTGATATGC-3′)购自上海生工生物工程有限公司。
1.1.3 主要仪器 全自动样品快速研磨仪JXFSTPRP-64(上海净信实业发展有限公司),微波炉G8023CTL-K3(佛山市格兰仕微波炉电器有限公司),干式恒温器DB10(杭州茂丰仪器有限公司),电子天平JA2003N(上海佑科仪器仪表有限公司),小型离心机3K15(德国希格玛实验室离心机有限公司),EPS 300 电泳仪(上海郎赋实业有限公司),全自动凝胶成像分析仪JS-680D(上海培清科技有限公司),恒温磁力搅拌器85-2(常州智博瑞仪器制造有限公司),酸度计PHS-3E(上海佑科仪器仪表有限公司)。
1.2 试验方法
1.2.1 野生食药用菌的采集 2020年9月,对河南省主要山区(大别山、伏牛山、太行山)及周边地区进行样品采集并拍照并进行编号,记录观察其形态学特征。
1.2.2 DNA提取 将51株食用菌菌株的子实体置于1.5 mL无菌离心管中,于全自动快速研磨仪中研磨成粉提取各样品的基因组DNA,基因组DNA提取方法采用微波炉法[16],1.0%的琼脂糖凝胶检测DNA样品的质量和浓度。
1.2.3 PCR扩增 PCR反应体系:DNA 模板1 μL,Taq PCR Master Mix聚合酶12.5 μL,上下游引物(10 μmol· L-1)各1 μL,超纯水4.5 μL。反应程序:95 ℃ 5 min,95 ℃ 1min,58 ℃ 30 s,72 ℃ 50 s,72 ℃ 10 min,30 个循环。
1.2.4 PCR产物检测及测序 扩增反应完毕后,取5 μL 的PCR产物与 1μL 4sGelRed染液混合,加样于琼脂糖凝胶点样孔中进行电泳,电泳结束后,全自动凝胶成像分析仪JS-680D下检测扩增条带。回收正确的扩增条带(500~800 bp)连于pMD-18T载体上,将连接产物转化于大肠杆菌(Escherichia coli)DH5α菌株感受态细胞,涂布在含有Amp的LB固体培养基上,37 ℃培养过夜,菌落PCR法验证单克隆大肠杆菌菌株,将验证正确的大肠杆菌菌株送华大基因测序。
1.2.5 同源性分析和系统发育树的构建 测序获得的序列经DNAMAN软件多序列比对重排后,于NCBI数据库上比对,MEGA-X软件构建同种菌株的系统发育树[17]。
2 结果与分析
2.1 形态学鉴定
共采集51株野生食用菌(图1),根据其形态特征,结合查阅中国大型菌物资源图鉴[15]鉴定标本51株,51个菌株隶属于2个亚纲5个目14个科14个属(表2),表现出了较高的资源丰富度。部分标本形态学鉴定如下:编号20LD014、20LD015大型真菌菌盖直径5~10 cm,发生在三门峡市卢氏县大沟山的针阔混交林地上,菌肉淡黄色至黄色,菌柄长5~10 cm,圆柱形,基部有暗褐色硬毛,初步鉴定为牛肝菌目的物种;编号20XS001大型真菌发生在信阳市浉河区一块腐朽木头上,菌盖半圆形,表面颜色淡黄色至蓝灰色,边缘锐,菌肉乳白色,初步鉴定为多孔菌目的物种;编号20XH002大型真菌发生在辉县市枣树沟一块腐木上,菌盖扇形,菌肉木栓质,边缘薄,全缘,具漆样光泽,菌管多层,分层不明显,初步鉴定为多孔菌目灵芝科灵芝属的物种;编号20LD009大型真菌发生在三门峡市卢氏县大沟山的阔叶林中地上,子实体高3~8 cm,近球形,表面覆盖疣状和锥形突起,脱落后在表面形成淡色圆点,连接成网纹,初步鉴定为蘑菇科马勃属的物种;编号20XH005大型真菌发生在辉县市轿顶山景区针叶林中落叶层上,子实体高5~7.5 cm,黄色,整体近球形至倒圆锥形,主支长1~4 cm,分支3~5回,初步鉴定为枝瑚菌属的物种。
2.2 rDNA ITS区段PCR扩增
51株野生食用菌菌株的rDNA ITS 区段 PCR扩增均得到了目的条带,经检测,扩增条带在500~800 bp之间,符合测序要求。
2.3 测序及NCBI比对结果
Renske等[18]提出,供试的rDNA ITS序列通过与NCBI的 GenBank数据库进行BLAST检索比对,序列相似性为95%~99%,鉴别为相同属;序列相似性≤95%,鉴别为相同科。将经检验条带正确的阳性克隆菌液测序后,得到的rDNA ITS序列在GeneBank核酸序列数据库进行BLAST比对,得到与其最相似的物种名、登记号及相似率,结果表明,51个菌株核酸序列在NCBI上的序列相似性在92.9%~100%之间(表3),序列相似性≥99%的有42个菌株,序列相似性为95%~99%的有8个菌株,序列相似性≤95%的有1个菌株。
2.4 分子系统进化分析
将测序得到的rDNA ITS1序列经DNAMAN软件比对重排后,MAGA X软件基于NJ法构建51株野生菌株的rDNA ITS1序列的分子进化系统树(图2)。由图2可知,整个进化树的51个菌株分为3个分支。侧耳属(Pleurotus)盖囊侧耳(P. cystidiosus)菌株编号37、40、42、43、44聚为一支,分支中菌株编号37、43为一组,其自展支持强度为58;菌株编号40、42、44组成一个单系类群,其自展支持强度为60。侧耳属肺形侧耳(P. pulmonarius)、哥伦比亚侧耳(P. columbines)、糙皮侧耳(P. ostreatus)和美味侧耳(P. sapidus)聚为第二支,其中美味侧耳、哥伦比亚侧耳和糙皮侧耳聚为一个类群,除菌株编号21肺形侧耳外,其他肺形侧耳菌株单独聚为一个类群,菌株编号33、34、35、36、50聚为一组,菌株编号22、23、24、26、27、28、29、30、31、47、49聚为一组,菌株编号21、25、32、48分别各自为一组。丝膜菌属(Cortinarius)、马勃属(Lycoperdon)、口蘑属(Tricholoma)、乳牛肝菌属(Suillus)、灵芝属(Ganoderma)、乳菇属(Lactarius)、栓孔菌属(Trametes)、丛枝菌属(Ramaria)等聚为第三支,丝膜菌属的菌株编号2、17和18聚为一组,组成一个单系类群,其自展支持强度为92;马勃属的8、9和10三个菌株组成一个单系类群,其自展支持强度为100;口蘑属的两个菌株13和20组成姐妹群,其自展支持强度为100;灵芝属的两个菌株38和39及栓孔菌属的1个菌株45组成一个单系类群,其自展支持强度为94;乳菇属的三个菌株4、5和7与丛枝菌属的菌株(菌株41)聚为一组;乳牛肝菌属的三个菌株6、14和15聚为一组,组成一个单系类群,其自展支持强度为100;Rhodocollybia butyracea菌株1、Tricholoma sulphurescens菌株3、Armillaria gallica菌株11、Tricholoma bakamatsutake菌株12、Lepista nuda菌株16、Amanita citrine菌株19、Psathyrella SP.菌株46、Volvariella bombyvina菌株51各自單独聚为一组。2.5 属间各菌株ITS序列比对分析
2.5.1侧耳属ITS序列比对分析 将经测序的侧耳属的25个株菌株之间进一步进行ITS序列比对分析(表未提供),通过表型与ITS测序相结合的方法,将菌株间序列相似性≥99.9%的定义为同一菌株,序列相似性99.0%~99.9%之间的定义为同一亚种。结果表明,菌株37和43序列相似性99.9%为同一菌株,菌株42和44序列相似性100%为同一菌株,菌株盖囊侧耳40与43、37、42、44序列相似性99.3%~99.8%,认定为同一亚种,表型上同为盖囊侧耳,与系统进化树的分析一致。同理分析结果表明,菌株21与其他菌株间的序列差异性在96.1%~96.5%之间,可认为肺形侧耳的13个菌株为2个不同菌株,22、23、24、26、27、28、29、30、31、47、48和49菌株序列相似性在99.1%~99.9%之间,可认为是肺形侧耳的不同亚种,此结果与图2肺形侧耳菌株21与其余菌株来源于不同祖先而分为两支的结果一致,此结果与表型结果基本一致;美味侧耳菌株36和50与哥伦比亚侧耳菌株32、33和35及糙皮侧耳菌株34间的序列相似性为在99.4%~99.9%之间,可认为是同一菌株的不同亚种,由图2可知,菌株33、34、35、36和50之间为姐妹群,且与菌株32之间可能存在共同祖先。哥伦比亚侧耳菌株25、32、33和35间的序列相似性在99.6%~100.0%,可认为是同一菌株。肺形侧耳菌株21、22、23、24、26、27、28、29、30、31、47、48和49间的序列相似性在96.1%~100.0%,糙皮侧耳菌株34与其他24株菌株间序列差异性在81.0%~99.7%,其中与美味侧耳菌株36和50及哥伦比亚侧耳菌株25,32,33和35间的序列差异性在99.4%~99.7%之间,可认为是同一菌株;与肺形侧耳菌株21、22、23、24、26、27、28、29、30、31、47、48和49间序列差异性在96.9%~98.9%之间;与盖囊侧耳菌株37、40、42、43和44间序列差异性在80.9%~81.2%之间,且由图2可知,肺形侧耳与哥伦比亚侧耳菌株25及肺形侧耳、盖囊侧耳聚在不同分支,亲缘关系较远。根据ITS序列比对分析,侧耳属的25株菌株被确定为6株菌株,分别为盖囊侧耳、美味侧耳、哥伦比亚侧耳、肺形侧耳(菌株21)、肺形侧耳(菌株22、23、24、26、27、28、29、30、31、47、48和49)、糙皮侧耳,此鉴定结果与表型结果基本一致。
2.5.2丝膜菌属、马勃属、口蘑属、乳牛肝菌属、灵芝属、乳菇属ITS序列比对分析 将经测序的丝膜菌属、马勃属、口蘑属、乳牛肝菌属、灵芝属、乳菇属的18株菌株进一步进行ITS序列比对分析,结果表明,丝膜菌属的菌株2、17和18间的序列相似性分别为83.71%(菌株2和17),83.71%(菌株2和18),89.28%(菌株17和18),可认为分别为3种菌株,由图2可知,菌株17与18为姐妹群且与菌株2可能来源于共同的祖先;马勃属菌株8、9和10间的序列相似性分别为96.76%(菌株8和9),98.92%(菌株8和10),96.89%(菌株9和10),可认为分别为3种菌株,其中菌株9与10为姐妹群且与菌株8可能来源于共同的祖先(图2);口蘑属的菌株3、12、13和20)间的序列相似性分别为73.89%(菌株3和12),80.28%(菌株3和13),80.70%(菌株3和20),77.11%(菌株12和13),77.53%(菌株12和20),99.57%(菌株13和20),可认为分别为3株菌株,由图2可知,菌株3和12各自独立成支,与菌株13和20亲缘关系较远;乳牛肝菌属的菌株6、14和15间的序列相似性分别为99.42%(菌株6和14),99.42%(菌株6和15),99.14%(菌株14和15),可认为1种菌株,且菌株6与15为姐妹群与菌株14有共同祖先(图2);灵芝属的菌株38和39为姐妹群,之间的序列相似性分别为99.53%,可认为是1种菌株且可能与菌株45来源于共同祖先(图2);乳菇属的菌株4、5和7间的序列相似性分别为80.13%(菌株4和5),80.35%(菌株4和7),91.20%(菌株5和7),其中菌株5与7为姐妹群,与菌株4可能起源于共同祖先(图2),可认为是3种菌株。丝膜菌属、马勃属、口蘑属、乳牛肝菌属、灵芝属、乳菇属的18株菌株可最终确定为14株菌株。
3 结论与讨论
ITS序列分析表明51个野生菌株隶属于2个亚纲5个目14个科14个属的28个种。分子进化系统树将51个菌株分为3个大支,侧耳属(Pleurotus)的盖囊侧耳(Pleurotus cystidiosus)单独聚为一支,肺形侧耳(Pleurotus pulmonarius)、哥伦比亚侧耳(Pleurotus columbines)和糙皮侧耳(Pleurotus ostreatus)等聚为第二支,丝膜菌属(Cortinarius)、马勃属(Lycoperdon)、口蘑属(Tricholoma)、乳牛肝菌属(Suillus)、灵芝属(Ganoderma)、乳菇属(Lactarius)等聚为第三支,包含了鬼笔亚纲(Phallomycetidae)的红菇目(Russulales)、针菇目(Gomphales)和多孔菌目(Polyporales) 3个目,蘑菇亚纲(Basidiomycetidae)伞菌目(Agaricales)中的鹅膏属(Amanita)、小脆柄菇属(Lyophyllum)、蜜环菌属(Armillaria)、粉金钱菌属(Rhodocollybia)、口蘑属(Tricholoma)、香蘑属(Lepista)、马勃属(Lycoperdon)、丝膜菌属(Cortinarius)8个属。糙皮侧耳(P. ostreatus)与美味侧耳(P. sapidus)菌株和哥伦比亚侧耳(P. columbines)间的序列相似性在99.4%~99.7%之间,与肺形侧耳(P. pulmonarius)菌株间序列相似性在96.9%~98.9%之间,与盖囊侧耳(P. cystidiosus)5个菌株间序列相似性在80.9%~81.2%之间,根据Renske等[18]提出的观点,侧耳属的25株菌株被确定为6株菌株,分别为盖囊侧耳、美味侧耳、哥伦比亚侧耳、2株肺形侧耳(菌株21和菌株23)、糙皮侧耳;丝膜菌属的3株菌株间的序列相似性<90%,鉴定为差异菌株;马勃属3株菌株间的序列相似性<99%,鉴定为3种菌株;口蘑属的4株菌株鉴定为3株菌株;乳牛肝菌属的3株菌株相似性大于99%鉴定为同1种菌株;灵芝属的2个菌株鉴定为1种菌株;乳菇属的3株菌株间鉴定为3种菌株,因此51株菌株鉴定为28个差异菌株。因此该方法可以作为野生资源的分子鉴定及系统地位分类的研究手段之一[19],同时该方法还可以可有效的鉴定出食用菌不同属间及同属不同种之间的物种的真实性,已经在羊肚菌属物种[20]的鉴定及54个栽培平菇种间鉴定进行应用[21]。
通过该方法可以确定不同属间的亲缘差异程度,糙皮侧耳与盖囊侧耳菌株间序列差异性较大(相似性在80.9%~81.2%之间),而与美味侧耳菌株和哥伦比亚侧耳间的序列差异性较小(相似性在99.4%~99.7%之间),而远缘杂交就是把不同种属的物种进行杂交,有的甚至会把亲缘关系更远的物种做以杂交,从而获得类型多样、特性丰富的新种质或变异[22]。该研究可以糙皮侧耳(菌株34)和盖囊侧耳(菌株37、40、42、43和44)任一菌株为亲本材料进行远缘杂交,为后续研究糙皮侧耳与盖囊侧耳远缘杂交提供了理论依据。因野生资源具有抗逆性强的特性,可将已经得到人工种植的菌类,如糙皮侧耳、盖囊侧耳、口蘑属、Volvariella bombycina、Ganoderma lucidum等进行野生驯化,得到性状优良的可栽培菌株,丰富现有可食用食用菌种类;未经人工栽培的野生菌株,如Suillus luteus、Ramaria abietina、马勃属等,可作为遗传种质材料,开发利用其功能基因,也可直接作为研究材料,研究其生理生化特性、开发利用其有效功能成分。
参考文献:
[1] PERA J, ALVAREZ I F. Ectomycorrhizal fungi of Pinus pinaster[J]. Mycorrhiza, 1995, 5(3): 193-200.
[2] RINC?譫N A, ?譧LVAREZ I F, PERA J. Ectomycorrhizal fungi of Pinus pinea L. in northeastern Spain[J]. Mycorrhiza, 1999, 8(5): 271-276.
[3] 李碧琼, 陈政明, 卢翠香, 等. 莆田老鹰尖自然保护区野生食用菌资源调查[J]. 中国食用菌, 2012, 31(1): 8-10.
[4] 徐凌川, 張华, 李自发, 等. 山东省大型真菌生物多样性及资源保护与可持续利用[J]. 中国食用菌, 2006, 25(2): 12-16.
[5] 陈光富, 朱文浩, 刘德团. 玉龙雪山云冷杉针叶林野生食用菌资源调查[J]. 贵州农业科学, 2019, 47(11): 75-79.
[6] 张根伟, 张丽萍, 李书生, 等. 承德坝上松林野生食用菌资源[J]. 中国农学通报, 2012, 28(28): 286-289.
[7] 申进文, 决超, 徐柯, 等. 伏牛山大型真菌资源(V)[J]. 食用菌, 2011, 33(4): 12-13.
[8] 杜敏华, 高立献, 李景照, 等. 河南伏牛山自然保护区大型真菌物种的多样性初步调查[J]. 食药用菌, 2016, 24(5): 306-311.
[9] 桂明英, 何容, 郭永红, 等. 基于形态特征和ITS序列对新疆芦苇根蘑菇的分类鉴定[J]. 食用菌, 2014(4): 14-16.
[10] 白树猛, 田黎. ITS序列分析在真菌分类鉴定和分子检测中的应用[J]. 畜牧与饲料科学, 2009, 30(1): 52-53, 189.
[11] SONG J Y, SHI L C, LI D Z, et al. Extensive pyrosequencing reveals frequent intra-genomic variations of internal transcribed spacer regions of nuclear ribosomal DNA[J]. PLOS One, 2012, 7(8): e43971.
[12] ZHU S, LI Q W, CHEN S C, et al. Phylogenetic analysis of Uncaria species based on internal transcribed spacer (ITS) region and ITS2 secondary structure[J]. Pharmaceutical Biology, 2018, 56(1): 548-558.
[13] CHIOU S J, YEN J H, FANG C L, et al. Authentication of medicinal herbs using PCR-amplified ITS2 with specific primers[J]. Planta Medica, 2007, 73(13): 1421-1426.
[14] GRAJALES A, AGUILAR C, S?魣NCHEZ J A. Phylogenetic reconstruction using secondary structures of Internal Transcribed Spacer 2 (ITS2, rDNA): finding the molecular and morphological gap in Caribbean gorgonian corals[J]. BMC Evolutionary Biology, 2007, 7: 90.
[15] 李玉. 中国大型菌物资源图鉴[M]. 郑州: 中原农民出版社, 2015.
[16] 田永强, 苏敏, 赵洪林, 等. 微波法快速提取丝状真菌基因组DNA[J]. 中国抗生素杂志, 2008, 33(11): 703-705.
[17] 张丽娜, 荣昌鹤, 何远, 等. 常用系统发育树构建算法和软件鸟瞰[J]. 动物学研究, 2013, 34(6): 640-650.
[18] LANDEWEERT R, LEEFLANG P, KUYPER T W, et al. Molecular identification of ectomycorrhizal mycelium in soil horizons[J]. Applied and Environmental Microbiology, 2003, 69(1): 327-333.
[19] 李秋玲, 吳智艳, 乔洁, 等. 基于ITS序列对野生大型真菌进行分子鉴定及系统地位研究[J]. 廊坊师范学院学报(自然科学版), 2017, 17(4): 53-57.
[20] 刘文丛, 刘颖, 郭相, 等. 滇西北地区羊肚菌的分子鉴定及ITS序列分析[J]. 江苏农业科学, 2012, 40(3): 31-34.
[21] 崔筱, 刘芹, 段亚魁, 等. 平菇种质资源的遗传多样性分析[J]. 河南农业科学, 2020, 49(3): 138-144.
[22] 李振声. 小麦远缘杂交[M]. 北京: 科学出版社, 1985.

