基于iTRAQ技术分析模拟空间诱变酵母菌株的差异蛋白表达
郑世茂 齐文武 陈晓云 白云昇 庞欣





摘 要:目的:探究模拟空间诱变处理后,生长速率提高的酿酒酵母细胞的诱变机制。方法:采用同位素标记相对和绝对定量(iTRAQ)技术对模拟空间诱变处理前后的酿酒酵母进行定量蛋白质组学研究,探讨对照组与诱变组在蛋白质组表达水平上的差异,以及受影响的生物功能。结果:共鉴定到764个差异蛋白,占检测蛋白总数的15.54%。其中,上调差异表达蛋白378个、下调差异表达蛋白386个,主要涉及細胞核、线粒体、核糖体和质膜等重要细胞器和细胞结构。结论:提高重要代谢的效率、降低DNA修复功能与环境防御能力,可能是模拟空间诱变提高酿酒酵母细胞生长速率的重要机制之一。
关键词:模拟空间诱变;酿酒酵母;蛋白质组学;iTRAQ
空间诱变育种是近年来兴起的一种新型诱变技术。空间环境能够影响微生物的生物学形态和表型,是微生物诱变育种的有效方法[1],但现阶段实施空间诱变仍有诸多限制,此时模拟空间诱变就成为一个非常好的选择。酿酒酵母是与人类关系最广泛的一种酵母,传统上它用于制作面包和馒头等食品及酿酒,是发酵中最常用的菌株。同位素标记相对与绝对定量(iTRAQ)技术是研究蛋白质组学的重要技术之一,该方法几乎可以对任何蛋白样品进行定量分析,具有可定量、高精度、高通量等特点,可在一次试验中对8个样品进行定量比较,已经广泛地应用于微生物、植物、动物、人体细胞定量蛋白质组学的研究[2-5]。但是,目前针对模拟空间诱变前后的酵母蛋白组学分析与研究国内外鲜见报道。本试验采用iTRAQ技术对模拟空间诱变后样品与对照组样品进行定量蛋白质组学研究,通过对鉴定到的蛋白质进行差异比对,进行GO功能注释、KEGG Pathway代谢通路分析、KOG注释与亚细胞定位预测,比较模拟空间诱变前后酿酒酵母在蛋白质组表达水平的差异,进而分析引起诱变后酵母生长速率提高的可能的机制。
1 材料与方法
1.1 材料
出发菌株为酿酒酵母(Saccharomyces cerevisiae)AS 2.430,诱变菌株为出发菌株在模拟空间诱变环境参数(包括但不限于真空、振动、加速度等)下筛选出的生长速率提高12.27%的菌株S.c_11。YPD液体培养基:酵母膏1%、蛋白胨2%、葡萄糖2%。
岛津LC-20AB液相系统(分离柱为5 μm 4.6 mm×250 mm Gemini C18柱);Thermo公司UltiMate 3 000 UHPLC、trap柱、自装C18柱(75 μm内径,3 μm>粒径,25 cm柱长);质谱仪Q-Exactive HF X。SDSL3、含EDTA的蛋白酶抑制剂Cocktail、DTT、丙酮、碘乙酰胺溶液、Tissue lyser、考马斯亮蓝G-250、乙醇、乙酸、胰蛋白酶、iTRAQ Reagents - 8plex kits 异丙醇、TRAB溶液、流动相A(5%ACN,pH 9.8)、流动相B(95% ACN,pH 9.8)、流动相C(2% ACN,0.1%FA)、流动相D(98%ACN,0.1% FA)。
1.2 蛋白质提取
将酵母菌出发菌株和诱变菌株于斜面试管培养后分别接种于YPD液体发酵培养基中,各设置4个平行样,在32 ℃、200 r/min 条件下培养24 h得对照组与诱变组发酵液。将发酵液经低温离心后收集酵母细胞,用灭菌预冷的PBS洗涤3次,获得8个菌体样品。提取蛋白过程中如未特殊说明,试验则在4 ℃下进行操作。称取适量样品,加500 μL含SDSL3的Cocktail,静置反应 5 min,加入终浓度10 mmol/L DTT溶液;超声、离心取上清液;加入5倍体积预冷丙酮,-20 ℃冰箱放置过夜,离心去上清液获取沉淀;风干沉淀,加入适量的Cocktail溶解蛋白;离心后取上清液;加终浓度为10 mmol/L的DTT溶液,56 ℃中水浴1 h,保证充分反应;加入终浓度55 mmol/L的碘乙酰胺溶液,暗室放置45 min;加入5倍体积预冷丙酮,-20 ℃冰箱放置2 h,离心获取沉淀;风干沉淀,加入适量的Cocktail溶解蛋白,离心后取蛋白质上清液[6]。
1.3 蛋白定量
采用Bradford定量方法定量[7]:在96孔酶标板A1~A10位置依次加入标准蛋白 (0.2 μg/μL BSA)0、2、4、6、8、10、12、14、16、18 μL,之后依次加入纯水20、18、16、14、12、10、8、6、4、2 μL,之后各孔加入考马斯亮蓝G-250定量工作液180 μL。用酶标仪测量OD595,依据OD595与蛋白浓度制作线性标准曲线。稀释待测蛋白质溶液若干倍,在20 μL蛋白溶液中加入180 μL定量工作液,读取OD595。依据标准曲线以及样品OD595计算样品蛋白浓度。其中,对照组4个生物重复样品为S.c_1、S.c_2、S.c_3、S.c_4,诱变组4个生物重复样品为S.c_11_1、S.c_11_2、S.c_11_3、S.c_11_4。
1.4 蛋白酶解
8个样品各取100 μg蛋白溶液;按蛋白∶酶=40∶1的比例加入胰蛋白酶2.5 μg,37 ℃酶解4 h;加酶循环1次,37 ℃继续酶解8 h;肽段除盐并真空抽干。
1.5 肽段标记
取出一定量iTRAQ标签试剂并恢复至室温,各管试剂加入50 μL异丙醇,涡旋振荡混匀后低速离心;用0.5 mol/L TEAB溶解肽段样品,并加入到对应iTRAQ标签试剂中,不同样品肽段选用不同的iTRAQ标签,静置2 h充分反应。
1.6 肽段分离
采用岛津LC-20AB液相系统对样品进行液相分离。用2 mL流动相A复溶抽干的肽段样品并进样,以1 mL/min的流速梯度洗脱:5%流动相B 10 min,20%流动相B 40 min,95%流动相B 1 min,流动相B持续3 min,5%流动相B平衡10 min。在214 nm波长下监测洗脱峰并每分钟收集1个组分,结合色谱洗脱峰图合并样品得到20个组分,然后冷冻抽干[8]。
1.7 纳升高效液相分离
将抽干的肽段样品用流动相C复溶,20 000 g离心10 min后,取上清进样。通过HPLC进行分离。样品首先进入trap柱富集并除盐,随后与自装C18柱串联,以300 nL/min流速通过如下有效梯度进行分离:0~5 min,5% 流动相D;5~45 min,流动相D从5%升至25%;45~50 min,流动相D从25%升至35%;50~52 min,流动相D从35%升至80%;52~54 min,80%流动相D;54~60 min,5%流动相D。
1.8 质谱检测
经过HPLC分离的肽段离子化后进入到与之串联质谱仪Q-Exactive HF X进行DDA(data-dependent acquisition)模式检测。
1.9 蛋白质鉴定与定量分析
使用软件Mascot 2.3.02进行蛋白鉴定,质谱数据与UniProt数据库比对搜索得到最终的蛋白鉴定结果。筛选包含至少1个可信的特异性肽段的蛋白。采用IQuant软件[9]以Mascot Percolator算法[10]进行iTRAQ数据定量。分别在谱图/肽段水平与蛋白质水平上进行1% FDR的过滤(PSM-level FDR≤0.01),从而获得显著性鉴定的谱图和肽段列表。利用肽段进行蛋白组装,并产生一系列的蛋白组。
1.10 生物信息学分析
对差异表达蛋白进行GO、KEGG Pathway富集分析与KOG注释,使用WoLF PSORT软件[11]实现蛋白质的亚细胞定位预测。
2 结果与分析
2.1 蛋白质定量结果
对照组与诱变组经Bradford定量法定量,结果见表1。
2.2 蛋白质鉴定结果
质谱数据经Mascot 2.3.02利用UniProt数据库比对鉴定样本中的蛋白质。对照组4个生物重复样品与诱变组4个生物重复样品,共进行1次重复试验。对有生物重复的比较而言,在生物重复配对情况下,最终差异蛋白需至少在1次配对重复中被定义为差异蛋白。此次试验共鉴定到4 916个蛋白质,包含764个差异蛋白质,占检测蛋白总数的15.54%。其中,上调蛋白数目378个、下调蛋白数目386个。鉴定到的所有蛋白的分子量分布情况见附图,其中X轴为蛋白质分子量范围(kDa),Y轴为相应蛋白比例。
2.3 生物信息学分析
2.3.1 差异蛋白基因本体论(GO)富集分析 GO富集分析给出了差异蛋白显著富集的GO条目,对差异蛋白涉及的分子功能、细胞组分、生物过程进行分析。检测到的764个差异蛋白,参与的分子功能有10个,其中主要功能有催化活性、结合、结构分子活性、转运活性、分子功能调节等;参与构成的细胞组分有12个,主要有细胞、细胞组分、细胞器、细胞器组分、大分子复合物等;参与的生物过程有19个,主要有细胞过程、代谢过程、细胞成分组织或生物发生、生物调节、定位、应激反应、生物过程调控等(表2、表3)。
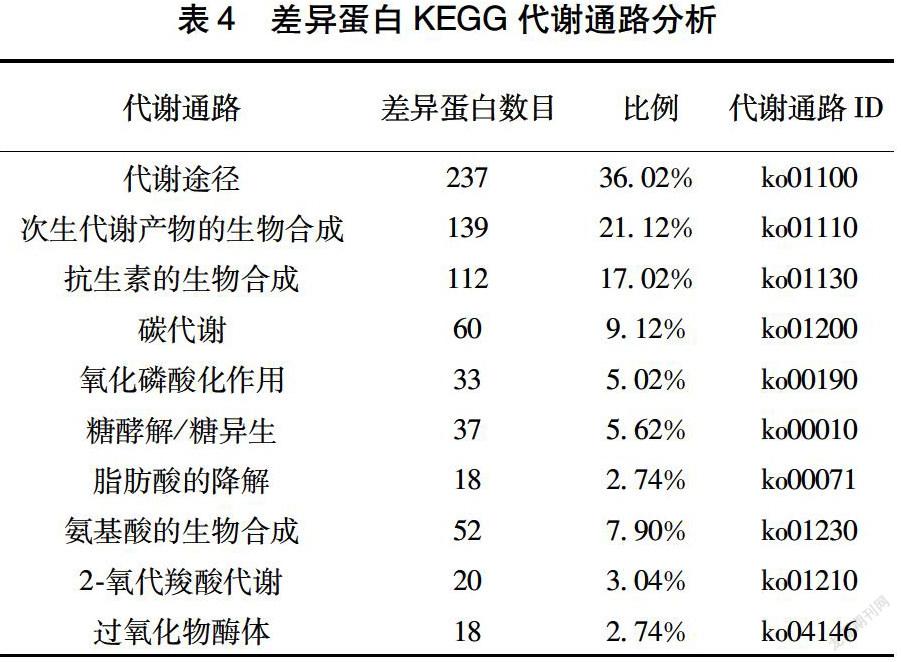
2.3.2 差异蛋白KEGG Pathway富集分析 KEGG数据库中主要的数据库有4个,分别为Pathway、Genes、Ligand、Brite[12],其中KEGG Pathway以图形代替文字直观地描述各种代谢途径以及各途径之间的关系。经KEGG Pathway分析,本次试验有658个差异蛋白参与了111条生物信号通路(表4)。
2.3.3 差异蛋白KOG注释分析 KOGs数据库是一种常用的蛋白功能注释数据库,每个KOG条目都包含一系列直系同源蛋白或旁系同源蛋白。此次KOG注释分析将所有鉴定到的差异蛋白BLAST比对KOG数据库,得到相应的KOG注释结果。750个显著差异蛋白参与了能量产生与转化,氨基酸转运和代谢,糖类运输和代谢,脂质转运和代谢,核苷酸转运与代谢,DNA的复制、重组和修复、转录、翻译,RNA加工和修饰,辅酶运输与代谢,无机离子的运输与代谢,次级代谢产物的生物合成、运输和分解代谢,信号转导等重要生物反应过程(表5)。
2.3.4 差异表达蛋白亚细胞定位预测 蛋白质合成后,经信号引导转运被分泌到细胞外部、细胞质基质或者进入特定的细胞器中,翻译后的蛋白只有到达正确的部位才能发挥其功能,參与细胞的各种生命活动。因此,进行蛋白质的亚细胞定位是对差异蛋白功能注释的重要部分。本次试验采用WoLF PSORT软件实现蛋白质的亚细胞定位预测。764个差异蛋白被预测出现在12个亚细胞结构中,其中最主要的结构有细胞核(34.82%)、细胞液(27.23%)、线粒体(18.98%)、细胞外(5.37%)、质膜(4.58%)等。
3 结论
经过模拟空间诱变,酿酒酵母的差异表达蛋白主要分布在细胞核、线粒体、核糖体和质膜等主要细胞结构中,涉及氨基酸、蛋白质、糖类、脂类、核苷酸、核酸等重要代谢过程;通过提高DNA复制,mRNA转录,蛋白质翻译、加工、转运,信号转导,细胞骨架结构维持等代谢过程效率。Rad50是一种参与DNA双链断裂修复的蛋白质,对于DNA双链断裂修复非常重要。Rad50蛋白在模拟空间诱变后的菌种中含量提升,这可能是酿酒酵母经模拟空间环境筛选出的优势菌株,但是在正常生产条件下并无辐射等诱变因素使得DNA双链断裂,因此,Rad50蛋白含量的增加对正常生产条件下的酿酒酵母菌株影响并无积极影响[13]。蛋白HSM3与DNA裂解酶APN1分别在DNA错配修复与切除修复中起重要作用,这两种蛋白的下调可能会引起突变率的提高,但同时减少了DNA修复时间,使得DNA复制速率有所提高[14]。海藻糖作为酵母重要的抗逆因子具有重要的抗逆作用,生物合成海藻糖的关键酶是海藻糖-6-磷酸合成酶TPS1[15]。诱变后酵母细胞TPS1下调,其抗逆能力也随之下降。由此可见,细胞是通过降低其对DNA复制后的修复能力与环境防御能力来提高生长速率的。但是诱变后的酿酒酵母有关DNA修复和环境防御部分的相关蛋白含量下调,这一变化对其突变与生长的意义及所带来的影响还需要进一步研究。
4 讨论
从20世纪60年代开始,国内外研究人员就利用返回式卫星或飞船搭载微生物菌种,获得了多种地面难于得到的优良菌株[16]。实验室近些年通过模拟空间诱变获得了一些生长速率加快的菌株,但均停留在现象研究上,深入的机理研究十分匮乏。本研究检测到的764个差异表达蛋白,涉及的多为较重要细胞结构物质、能量代谢与信号过程。上调蛋白涉及细胞核中染色质、核仁核孔复合体等重要成分,还有细胞质与细胞核中核糖体的大亚基与小亚基成分、内质网膜重要成分、高尔基体膜组成成分、细胞骨架成分、细胞质膜等,这些蛋白的上调为酿酒酵母的快速生长提供了物质基础。上调蛋白还涉及线粒体中线粒体双层膜结构、膜间空间、呼吸链中电子传递的酶复合物、V-型与F-型质子泵等重要成分,这些蛋白上调为酿酒酵母的快速生长提供了能量基础。上调蛋白中涉及的重要生物过程包括糖类代谢过程、氨基酸合成过程、核苷酸代谢过程、脂类的合成与转运、辅助因子与中间产物生物合成、产能代谢过程;蛋白质转录、翻译、折叠、转运的正调节,其中包括组蛋白乙酰化与H2B泛素化的正调控相关蛋白,组蛋白乙酰化包含H2A、H2B、H3、H4的10个以上的赖氨酸修饰位点[17],组蛋白H2B泛素化是RNA聚合酶转录Ⅱ所必需的[18];DNA复制,RNA加工处理,细胞骨架形成与维持,信号转导途径过程;cAMP合成过程,Ras蛋白信号转导、G蛋白偶联的信号传导途径。
下调蛋白包括转录负调控蛋白、染色质沉默过程相关蛋白;DNA修复蛋白,含切除修复与错配修复等相关蛋白,如蛋白HSM3与DNA裂解酶APN1,DNA修复相关蛋白减少,这可能是诱变后酵母DNA复制过程中以牺牲复制准确率为代价来快速复制DNA的机制;细胞骨架的解聚过程相关蛋白,有助于维持细胞骨架结构、细胞形态以及保证各种细胞器与生物大分子分布正常;环境防御相关蛋白如TPS1。这些蛋白涉及的生物过程与细胞的生存有关,但是并非细胞所必需,这可能是酿酒酵母细胞为应对模拟空间诱变这一逆境而启动的生物效应。
参考文献
[1]涂杜,罗佳,徐志宏.空间诱变后嗜酸乳酸菌对小鼠肠道功能的影响[J]. 安徽农业科学,2016,44(24):102-105.
[2]李军,冯世文,曾芸,等.大肠杆菌O157:H7氟苯尼考耐药菌株与敏感菌株的蛋白组学差异[J]. 南方农业学报,2019,50(4):875-882.
[3]曾睿,张果,郑腾达,等.基于 iTRAQ 技术的天然富硒与非富硒水稻差异蛋白分析[J]. 中国粮油学报,2019,34 (11):1-9.
[4]马晓聪,郑景辉,赵媚,等.基于iTRAQ技术的黄连解毒汤对自发性高血压大鼠主动脉差异蛋白的研究[J]. 中华中医药学刊,2019,37(9):2109-2112、2311.
[5]马晓丽,刘娟,曹雷雨,等.应用 iTRAQ 技术筛选不同生存期食管癌患者的血清差异蛋白[J]. 现代肿瘤医学,2019,27(20):3611-3616.
[6]Estéfani García-Ríos,Amparo Querol,José Manuel Guillamón.iTRAQ-based proteome profiling of Saccharomyces cerevisiae and cryotolerant species Saccharomyces uvarum and Saccharomyces kudriavzevii during low-temperature wine fermentation[J]. Journal of Proteomics,2016(146):70-79.
[7]崔为同,薛华儒,成洪达,等.Bradford定量法在蛋白质组学中应用的优化研究[J]. 生物技术进展,2019,9(3):309-315.
[8]许洁,王红旗,孔德康.基于iTRAQ技术荧蒽降解菌的比较蛋白质组学分析[J]. 中国环境科学,2018,38(1):284-292.
[9]Wen B,Zhou R,Feng Q,et al.IQuant:an automated pipeline for quantitative proteomics based upon isobaric tags[J]. Proteomics,2014,14(20):2280-2285.
[10]Brosch M,Yu L,Hubbard T,et al.Accurate and sensitive peptide identification with mascot percolator[J]. Journal of Proteome Research,2009,8(6):3176-3181.
[11]Paul H,Keun-Joon P,Takeshi O,et al.Wolf psort:protein localization predictor[J]. Nucleic Acids Research,2007,35(suppl 2):W585-W587.
[12]韓增叶,田平芳.KEGG数据库在生物合成研究中的应用[J]. 生物技术通报,2011(1):76-82.
[13]Malone R E,Ward T,Lin S,et al.The RAD50 gene,a member of the double strand break repair epistasis group,is not required for spontaneous mitotic recombination in yeast[J]. Current Genetics,1990,18(2):111-116.
[14]Fedorova I V,Gracheva L M,Kovaltzova S V,et al.The yeast HSM3 gene acts in one of the mismatch repair pathways[J]. Genetics,1998,148(3):963973.
[15]郭永豪.酵母菌基因tps1的克隆与表达分析[D]. 郑州:河南农业大学,2005.
[16]印红,陆维,谢申猛.搭载后红曲霉菌突变株的染色体DNA随机扩增多态性分析研究[J]. 航天医学与医学工程,2004,17(5):374-376.
[17]C.D.Allis,朱兵,等,译.表观遗传学[M].北京:科学出版社,2009:231-247.
[18]董强.组蛋白H2B泛素化修饰研究进展[J]. 安徽农业科学,2009,37(20):9380-9383.

