一株具有滑行能力的革兰氏阴性细菌的遗传操作体系构建
谢云轩


摘 要:开发全新来源的抗生素菌种资源在当前临床可用抗生素资源紧缺的情况下具有十分重要的意义。具有滑行能力的捕食细菌拥有丰富的次生代谢资源,是开发新型抗生素的理想菌株。在本研究中,一株从渤海沿岸石油烃污染土壤中分离得到的嗜麦芽窄食单胞菌展示出具有合成丰富代谢物的能力。对该菌株的基因组成分析显示基因组中存在新颖聚酮合成酶-非核糖体多肽合成酶(PKS-NRPS)基因簇。本研究探讨了利用接合转移方式进行聚酮合成酶基因簇的原位敲除的可行性,发现利用该方法进行基因敲除有约2.5%的概率获得正确重组子。
关键词:嗜麦芽窄食单胞菌;聚酮合成酶-非核糖体多肽合成酶;生物信息学分析;接合转移
微生物来源的天然药物及其衍生物在对抗微生物感染中占据着很大的比例1。微生物在长期进化中保留下来的抗生素具有很强的靶位点特异性和很高的生物活性2。近年来,抗生素的普遍使用在全球范围内导致一些具有抗生素抗药性的人类致病菌的产生,给临床治疗带来了新的挑战3。而新批准的抗生素大部分具有与原有抗生素相似的骨架结构,极易造成靶向致病菌再次培育完善的抗药机制以及抗生素的二次失效。较有名的实例包括ceftobiprole、daptomycin、tigecycline这些天然抗生素药物在对多重抗药性的金黄色葡萄球菌S.aureus和多重抗药性的肠球菌Enterococci治疗中的逐渐失效4。耐药型致病菌株的快速传播,已经导致临床上出现能对抗多种抗生素的“超级细菌”,给病人带来致命威胁。而拥有13亿人口的中国是世界上滥用抗生素最为严重的国家之一,细菌整体耐药率远远高于发达国家。针对耐药型致病菌株来研发新的抗生素目前变得刻不容缓。然而在全球抗生素资源短缺的同时,很多国际知名的制药行业在近些年却逐渐减少研发新型抗生素的投资,造成了抗生素供求关系矛盾的加剧。哈佛大学医学院著名天然产物学家Christopher T .Walsh教授指出,如果不增加抗生素的研发力度,人类对抗致病菌感染的能力将会迅速退化到抗生素發明之前的水平5。充分挖掘已有的抗生素主要来源菌细胞内的次生代谢物资源、开发全新的抗生素菌种资源迫在眉睫6,7。人们从滑行细菌中分离得到很多生物活性物质,部分显示出良好的成药性,进入临床试用阶段。例如具有抗肿瘤活性的curacin A和抗金黄色葡萄球菌活性的TAN-1057A是具有良好开发前景的先导化合物8,9,抗肿瘤化合物epothilone和dolastatin10,11已经进入临床试验期。
本文对分离自沿海潮间带区域嗜麦芽窄食单胞菌进行代谢产物分析测试并进行基因组成分析。通过比较已发表的嗜麦芽窄食单胞菌的基因序列,对代谢物较丰富的嗜麦芽窄食单胞菌P3-15的基因组进行初步分析,获取新颖聚酮合成酶-非核糖体多肽合成酶(PKS-NRPS)基因簇3个(图1A,B,C),针对图1A的基因簇设计引物并通过接合转移的方法进行基因簇敲除。本研究建立了嗜麦芽窄食单胞菌遗传操作体系,并成功地对一新颖聚酮合成酶-非核糖体多肽合成酶进行基因敲除。研究证实嗜麦芽窄食单胞菌具有基因敲除可操作性,能够通过基因修饰获取新颖代谢产物。
实验方法:
1.采集渤海沿岸石油烃污染土壤(5厘米深度),以无菌蒸馏水清洗土壤并在1.5%几丁质的琼脂糖平板上进行筛选,将单克隆转移至LB(含氨苄霉素、卡那霉素)液体培养基内进行富集及进一步筛选。通过碱裂解法提取单克隆总DNA,利用特异性引物进行16SrRNA可变区扩增,通过454测序分析获取十二株菌株的16SrRNA组成。
2.选取代谢物较丰富的P3-15菌株进行总DNA提取及细菌框架图的测序分析(凌恩生物),获取长度4.2Mb的总DNA序列。通过ANTI-SMASH分析获得新颖聚酮合成酶-非核糖体多肽合成酶(PKS-NRPS)基因簇3个。设计引物(CATTAGCT ACAGCGCGCCGGACC/CAGTTACCGCATCAAGCCCATTAC)进行靶序列PKS1扩增(预期片段大小:815bp)。
3.利用多聚酶链式反应(天根生物PCR扩增试剂盒)获取200ngPKS1靶序列片段。培养大肠杆菌S17-1细胞(200毫升LB液体培养基),提取接合转移载体pJQ200SK共200ng(图2)(天根生物质粒提取试剂盒)。采用SpeI及PstI双酶切载体与PKS1序列(NOVOZYME),纯化酶切片段达到50ng/l浓度。用不少于200ngPKS1与100ng载体在16°C连接过夜(12h)。冰上解冻100L大肠杆菌DH5细胞,在解冻的细胞液内加入10l转化液冰浴30分钟,随后42°C热激90秒。继续冰浴5分钟后加入1ml无抗性LB液体培养基,37°C松弛培养60分钟。在12300rpm转速下离心1分钟除去上清,将剩余细胞悬浮于约100l的无抗LB培养基中。将细胞悬浮液平铺到LB(含100g/ml庆大霉素)固体平板培养基上,37°C生长16-24h(同时用不含PKS1的空白质粒转化大肠杆菌DH5细胞作为阴性对照)。将有抗性的单克隆转移至10mlLB(含100g/ml庆大霉素)液体培养基中,37°C生长16-24h。对质粒进行小量制备及PCR验证(图1)。
4.对正确的PKS1-pJQ200SK载体进行制备(100mlLB液体培养基),浓缩至200ng/ml。取2 l转化大肠杆菌S17-1(热激法),挑取单克隆在10mlLB(含100g/ml庆大霉素)液体培养基内培养至O.D.0.7,取1ml培养液进行接合转移。同时培养野生P3-15菌株至O.D.0.7,取1ml培养液进行接合转移。
5.将1mlS17-1及P3-15菌株离心,弃上清。将菌体悬浮于1ml100mg/L的硫酸镁溶液中,12300rpm离心后弃全部上清。加入1ml硫酸镁溶液继续悬浮,再次离心后留约100l硫酸镁溶液,将细胞悬浮后平铺于1/10TSA(含100g/ml庆大霉素和25g/ml卡那霉素)固体培养基上,30°C培养4-5天。
6.挑取具有抗性的單克隆在3ml1/10TSB(含100g/ml庆大霉素和25g/ml卡那霉素)培养基中30°C培养4-5天。提取单克隆总DNA并进行PKS1片段的PCR验证。
实验结果:
1.P3-15的新颖聚酮合成酶-非核糖体多肽合成酶(PKS-NRPS)的antiSMASH生物信息学分析:
PKS合成基因簇及其他次级代谢产物相关的合成基因簇通常使用antiSMASH软件进行注释12。对P3-15的antiSMASH注释证实其基因组内含有包括Bacteriocin、arylpolyene、未知Nrps等次级代谢产物合成基因簇(图2)。针对图2A中酮基合成酶序列设计引物,扩增长度815bp的片段。
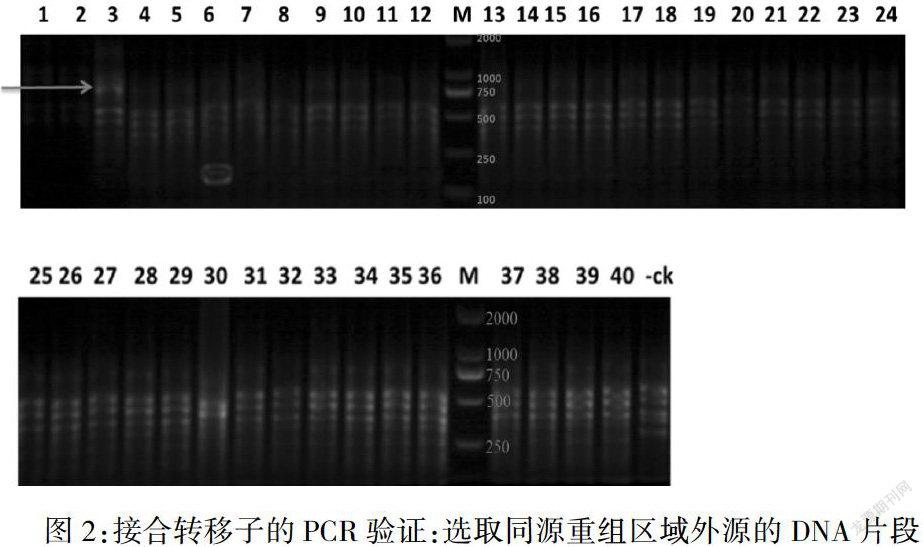
2.转化大肠杆菌S17-1并对转化子进行基因型鉴定。对鉴定正确的转化子进行扩大培养,并获取对数增长期细胞(O.D.0.5-0.6)。同时培养嗜麦芽窄食单胞菌至对数增长期。混合等体积的大肠杆菌S17-1和嗜麦芽窄食单胞菌细胞并涂布平板,采用庆大霉素和卡那霉素进行抗生素筛选。28°C培养2-4天。挑取40个具有双重抗生素抗性的单克隆进行PCR验证(918bp)(图2)。通过抗生素筛选和PCR验证,#3号克隆的基因组模板扩增产生与预期扩增大小相同的条带,是正确的重组子。
图2:接合转移子的PCR验证:选取同源重组区域外源的DNA片段作为设计引物的序列参考,对重组子进行基因型的验证。预期条带大小:918bp(蓝箭头)。
参考文献:
1 Newman, D.J., Cragg, G.M., 2012. Natural products as sources of new drugs over the 30 years from 1981 to 2010. J. Nat. Prod. 75(3), 311–335.
2 Koehn, F.E., Carter, G.T., 2005. The evolving role of natural products in drug discovery. Nat. Rev. Drug. Discov. 4(3), 206–220.
3 Woodford, N., Turton, J.F., Livermore, D.M., 2011. Multiresistant Gram-negative bacteria: the role of high-risk clones in the dissemination of antibiotic resistance. FEMS Microbiol. Rev. 35(5), 736–755.
4 Boucher, H.W., Talbot, G.H., Bradley, J.S., Edwards, J.E., Gilbert, D., Rice, L.B., Scheld,M., Spellberg, B., Bartlett, J., 2009. Bad bugs no drugs: no ESKAPE! An update from the infectious diseases society of America. Clin. Infect. Dis. 48(1), 1–12.
5 Walsh, C.T., Fischbach, M.A., 2010. Natural products version 2.0: connecting genesto molecules. J. Am. Chem. Soc. 132(8), 2469–2493.
6 Appelbaum, P.C., 2012. 2012 and beyond: potential for the start of a second pre-antibiotic era? J. Antimicrob. Chemother. 67(9), 2062–2068.
7 Bush, K., Courvalin, P., Dantas, G., Davies, J., Eisenstein, B., Huovinen, P., Jacoby, G.A., Kishony, R., Kreiswirth, B.N., Kutter, E., Lerner, S.A., Levy, S., Lewis, K., Lomovskaya, O., Miller, J.H., Mobashery, S., Piddock, L.J., Projan, S., Thomas, C.M., Tomasz, A., Tulkens, P.M., Walsh, T.R., Watson, J.D., Witkowski, J., Witte, W., Wright, G., Yeh, P., Zgurskaya, H.I., 2011. Tackling antibiotic resistance. Nat. Rev.Microbiol. 9, 894–896.
8 Verdier-Pinard, P., Lai, J., Yoo, H., Yu, J.,Marquez, B., Nagle, D.,Nambu, M., White, J., Falck, J., Gerwick, W., Day, B., Hamel, E., 1998. Structure-activity analysis of the interaction of curacin A, the potent colchicine site antimitotic agent, with tubulin and effects of analogs on the growth of MCF-7 breast cancer cells. Mol. Pharmacol. 53(1), 62-76.
9 Aguilar, N., Kruger, J., 2002. Toward a library synthesis of the natural dipeptide antibiotic TAN 1057 A, B. Molecules. 7(6), 469-474.
10 Larkin, J., Kaye, S., 2006. Epothilones in the treatment of cancer. Expert Opin. Invest. Drugs. 15(6),691-702.
11 Simmons, T., Andrianasolo, E., McPhail, K., Flatt, P., Gerwick, W., 2005. Marine natural products as anticancer drugs. Mol. Cancer Ther.4(2), 333-342.
12 Medema M H, Kai B, Cimermancic P, et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research, 2011, 39(8):339-46.

