基于光学-ALS变量组合和非参数模型的天然次生林地上生物量估算
赵颖慧,郭新龙,甄 贞*
(1. 东北林业大学林学院,黑龙江 哈尔滨 150040;2. 东北林业大学森林生态系统可持续经营教育部重点实验室,黑龙江 哈尔滨 150040)
森林是重要的陆地生态系统,也是地球资源中最为重要的自然资源之一。近年来,陆地植被生物量的监测已成为一个重要的研究课题,它旨在减少与森林破坏、退化相关的温室气体排放[1],以及更好地量化全球碳循环[2]。干、枝和叶生物量合称为森林地上生物量[3](aboveground biomass, AGB)。全球气候变化研究迫切需要快速、高效的森林AGB制图[4-5]。由于遥感技术具有宏观、大面积、快速和可重复等特点,遥感估算法已经成为区域森林AGB估算的主要方法。
天然次生林占我国森林总面积的70%,是我国森林资源的主体和重要基地[6]。天然次生林一般在土层较深厚、阳坡且处于下坡位时生物量较高[7],生物量空间分配特征因不同龄组也有所不同,一般成熟林生物量较高,而幼龄林生物量较低[8]。近年来,有学者进行天然次生林生物量遥感估算的研究,申鑫等[9]利用高光谱与高空间分辨率遥感数据对亚热带天然次生林的生物量进行反演,结果表明,AGB模型的精度要高于地下生物量模型,而且综合两种数据的模型精度要高于仅使用高光谱数据模型,可以有效地估算亚热带天然次生林生物量。Li等[10]使用Sentinel-1A和Landsat 8数据对湖南省郴州天然次生林AGB进行估算,研究表明极端梯度提升模型(extreme gradient boosting, XGBoost)结合Sentinel-1A和Landsat 8图像作为预测变量,可以提供最佳的森林AGB估算,并且XGBoost模型可以减少高估和低估的问题。由此可见,采用多源遥感数据结合可有效地估算天然次生林AGB。
机载激光雷达(airborne laser radar, ALS)发射的激光脉冲可以穿透植被冠层获得其三维结构和能量信息,可用于估算森林参数。学者们利用激光雷达(light detection and ranging, LiDAR)衍生的森林结构参数与统计建模估算北方森林[11-13]、软木为主的温带混交林[14-16],以及热带、亚热带硬木林[17-19]的AGB。虽然ALS能够表征森林垂直结构参数,但在时空分布上不具有优势,也不能提供估算森林AGB所需要的光谱信息。Sentinel-2A卫星对植被的监测十分有效。因此,综合利用多源遥感数据源是获取森林AGB精确分析结果的有效途径,Brovkina等[20]研究表明无论是单木尺度还是样地尺度都是二者融合估算精度最高。Ediriweera等[21]研究表明单独使用LiDAR数据可以较为准确地估算桉树林的生物量,但是加上Landsat 5 TM数据更能提高估算的精度。郭艺歌等[22]研究表明多光谱图像HJ1B进行混合像元分解后的组分图像与雷达图像ALOS/PALSAR进行图像融合建立的生物量回归模型,其估算生物量与实测生物量一致性较好。虽然这些研究都是使用的多源遥感数据,但大多是多源数据的协同,很少有将多源遥感数据提取的变量再组合之后用于估算生物量。因此,本研究采用ALS数据和Sentinel-2A数据提取特征变量,并进行变量结合来估算森林AGB。
传统的如多元线性逐步回归(stepwise multiple linear regression, SMLR)[23-24]模型能很好地解释自变量与因变量之间的线性关系,常用于建立森林参数和遥感预测因子的关系模型。近年来,非参数模型的应用更加普遍,与线性回归相反,非参数模型能够解释复杂的非线性关系,从而被应用到许多研究中,如K近邻(K-nearest neighbor,K-NN)[25]、随机森林(random forest, RF)[26]、支持向量回归(support vector regression, SVR)[27]、堆叠稀疏自动编码器(stacked sparse auto-encoder, SSAE)模型[28]等。随着非参数模型的发展,这些模型不仅应用到目标检测和图像分类中,很多研究[28-29]也表明非参数模型在估算森林植被参数方面十分有效。
本研究以东北林业大学帽儿山林场为研究区域,以ALS数据、Sentinel-2A数据和2016年森林资源连续清查固定样地为数据源,分别以ALS和Sentinel-2A数据结合变量(combined optical and LiDAR index, COLI)结合成新的变量COLIs,构建5种方法(SMLR、K-NN、SVR、RF和SSAE)的天然次生林AGB估算模型并对比分析,探讨了COLIs对天然次生林AGB估算精度的影响,为大范围森林AGB估算提供参考。
1 材料与方法
1.1 研究区概况
研究区为东北林业大学帽儿山实验林场,位于黑龙江省尚志市西北部(127°29′~127°44′E,45°14′~45°29′N),总面积26 496 hm2,共10个施业区。地貌属低山丘陵区,地势由南向北逐渐升高,平均海拔300 m,年平均气温2.8 ℃左右,年降水量为723 mm左右,土壤以暗棕壤为主。帽儿山地区的植被属长白植物区系,是典型的东北天然次生林区,乔木树种主要包括:白桦(Betulaphatyphylla)、山杨(Populusdavidiana)、五角槭(Acermono)、柞树(Querusmongolica)、椴树(Tiliaamurenisis)、榆树(Ulmuspumila)、水曲柳(Fraxinusmandshurica)、胡桃楸(Juglansmandshurica)、黄菠萝(Phellodendronamurense)、落叶松(Larixgmelinii)、红松(Pinuskoraiensis)、樟子松(Pinussylvestris)、云杉(Piceakoraiensis)等。
1.2 研究数据获取
1.2.1 ALS数据
数据于2015年9月由中国林业科学研究院设计的LiCHy系统搭载LiDAR传感器(Riegl LMS-Q680i)获取,传感器波长为1 550 nm,激光离散束角0.5 mrad,激光脉冲长度3 ns,最大频率400 kHz,扫描角±30°,采样间隔1 ns,垂直精度0.15 m,平均点云密度3.6点/m2。
1.2.2 Sentinel-2A数据
Sentinel-2A数据是通过ESA共享网站(https://scihub.copernicus.eu/)获取,成像时间为2016年8月9日,与外业调查时间一致。L1C级标准产品,共包含13个波段:波段2、3、4、8分别为蓝、绿、红、近红外波段,分辨率均为10 m;波段5、6、7为3个红边波段,波段8a为窄近红外波段,波段11、12为两个短波红外波段,分辨率为20 m;其余3个波段分辨率为60 m,分别是气溶胶波段、水汽波段和卷云波段。
1.2.3 样地数据
样地数据为2016年森林资源连续清查的263块固定样地,面积为0.06 hm2,分布在1 km×1 km的网格上,其中有林地193块,去掉一部分由于调查产生误差较大的数据,剩余161块,对样地内树木每木检尺,起测径阶为5 cm,记录胸径(DBH)、树种等,手持GPS记录样地中心点坐标,精度在5 m以内,样地信息如表1所示。
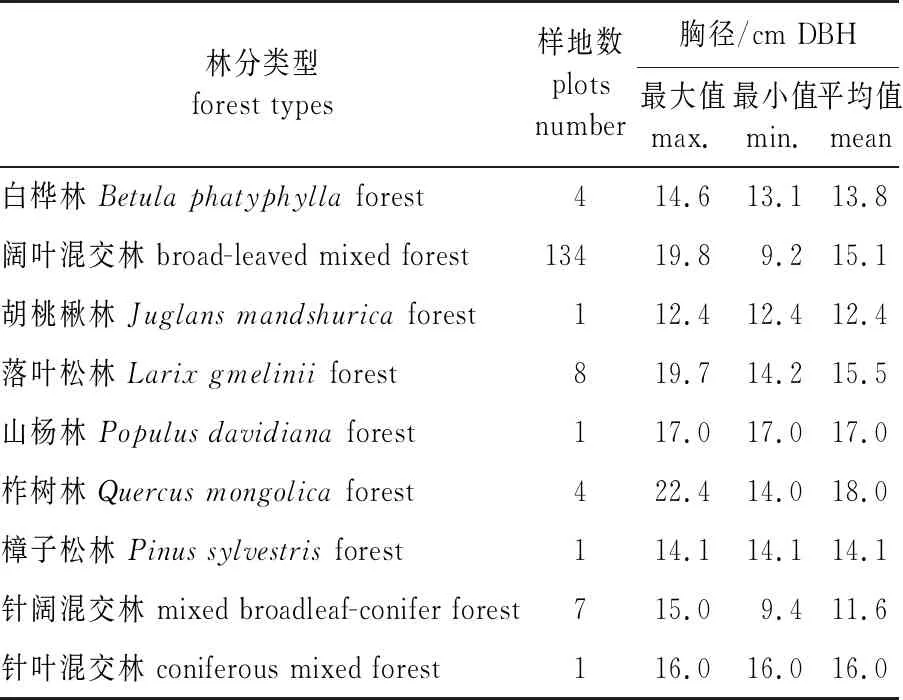
表1 帽儿山固定样地统计信息
1.3 数据预处理
ALS数据预处理包括:对点云数据按样地边界进行裁剪;将样地内点云数据滤波分类,分成地面点和非地面点;由地面点插值生成数字高程模型(digital elevation model, DEM);以DEM对植被回波点的高程进行归一化处理。
Sentinel-2A数据预处理包括:大气校正、重采样和几何精校正。首先利用ESA提供的SNAP软件中sen2cor插件对数据进行大气校正,得到各个波段的地表反射率;其次,将所需波段全部重采样至10 m分辨率;最后,将各个波段融合后,以帽儿山数字正射影像图(digital orthophoto map, DOM)为基准影像,选取显著标志点进行几何精校正,误差控制在0.2个像元内。
1.4 样地AGB计算
利用东北林区主要树种的生物量异速生长方程[30]分别对树干、树枝和树叶部分的生物量进行估算,相加后得到AGB,公式如下:
W=a×Db。
(1)
式中:W为生物量;D为胸径;a、b为异速生长方程的模型参数,依据各树种参数值[30]。按树种分别计算样地内每株树的生物量,然后将样地内单木生物量求和,与样地面积相除,得到单位面积生物量。
1.5 特征变量提取
1.5.1 ALS数据特征变量的提取
以预处理后的ALS点云数据提取首回波高度的统计量:包括最大值(hmax)、最小值(hmin)、平均值(hmean)、偏斜度(hskew)、峰度(hkurt)、标准差(hstd)、方差(hvar)、变异系数(hcv)、众数(hmode)和分位数(h10、h20、h30、h40、h50、h60、h70、h80、h90、h99)[31]。很多研究以10%为间隔提取高度分位数[32-35],表明分位数以10%为间隔与生物量高度相关,因此本研究的高度分位数也是以10%为间隔提取。另外,为排除灌木层的影响,一般取归一化处理后高于2 m的回波点,因此设本研究提取密度变量为C,即为高于2 m的回波点在所有回波点中所占的比例。
1.5.2 Sentinel-2A数据特征变量的提取
Sentinel-2A数据包含了3个红边波段(5、6和7波段),对植被的监测十分有效,因此本研究以Sentinel-2A数据提取植被指数为主要特征变量。有研究表明中心波长在705 nm处的反射率与叶绿素含量的相关性优于740和783 nm处的反射率[36]。因此,本研究选取中心波长在705 nm处(即波段5)计算红边植被指数。Sentinel-2A提取的植被指数包括归一化植被指数[37]、差值植被指数[38]、增强植被指数[39]、比值植被指数[40]、叶绿素指数[41]、最佳土壤调节植被指数[42]、红边归一化植被指数[43]、红边比值植被指数[44]、红边叶绿素指数[41]。
1.6 特征变量的组合
由于光学数据可以提供植被冠层的光谱信息,而ALS数据可以获得森林的垂直结构信息,因此,ALS与光学遥感数据的协同对于估算森林AGB具有很高的价值。由Zhang等[29]研究发现,以ALS数据和Landsat 8 OLI数据提取的特征变量为基础计算光学-ALS的结合变量(combined optical and LiDAR index, COLI,即COLIs,式中记为ICOLs)在估算森林生物量方面取得了较高的精度。因此本研究也采用这种光学-ALS变量的结合方式,即以ALS数据提取的特征变量(AL)和Sentinel-2A数据提取的特征变量(AO)来计算ICOL1和ICOL2,公式为:
ICOL1=Isvi×BLV;
(2)
ICOL2=Isvi_BLV=(BLV-Isvi)/(BLV+Isvi)。
(3)
式中:BLV为与AGB拟合精度最高的ALS变量;Isvi为Sentinel-2A数据提取的各个植被指数。由于BLV和Isvi二者的量纲不同,不能直接进行运算,因此在运算之前需要对BLV和Isvi进行归一化处理。
构建6组特征变量组合,即AO+AL、ICOL1、ICOL2、ICOL1+AO+AL、ICOL2+AO+AL、ICOL1+ICOL2+AO+AL,进行森林AGB估算。
1.7 建模方法
1.7.1 多元线性逐步回归
多元线性逐步回归(stepwise multiple linear regression, SMLR)是比较传统的回归模型,主要是在考虑的全部自变量中按其对逐步回归作用大小、显著程度大小或贡献大小,由大到小地逐个引入回归方程,而对逐步回归作用不显著的变量可能始终不被引入回归方程。本研究将非参数估算方法与该传统的SMLR回归模型进行对比分析。
1.7.2K-最近邻
K-最近邻[45](K-nearest neighbor,K-NN)算法是较简单的机器学习算法之一,不仅可以用于分类还可以用于回归。通过找出一个样本的K个最近邻样本,将不同距离对样本产生的影响给予样本不同的权值,得到样本的属性。使用该方法需要设置:计算预测值时考虑的最近邻个数K、选择近邻样本的距离参数及计算预测值时对近邻样本的加权方案等几个参数。本研究经多次测试,最终选择的近邻个数K为10,距离参数为欧氏距离,加权方案为计算预测值时与所有的相邻样本权重相等。
1.7.3 支持向量回归
支持向量回归[46](support vector regression, SVR)对于低维输入空间中非线性可分训练样本,通过核函数将其映射到高维空间中使其线性可分,并保证良好的泛化能力。本研究经多次测试获得最优参数,即核函数为多项式核函数,多项式的系数为1,误差项的惩罚参数C=10,损失函数的值为0.5,使用Python的sklearn包来实现。
1.7.4 随机森林
随机森林(random forest, RF)[47]是建立在决策树基础上,通过多次bootstrap抽样获得多个随机样本,并以这些样本分别建立相应的决策树,从而构成RF。由于RF选取样本的方法是放回随机抽样,因此,建立回归树时有一部分样本数据不会被选中而作为检验样本出现,起到了样本内部交叉验证的作用,两个随机性的引入,可减少过拟合情况的出现。同样RF也需要设置几个参数,本研究经测试,基础决策树的数量设置为500,每个基础决策树模型分裂所需最小样本数为4,每个决策树模型叶节点所包含的最小样本数设为10。
1.7.5 堆叠稀疏自编码器模型
堆叠稀疏自编码器(stack sparse auto-encoder,SSAE)是一种深度学习模型,最早由Shao等[28]提出并用于估算森林AGB。一个简单的自编码器(auto-encoder,AE)模型包含编码器和解码器两部分,本质上是对输入信号经过中间层的激活函数进行变换后输出,相当于一个3层的神经网络,而AE的目的是让输出信号尽可能地复现输入信号,从而使中间的编码层获得一定的学习能力,通常会对中间的编码层进行一定约束。从数据维度来看,当中间编码层的维度大于输入数据的维度时,编码层的表达会变得稀疏,此时的自编码器便是稀疏自编码器(sparse auto-encoder,SAE)。SSAE就是包含了多个SAE的模型,其本质就是将前一个SAE的输出作为下一个SAE的输入,建立深度神经网络,进行更深层次的学习以产生数据的深层特征,最后再通过解码层输出结果。本研究采用Python语言实现SSAE模型,并且使用4层编码层进行训练,再由4层解码层将数据解码成原来的维度,其中激活函数为relu。
1.8 模型精度评价
采用十折交叉验证法进行模型的精度验证,首先将数据集随机分成10份,然后轮流将其中9份作为训练数据,1份作为测试数据进行试验,最后取10次验证之后得到的评价指标均值作为最终的评价指标。将6组特征变量组合分别代入估算模型,得到估算值与森林AGB实测值的关系模型,使用验证数据集的决定系数(R2)[48]、均方根误差[RMSE,式中记为σ(RMSE)]、相对均方根误差[rRMSE,式中记为σ(rRMSE)]作为模型精度评价指标。计算公式为:
R2=
(4)
(5)
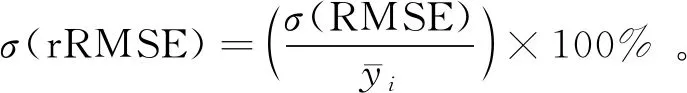
(6)
2 结果与分析
2.1 ALS特征变量与AGB拟合结果
用统计回归分析方法建立了161块样地的ALS特征变量与AGB之间的关系模型,精度评价如表2所示。
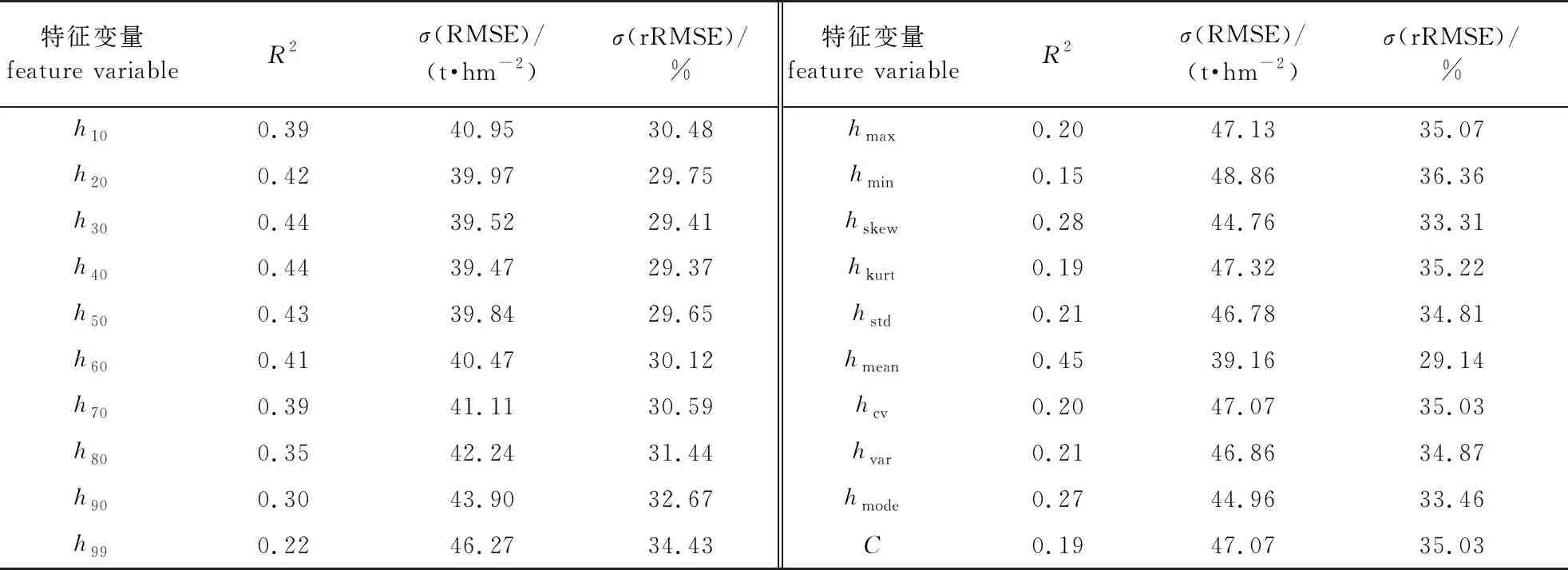
表2 ALS特征变量与AGB拟合的精度
从表2中可以看出:首回波高度均值(hmean)与AGB的相关系数最高,因此使用hmean作为COLI中的ALS特征变量(BLV)。
2.2 估算森林AGB结果
以ALS提取的特征变量(AL)、Sentinel-2A数据提取的特征变量(AO)以及结合变量(ICOL1和ICOL2),构建6组特征变量组合,即AO+AL、ICOL1、ICOL2、ICOL1+AO+AL、ICOL2+AO+AL、ICOL1+ICOL2+AO+AL,分别输入SMLR、K-NN、SVR、RF和SSAE模型中估算森林AGB,各模型精度结果如表3所示。
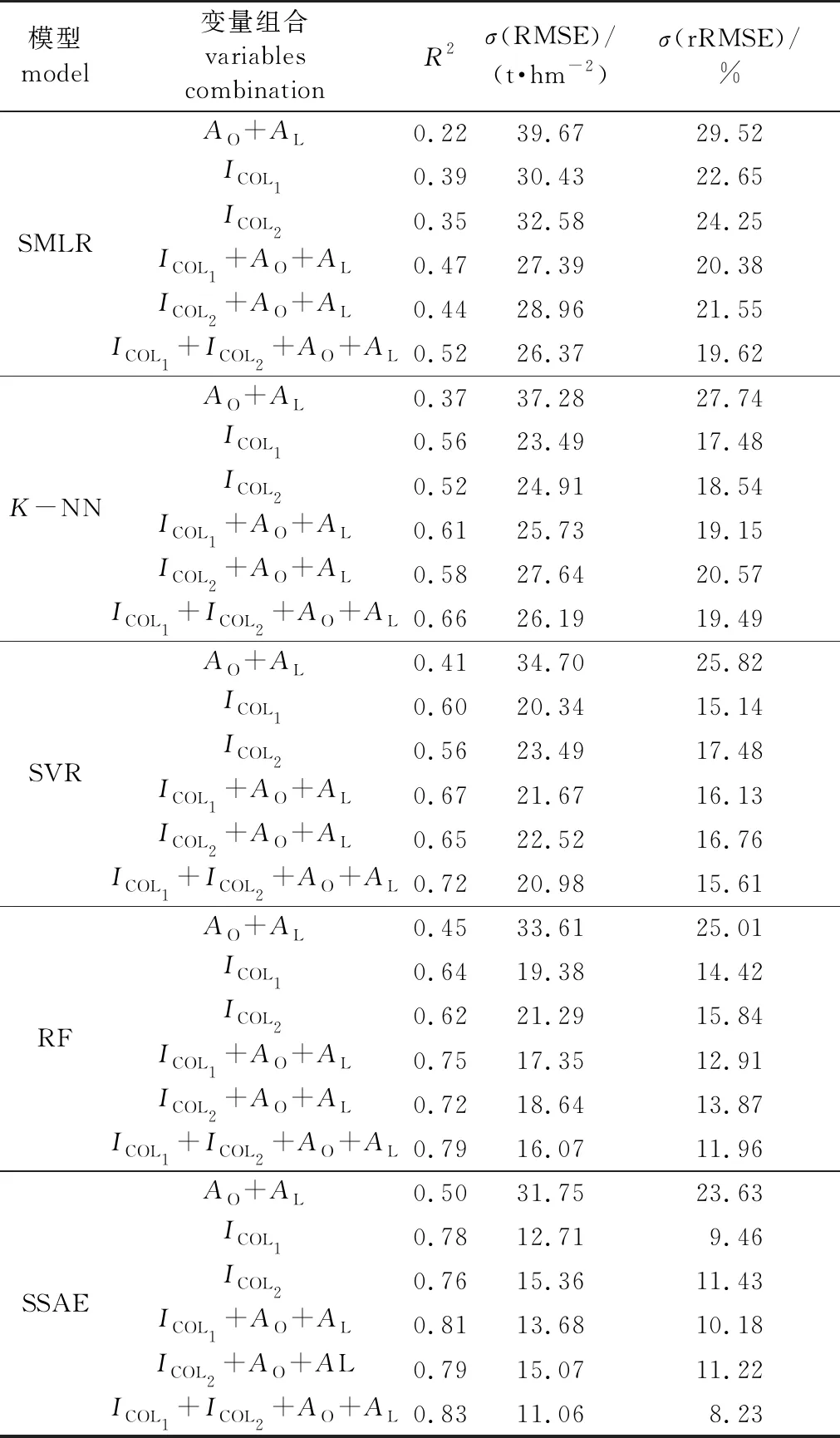
表3 各个模型的精度结果
由表3可以看出:无论是哪一组特征变量输入模型,SSAE模型的估算精度均最高(R2=0.83;RMSE为11.06 t/hm2; rRMSE为8.23%),其次是RF、SVM和K-NN模型,仅应用ALS提取的特征变量(AL)和以Sentinel-2A数据提取特征变量(AO)的SMLR模型精度最低(R2=0.22; RMSE为39.67 t/hm2;rRMSE为29.52%)。随着变量的增加,各模型的精度也有所提高。此外,结合变量ICOL1相比于ICOL2表现出更好的性能,无论是单独使用ICOL1变量还是ICOL1与AL、AO的变量组合都比单独应用ICOL2变量和应用ICOL2+AO+AL变量组合时的精度略高。
3 讨 论
3.1 基于Sentinel-2A的生物量估算
在光学遥感数据的可见光至红外波段,植物的反射光谱曲线具有显著的特征,但是不同类型植物之间反射光谱存在一定的差异,即使同一种植物,也会由于其生长状态的不同而发生变化,因此可以利用植被的光谱特征来监测其生长状况。而Sentinel-2A数据的传感器可捕获红光到近红外区间的红边信息,因而更有利于反映植被变化情况。Astola等[49]研究Sentinel-2A估算森林参数的性能优于Landsat 8数据。郑阳等[50]利用Sentinel-2A数据估算了冬小麦的地上生物量,结果表明所选的植被指数均与生物量显著相关且红边叶绿素指数与生物量估算精度最高。因此,本研究选用Sentinel-2A数据作为光学遥感变量。由于本研究尚未考虑到Sentinel-2A数据地形校正,模型估算精度在一定程度上受到影响,因此对Sentinel-2A数据地形校正后的估算还需进一步研究。
3.2 结合变量ICOLs对估算森林AGB的影响
本研究中,将ALS数据提取的特征变量与AGB拟合后精度最高的首回波高度均值(hmean)及Sentinel-2A提取的特征变量重新结合成新的特征变量,ICOL1和ICOL2相较于仅使用ALS和Sentinel-2A数据提取的变量,在森林AGB估算中都具有较好的表现,即加入ICOL1和ICOL2都取得了更高的精度,而且当所有变量输入模型时精度最高,这也与Zhang等[29]的研究结果一致。
本研究的变量ICOLs是根据植被指数的计算方式,将ALS数据的首回波高度参数和Sentinel-2A的植被指数相结合,使ICOLs既包含了ALS数据的高度信息,又包含了Sentinel-2A数据的光谱信息,明显地提高了生物量模型的精度,ICOLs的表现可归因于多光谱图像提供的地表郁闭密度和覆盖度信息的能力,以及ALS数据对于树木枝干的森林信息的表达能力。与单独的主被动遥感特征变量(AO+AL)相比,ICOLs既包含了与AGB高度相关的高度变量,也包含了能够反映植被光谱特性的植被指数变量,二者结合的ICOLs变量对森林AGB具有更强的解释能力,因此,无论是单独使用ICOLs还是加入ICOLs变量都使模型获得了更高的精度。此外,虽然ICOL1拟合模型一般比ICOL2拟合模型能够得到更小的RMSE和更高的R2,但是它们之间的模型精度相差不大,都可以对生物量进行较准确的估算。总的来说,ICOLs可以提高估算生物量模型的精度,由于单一数据源的局限性可以通过另一种数据得到补偿。另外,在广泛应用之前,ICOLs应该需要进一步的调查和验证。
鉴于提取的AO和AL变量以及ICOLs的计算方式,各个变量之间可能会存在共线性关系,在线性模型中,自变量的共线性会导致结果不准确,影响模型的精度,但是在机器学习等非线性模型中,尤其是RF模型,受自变量的共线性影响较小[51]。而在本研究中用到的线性模型只有多元线性逐步回归,该方法本身具有逐步回归的过程,可以消除共线性的影响。因此在本研究中变量的共线性对整体预测结果影响不大。
3.3 基于不同模型的生物量估算
本研究中深度学习模型SSAE的估算精度要高于其他4种模型(RF、SVR、K-NN和SMLR),这是由于SSAE模型隐含层的维度大于输入数据的维度,通过给隐含层的神经元加入稀疏性限制来发现输入数据中的结构,因此SSAE模型能够自动逐层学习数据更深层次的空间稀疏特征。在SSAE模型中,输入变量被映射到另一个特征空间中,在这个特征空间中AGB可以被准确预测。由于隐含层的稀疏性,得到了更完备的稀疏特征,且SSAE模型具有多层次的结构,因此学习到的特征对AGB具有更强的解释能力。模型中的编码层也获得了一定的学习能力,最后通过解码层将数据输出,从而得到更准确的森林AGB估算结果。在今后的研究中,可以针对不同的森林类型分别应用SSAE建立模型,以期提高森林AGB的估算精度。
此外,除了SSAE模型,RF模型的精度要高于其他模型(SVR、K-NN和SMLR),应用RF模型估算森林参数也能够得到较好的结果,如孙雪莲[52]的研究表明RF模型的拟合效果较好。RF相对突出的性能取决于其独特的回归技术和算法设计,这对于噪声和异常值具有良好的稳健性和灵活性。
在本研究中,SVR模型的估算精度较RF差,原因可能是由于噪声变量对SVR的最优超平面产生了偏差,从而降低了模型的精度。对于所有变量组合,SVR比K-NN的精度更高,说明SVR在处理高维和非线性问题上比K-NN更有优势,这与Shao等[28]的研究结果一致。此外,K-NN模型的精度相对较低,可能是与K-NN模型在实现过程中缺少一种有效的方法确定相邻样本间的权重有关。一般情况下,SMLR模型的精度都是最低的[53],因为SMLR只能够较好地解释变量之间的线性关系,然而研究中不是所有的变量都与AGB呈线性相关,因此SMLR模型精度相对较低。
参考文献(reference):
[1]GOETZ S, DUBAYAH R. Advances in remote sensing technology and implications for measuring and monitoring forest carbon stocks and change[J]. Carbon Manag, 2011, 2(3): 231-244. DOI:10.4155/cmt.11.18.
[2]ZOLKOS S G, GOETZ S J, DUBAYAH R. A meta-analysis of terrestrial aboveground biomass estimation using lidar remote sensing[J]. Remote Sens Environ, 2013, 128: 289-298. DOI:10.1016/j.rse.2012.10.017.
[3]娄雪婷, 曾源, 吴炳方. 森林地上生物量遥感估测研究进展[J]. 国土资源遥感, 2011, 23(1): 1-8. LOU X T, ZENG Y, WU B F. Advances in the estimation of above-ground biomass of forest using remote sensing[J]. Remote Sens Land Resour, 2011, 23(1): 1-8. DOI:10.1016/j.ejphar.2011.07.001.
[4]TSUI O W, COOPS N C, WULDER M A, et al. Integrating airborne LiDAR and space-borne radar via multivariate Kriging to estimate above-ground biomass[J]. Remote Sens Environ, 2013, 139: 340-352. DOI:10.1016/j.rse.2013.08.012.
[5]LI W, NIU Z, LIANG X L, et al. Geostatistical modeling using LiDAR-derived prior knowledge with SPOT-6 data to estimate temperate forest canopy cover and above-ground biomass via strati-fied random sampling[J]. Int J Appl Earth Obs Geoinformation, 2015, 41: 88-98. DOI:10.1016/j.jag.2015.04.020.
[6]CHEN X W, LI B L, LIN Z S. The acceleration of succession for the restoration of the mixed-broadleaved Korean pine forests in Northeast China[J]. For Ecol Manag, 2003, 117(1/2/3): 503-514. DOI:10.1016/S0378-1127(02)00455-3.
[7]安利波, 毕会涛, 杨红震, 等. 河南不同立地条件下栓皮栎天然次生林群落生物量结构特征[J]. 河南农业大学学报, 2016, 50(5): 624-628. AN L B, BI H T, YANG H Z, et al. Structure characteristics of community biomass ofQuercusvariabilis natural secondary forest under different site conditions in Henan Province[J]. J Henan Agric Univ, 2016, 50(5): 624-628. DOI: 10.16445/j.cnki.1000-2340.2016.05.008.
[8]赵金龙, 王泺鑫, 韩海荣, 等. 辽河源不同龄组油松天然次生林生物量及空间分配特征[J]. 生态学报, 2014, 34(23): 7026-7037. ZHAO J L, WANG L X, HAN H R, et al. Biomass and spatial distribution characteristics ofPinustabulaeformisnatural secondary forest at different age groups in the Liaoheyuan Nature Reserve, Hebei Province[J]. Acta Ecol Sin, 2014, 34(23): 7026-7037. DOI:10.5846/stxb201303060357.
[9]申鑫, 曹林, 佘光辉. 高光谱与高空间分辨率遥感数据的亚热带森林生物量反演[J]. 遥感学报, 2016, 20(6): 1446-1460. SHEN X, CAO L, SHE G H. Subtropical forest biomass estimation based on hyperspectral and high-resolution remotely sensed data[J]. J Remote Sens, 2016, 20(6): 1446-1460. DOI:10.11834/jrs.20165210.
[10]LI Y C, LI M Y, LI C, et al. Forest aboveground biomass estimation using Landsat 8 and Sentinel-1A data with machine learning algorithms[J].Sci Rep, 2020, 10: 9952. DOI:10.1038/s41598-020-67024-3.
[11]HUANG W L, SUN G Q, DUBAYAH R, et al. Mapping biomass change after forest disturbance: applying LiDAR footprint-derived models at key map scales[J]. Remote Sens Environ, 2013, 134: 319-332. DOI:10.1016/j.rse.2013.03.017.
[12]MONTESANO P M, NELSON R F, DUBAYAH R O, et al. The uncertainty of biomass estimates from LiDAR and SAR across a boreal forest structure gradient[J]. Remote Sens Environ, 2014, 154: 398-407. DOI:10.1016/j.rse.2014.01.027.
[13]NAESSET E, GOBAKKEN T, SOLBERG S, et al. Model-assisted regional forest biomass estimation using LiDAR and InSAR as auxiliary data: a case study from a boreal forest area[J]. Remote Sens Environ, 2011, 115(12): 3599-3614. DOI:10.1016/j.rse.2011.08.021.
[14]HUDAK A T, STRAND E K, VIERLING L A, et al. Quantifying aboveground forest carbon pools and fluxes from repeat LiDAR surveys[J]. Remote Sens Environ, 2012, 123: 25-40. DOI:10.1016/j.rse.2012.02.023.
[15]PFLUGMACHER D, COHEN W B, KENNEDY R E. Using Landsat-derived disturbance history (1972-2010) to predict current forest structure[J]. Remote Sens Environ, 2012, 122: 146-165. DOI:10.1016/j.rse.2011.09.025.
[16]ZHAO K G, POPESCU S, NELSON R. Lidar remote sensing of forest biomass: a scale-invariant estimation approach using airborne lasers[J]. Remote Sens Environ, 2009, 113(1): 182-196. DOI:10.1016/j.rse.2008.09.009.
[17]ASNER G P, POWELL G V, MASCARO J, et al. High-resolution forest carbon stocks and emissions in the Amazon[J]. PNAS, 2010, 107(38): 16738-16742. DOI:10.1073/pnas.1004875107.
[18]CAO L, COOPS N C, INNES J L, et al. Estimation of forest biomass dynamics in subtropical forests using multi-temporal airborne LiDAR data[J]. Remote Sens Environ, 2016, 178: 158-171. DOI:10.1016/j.rse.2016.03.012.
[19]FERRAZ A, SAATCHI S, MALLET C, et al. Airborne lidar estimation of aboveground forest biomass in the absence of field inventory[J]. Remote Sens, 2016, 8(8): 653. DOI:10.3390/rs8080653.
[20]BROVKINA O, NOVOTNY J, CIENCIALA E, et al. Mapping forest aboveground biomass using airborne hyperspectral and LiDAR data in the mountainous conditions of Central Europe[J]. Ecol Eng, 2017, 100: 219-230. DOI:10.1016/j.ecoleng.2016.12.004.
[21]EDIRIWEERA S, PATHIRANA S, DANAHER T, et al. Estimating above-ground biomass by fusion of LiDAR and multispectral data in subtropical woody plant communities in topographically complex terrain in north-eastern Australia[J]. J For Res, 2014, 25(4): 761-771. DOI:10.1007/s11676-014-0485-7.
[22]郭艺歌, 王新云, 何杰, 等. 基于多源遥感数据的森林生物量估算模型研究[J]. 人民长江, 2016, 47(3): 17-22. GUO Y G, WANG X Y, HE J, et al. Study on forest biomass estimation model based on multisource remote sensing data[J]. Yangtze River, 2016, 47(3): 17-22. DOI:10.16232/j.cnki.1001-4179.2016.03.005.
[23]李明阳, 吴军, 余超, 等. 福建武夷山自然保护区森林碳储量遥感估测方法与空间分析[J]. 南京林业大学学报(自然科学版), 2014, 38(6): 6-10. LI M Y, WU J, YU C, et al. Estimation and spatial analysis of forest carbon stocks based on remote sensing technology in Wuyi Mountain National Reserve of Fujian Province[J]. J Nanjing For Univ (Nat Sci Ed), 2014, 38(6): 6-10. DOI:10.3969/j.issn.1000-2006.2014.06.002.
[24]潘磊, 孙玉军, 王轶夫, 等. 基于Sentinel-1和Sentinel-2数据的杉木林地上生物量估算[J]. 南京林业大学学报(自然科学版), 2020, 44(3): 149-156. PAN L, SUN Y J, WANG Y F, et al. Estimation of aboveground biomass in a Chinese fir(Cunninghamialanceolata)forest combining data of Sentinel-1 and Sentinel-2[J]. J Nanjing For Univ (Nat Sci Ed), 2020, 44(3): 149-156. DOI:10.3969/j.issn.1000-2006.201811012.
[25]TIAN X, LI Z Y, SU Z B, et al. Estimating montane forest above-ground biomass in the upper reaches of the Heihe River Basin using Landsat-TM data[J]. Int J Remote Sens, 2014, 35(21): 7339-7362. DOI:10.1080/01431161.2014.967888.
[26]FASSNACHT F E, HARTIG F, LATIFI H, et al. Importance of sample size, data type and prediction method for remote sensing-based estimations of aboveground forest biomass[J]. Remote Sens Environ, 2014, 154: 102-114. DOI:10.1016/j.rse.2014.07.028.
[27]蒙诗栎, 庞勇, 张钟军, 等. WorldView-2纹理的森林地上生物量反演[J]. 遥感学报, 2017, 21(5): 812-824. MENG S L, PANG Y, ZHANG Z J, et al. Estimation of aboveground biomass in a temperate forest using texture information from WorldView-2[J]. J Remote Sens, 2017, 21(5): 812-824. DOI:10.11834/jrs.20176083.
[28]SHAO Z F, ZHANG L J, WANG L. Stacked sparse autoencoder modeling using the synergy of airborne LiDAR and satellite optical and SAR data to map forest above-ground biomass[J]. IEEE J Sel Top Appl Earth Obs Remote Sens, 2017, 10(12): 5569-5582. DOI:10.1109/JSTARS.2017.2748341.
[29]ZHANG L J, SHAO Z F, LIU J C, et al. Deep learning based retrieval of forest aboveground biomass from combined LiDAR and landsat 8 data[J]. Remote Sens, 2019, 11(12): 1459. DOI:10.3390/rs11121459.
[30]董利虎. 黑龙江省主要树种相容性生物量模型研究[D]. 哈尔滨: 东北林业大学, 2012. DONG L H. Compatible biomass models of main tree species in Heilongjiang Province[D]. Harbin: Northeast Forestry University, 2012.
[31]ANDERSEN H E, MCGAUGHEY R J, REUTEBUCH S E. Estimating forest canopy fuel parameters using LIDAR data[J]. Remote Sens Environ, 2005, 94(4): 441-449. DOI:10.1016/j.rse.2004.10.013.
[32]LU D S, CHEN Q, WANG G X, et al. Aboveground forest biomass estimation with landsat and LiDAR data and uncertainty analysis of the estimates[J]. Int J For Res, 2012(2):1-16. DOI:10.1155/2012/436537.
[33]CHEN Q, LAURIN G V, BATTLES J J, et al. Integration of airborne lidar and vegetation types derived from aerial photography for mapping aboveground live biomass[J]. Remote Sens Environ, 2012, 121: 108-117. DOI:10.1016/j.rse.2012.01.021.
[34]LUO S Z, WANG C, XI X H, et al. Fusion of airborne LiDAR data and hyperspectral imagery for aboveground and belowground forest biomass estimation[J]. Ecol Indic, 2017, 73: 378-387. DOI:10.1016/j.ecolind.2016.10.001.
[35]徐婷, 曹林, 申鑫, 等. 基于机载激光雷达与Landsat 8 OLI数据的亚热带森林生物量估算[J]. 植物生态学报, 2015, 39(4): 309-321. XU T, CAO L, SHEN X, et al. Estimates of subtropical forest biomass based on airborne LiDAR and Landsat 8 OLI data[J]. Chin J Plant Ecol, 2015, 39(4): 309-321. DOI:10.17521/cjpe.2015.0030.
[36]曹霖, 彭道黎, 王雪军, 等. 应用Sentinel-2A卫星光谱与纹理信息的森林蓄积量估算[J]. 东北林业大学学报, 2018, 46(9): 54-58. CAO L, PENG D L, WANG X J, et al. Estimation of forest stock volume with spectral and textural information from the Sentinel-2A[J]. J Northeast For Univ, 2018, 46(9): 54-58. DOI:10.13759/j.cnki.dlxb.2018.09.012.
[37]ROUSE J W J, HAAS R H, SCHELL J A, et al. Monitoring ve-getation systems in the great plains with ERTS[EB/OL]. https://ntrs.nasa.gov/api/citations/19740022614/downloads/19740022614.pdf(accessedon 16 Apri 2021),1974.
[38]RICHARDSON A J, WIEGAND C L. Distinguishing vegetation from soil background information[J]. Photogramm Eng Remote Sens, 1977, 43(12): 1541-1552. DOI:10.1109/TGE.1977.294499.
[39]HUETE A, DIDAN K, MIURA T, et al. Overview of the radiometric and biophysical performance of the MODIS vegetation indices[J]. Remote Sens Environ, 2002, 83(1/2): 195-213. DOI:10.1016/S0034-4257(02)00096-2.
[40]JORDAN C F. Derivation of leaf-area index from quality of light on the forest floor[J]. Ecology, 1969, 50(4): 663-666. DOI:10.2307/1936256.
[42]RONDEAUX G, STEVEN M, BARET F. Optimization of soil-adjusted vegetation indices[J]. Remote Sens Environ, 1996, 55(2): 95-107. DOI:10.1016/0034-4257(95)00186-7.
[43]GITELSON A A, MERZLYAK M N. Remote estimation of chlorophyll content in higher plant leaves[J]. Int J Remote Sens, 1997, 18(12): 2691-2697. DOI:10.1080/014311697217558.
[44]GITELSON A A, GRITZ Y, MERZLYAK M N. Relationships between leaf chlorophyll content and spectral reflectance and algorithms for non-destructive chlorophyll assessment in higher plant leaves[J]. J Plant Physiol, 2003, 160(3): 271-282. DOI:10.1078/0176-1617-00887.
[45]谢福明, 舒清态, 字李, 等. 基于K-NN非参数模型的高山松生物量遥感估测研究[J]. 江西农业大学学报, 2018, 40(4): 743-750. XIE F M, SHU Q T, ZI L, et al. Remote sensing estimation ofPinusdensataaboveground biomass based on K-NN nonparametric model[J]. Acta Agric Univ Jiangxiensis, 2018, 40(4): 743-750. DOI:10.13836/j.jjau.2018094.
[46]GLEASON C J, IM J. Forest biomass estimation from airborne LiDAR data using machine learning approaches[J]. Remote Sens Environ, 2012, 125: 80-91. DOI:10.1016/j.rse.2012.07.006.
[47]PANDIT S, TSUYUKI S, DUBE T. Estimating above-ground biomass in sub-tropical buffer zone community forests, Nepal, using Sentinel-2 data[J]. Remote Sens, 2018, 10(4): 601. DOI:10.3390/rs10040601.
[48]STEPHEN A B, PHILIP H Y. The trouble withR2[J]. Journal of Parametrics, 2006, 25(1):87-114. DOI: 10.1080/10157891.2006.10462273.
[50]郑阳, 吴炳方, 张淼. Sentinel-2数据的冬小麦地上干生物量估算及评价[J]. 遥感学报, 2017, 21(2): 318-328. ZHENG Y, WU B F, ZHANG M. Estimating the above ground biomass of winter wheat using the Sentinel-2 data[J]. J Remote Sens, 2017, 21(2): 318-328. DOI:10.11834/jrs.20176269.
[51]雷相东. 机器学习算法在森林生长收获预估中的应用[J]. 北京林业大学学报, 2019, 41(12): 23-36. LEI X D. Applications of machine learning algorithms in forest growth and yield prediction[J]. J Beijing For Univ, 2019, 41(12): 23-36. DOI:10.12171/j.1000-1522.20190356.
[52]孙雪莲. 基于Landsat 8-OLI的香格里拉高山松林生物量遥感估测模型研究[D]. 昆明: 西南林业大学,2016. SUN X L. Biomass estimation model ofPinusdensataforests in Shangri-La City based on Landsat 8-OLI by remote sensing[D]. Kunming: Southwest Forestry University, 2016.
[53]HAME T, RAUSTE Y, ANTROPOV O, et al. Improved mapping of tropical forests with optical and SAR imagery, part II: above ground biomass estimation[J]. IEEE J Sel Top Appl Earth Obs Remote Sens, 2013, 6(1): 92-101. DOI:10.1109/JSTARS.2013.2241020.

