高通量测序分析不同采集保存温度下甘南牧区牦牛酸奶真菌多样性
张文齐,麻和平,刘彩云,王 洁,彭章普,邵建宁,
(1.甘肃省科学院生物研究所 甘肃省微生物资源开发利用重点实验室,甘肃兰州 730000;2.甘肃省科学院,甘肃兰州 730000)
传统发酵乳制品制备多采用自然发酵,原料的不同、独特的制备方法和环境气温的差异等造就了传统发酵乳制品微生物群落的丰富性和多样性[1−3]。近年来,有关传统发酵乳制品微生物多样性研究,既有传统的分离培养研究,也有高通量测序、宏基因组学分析等非培养研究[4−5]。利用传统的分离技术从样品中获得的微生物,代表具体培养条件所能分离到的微生物,无法分离到样品中所有微生物,不能完整地揭示样品的菌群组成[6−7]。高通量测序、宏基因组学分析等非培养研究无需分离培养微生物,能直接反映样品中微生物群落构成和多样性[8−11]。
目前,对传统发酵乳制品中参与发酵微生物的分离和菌群多样性相关研究较多[12−14],然而,通过高通量测序分析比较不同采集保存温度下的传统发酵乳制品微生物多样性,优化传统发酵乳制品采集保存温度却鲜有报道。Sun等[15]通过高通量焦磷酸测序对17 个自然发酵乳制品中的细菌和真菌群落多样性进行研究,所有样品中细菌属47 个,真菌属43 个;王远微等[16]对川西北部分牧区的10 份传统发酵牦牛酸奶样品进行酵母菌的分离,通过常规形态特征和26S rRNA基因测序分析鉴定出16 株马克斯克鲁维酵母菌;袁凤霞等[17]将前期从新疆、西藏和青海等地区传统自制乳制品中分离的27 株酵母菌株分别与保加利亚乳杆菌发酵酸乳,筛选优良共生酵母菌株。还有研究表明传统发酵奶制品在发酵过程中乳酸菌和酵母菌的共同作用使得发酵后的奶制品具有独特的风味和口感[18−20]。
本研究以常温、冰袋、干冰和液氮不同保存条件下采集的甘南牧区牧民自制牦牛酸奶样品为研究对象,采用Illumina NovaSeq高通量测序技术,对牦牛酸奶扩增真菌ITS2 区进行测序,通过分析比较不同采集保存温度下样品的真菌群落结构和真菌多样性,探究基于高通量测序不同采集保存温度对传统发酵乳制品微生物真菌多样性分析的影响,为传统发酵乳制品采样条件优化提供理论指导,并为科学保护、开发与利用传统发酵乳制品中优良微生物资源提供参考依据。
1 材料与方法
1.1 材料与仪器
牦牛酸奶样品 20 份甘南牧区牦牛酸奶样品的采样具体信息见表1;QIAamp Fast DNA Stool Mini Kit、QIAquick Gel Extraction Kit德国QIAGEN公司;PowerSoil DNA Isolation Kit美国MOBIO公司;KAPA HiFi Hotstart ReadyMix Kit美国KAPA Biosystems公司;2× Phanta Master Mix南京诺唯赞生物科技股份有限公司;AxyPrep DNA凝胶回收试剂盒 美国AXYGEN公司。
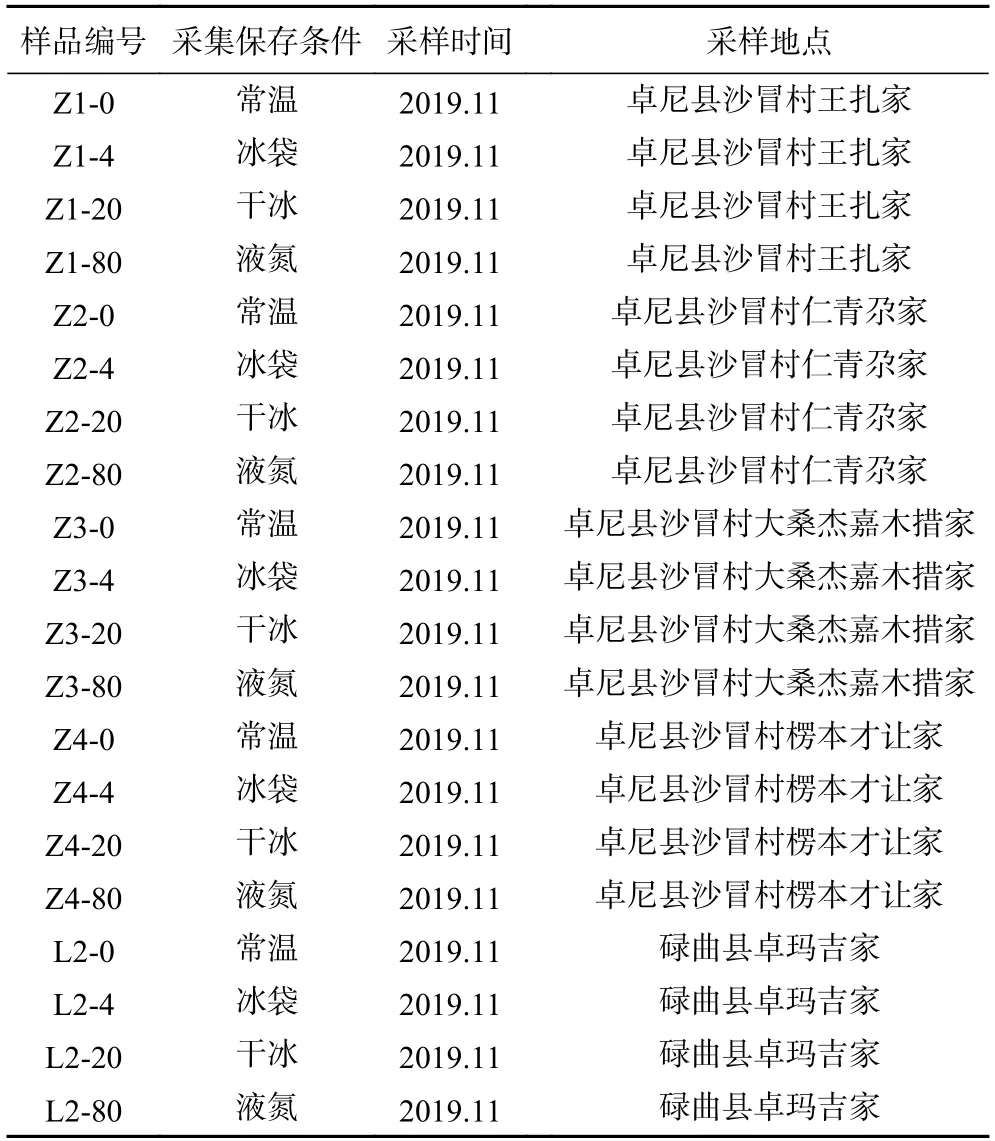
表1 甘南牧区牦牛酸奶采样信息Table 1 Information of yak yogurt samples collected from Gannan pastoral area
5424R低温高速离心机 德国Eppendorf公司;VDRTEX-5 涡旋混合器 海门市其林贝尔仪器制造有限公司;Tanon 3500 凝胶成像系统 上海天能科技有限公司;DYY-6C电泳仪 上海天能科技有限公司;ND-2000C微量紫外分光光度计 美国NanoDrop公司;Applied Biosystems VeritiPro PCR仪 美国Life Technologies公司。
1.2 实验方法
1.2.1 牦牛酸奶样品的采集 实地采集牧民自制牦牛酸奶,每份样品采集4 管,每管30 mL,密封后标记样品号,分别置于常温、冰袋、干冰和液氮容器中保存,运回实验室后,分别于常温、4、−20 ℃和−80℃保存备用。样品采集自甘肃省甘南藏族自治州卓尼县(Z)、碌曲县(L),常温采集实验室常温保存样品分组为Z-0,冰袋采集实验室4 ℃保存样品分组为Z-4,干冰采集实验室−20 ℃保存样品分组为Z-20,液氮采集实验室于−80 ℃保存样品分组为Z-80。
1.2.2 牦牛酸奶样品中总DNA提取 取2 mL牦牛酸奶样品,采用试剂盒QIAamp Fast DNA Stool Mini Kit,按照说明书的要求提取牦牛酸奶的总DNA,利用微量紫外分光光度计和1%琼脂糖凝胶电泳,进行总DNA完整性和浓度质检。
1.2.3 PCR扩增与测序 使用KAPA HiFi Hotstart ReadyMix PCR kit将提取到的总DNA作为模板进行PCR扩增,采用引物为ITS3 GCATCGATGAAGAACGCAGC(F2045)和ITS4 TCCTCCGCTTATT-GATATGC(R2390)扩增真菌ITS2 区(引物中已带特定barcode),PCR反应条件为:95 ℃ 3 min;98 ℃20 s,58 ℃ 15 s,72 ℃ 20 s,30 个循环;72 ℃ 5 min。PCR反应体系为:2×KAPA Library Amplification Ready Mix 15 μL,引物(10 μmol/L)各为1 μL,模板DNA为50 ng,加ddH2O补齐至30 μL。
扩增PCR产物用2%琼脂糖凝胶电泳检测,使用AxyPrep DNA凝胶提取试剂盒切胶回收PCR产物,文库质检合格后,使用Qubit 2.0 进行文库定量,并根据每个样品的数据量要求,进行相应比例的混合,采用Illumina NovaSeq PE250 进行上机测序,Illumina NovaSeq高通量测序由上海锐翌生物科技有限公司完成。
1.2.4 数据处理 ITS2 序列被控制在220~500 bp之间,保证每条reads平均质量值不低于20,且含N碱基数不超过3 个。将序列完全一样的Clean Reads根据其丰度大小进行排序,将其中的Singletons过滤掉。使用UPARSE(http://drive5.com/uparse/)根据97%相似度进行OTU聚类,并使用Userach(版本7.0.1090)鉴定和移除嵌合体序列。每个OTU都有一个代表性的序列,使用RDP数据库(http://rdp.cme.msu.edu/),置信度阈值设置为0.8,利用RDP Classifer(http://rdp.cme.msu.edu/)将每个代表性序列进行物种注释。不同样本对应的Reads数量差距较大,为避免因样品数据大小不同而造成分析时的偏差,在样品达到足够的测序深度的情况下,对每个样品进行随机抽平处理。测序深度使用Alpha多样性中的coverage指数来衡量,OTU profiling table、Alpha多样性指数和Beta多样性指数通过QIIME(version 1.9.1)的python脚本实现。
2 结果与分析
2.1 样品序列信息及OTU分析
对从采集至实验室8 d后的牦牛酸奶样品提取总DNA,经质检合格,通过样品真菌ITS2 区测序,20 份牦牛酸奶样品质控过滤后得到的有效序列为728022 条。采用稀释曲线评估各样品测序量是否足够,结果如图1 所示,由稀释曲线可知,随着序列数的增加,chao1 指数趋于平缓,说明本次样本测序的深度足够,每个样品随机抽取31495 条reads,抽平足以反映各样品中真菌物种多样性,测序量满足后续生物信息学分析的要求。各个样品及分组信息,质控后序列数,可观测的物种数量,chao1、shannon、simpson和coverage指数见表2,所有样本的测序深度均大于0.99,说明样本测序结果可以反映样品的真实情况。所有序列按97%的相似度进行OTU聚类,从20 份样品中共检测到702 个OTU,隶属于6 门、24 纲、61 目、126 科、198 属。

图1 所有样品稀释曲线图Fig.1 Rarefaction curve of all samples
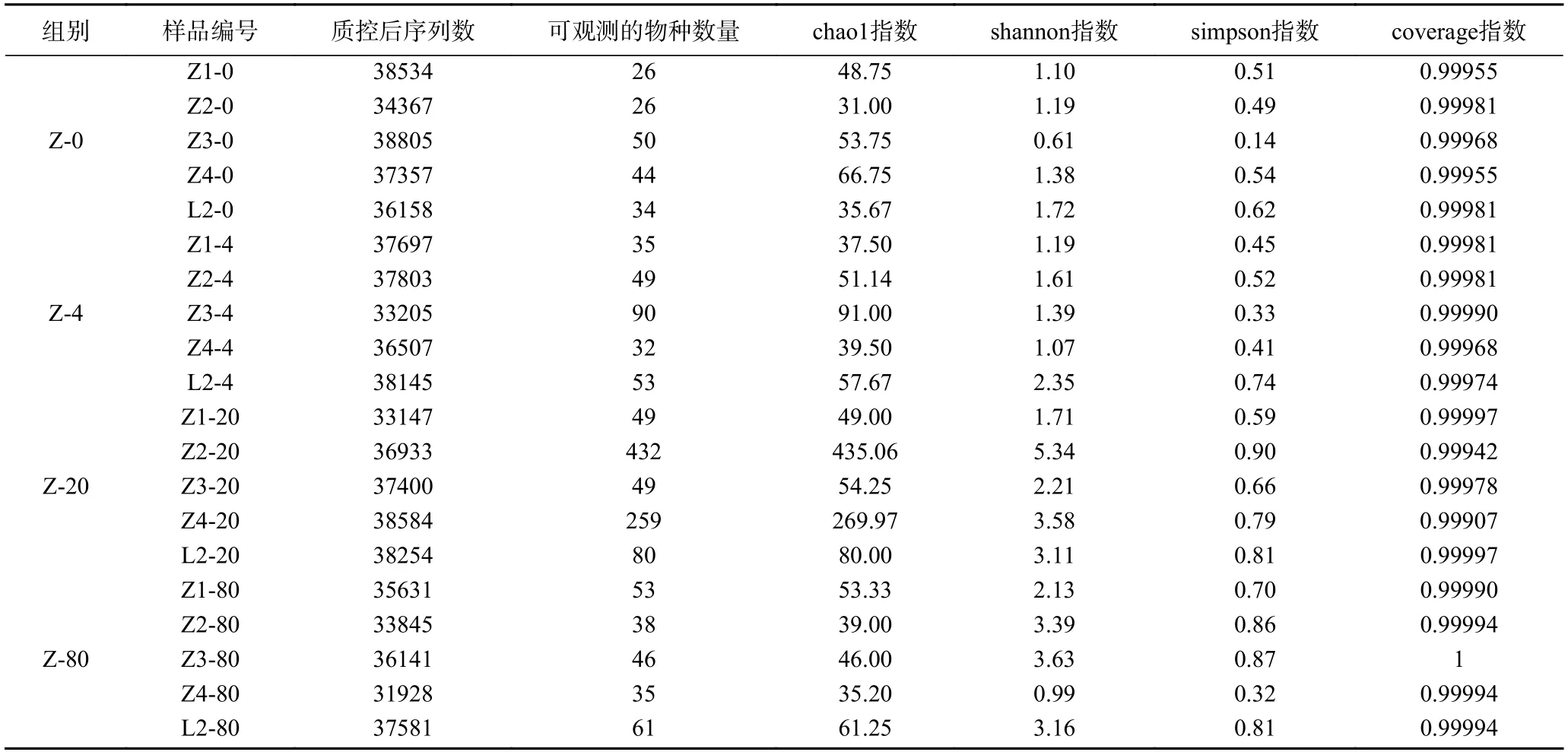
表2 20 份牦牛酸奶样品的序列信息及α 多样性指数Table 2 Sequence information and α diversity of 20 yak yoghurt samples
2.2 不同采集保存温度下样品真菌群落结构的比较
2.2.1 组间真菌群落结构比较 Z2-20、Z4-20 样品可观测的物种数量分别为432、259,明显高于其同一牦牛酸奶不同采集保存温度下的其它样品,但在干冰采集实验室−20 ℃保存的其它样品Z1-20、Z3-20、L2-20 可观测的物种数量未出现明显高于同一牦牛酸奶不同采集保存温度下的其它样品,故排除Z2-20、Z4-20 异常样本,对不同采集保存温度样品组间α多样性指数进行了kw检验分析,结果见表3,组间α多样性各指数均无显著差异(P>0.05)。对不同采集保存温度样品的真菌群落进行了主成分(PCA)分析,PCA可以初步反映出不同样品可能表现出分散和聚集的分布情况,结果见图2,常温采集组(Z-0)、冰袋采集组(Z-4)、干冰采集组(Z-20)、液氮采集组(Z-80)的样品分布均较为聚集,从而可以判断出不同采集保存温度样品真菌群落组成差异不大,群落组成较为接近。

表3 组间样品α 多样性指数比较Table 3 Comparison of α diversity indices among groups
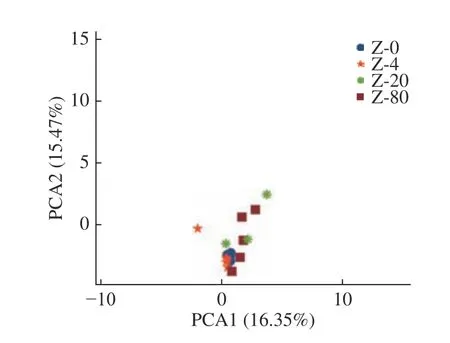
图2 各组样品的PCA分析Fig.2 PCA analysis of the different groups
2.2.2 不同采集保存温度样品门水平物种丰度分析 在真菌门水平,样品中一共鉴定出6 个门水平物种,图3 显示不同采集保存温度样品组在门水平真菌相对丰度物种,主要5 个门,其余相对丰度太低的合并为other。常温采集组的牦牛酸奶真菌ITS2 区测序,共检测出4 个真菌菌门,其中子囊菌门(Ascomycota)平均丰度值为99.25%,担子菌门(Basidiomycota)平均丰度值为0.73%,接合菌门(Zygomycota)平均丰度值为0.0052%,未分类菌门平均丰度值为0.010%。冰袋采集组的牦牛酸奶经高通量测序共检测出5 个真菌菌门,其中子囊菌门(Ascomycota)平均丰度值为 98.39%,担子菌门(Basidiomycota)平均丰度值为1.26%,接合菌门(Zygomycota)平均丰度值为0.043%,壶菌门(Chytridiomycota)平均丰度值为0.022%,未分类菌门平均丰度值为0.28%。干冰采集组的牦牛酸奶经高通量测序共检测出5 个真菌菌门,其中子囊菌门(Ascomycota)平均丰度值为 96.04%,担子菌门(Basidiomycota)平均丰度值为3.28%,接合菌门(Zygomycota)平均丰度值为 0.19%,壶菌门(Chytridiomycota)平均丰度值为0.057%,未分类菌门平均丰度值为0.44%。液氮采集组的牦牛酸奶经高通量测序共检测出5 个真菌菌门,其中子囊菌门(Ascomycota)平均丰度值为 82.09%,担子菌门(Basidiomycota)平均丰度值为16.90%,接合菌门(Zygomycota)平均丰度值为 0.36%,壶菌门(Chytridiomycota)平均丰度值为0.013%,未分类菌门平均丰度值为0.64%。ITS2 区测序分析的甘南牧区牦牛酸奶样品中子囊菌门(Ascomycota)丰度占总数的82.09%~99.25%,为样品中真菌优势菌门。通过门水平真菌群落结构分析比较,不同采集保存温度样品组间真菌群落组成基本相同,仅组成比例不同。

图3 门分类水平真菌相对丰度Fig.3 Relative abundance of fungi at phylum level
2.2.3 不同采集保存温度样品属水平物种丰度分析 在真菌属水平,图4 显示了不同采集保存温度牦牛酸奶样品在属水平真菌相对丰度前20 的物种,其它物种合并为other。常温采集组的牦牛酸奶相对丰度大于1%的菌属包括酵母菌属(Saccharomyces,63.49%)、克鲁维酵母菌属(Kluyveromyces,31.89%)、毕赤酵母属(Pichia,3.39%)。冰袋采集组的牦牛酸奶相对丰度大于1%的菌属包括酵母菌属(Saccharomyces,61.59%)、克鲁维酵母菌属(Kluyveromyces,16.08%)、毕赤酵母属(Pichia,10.01%)、假丝酵母属(Candida,9.48%)。干冰采集组的牦牛酸奶相对丰度大于1%的菌属包括酵母菌属(Saccharomyces,23.98%)、克鲁维酵母菌属(Kluyveromyces,27.35%)、毕赤酵母属(Pichia,35.21%)、Davidiella属(3.12%)、马拉色氏霉菌属(Malassezia,1.76%)。液氮采集组的牦牛酸奶相对丰度大于1%的菌属包括酵母菌属(Saccharomyces,37.58%)、克鲁维酵母菌属(Kluyveromyces,22.68%)、毕赤酵母属(Pichia,13.91%)、Davidiella属(2.53%),马拉色氏霉菌属(Malassezia,12.28%)。测序分析的甘南牧区牦牛酸奶样品中酵母菌属(Saccharomyces)丰度占总数的23.98%~63.49%,克鲁维酵母菌属(Kluyveromyces)丰度占总数的16.08%~31.89%,毕赤酵母属(Pichia)丰度占总数的3.39%~35.21%,是相对丰度较高的真菌属,为样品中的优势菌属。通过属水平真菌群落结构分析比较,不同采集保存温度下牦牛酸奶样品组间真菌群落组成相似,仅组成比例不同。
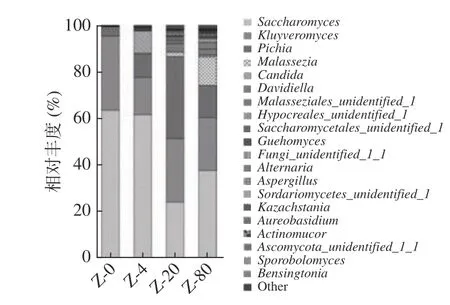
图4 属水平前20 名真菌的相对丰度Fig.4 Relative abundance of the top 20 fungi at genus level
2.2.4 组间物种差异分析 属水平的组间物种差异分析,采用秩和检验得到不同采集保存温度下牦牛酸奶样品组间的差异细菌属共2 个,其中液氮采集组,Davidiella、Hypocreales unidentified2 个菌属显著高于其它组别的真菌群落菌属(P<0.05),结果见表4。为了直观展示各组及各样品间真菌属差异,对差异真菌属进行热图(Heatmap)分析和主成分分析(PCA),Heatmap图是以颜色梯度来表征二维矩阵或表格中的数据大小,反映不同分组或样本在各分类水平上群落组成的相似性与差异性,由图5 可知,液氮采集组样品的群落组成和其它样品组差异较大。PCA图能够直观地把样品之间相似或差异的程度呈现在二维坐标系上,在图6 中常温采集组和冰袋采集组样品表现出聚集的分布,表明常温、冰袋采集组样品的群落组成更为接近;干冰采集组和液氮采集组样品表现出聚集的分布,表明干冰、液氮采集组样品的群落更为接近。
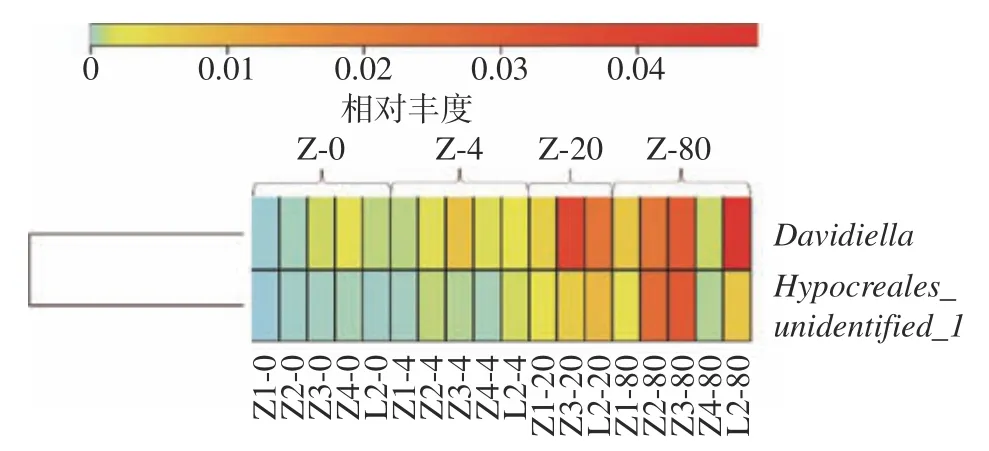
图5 属水平差异物种的HeatmapFig.5 Heatmap of different genus among groups

图6 属水平差异物种的PCA分析Fig.6 PCA analysis of different genus among groups
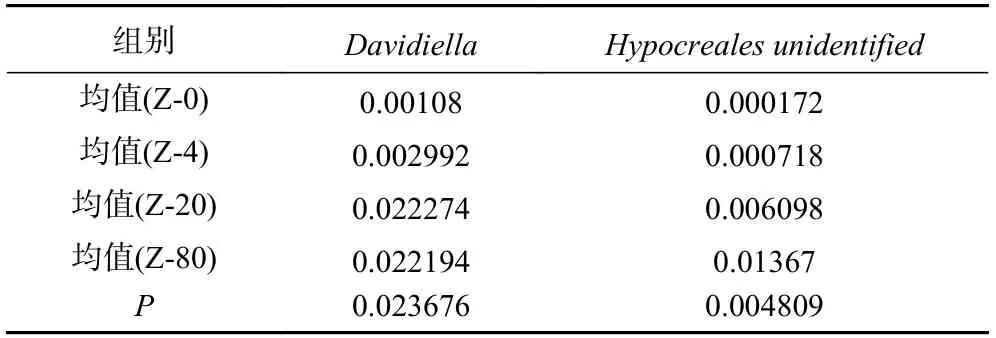
表4 差异显著物种Table 4 Significantly different genus among groups
3 讨论与结论
牦牛酸奶是由牦牛奶通过传统发酵方法制得,是经过长期自然驯化优良食品微生物的天然载体,含有极具开发利用价值的微生物资源[21]。本研究采用Illumina NovaSeq高通量测序技术测序甘南牧区牦牛酸奶样品真菌ITS2 区,分析牦牛酸奶中真菌菌群组成,比较不同采集保存温度样品真菌多样性,对20 份甘南牧区牦牛酸奶样品高通量测序结果分析表明,子囊菌门(Ascomycota)为样品中真菌优势菌群门,酵母菌属(Saccharomyces)、克鲁维酵母菌属(Kluyveromyces)、毕赤酵母属(Pichia)为优势菌属。Zhang等[22]报道青海地区牦牛酸奶与其它发酵酸奶相比含有更多的乳酸菌和酵母菌,其中青海西北部的高原牦牛酸奶和青海东部的环湖牦牛酸奶中乳酸菌是优势菌,而青海南部的环湖牦牛酸奶中酵母菌是优势菌。Wu等[23]研究发现,3 种乳酸菌以及5 种酵母菌是西藏不同海拔地区牦牛酸奶中主要发酵菌群。孙志宏等[24]运用宏基因组学技术对西藏地区自然发酵乳中微生物多样性进行分析,结果表明在真菌门水平上子囊菌门(Ascomycota)为最优势菌门,在属水平上优势菌属为毕赤酵母属(Pichia)、耐碱酵母属(Galactomyces)、酵母菌属(Saccharomyces)。乌仁图雅[25]、张冬蕾[26]、杨彦荣[27]通过焦磷酸测序技术对传统发酵乳制品酸牦牛奶、酸牛奶进行了真菌多样性研究,结果表明,酸牦牛奶、酸牛奶中真菌优势菌门均为子囊菌门。从以上研究结果中可以看出牦牛乳制品中的真菌菌群尽管比较复杂,但是都以酵母菌为主,只是优势的酵母菌的种类有所不同,本次甘南牧区牦牛酸奶样品真菌菌群组成分析结果与上述学者研究结论较为一致。
采用Illumina高通量测序技术分析比较了4 种不同采集保存温度下牦牛酸奶样品真菌多样性。目前,尚未见不同采集保存温度对传统发酵乳制品微生物多样性分析影响的报道,呼斯楞等[28]在内蒙古呼伦贝尔地区传统发酵乳中乳酸菌的多样性分析中,样品采集采用低温保存,带回实验室于−80 ℃超低温保藏;陈芝兰等[29]在西藏地区传统发酵乳中乳酸菌多样性及微生物数量分析中,将牧民自然发酵制作的酸奶放入冰盒内保存;刘振东等[30]在西藏不同产区曲拉细菌群落结构的比较分析中将采集样品放入冰盒保存并立即运送到实验室,置于−80 ℃冰箱中备用;张敏等[31]在基于16S rDNA高通量测序方法比较新疆西北部地区乳品中微生物的多样性中,将采集样品放入车载冰箱运回实验室,在−80 ℃超低温冰箱保存;马江等[32]在甘南牦牛曲拉中真菌群落结构研究中,将样品置于冷藏箱中,运输至实验室。以上乳制品微生物多样性研究采样均以低温采集为主。在未来研究中,为了更大可能的获得牦牛酸奶中不同种类的微生物多样性信息和定向的分离出目标微生物,可在借鉴已有最新测序技术在传统发酵乳制品微生物基因组和生物多样性中的研究结果,进一步优化不同的采样方法与分离策略,更好地开展传统发酵乳制品中优良微生物资源的保护与发掘研究工作。
采用Illumina NovaSeq高通量测序技术,对4 种不同保存温度条件下采集自甘南牧区20 份牦牛酸奶样品真菌ITS2 区进行测序,经OTU聚类分析,采集的甘南牧区牦牛酸奶样品中的真菌分属于6 门、24 纲、61 目、126 科、198 属。子囊菌门(Ascomycota)在采集牦牛酸奶样本真菌中占比最高,其丰度值变化范围为82.09%~99.25%,是20 份样品中的真菌优势菌门。酵母菌属(Saccharomyces)丰度值变化范围为23.98%~63.49%,克鲁维酵母菌属(Kluyveromyces)丰度值变化范围为16.08%~31.89%,毕赤酵母属(Pichia)丰度值变化范围为3.39%~35.21%,是样品中的真菌优势菌属。基于Illumina NovaSeq高通量测序分析比较,常温、冰袋、干冰和液氮4 种不同保存温度条件下采集自甘南牧区牦牛酸奶真菌多样性之间无显著差异,真菌群落组成基本相同,仅组成比例不同。

