circ-SFMBT2通过miR-224-5p/USP3轴对肝癌细胞增殖及迁移能力的影响*
伍玉海, 戴大飞, 叶蕊, 于北北, 张书军, 陈晓鹏***
(1.皖南医学院 研究生院, 安徽 芜湖 241002; 2.皖南医学院第一附属医院弋矶山医院 肝胆一科, 安徽 芜湖 241001)
目前越来越多的研究发现非编码RNA(non-coding RNA,ncRNA)在对疾病的调控中发挥重要作用,其中包括环状RNA( circular RNA,circRNA)和微小RNA(microRNA,miRNA)[1]。circRNA是通过部分基因外显子环化而成的,由于没有3′-尾巴和5′-帽子结构,进而避免了RNA酶消化,可稳定存在于细胞内[2],因此circRNA可被用作肿瘤的诊断探针[3]。研究发现circRNA可通过靶向吸附miRNA来调控基因表达,在癌症有关的各种生物学过程中充当重要调节剂,包括细胞增殖、侵袭及转移,进而影响疾病进程[4-6]。本研究circRNA名为hsa_circ_0017639(Geo数据库中的GSE78092),hsa_circ_0017639源自基因SFMBT2(scm-like with four mbt domains 2),因此被命名为circ-SFMBT2。既往研究发现circ-SFMBT2在胃癌组织中表达上调,沉默circ-SFMBT2会显著抑制胃癌细胞的增殖[7],circ-SFMBT2通过mirR-182-5p/MTSS1途径抑制神经胶质瘤细胞的增殖与转移[8],circ-SFMBT2在众多circRNA中属于一个研究较新的基因,在其他肿瘤等疾病中的作用及机制尚不明确。肝细胞癌(hepatocellular carcinoma,HCC)是世界上第5大常见的癌症[9],circ-SFMBT2在肝癌发生发展的过程中具有研究价值。本研究利用实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction ,qRT-PCR)发现肝癌细胞高表达circ-SFMBT2,并进一步探究其对肝癌细胞侵袭和迁移的影响以及可能的调控机制,为肝癌治疗提供潜在治疗靶点。
1 材料与方法
1.1 材料
1.1.1细胞来源 肝癌细胞系HepG2、SMMC7721、Huh-7、Sk-Hep-1及人正常肝细胞细胞系LO2购于中国科学院细胞库。
1.1.2主要试剂与仪器 胎牛血清、氨基酸葡萄糖培养基(dulbecco′s modified eagle medium,DMEM)、Lipo2000转染试剂及OptiMEM低血清培养基(美国Gibco),circ-SFMBT2干扰载体和泛素特异性蛋白酶3(Ubiquitin-specific protease 3,USP3)表达载体(上海吉玛基因生物),RNA TRIzol提取试剂盒(美国Invitrogen),pTRUEscript the First Strand cDNA Synthesis Kit和2×SYBR Green qPCR Mix(北京Aidlab),细胞增殖检测(cell counting kit-8,CCK-8)试剂盒(上海碧云天),Transwell小室(美国Corning),Cy5-标记的circ-SFMBT2探针(上海生工合成),qRT-PCR仪(瑞士Roche)。
1.2 方法
1.2.1细胞培养与转染 肝癌细胞系HepG2、SMMC7721、Huh-7、Sk-Hep-1及人正常肝细胞LO2置于含10%胎牛血清DMEM培养基,37 ℃的5%CO2细胞培养箱中培养;将circ-SFMBT2干扰载体、circ-SFMBT2 NC载体、miR-224-5p抑制剂、USP3过表达载体及miR-224-5p模拟物按照Lipo-2000转染试剂说明书进行转染,转染后分别构建circ-SFMBT2干扰组(si-circ-SFMBT2组)、对照组(circ-SFMBT2 NC,NC组),在si-circ-SFMBT2干扰组的成功模型后继续转染miR-224-5p抑制剂构建miR-224-5p抑制组(si-circ-SFMBT2+inhibitor组)、在si-circ-SFMBT2干扰组的成功模型后继续转染USP3过表达构建USP3过表达组(si-circ-SFMBT2+USP3组),HepG2细胞转染miR-224-5p过表达模拟物成功构建miR-224-5p mimic组(mimic组),在miR-224-5p mimic组成功模型后继续转染USP3过表达载体构建 mimic+USP3组。具体方法是将转染片段(50 ng)与Lipo2000混合后加OptiMEM培养基培养6 h,换正常DMEM培养基继续培养,转染48 h,细胞经qRT-PCR实验检测后可进行后续实验。
1.2.2qRT-PCR 细胞总RNA利用TRIzol试剂进行抽提,利用pTRUEscript the First Strand cDNA Synthesis Kit 进行互补DNA(complementary DNA,cDNA)合成,利用2×SYBR Green qPCR Mix进行qRT-PCR检测;利用2-ΔΔCt计算方法对结果进行统计分析。实验重复3次。引物见表1。

表1 目的基因及阴性对照基因引物序列
1.2.3生物信息学分析 circRNA和miRNA之间的靶向作用关系通过Circular RNA Interactome网站进行预测,miRNAs和信使RNA(messger RNAs,mRNAs)之间的靶向作用关系利用TargetScan (http://www.targetscan.org/) 网站进行预测。
1.2.4荧光原位杂交技术和双荧光素实验 荧光原位杂交(fluorescence in situ hybridization,FISH)分析Cy5-标记的circ-SFMBT2探针(5′-CTCCTGCGTCGGTGACTAAGCAATCAAAGAGAAGTAC-3′-biotin)由上海生工合成,细胞核用4′,6-二脒基-2-苯基吲哚(4′,6-diamidino-2-phenylindole,DAPI)染色。人正常肝细胞细胞系LO2标记为Normal组,肝癌细胞HepG2细胞株标记为HCC组。该实验按照说明书要求做,观察并记录荧光结果。
1.2.5CCK-8 取“1.2.1”项下6组HepG2细胞利用CCK-8试剂盒进行增殖检测。含有2 000细胞的DMEM培养基100 μL加96孔板,培养0、24、48及72 h,吸弃培养基,分别替换成含有10%CCK-8试剂的DMEM培养基孵育1 h,酶标仪上于 450 nm下测定各组HepG2细胞吸光值(optical density,OD)。
1.2.6克隆斑点形成实验 取“1.2.1”项下6组HepG2细胞按200个细胞/孔密度接种于6孔板,培养10 d,4%多聚甲醛固定20 min,0.1%结晶紫染色5 min,拍照统计分析。
1.2.7细胞迁移分析 Transwell上室种无血清的DMEM培养基200 μL,其中分别含“1.2.1”项下6组HepG2细胞1×105个,下室加含15%胎牛血清的DMEM培养基500 μL,置于37 ℃的5%CO2细胞培养箱培养24 h,4%多聚甲醛固定20 min,0.1%结晶紫染色5 min,拍照统计分析。
1.3 统计学分析
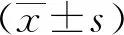
2 结果
2.1 circ-SFMBT2的表达
结果显示,与LO2细胞比较,Huh-7、Sk-Hep-1、SMMC7721及HepG2细胞中circ-SFMBT2表达均上调,其中HepG2细胞中表达上调最为明显(P<0.05),因此选择HepG2细胞作为后续研究对象。见图1。
注:(1)与LO2比较,P<0.05。
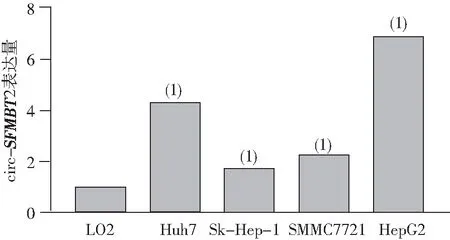
2.2 circ-SFMBT2表达和亚细胞定位检测
结果表明,HCC组细胞circ-SFMBT2表达较Normal组上调,且circ-SFMBT2主要定位在细胞质中。见图2。
Normal组 HCC组
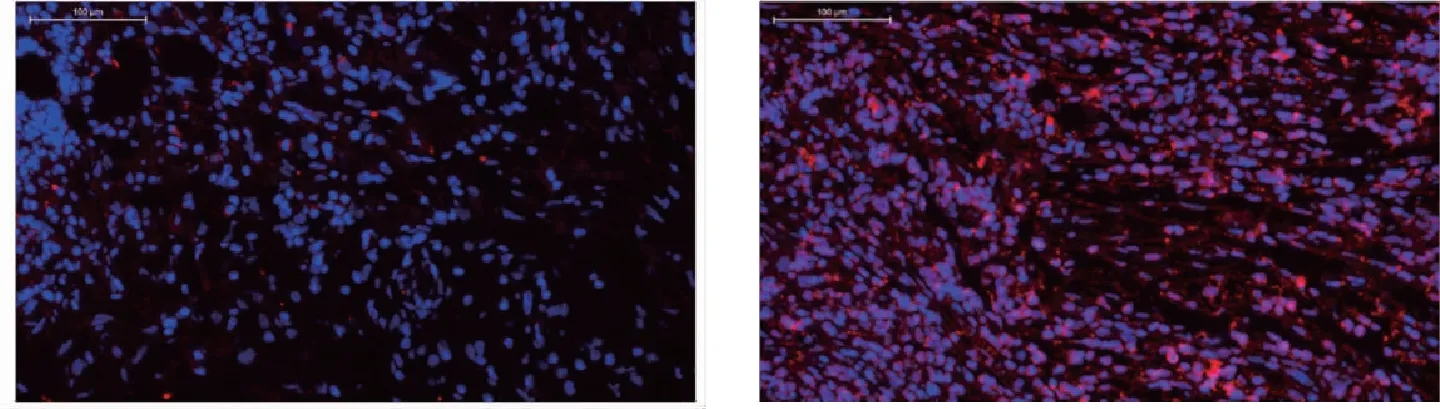
2.3 干扰circ-SFMBT2表达后肝癌细胞增殖和迁移能力
qRT-PCR检测结果表明,与NC组比较,si-circ-SFMBT2组 HepG2细胞 circ-SFMBT2表达下调(P<0.05);转染circ-SFMBT2干扰载体后,CCK-8和克隆斑点形成实验检测结果显示,与NC组比较,circ-SFMBT2干扰组HepG2细胞增殖能力降低(P<0.05);Transwell实验检测结果显示,与NC组比较,circ-SFMBT2干扰组HepG2细胞迁移能力降低(P<0.05)。见图3。
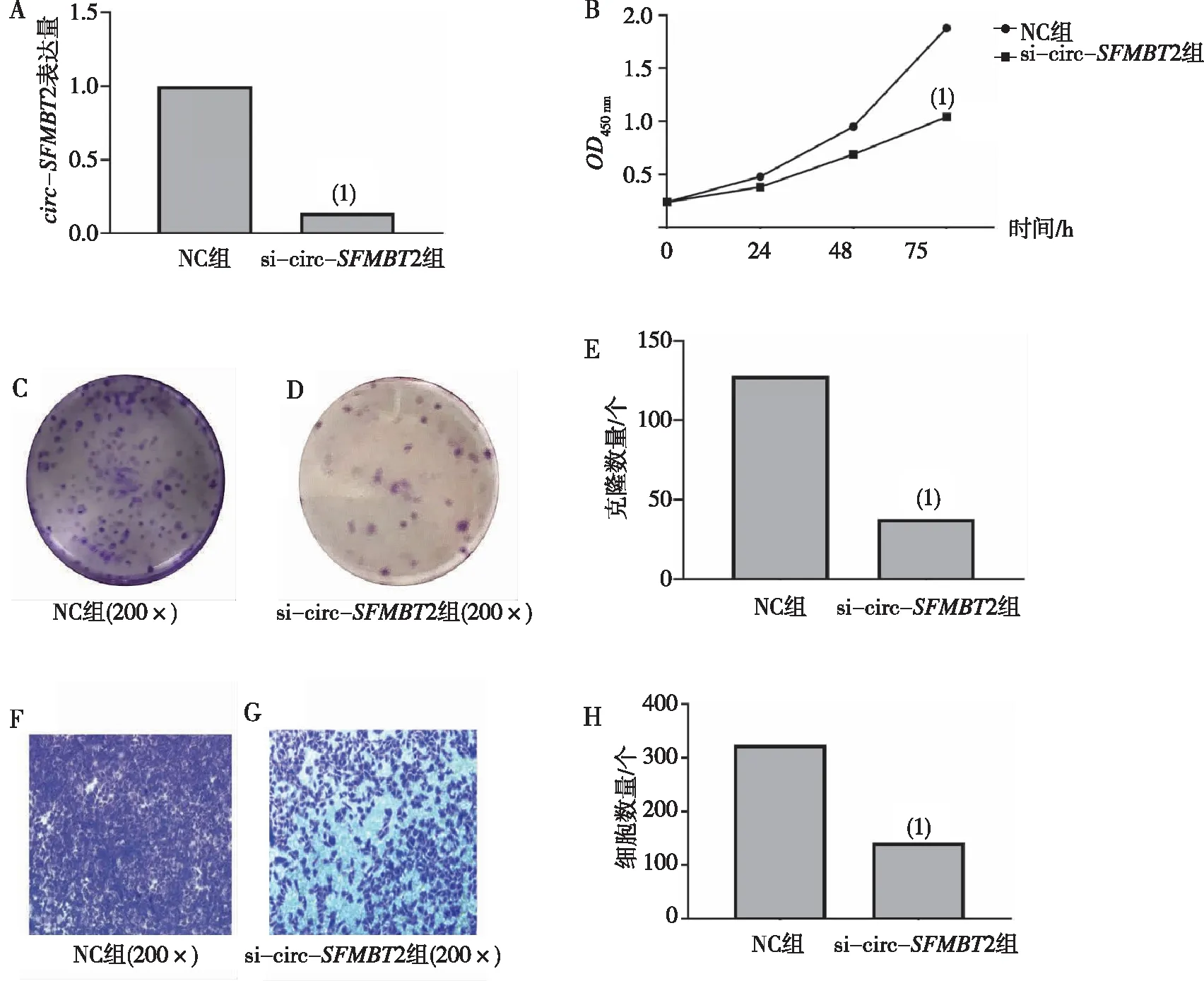
注:A为转染后HepG2细胞circ-SFMBT2的表达,B为CCK-8的结果,C~E分别为 NC组和si-circ-SFMBT2组细胞平板克隆实验形态学和定量结果,F~H分别为Transwell法检测细胞迁移能力形态学和定量结果;(1)与NC组比较,P<0.05。
2.4 双荧光素实验
野生型circ-SFMBT2载体上荧光活力检测显示,转染miR-224-5p过表达mimic片段的293T细胞相较于转染miR-224-5p NC片段的293T细胞荧光活力下降(P<0.05);USP3-3′UTR野生型荧光素报告载体上,转染miR-224-5p过表达mimic片段的293T细胞相较于转染miR-224-5p NC片段的293T细胞荧光活力下降,差异有统计学意义(P<0.05)。见图4。
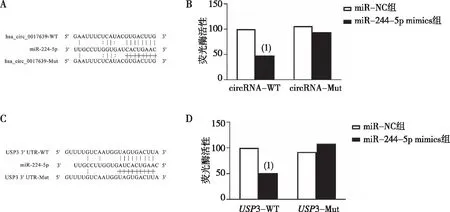
注:A为circ-SFMBT2荧光载体序列和miR-224-5p转染片段序列,B为circ-SFMBT2载体上双荧光实验定量结果,C为USP3-3′UTR荧光载体序列和miR-224-5p转染片段序列,D为USP3-3′UTR载体上双荧光实验定量结果;(1)与miR-NC组比较,P<0.05。
2.5 细胞转染实验和Transwell实验
qRT-PCR检测结果显示,circ-SFMBT2干扰组(si-circ-SFMBT2组)、miR-224-5p干扰组(si-circ-SFMBT2+inhibitor组)及USP3过表达组(si-circ-SFMBT2+USP3组)HepG2细胞circ-SFMBT2的表达均较NC组下调(P<0.05);与NC组比较,miR-224-5p干扰组HepG2细胞的miR-224-5p表达下调(P<0.05),circ-SFMBT2干扰组和USP3过表达组HepG2细胞的miR-224-5p表达上调(P<0.05);与NC组、circ-SFMBT2干扰组及miR-224-5p干扰组比较,USP3过表达组HepG2细胞的USP3表达上调(P<0.05)。克隆斑点形成实验检测表明,与NC组比较,circ-SFMBT2干扰组HepG2细胞克隆数量下降、USP3过表达组HepG2细胞克隆数量上升(P<0.05)。Transwell实验检测结果表明,与NC组比较,circ-SFMBT2干扰组HepG2细胞迁移细胞数量下降(P<0.05),USP3过表达组HepG2细胞迁移细胞数量上升(P<0.05)。见图5。
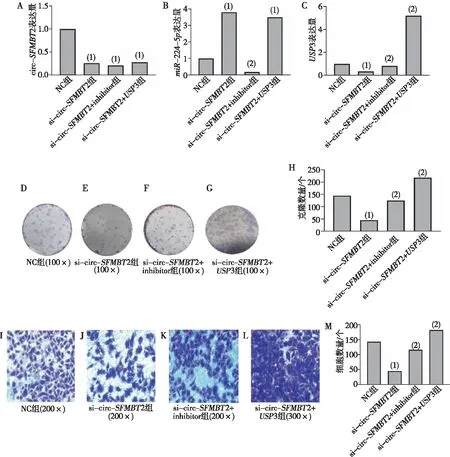
注:A~C分别为转染后HepG2细胞circ-SFMBT2、miR-224-5p及USP3的表达,D~H分别为细胞平板克隆实验形态学和定量的结果,I~M分别为Transwell细胞迁移能力实验形态学和定量结果;(1)与NC组比较,P<0.05;(2)与si-circ-SFMBT2组比较,P<0.05。
2.6 克隆斑点实验和Transwell实验
qRT-PCR检测结果显示,与NC组比较、转染miR-224-5p过表达模拟物(mimic)后HepG2细胞miR-224-5p表达上调(P<0.05),同时miR-224-5p过表达组和USP3过表达组相较于mimic组HepG2细胞miR-224-5p表达上调(P<0.05);与NC组比较,USP3表达在mimic组HepG2细胞中表达下调,在USP3过表达组HepG2细胞中上调(P<0.05)。克隆斑点形成实验检测结果表明,与NC组比较,mimic组HepG2细胞克隆细胞数下降、USP3过表达组HepG2细胞克隆细胞数上升(P<0.05)。Transwell实验检测检测结果表明,与NC组比较,mimic组HepG2细胞迁移细胞数下降、USP3过表达组HepG2细胞迁移细胞数上升(P<0.05)。见图6。
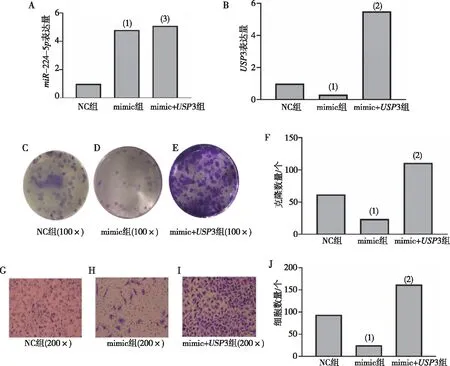
注:A、B分别为转染后HepG2细胞miR-224-5p和USP3的表达;C、D、E及F为细胞平板克隆实验形态学和定量的结果,G、H、I及J分别为Transwell细胞迁移能力实验形态学和定量结果;(1)与NC组比较,P<0.05;(2)与mimic组比较,P<0.05。
3 讨论
越来越多的研究表明,circRNA/miRNA/mRNA调控系统在胃癌、乳腺癌、胰腺癌、宫颈癌、卵巢癌及肝癌等多种癌症的进展中起着重要的调控作用,均被证实可促进增殖、转移和化疗耐药[10-13]。既往研究发现,circRNA ABCB10通过吸附miR-670-3p来促进HMG20A表达[14],circ-FOXP1可通过吸附miR-875-3p来促进SOX9基因表达,进而促进肝癌发生[15]。circ-SFMBT2吸附miR-182-5p,通过调节cAMP反应元件结合蛋白1(cyclic adenosine monophosphate responsive element binding protein 1,CREB1)mRNA表达,从而参与胃癌进展[7]。本研究qRT-PCR检测结果发现circ-SFMBT2在肝癌细胞中的表达也显著升高,为进一步探究其在肝癌细胞增殖和迁移中的作用,通过将circ-SFMBT2干扰载体瞬时转染进HepG2细胞,验证转染效率满意,顺利构建si-circ-SFMBT2干扰模型,CCK-8和迁移实验发现下调circ-SFMBT2后抑制了肝癌细胞增殖和迁移。竞争内源性RNA (competing endogenous RNA, ceRNA)假说认为,circRNAs主要是由外显子组成,并分布于细胞质中,这与circ-SFMBT2的FISH实验结果一致,circRNAs能够通过与AGO2蛋白结合,利用自身含有的miRNA反应元件(microRNA response element, MRE)吸附下游miRNAs来调控基因表达[16]。此外,最近有研究指出circRNAs可以受到m6A甲基化修饰,这也将为circRNAs的机制研究打开新的篇章[17]。上述这些特征表明 circRNAs具有成为疾病诊断的生物标志物和治疗靶点的潜力。
生物信息学分析发现circ-SFMBT2下游结合靶点有很多,包括hsa-miR-1290、miR-1296、miR-1305、miR-182、miR-188-3p、miR-198、miR-224-5p、miR-582-3p、miR-587、miR-622、miR-626及miR-885-3p等,但miR-224-5p与circ-SFMBT2之间在结合位点上具有更高的保守性,为了进一步确定circ-SFMBT2、miR-224-5p之间的靶向作用关系,将含有miR-224-5p结合位点的circ-SFMBT2野生型和突变体序列构建到了荧光素报告载体上,和miR-224-5p过表达mimic的片段一起转染到293T细胞中,结果显示转染野生型circ-SFMBT2和miR-224-5p mimic的荧光活力降低,说明circ-SFMBT2和miR-224-5p之间存在靶向作用关系。先前的研究表明,miR-224-5p的表达可以抑制许多癌症的增殖、迁移[18-20],并有研究报道miR-224-5p影响HepG2细胞脂质摄取与蓄积[21]。本研究中circ-SFMBT2下调促进了miR-224-5p的表达,而miR-224-5p下调则恢复了肝癌细胞的增殖、迁移;荧光素酶报告实验也证实circ-SFMBT2可以与miR-224-5p相互作用。
circRNA通过充当ceRNA来调控miRNA和靶基因表达。利用TargetScan推测发现USP3是miR-224-5p作用靶点。本研究中将含有miR-224-5p结合位点的USP3-3′UTR野生型和突变体序列构建到了荧光素报告载体上,和miR-224-5p过表达mimic的片段一起转染到293T细胞中,结果显示转染野生型USP3-3′UTR和miR-224-5p mimic的荧光活力降低,说明USP3-3′UTR和miR-224-5p之间存在靶向作用关系。同时根据研究报道,USP3可能作为一种致癌基因,促进多种类型癌症的进展[22],比如USP3基因敲除对乳腺癌细胞体外增殖和体内成瘤的具有抑制作用;USP3通过去泛素化可促进胃癌细胞迁移和侵袭;USP3可影响胃癌患者的预后[23]。在干扰circ-SFMBT2或过表达miR-224-5p条件下,过表达USP3可以恢复肝癌细胞的迁移和增殖能力,有研究发现海绵状 miR-224下调USP3作为竞争性内源性 RNA 促进大肠癌转移[24]。这与本次生物学分析和靶向实验结果相似。
综上,circ-SFMBT2可通过靶向miR-224-5p/USP3轴来调控肝癌细胞HepG2的增殖和迁移,下调circ-SFMBT2可抑制肝癌细胞HepG2的增殖和迁移能力,提示circ-SFMBT2是一个潜在的肝癌防治靶点,因此需要对circ-SFMBT2/miR-224-5p/USP3轴进行进一步的研究,可为开发新的肝癌治疗策略提供基础。

