利用食管类器官研究c-Myc在食管癌发生中的作用
陈友红,杨文豪,2,倪超
利用食管类器官研究在食管癌发生中的作用
陈友红1,杨文豪1,2,倪超1
1. 复旦大学生命科学学院,上海 200433 2. 四川大学华西第二医院小儿呼吸免疫科,成都 610041
基因在食管癌等多种恶性肿瘤中异常高表达,但其参与癌症发生发展机制尚不完全清楚。为探究在食管癌发生中的作用,本文成功构建了食管类器官作为研究模型,首先制备表达的慢病毒并通过高效侵染方法获得了稳定过表达的食管类器官。以正常食管类器官作为对照,使用ImageJ软件分析感染7代后食管类器官的形态,显示食管类器官的上皮形态并未出现异常。随后利用免疫荧光染色实验和CCK8试剂检测食管类器官的细胞增殖状态,结果显示细胞增殖速度也没有显著改变。最后通过实时荧光定量PCR实验(quantitative real-time PCR, qPCR)检测细胞周期、细胞代谢以及常见的在食管癌中高表达基因的表达情况,结果显示相关基因表达均未显著升高。这些结果初步表明在食管中单独过表达基因不足以诱导食管上皮细胞癌化。本文建立了食管类器官研究模型,通过高效的慢病毒过表达体系研究了原癌基因对食管类器官发育和增殖的潜在影响。这对于食管类器官模拟食管发育和食管癌发生相关研究具有一定参考意义。
食管癌;食管类器官;过表达;慢病毒侵染
食管癌预后差、生存率低,原因在于早期无症状而检测到的食管癌大多数都处于晚期高级别,极大增加了治疗难度[1~3]。探究食管癌发生过程中的分子学和细胞学机制能够改善早期诊断策略[4~6]。研究表明,在人类癌症中大部分肿瘤的发生与很多基因突变或异常表达密切相关(如、、等)[7~9]。
是一个常见的原癌基因,正常情况下其表达受到严格调控。主要通过作为转录因子与伴侣蛋白MAX形成二聚体,调控大量下游基因的表达。当其异常表达时往往导致细胞生长、细胞代谢等过程的异常以及肿瘤形成。研究发现在胰腺癌、肝癌、肺癌以及食管癌等肿瘤中均检测到的突变或异常表达[10~14],也有报道显示参与肝癌起始过程并且单独过表达就能诱发肝癌发生[15],但在食管癌发生过程的作用机制尚不清楚。
类器官是原始干细胞或成体干细胞在体外3D环境下培养形成的结构与功能均高度接近成体组织的结构,是新兴的体外研究模型[16,17]。传统的研究模型(主要有细胞系和小鼠品系)存在细胞系细胞类型单一和某些小鼠品系胚胎致死导致无法进行后续研究的问题,而类器官的出现则能弥补上述不足。类器官既具有与成体组织相同的细胞类型和结构,又能够特定地研究组织的发育和稳态[18]。由食管永生化上皮细胞与成纤维细胞共培养形成的器官类似物以及成体干细胞来源的食管类器官是近年来用于研究食管上皮的主要体外3D模型,已被广泛应用于研究食管的发育及疾病发生[19~22]。其中成体干细胞来源的食管类器官培养方法简单,同时能够通过单细胞形成,因而具有广阔的应用前景。
本文将小鼠成体组织来源的食管在基质胶中培养形成食管类器官,通过高效的慢病毒侵染方法在食管类器官中过表达。在此基础上,初步探究了在食管癌发生发展中的作用,发现的单独过表达对于食管类器官的形态、增殖和代谢以及食管癌相关的基因表达等无显著影响,提示基因的过表达不足以有效诱导食管癌化。这一发现有助于加深对食管癌发生过程的理解,为后续食管类器官研究提供一定方法参照。
1 材料与方法
1.1 实验材料
本实验所用小鼠为/品系6~8周雄性小鼠,购买自上海杰思捷实验动物有限公司;组装慢病毒所用293细胞由本实验室培养。
1.2 小鼠食管类器官培养
取得新鲜的小鼠食管组织,剪碎后移至1.5 mL Eppendorf管中加入已预热的消化液(消化液组成:0.25 mg/mL Pronase E,400 U/mL Collagenase I,20 U/mL DNase I),于37℃摇床100 r/min消化1 h。充分混匀,1000 r/min离心3 min,弃上清,用PBS洗2次。40 μm滤网过滤获得单细胞悬液,1000 r/min离心3 min,细胞沉淀置于冰上备用。取适量已解冻的基质胶液重悬细胞,混匀后铺于24孔板中(30 μL/孔),37℃静置15 min,待基质胶凝固充分加入500 μL体积液体培养基覆盖于表面,接着于37℃二氧化碳培养箱中培养,2~3天换液,通常培养10~12天后传代。食管类器官培养基[23]组成为:Advanced DMEM/F12 (12634028,Thermofisher,美国)中添加1xGlutamax (35050061,Gibco,美国);1xHEPES (15630080,Gibco,美国);1 mmol/L N- acetylcysteine (A9165-5g,Sigma,美国);10 mmol/L Nicotinamide (N0636,Sigma,美国);1xPenicillin- streptomycin (15140122,Gibco,美国);1xB27 (17504044,Gibco,美国);10 μmol/L Y27632 (13624,Cell Signaling Technology,美国);10 mmol/L SB202190 (8158,Cell Signaling Technology,美国),500 nmol/L A8301 (75073,Cell Signaling Technology,美国),100 μmol/L Gastrin (3006,R&D,美国),100 ng/mL R-sondin1 (4645-RS,R&D,美国),100 ng/mL Noggin (6057-NG,R&D,美国),50 ng/mL EGF (236-NG,R&D,美国)。
食管类器官传代方法:轻刮下类器官并转移至Eppendorf管中,1000 r/min离心3 min,去上清。用已预热的1xTryplE (2216499,Gibco,美国)消化液500 μL重悬,混匀后于37℃静置5~10 min。待消化充分后用终止消化,用PBS洗2次,以约5000个细胞接种到24孔板按上述步骤进行3D培养。基质胶(matrigel) (#356231)购自Corning公司。Pronase E (107433),Collagenase I (C0130),DNase I (10104159001)均购自美国Sigma公司。
1.3 过表达c-Myc慢病毒包装与感染
病毒过表达载体为pLVX-P2A-Puro-GFP,由本实验室改造而成。慢病毒包装质粒psPAX2 (#12260)、pMD2G (#12259)均购自美国 Addgene 公司。基因过表达的慢病毒包装时,在生长于10 cm 皿中密度为80%左右的293T细胞中,加入含10 μL vigofect、7 μg pLVX-、2 μg psPAX2、5 μg pMD2G的DMEM溶液,6 h后更换为完全培养基培养,48 h后收集上清,用0.45 μm滤膜过滤。收集上述慢病毒上清,45,000 r/min离心2 h,适量体积PBS溶解病毒沉淀,从而获得高浓度的慢病毒,于−80℃保存。此后,取1 μL慢病毒感染293T细胞,观察荧光强度并用qPCR实验检测下游基因表达情况,检验慢病毒的有效性。
感染食管类器官时,先将类器官消化成单细胞悬液。感染培养基为在食管类器官培养基中加入适量慢病毒(约20 μL浓缩病毒,聚凝胺浓度为10 μg/mL,总体积250 μL的混合液)。将感染培养基与食管单细胞悬液混合均匀,随后加入到48孔板中,封口膜密封后于孔板离心机中32℃条件下2000 r/min离心1 h。接着转移至37℃培养箱中继续培养6 h。室温1000 r/min离心3 min,去上清,用30 μL基质胶重悬细胞沉淀,按1.2所述方法进行3D培养。约72 h于荧光显微镜下观察绿色荧光所占比例,加入2 μg/mL的嘌呤霉素(puromycin)进行筛选,培养2代之后即可获得稳定高比例表达目的基因的食管类器官。
基因cDNA的来源于人肝实质细胞。NCBI中的转录本编号为Myc-206,全长1365 bp。上游克隆引物为:5′-GAATTCTGGATTTTTTTCGGGTAGTGGAAAACCAGCA-3′,下游克隆引物为:5′- ACTAGTTACGCACAAGAGTTCCGTAGCTGTTCAA- 3′,由苏州金唯智公司合成。Vigofect转染试剂购买自北京威格拉斯生物技术有限公司。嘌呤霉素(A610593)购自上海生工生物工程股份有限公司。
1.4 类器官免疫荧光染色
选取培养8~10天的成熟的食管类器官,收集于Eppendorf管里。以600~800 r/min离心3 min,用4%PFA 重悬混匀食管类器官,于4℃固定5 h。PBS洗一次,600 r/min离心后用0.25% tryton 100×500 μL重悬并透膜30 min。离心600 r/min,去上清。用5%BSA作为封闭液,用量500 μL重悬食管类器官,常温静置1 h。离心去上清,加入配置好的抗体液(c-Myc,Ki67均以1:100浓度配制) 50 μL与类器官混匀,随后4℃条件静置过夜。接着用PBST缓冲液洗3次,10 min/次。加入配置好的二抗(CY3抗体浓度1∶200,647二抗浓度为1∶400,Hoechst33342浓度为1∶1000),静置冰浴1 h。随后用PBST缓冲液洗3次,10 min/次,将类器官转移至粘附载玻片上,滴加抗荧光淬灭封片剂,盖上盖玻片完成封片。待凝固充分后用共聚焦显微镜观察并拍照,−20℃保存。
抗体(#18351)和Ki67抗体(#9449)购自美国Cell Signaling Technology公司,P63抗体(ab124762)与二抗CY3(A10520)、647(A32728)均购自美国Abcam公司,KRT13抗体(66684-1-Ig)购自美国Proteintech公司。Hoechst33342 (A3472)购自美国APExBIO公司。
1.5 定量PCR
选取培养8~10天的食管类器官,收集于Eppendorf管。1000 r/min离心3 min,去上清,用微量样品RNA提取试剂盒(天根生物科技)提取RNA。取1 μgRNA,用反转录试剂盒(Promega,美国)逆转录合成cDNA,稀释10倍至200 μL,并于–20℃长期保存,后续的定量PCR (qPCR)则在此浓度的情况下使用1 μL作为模板。在qPCR仪中以10 μL体系进行反应,每个样品设置3个复孔,所用2×SYBR Green qPCR Master Mix(B21702)为美国Bimake公司提供。反应的温度体系设置为:95℃ 15 min;95℃ 15 s,
60℃ 30 s,40个循环;95℃ 10 s,65℃~95℃(每隔5 s增加0.5℃)。反应引物序列如表1所示,所有引物均是在NCBI官网设计并进行Blast (https://www. ncbi.nlm.nih.gov/tools/primer-blast/),由金唯智公司合成。根据Ct值,以为内参,根据2–ΔΔCt法分析各基因的相对表达量。

表1 qPCR引物序列
“h-”表示human,“m-”表示mouse。“-总”为能同时检测内源和外源的引物;:cyclin;:cyclin dependent kinase;,ornithine decarboxylase;:nicotinamide phosphoribosyltransferase;:metastasis-associated protein 1;:lactate dehydrogenase A;:nuclear respiratory factor 1;:3-hydroxy-3-methylglutaryl-coenzyme a reductase。
1.6 CCK8试剂检测细胞增殖
取成熟的食管类器官,消化制备单细胞悬液。用luna-fl双荧光细胞计数仪(Luf-14-00980,韩国)计数确定细胞浓度。按1500细胞/孔接种到96孔板进行3D培养。对照组和实验组分别各铺9个孔。分别于Day1,Day4,Day5用CCK8试剂(APExBIO- K1018,美国)检测对应组3个孔的细胞活性。每个96孔添加10 μL的CCK8试剂(与90 μL培养基混合),随后于37℃培养箱中培养3 h,接着直接将96孔板于酶标仪检测480 nm处的吸光值,统计吸光值并用Graphpad8软件分析结果。
1.7 检测类器官形成能力
取成熟的食管类器官,消化制备单细胞悬液。用luna双荧光细胞计数仪计数确定细胞浓度。按500细胞/孔接种到96孔板进行3D培养。对照组和实验组各设置5个孔。于Day4时统计每个96孔中食管类器官的数量,此时的食管类器官通常直径大于30 μm。计算其由单细胞形成类器官数与细胞数的比值,定义为类器官形成率(%)。对照组与实验组各取3个孔的值(去掉最高值与最低值),比较类器官形成能力。
1.8 类器官形态与平均面积统计
选取成熟的食管类器官,消化制备单细胞悬液,使用细胞计数仪计数确定细胞悬液的浓度。以3000细胞/孔接种到24孔板中进行3D培养,每组设置3个复孔,利用显微镜观察并拍照。根据收集的JPEG格式的照片,使用图片分析软件ImageJ分析图中类器官的大小(平均面积),每个组累积统计100个类器官,选取同样的计算标准进行统计分析。根据面积统计结果用Graphpad8软件做出箱型图,并分析对照组与实验组的形态与大小的差异。
1.9 数据处理
对照组与实验组数据用Graphpa8处理并绘图,两组间的差异性评估采用检验评估。数据显著性用*标注。*:<0.05;**:<0.01;***:<0.001;ns:>0.05,无显著差异。
2 结果与分析
2.1 小鼠食管类器官培养
成熟的食管复层鳞状上皮主要由单层具有干性的基底细胞(basal cell)和多层不同程度分化的上基层细胞(suprabasal cell)组成,基底细胞和上基层细胞的主要标志物分别为和cytokeratin 13 ()。基底细胞被认为是食管上皮干细胞,能够不断增殖并向内移动形成不同程度分化的上基层细胞,最终高度角质化并从食管腔内脱落。通过这样的动态过程维持食管上皮的自我更新与稳态。
本研究分离了小鼠食管组织,将其消化制备成单细胞悬液,随后在基质胶中进行3D培养。通过调节培养基中Wnt信号通路激活剂的用量,我们获得了可以稳定传代(7代以上)的3D培养物。培养约10天形成的3D培养物如图1所示,从近乎单细胞状态开始,约3天时间逐渐增大形成透亮的小球状结构;到8~10天时,小球体逐渐长成清晰可见的实心囊状物,颜色逐渐加深。整个过程中生长状态良好,且形态与食管上皮形态高度相近。
为进一步证实食管培养物为食管类器官,我们通过免疫荧光实验鉴定3D培养物的细胞组成。结果如图2所示,3D培养物最外层细胞细胞核表达P63,而内层细胞细胞质则表达KRT13,这与成熟食管上皮细胞标志物一致。综上所述,形态分析和细胞类型鉴定结果表明本研究成功建立了食管类器官。
2.2 过表达c-Myc食管类器官的建立
为探究的功能,我们尝试借助于高效的慢病毒侵染方法在食管类器官中过表达。首先构建表达基因的慢病毒载体,随后利用高效侵染方法获得过表达的食管类器官,并通过qPCR和免疫荧光染色实验得到证实。
2.2.1 慢病毒构建与验证
过表达慢病毒载体的核心元件如图3A所示,如果慢病毒成功侵染类器官,则感染后的食管类器官会显示绿色荧光(coGFP)并具有嘌呤霉素(puromycin)抗性,能够非常方便我们进行观察和筛选。为检验构建的慢病毒的有效性,我们用慢病毒感染293T细胞,观察绿色荧光情况并用qPCR实验检测下游基因的表达。结果显示293T细胞均高表达绿色荧光,同时下游基因均显著高表达,说明慢病毒高效感染293T细胞并表达外源(图3,B和C),表明我们成功构建了表达的慢病毒。

图1 小鼠食管消化后的单细胞在3D培养条件下形成囊状实心结构
A~E:小鼠食管组织被消化成单细胞后培养9天形成实心囊状物的过程;F:图E的局部放大图。
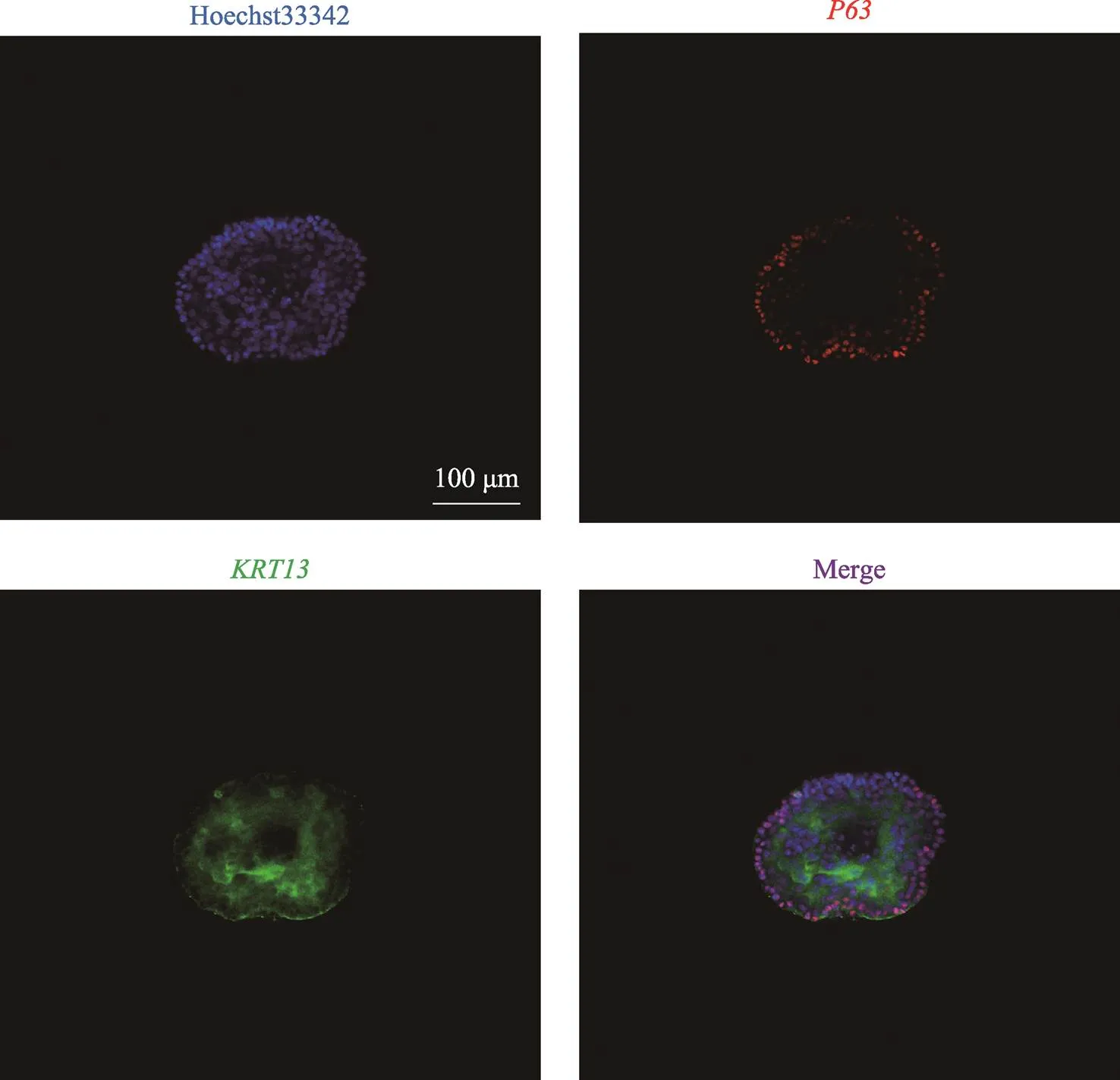
图2 食管类器官表达食管上皮细胞标志物P63和KRT13
培养至P1D9的食管类器官,使用抗体P63和KRT13进行免疫荧光染色结果。Hoechst33342试剂染细胞核,显示为蓝色;P63染基细胞细胞核,显示为红色;KRT13染上基层细胞细胞质,显示为绿色。

图3 成功构建表达外源基因的慢病毒
A:慢病毒质粒载体核心元件示意图。CMV为启动子,MCS表示多克隆位点(multiple cloning site)。B:慢病毒感染293T细胞72 h之后观察绿色荧光的结果。C:qPCR实验检测下游基因表达情况。*:<0.05;**:<0.01。
2.2.2 食管类器官的高效感染与筛选
类器官感染通常采用直接将慢病毒与单细胞混合培养的感染方法,但这种方法在食管类器官中感染效率不高且细胞存活率低[24~27]。为高效获得过表达的食管类器官,我们采用离心感染法。研究表明该方法能增加细胞与慢病毒接触几率并能提高细胞存活率。未进行慢病毒感染的正常食管类器官在含2 μg/mL嘌呤霉素的培养基中培养2天之内就会死亡。而慢病毒感染后类器官能够在含2 μg/mL嘌呤霉素的培养基中正常培养、传代,第1代第9天(记为P1D9)观察荧光情况如图4所示,初步显示慢病毒高效感染食管类器官。
进一步我们通过qPCR和免疫荧光染色实验检测总的表达情况,结果如图5A和B所示,均显示基因的显著高表达。综上所述,我们成功建立了过表达基因的食管类器官。
2.3 c-Myc过表达对食管类器官的影响
2.3.1过表达对食管的类器官形态的影响
组织形态紊乱是肿瘤的特征之一。为探究是否具有诱导食管癌发生的能力,我们首先分析了过表达食管类器官的形态体积的变化情况。感染后的类器官连续传代培养,对每一代的食管类器官生长状态进行详细观察和拍照记录,结合软件ImageJ分析类器官的大小(平均面积),利用软件Graphpad做出箱型图并分析差异。从图6A中可以看出,食管类器官长期稳定传代后,对照组与实验组相比同时期的形态没有显著差异;而从图6B和6C中对应的统计结果可以看出,对照组和实验组的食管类器官平均面积没有显著差异(>0.05)。综上所述,过表达后食管类器官的形态和平均面积均没有出现显著性变化。
2.3.2过表达对食管类器官增殖的影响
肿瘤组织通常表现出增殖异常现象。为探究过表达对食管增殖的影响,我们首先用Ki67抗体进行免疫荧光染色实验检测食管类器官细胞增殖水平。结果如图7所示,过表达组与对照组的Ki67表达量相同,且表达位置都在基底细胞,说明过表达后Ki67的表达并没有升高,即细胞的增殖水平没有增加。

图4 食管类器官的慢病毒感染与筛选
慢病毒感染后的食管类器官在含2 μg/mL嘌呤霉素的培养基中培养至P1D7(即第1代第7天)的绿色荧光观察结果。

图5 qPCR和免疫荧光染色实验显示高效获得过表达c-Myc的食管类器官
A:感染后培养9天(P1D9)用qPCR检测总的表达量;B:培养9天(P1D9)的食管类器官进行免疫荧光染色实验,用抗体检测的表达量。IF,Immunofluorescent staining method。***:<0.001。

图6 c-Myc过表达对食管类器官的形态和大小均无显著影响
A:慢病毒感染后的类器官进行连续传代培养的过程,每代培养8~10天,所示为第一代、第四代、第7代培养9天后的生长情况;B:对照组和实验组类器官平均面积比较(P4D9);C:对照组和实验组类器官平均面积比较结果(P7D9)。ns:>0.05,无显著差异。

图7 免疫荧光染色显示Ki67的表达无显著变化
慢病毒感染后的食管类器官,用抗体Ki67进行免疫荧光染色的结果。
食管复层鳞状上皮由食管上皮干细胞增殖分化形成并维持稳态,因此食管类器官的形成能力也反映了食管上皮细胞的增殖能力。我们通过将食管类器官消化成单细胞后,取相同数量的单细胞,计算其形成类器官的能力,评估过表达对食管上皮细胞增殖的影响。结果显示过表达不能促进食管类器官形成能力的增加,提示过表达不能导致食管上皮细胞增殖升高(图8A)。
为进一步探究过表达对食管细胞增殖影响,我们利用CCK8 试剂检测细胞增殖情况。图8B所示为不同时间点检测食管类器官的吸光值的统计结果。可以看出,过表达组与对照组食管类器官在480 nm处的吸光值强度没有显著性差异(>0.05),即CCK8试剂检测结果表明,过表达后食管类器官的增殖活性未出现异常增高情况。
综上所述,我们的研究结果表明过表达不能导致食管上皮细胞出现类似肿瘤的细胞增殖异常升高现象。
2.4 c-Myc过表达对细胞周期与细胞代谢相关基因表达的影响
的异常表达往往导致细胞生长、代谢相关基因的异常高表达,进而驱动肿瘤的发生。为探究过表达基因是否具有促进食管癌发生的能力,我们利用qPCR实验检测了下游与细胞周期、代谢相关基因的表达情况。其中细胞周期相关基因包括、、、、与[15];细胞代谢相关基因包括、、、、与[28~30]。结果表明在食管类器官中过表达后,细胞周期、细胞代谢相关的基因表达情况均未出现异常升高情况(图9)。
2.5 c-Myc过表达食对管癌相关基因表达的影响
肿瘤的发生往往伴随着常见肿瘤相关基因的异常表达。为探究是否具有诱导肿瘤发生的能力,我们利用qPCR实验检测了常见的食管癌中异常高表达基因的表达情况。其中食管鳞癌中报道高频过表达的基因包括和等;在食管腺癌中报道高频过表达的基因包括、和等[4,7]。qPCR检测结果表明,过表达后,食管癌相关基因的表达均未出现显著升高的情况(图10)。
3 讨论
食管癌是全球最高发的癌症之一。通过寻找相关肿瘤标志物和探究食管癌发生过程中相关的分子机制对于改善食管癌的早期诊断策略具有重要意义。由于通常情况下病人来源的样本不易获得,建立小鼠成体组织来源的食管类器官具有重要意义。本研()的食管类器官,为研究食管发育和疾病发生提供了一个良好的模型。

图8 食管类器官形成能力与用CCK8检测细胞增殖情况
A:相同细胞数培养形成的食管类器官数与对应细胞数的比例(食管类器官形成率);B:添加CCK8后测定吸光值(480 nm)。*:<0.05;ns:>0.05,无显著差异。

图9 q-PCR检测c-Myc下游与细胞周期、代谢相关的基因表达情况
A:下游与细胞周期相关基因的表达量;B:下游与细胞代谢相关基因的表达量。*:<0.05;**:<0.01;ns:>0.05,无显著差异。

图10 食管癌相关基因的表达情况
qPCR检测食管癌相关基因表达情况。结果显示均无显著性差异(ns:>0.05)。
究成功建立了小鼠成体组织来源的食管类器官,并且采用了高效的慢病毒侵染方法获得了过表达外源基因组织形态紊乱、细胞增殖与细胞代谢异常以及癌症相关基因的异常高表达是肿瘤的常见特征。在食管癌中异常高表达且有报道其参与肿瘤发生过程,但其在食管癌发生中的作用机制有待深入研究。为探究在食管癌发生中的作用机制,我们尝试在食管类器官中过表达,预期可能会诱导食管上皮癌化。然而我们多方面的检测实验结果均显示:过表达的食管类器官并未出现上述可能的癌化特征。
基因作为转录因子和原癌基因,它的异常高表达引起很多下游基因的异常表达如核糖体代谢、线粒体代谢、细胞周期等相关基因[28],从而导致疾病甚至癌症的发生[28,29,32,33]。据中国科学院分子细胞科学卓越创新中心惠利健课题组报道基因的过表达能够诱导肝癌的发生,在类器官水平就已显示细胞结构紊乱,异常增生以及线粒体代谢的异常活跃[15]。而基因作为一个在食管腺癌和食管鳞癌中都高频异常高表达的基因[13,14],有报道显示在食管鳞癌细胞系中过表达基因能够促进(programmed cell death ligand 1)[34,35]分子的表达,从而使得肿瘤细胞逃逸免疫系统的识别和攻击。然而这些研究都是以食管癌细胞系或食管癌来源的类器官作为研究模型,真正关于在食管癌起始过程中是否具有重要作用目前未见报道。通常在病变中检测到的异常高表达大多数只是3~4倍左右,少数达到10倍[36],即在这种情况下往往就会导致下游相关基因的异常表达并导致增殖、代谢异常。然而我们导入的外源基因检测显示显著高表达,却没有检测到相关基因的异常高表达。此外我们还检测了部分常见的在食管癌中异常高表达的基因的表达情况,过表达基因的食管类器官也未检测到这些基因异常高表达(尽管这些食管癌相关基因只具有有限的代表意义)。这些结果综合表明单独过表达基因不足以诱导食管癌化的发生,同时也提示食管癌的发生更有可能是多个基因突变或异常表达积累的结果。因此,为建立食管癌诱导模型,我们正在开展在现有过表达基因的食管类器官基础上引入更多的基因突变的工作。例如在食管腺癌和鳞癌中均有出现高频突变的基因和基因等。
此外,需要指出的是图5B显示似乎在基底细胞中的表达较低。为验证是否在两类细胞中的表达模式有差异,我们又对成熟食管类器官(Day7)与早期食管类器官(Day3)进行了P63和的共定位染色(附图1,附图2)。结果显示在早期食管类器官中在所有干细胞中均高表达,而在后期成熟的食管类器官中基底细胞中表达比例较低;而在上基层细胞中的表达量则一直较高。这提示可能在食管基底细胞和上基层细胞中的蛋白稳定性有差异,进一步提示可能在基底细胞中存在抑制表达的信号。这可能是不能导致食管类器官癌化的原因之一。
综上所述,本文建立的食管类器官与所采用的高效慢病毒感染方法为研究食管癌及相关疾病发生的分子机制提供了一个良好模型。并且本文利用该模型探究了基因在食管癌发生中的作用,表明单独过表达基因不足以诱导食管癌化的发生。后续的研究将在现有过表达基因的基础上进一步探究多基因共同作用诱导食管癌形成的可能性。
附录:
附加材料详见文章电子版www.chinagene.cn。
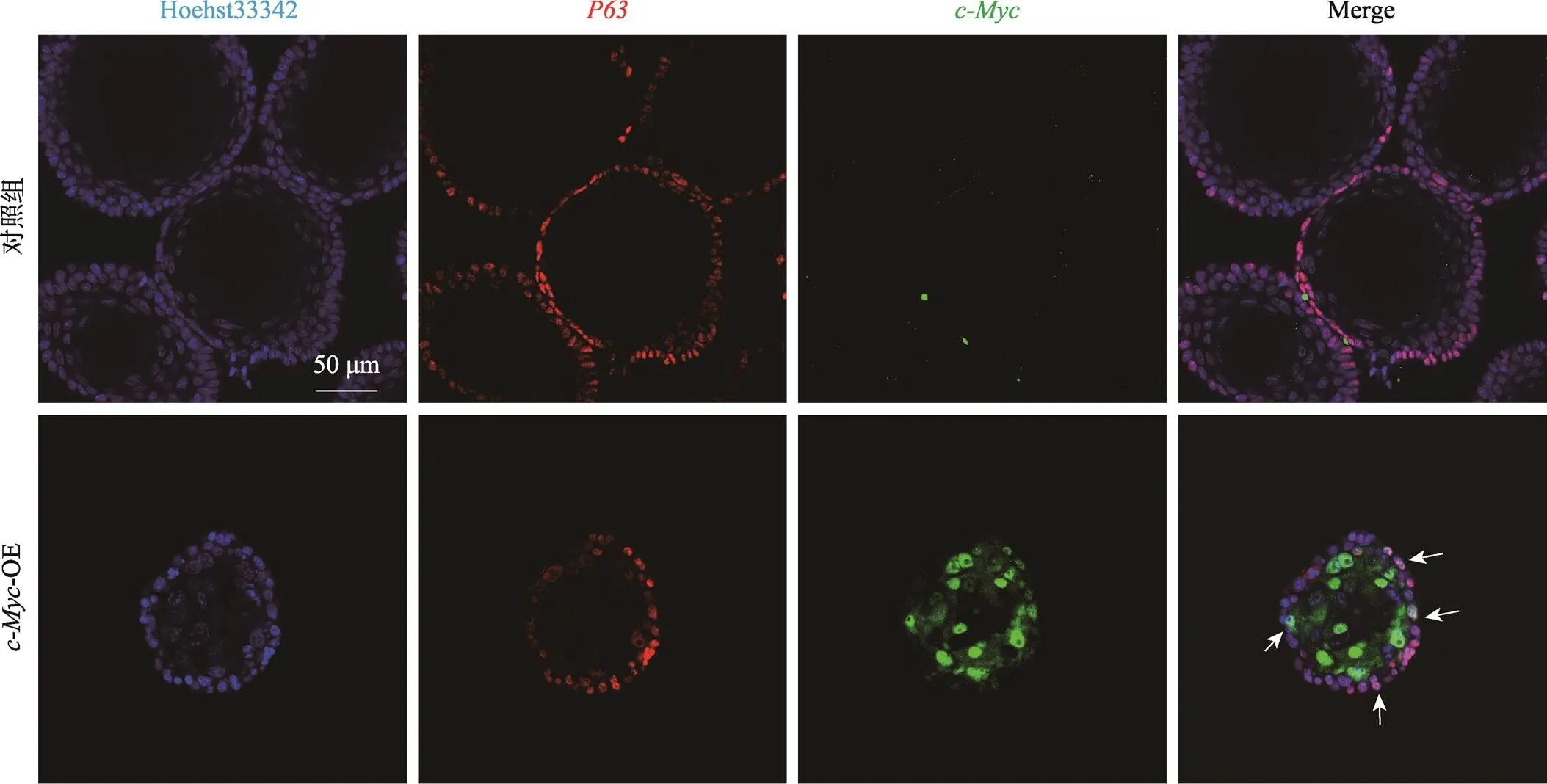
附图1 成熟食管类器官中与P63共定位结果
Supplemental Fig. 1 Co-localization ofand P63 in mature esophageal organoid
用抗体与P63对培养7天(Day7)的成熟食管类器官进行免疫荧光染色。图中白色箭头显示了同时表达-与P63的基底细胞。

附图2 早期食管类器官中c-与P63共定位结果
Supplemental Fig. 2 Co-localization of-and P63 in early esophageal organoid
用抗体-与P63对培养7天(Day7)的早期食管类器官进行免疫荧光染色。大部分基底细胞均表达-与P63。
[1] Lin YS, Totsuka Y, Shan BE, Wang CC, Wei WQ, Qiao YL, Kikuchi S, Inoue M, Tanaka H, He YT. Esophageal cancer in high-risk areas of China: research progress and challenges., 2017, 27(3): 215–221.
[2] Huang FL, Yu SJ. Esophageal cancer: risk factors, genetic association, and treatment., 2018, 41(3): 210–215.
[3] Sethi NS, Kikuchi O, Duronio GN, Stachler MD, McFarland JM, Ferrer-Luna R, Zhang YX, Bao CY, Bronson R, Patil D, Sanchez-Vega F, Liu JB, Sicinska E, Lazaro JB, Ligon KL, Beroukhim R, Bass AJ. Early TP53 alterations engage environmental exposures to promote gastric premalignancy in an integrative mouse model., 2020, 52(2): 219–230.
[4] Testa U, Castelli G, Pelosi E. Esophageal cancer: genomic and molecular characterization, stem cell compartment and clonal evolution., 2017, 4(3): 67.
[5] di Pietro M, Canto MI, Fitzgerald RC. Endoscopic management of early adenocarcinoma and squamous cell carcinoma of the esophagus: screening, diagnosis, and therapy., 2018, 154(2): 421–436.
[6] Talukdar FR, di Pietro M, Secrier M, Moehler M, Goepfert K, Lima SSC, Pinto LFR, Hendricks D, Parker MI, Herceg Z. Molecular landscape of esophageal cancer: implications for early detection and personalized therapy., 2018, 1434(1): 342–359.
[7] Cancer Genome Atlas Research Network, Analysis Working Group: Asan University, BC Cancer Agency, Brigham and Women’s Hospital, Broad Institute, Brown University, Case Western Reserve University, Dana-Farber Cancer Institute, Duke University, Greater Poland Cancer Centre, Harvard Medical School, Institute for Systems Biology, KU Leuven, Mayo Clinic, Memorial Sloan Kettering Cancer Center, National Cancer Institute, Nationwide Children’s Hospital, Stanford University, University of Alabama, University of Michigan, University of North Carolina, University of Pittsburgh, University of Rochester, University of Southern California, University of Texas MD Anderson Cancer Center, University of Washington, Van Andel Research Institute, Vanderbilt University, Washington University, Genome Sequencing Center: Broad Institute, Washington University in St. Louis, Genome Characterization Centers: BC Cancer Agency, Broad Institute, Harvard Medical School, Sidney Kimmel Comprehensive Cancer Center at Johns Hopkins University, University of North Carolina, University of Southern California Epigenome Center, University of Texas MD Anderson Cancer Center, Van Andel Research Institute, Genome Data Analysis Centers: Broad Institute, Brown University, Harvard Medical School, Institute for Systems Biology, Memorial Sloan Kettering Cancer Center, University of California Santa Cruz, University of Texas MD Anderson Cancer Center, Biospecimen Core Resource: International Genomics Consortium, Research Institute at Nationwide Children’s Hospital, Tissue Source Sites: Analytic Biologic Services, Asan Medical Center, Asterand Bioscience, Barretos Cancer Hospital, BioreclamationIVT, Botkin Municipal Clinic, Chonnam National University Medical School, Christiana Care Health System, Cureline, Duke University, Emory University, Erasmus University, Indiana University School of Medicine, Institute of Oncology of Moldova, International Genomics Consortium,Invidumed, Israelitisches Krankenhaus Hamburg, Keimyung University School of Medicine, Memorial Sloan Kettering Cancer Center, National Cancer Center Goyang, Ontario Tumour Bank, Peter MacCallum Cancer Centre, Pusan National University Medical School, Ribeirão Preto Medical School, St. Joseph’s Hospital &Medical Center, St. Petersburg Academic University, Tayside Tissue Bank, University of Dundee, University of Kansas Medical Center, University of Michigan, University of North Carolina at Chapel Hill, University of Pittsburgh School of Medicine, University of Texas MD Anderson Cancer Center, Disease Working Group, Duke University, Memorial Sloan Kettering Cancer Center, National Cancer Institute, University of Texas MD Anderson Cancer Center, Yonsei University College of Medicine, Data Coordination Center: CSRA Inc, Project Team: National Institutes of Health. Integrated genomic characterization of oesophageal carcinoma., 2017, 541(7636): 169–175.
[8] Lin DC, Dinh HQ, Xie JJ, Mayakonda A, Silva TC, Jiang YY, Ding LW, He JZ, Xu XE, Hao JJ, Wang MR, Li CQ, Xu LY, Li EM, Berman BP, Koeffler HP. Identification of distinct mutational patterns and new driver genes in oesophageal squamous cell carcinomas and adenocarcinomas., 2018, 67(10): 1769–1779.
[9] Forbes SA, Beare D, Boutselakis H, Bamford S, Bindal N, Tate J, Cole CG, Ward S, Dawson E, Ponting L, Stefancsik R, Harsha B, Kok CY, Jia MM, Jubb H, Sondka Z, Thompson S, De T, Campbell PJ. COSMIC: somatic cancer genetics at high-resolution., 2017, 45(D1): D777–D783.
[10] Hoffman B, Amanullah A, Shafarenko M, Liebermann DA. The proto-oncogenein hematopoietic development and leukemogenesis., 2002, 21(21): 3414–3421.
[11] Kim EY, Kim A, Kim SK, Chang YS. MYC expression correlates with PD-L1 expression in non-small cell lung cancer., 2017, 110: 63–67.
[12] Dolezal JM, Wang HB, Kulkarni S, Jackson L, Lu J, Ranganathan S, Goetzman ES, Bharathi SS, Beezhold K, Byersdorfer CA, Prochownik EV. Sequential adaptive changes in a c-Myc-driven model of hepatocellular carcinoma., 2017, 292(24): 10068–10086.
[13] Huang J, Jiang DX, Zhu T, Wang YQ, Wang H, Wang Q, Tan LJ, Zhu HG, Yao JX, Hou YY. Prognostic significance ofamplification in esophageal squamous cell carcinoma., 2019, 107(2): 436–443.
[14] von Rahden BHA, Stein HJ, Puhringer-Oppermann F, Sarbia M.amplification is frequent in esophageal adenocarcinoma and correlated with the upregulation of VEGFA expression., 2006, 8(9): 702–707.
[15] Sun LL, Wang YQ, Cen J, Ma XL, Cui L, Qiu ZX, Zhang ZT, Li H, Yang RZ, Wang CH, Chen XT, Wang L, Ye Y, Zhang HB, Pan GY, Kang JS, Ji Y, Zheng YW, Zheng S, Hui LJ. Modelling liver cancer initiation with organoids derived from directly reprogrammed human hepatocytes., 2019, 21(8): 1015–1026.
[16] Clevers H. Modeling development and disease with organoids., 2016, 165(7): 1586–1597.
[17] Huch M, Koo BK. Modeling mouse and human development using organoid cultures., 2015, 142(18): 3113–3125.
[18] Schutgens F, Clevers H. Human organoids: tools for understanding biology and treating diseases., 2020, 15: 211–234.
[19] Trisno SL, Philo KED, McCracken KW, Catá EM, Ruiz-Torres S, Rankin SA, Han L, Nasr T, Chaturvedi P, Rothenberg ME, Mandegar MA, Wells SI, Zorn AM, Wells JM. Esophageal organoids from human pluripotent stem cells delineate SOX2 functions during esophageal specification., 2018, 23(4): 501–515.e7.
[20] Zhang YC, Yang Y, Jiang M, Huang SX, Zhang WW, Alam DA, Danopoulos S, Mori M, Chen YW, Balasubramanian R, de Sousa Lopes SMC, Serra C, Bialecka M, Kim E, Lin SJ, de Carvalho ALRT, Riccio PN, Cardoso WV, Zhang X, Snoeck HW, Que JW. 3D Modeling of esophageal development using human PSC-derived basal progenitors reveals a critical role for notch signaling., 2018, 23(4): 516–529.e5.
[21] Bailey DD, Zhang YC, van Soldt BJ, Jiang M, Suresh S, Nakagawa H, Rustgi AK, Aceves SS, Cardoso WV, Que JW. Use of hPSC-derived 3D organoids and mouse genetics to define the roles of YAP in the development of the esophagus., 2019, 146(23): dev178855.
[22] Fujii M, Clevers H, Sato T. Modeling human digestive diseases with CRISPR-Cas9-modified organoids., 2019, 156(3): 562–576.
[23] DeWard AD, Cramer J, Lagasse E. Cellular heterogeneity in the mouse esophagus implicates the presence of a nonquiescent epithelial stem cell population., 2014, 9(2): 701–711.
[24] Koo BK, Sasselli V, Clevers H. Retroviral gene expression control in primary organoid cultures., 2013, 27: Unit 5A.6.
[25] Maru Y, Orihashi K, Hippo Y. Lentivirus-based stable gene delivery into intestinal organoids., 2016, 1422: 13–21.
[26] Wei JS, Ran G, Wang X, Jiang N, Liang JQ, Lin XH, Ling C, Zhao B. Gene manipulation in liver ductal organoids by optimized recombinant adeno-associated virus vectors., 2019, 294(38): 14096–14104.
[27] Sachs N, Papaspyropoulos A, Zomer-van Ommen DD, Heo I, Böttinger L, Klay D, Weeber F, Huelsz-Prince G, Iakobachvili N, Amatngalim GD, de Ligt J, van Hoeck A, Proost N, Viveen MC, Lyubimova A, Teeven L, Derakhshan S, Korving J, Begthel H, Dekkers JF, Kumawat K, Ramos E, van Oosterhout MF, Offerhaus GJ, Wiener DJ, Olimpio EP, Dijkstra KK, Smit EF, van der Linden M, Jaksani S, van de Ven M, Jonkers J, Rios AC, Voest EE, van Moorsel CH, van der Ent CK, Cuppen E, van Oudenaarden A, Coenjaerts FE, Meyaard L, Bont LJ, Peters PJ, Tans SJ, van Zon JS, Boj SF, Vries RG, Beekman JM, Clevers H. Long-term expanding human airway organoids for disease modeling., 2019, 38(4): e100300.
[28] Dang CV.on the path to cancer., 2012, 149(1): 22–35.
[29] Dang CV., metabolism, cell growth, and tumorigenesis., 2013, 3(8): a014217.
[30] Li J, Liang Y, Lv H, Meng H, Xiong G, Guan XY, Chen XD, Bai Y, Wang K. miR-26a and miR-26b inhibit esophageal squamous cancer cell proliferation through suppression ofpathway., 2017, 625: 1–9.
[31] McCauley HA, Wells JM. Pluripotent stem cell-derived organoids: using principles of developmental biology to grow human tissues in a dish., 2017, 144(6): 958–962.
[32] Pan XN, Chen JJ, Wang LX, Xiao RZ, Liu LL, Fang ZG, Liu QT, Long ZJ, Lin DJ. Inhibition ofovercomes cytotoxic drug resistance in acute myeloid leukemia cells by promoting differentiation., 2014, 9(8): e105381.
[33] Chan JC, Hannan KM, Riddell M, Ng PY, Peck A, Lee RS, Hung S, Astle MV, Bywater M, Wall M, Poortinga G, Jastrzebski K, Sheppard KE, Hemmings BA, Hall MN, Johnstone RW, McArthur GA, Hannan RD, Pearson RB. AKT promotes rRNA synthesis and cooperates withto stimulate ribosome biogenesis in cancer., 2011, 4(188): ra56.
[34] Liang MQ, Yu FQ, Chen C.-regulates PD-L1 expression in esophageal squamous cell carcinoma., 2020, 12(2): 379–388.
[35] Hong J, Maacha S, Belkhiri A. Transcriptional upregulation ofby AXL confers epirubicin resistance in esophageal adenocarcinoma., 2018, 12(12): 2191–2208.
[36] Rygiel AM, Milano F, Ten Kate FJ, Schaap A, Wang KK, Peppelenbosch MP, Bergman JJGHM, Krishnadath KK. Gains and amplifications of-, EGFR, and 20.q13 loci in the no dysplasia-dysplasia-adenocarcinoma sequence of Barrett's esophagus., 2008, 17(6): 1380–1385.
Using esophagus organoid to explore the role ofin esophageal cancer initiation
Youhong Chen1, Wenhao Yang1,2, Chao Ni1
gene is aberrantly highly expressed and participates in cancer initiation and development in various malignant tumors including esophageal cancer, while the underlying mechanism(s) still remains unclear. In order to explore the role ofin the occurrence of esophageal cancer, we successfully established the esophageal organoids (EOs) as the research model. By constructing a lentivirus overexpressingand developing more effective infection method, EOs with stable overexpression ofwere efficiently obtained. The morphologies of EOs with or without overexpressingwere first analyzed with ImageJ, which showed no difference between two groups during continuous subculture. Subsequently, we applied immunofluorescence and CCK8 assays to evaluate the cell proliferation, and the results showed no change in the-overexpressed group as compared to control EOs. Furthermore, qPCR was used to detect the expression of genes that are related to cell cycle, cell metabolism as well as esophageal cancer. The results indicated the expression of these genes was not significantly increased in theoverexpressing EOs. In conclude, we discovered that overexpression ofgene alone in the esophagus organoid is not sufficient to induce carcinogenesis in esophageal carcinoma. In this study, we successfully established an esophagus organoid culture system and together with efficient lentivirus-infection method for investigation on the effects of overexpressingin esophageal cancer. Our work demonstrated a promising research model for the study of esophagus development and esophageal cancer.
esophagus cancer; esophageal organoid;overexpression; lentivirus infection
2021-01-08;
2021-04-06
国家自然科学基金青年项目(编号:31900581)资助[Supported by the National Natural Science Foundation of China (No. 31900581)]
陈友红,在读硕士研究生,专业方向:利用食管类器官模拟食管癌发生。E-mail: 15216686231@163.com
倪超,博士,助理研究员,研究方向:利用肺类器官模拟疾病发生。E-mail: chaoni@fudan.edu.cn
10.16288/j.yczz.21-010
2021-04-26 16:24:46
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210426.1101.002.html
(责任编委: 方向东)

