基于深度学习的中文生物医学实体关系抽取系统
丁泽源,杨志豪,罗 凌,王 磊,张 音,林鸿飞,王 健
(1. 大连理工大学 计算机科学与技术学院,辽宁 大连 116024;2. 军事医学科学院,北京 100850)
0 引言
伴随着生物医学领域的飞速发展,生物医学领域的相关文献数量也呈现指数级别增长。由于文献中蕴含着海量的生物医学知识,人工提取文献信息需要耗费大量的时间,而且很难满足相关的研究人员的研究需求。因此,如何自动地从生物医学文献中抽取结构化信息成为一个重要的研究领域。
在当前自然语言处理(natural language processing,NLP)研究中,文本挖掘技术的兴起给上述难题提供了相应的解决方案。文本挖掘技术是指从非结构化的文本数据中自动发现和抽取有价值知识的过程,而命名实体识别和关系抽取任务是文本挖掘技术中关键的步骤。对于给定的一段文本,实体识别和关系抽取技术需要在辨别实体的基础上,抽取出实体之间的关系。如今实体识别和关系抽取越来越多地被应用于专业领域,如医疗、教育、生物等领域。由于生物医学与人类的健康密切相关,因此该领域的信息抽取技术也受到学界的广泛关注。然而生物医学领域中有大量的专业名词的缩写,并且名词与名词之间也存在着不同种类的关系,这给生物医学领域的信息抽取任务带来了挑战。基于此,本文构建了一个基于深度学习的中文生物医学实体关系抽取系统,该系统可以自动地识别中文生物医学文献中的实体以及实体间关系。
生物医学命名实体识别(biomedical named entity recognition,Bio-NER)是生物医学文本挖掘的基本步骤,Bio-NER 任务的目标是从给定的非结构化医学文本中识别出相关的实体 (如疾病、药物、蛋白质、症状等) 。由于生物医学领域的以下特性: (1) 在生物医学领域常常出现专业名词的缩写。不同的实体常常对应着同一个缩写,这些缩写会导致歧义问题。(2) 在生物医学领域,同一个实体可能有不同的命名方式,这些实体缺乏统一的命名方式,导致实体名的稀疏,给实体识别带来了很多困难。以上的这些特性使得生物医学领域的实体识别具有很大的挑战性。
生物医学领域的关系抽取 (relation extraction, RE)是要实现从生物医学文本中识别出生物医学实体(如疾病、药物、基因、蛋白质等)之间的语义关系并形成关系网络。当前,生物医学领域的关系抽取研究主要集中在基因与基因的关系、蛋白质与蛋白质互相作用关系、基因与疾病的关系、基因与治疗药物之间的关系等方面。
本文提出的基于深度学习的中文生物医学实体关系抽取系统是流水线结构。首先利用公开的英文生物医学实体关系标注语料,结合翻译技术和人工标注方法构建中文生物医学实体关系语料;然后将预训练好的ELMo[1]作为新的特征输入到 BiLSTM+CRF 模型中[2],与现有的中文实体识别模型相比,我们的模型可以很好地缓解专业名词缩写引起的歧义问题和实体名稀疏的问题,从而提高实体识别的性能。为了捕获的语义信息,本文使用结合注意力 (Attention) 机制的双向长短期记忆网络 (BiLSTM) 抽取实体间的关系[3]。不同于英文,汉字往往具有很强的语义信息。本文对笔画信息进行建模,将其作为中文独有的特征加入关系抽取的模型中,以此来提高关系抽取的性能。
1 生物医学语料构建
1.1 语料库的标注体系
语料库标注是对原始语料进行预处理,使用便于计算机存储以及读取的标注格式,并结合语料本身特殊需求进行标注。TEI(text encoding initiative)是机器可读文本的国际信息编码规范[4]。TEI标注模式是由计算语言学学会、文学与语言学计算协会和计算机与人文科学学会三家学术团体共同参与制订的。目前许多大型语料库都是基于TEI标注准则的,如 “英国国家语料库 ”等。
本文结合生物医学语料本身特点以及TEI便于计算机存储及读取等特点,采用TEI标注与自定义标注相结合的方式进行标注。标注体系包括以下内容:蛋白质(proteins)、化学物(chemicals)、疾病(diseases)、药名(drug)、脱氧核糖核酸(DNA)以及核糖核酸(RNA)。实体间关系标注为存在关系或者不存在关系。
1.2 语料库的构建
本文构建的中文生物医学语料来源于英文生物医学实体关系标注语料 BioCreative CDR (Chemical-Disease Relation, CDR)[5]语料和JNLPBA (International Joint Workshop on Natural Language Processing in Biomedicine and Its Applications)[6]语料。 BioCreative 评测是国际上用于生物自然语言处理研究的重要评测。 BioCreative V[7]任务中化学物-疾病关系(CDR)语料作为本系统中的英文语料库来源之一。JNLPBA是与国际计算语言学会议同时召开的公开评测会议, 其主要评测任务是生物命名实体识别。
本文结合翻译技术和人工标注方法构建中文生物医学实体关系语料。在使用翻译技术构建语料的过程中,主要遇到以下两个问题: ①确定译文中实体位置的问题。在翻译过程中,原文中的实体位置与译文中实体位置不是相对应的,因此如何确定译文中实体的位置就成了一个难点。②实体翻译不准确问题。在生物医学实体中包含着大量的缩写、特殊符号和数字,这些符号对英文翻译的质量造成很大影响。统计发现,常常由于DNA、RNA等缩写的翻译不准确,导致得到的中文生物医学实体关系语料质量不高。
因此,本文在构建中文生物医学的实体关系语料过程中,采用以下方式解决上文描述的问题: 由于生物医学语料中的实体都是专有名词,如果将这些实体用特殊符号代替,并不会对句子产生很大影响。经过人工检查发现,这种方法不仅能确定译文中的实体位置,还能改善由缩写、符号和数字带来的翻译不准确问题。所以问题 ①的解决方法为:先将英文语料中的实体用特殊字符代替,然后单独处理实体,最后将处理好的实体代替译文中的特殊符号。针对“实体翻译不准确”的问题,本文通过百度文库、博客和人工积累的英文实体,建立了一个包含3 291个中英文医学实体的对照表。后续用到的英文实体直接进行查表,这样不仅提高翻译的准确度,还进一步改善了中文生物医学语料的质量。翻译好的中文数据集与英文数据集对比如表1所示,中英文实体对照表如表2所示。

表1 中英文生物医学数据集对比

表2 中英文实体对照表
2 生物医学实体识别
生物医学命名实体识别 (Bio-NER)是指从给定的非结构化医学文本中识别出相关的实体 (例如疾病、药物、蛋白质、症状等) 。生物医学实体识别过程主要包括以下两个部分: ①实体边界识别; ②实体类别确定。命名实体识别通常是知识挖掘、信息抽取的第一步,被广泛应用在自然语言处理领域。
由于词嵌入 (word embedding) 在自然语言处理任务中普遍获得很好的效果[8],所以几乎所有的自然语言处理任务中都会添加word embedding。目前常用的获取word embedding方法都是通过训练语言模型 (language model),将语言模型中预测的隐层状态(hidden state)作为词的表示,在给定N个字的序列(t1,t2,…,tN)中,前向语言模型就是通过前k-1个输入序列(t1,t2,…,tk-1)的hidden 表示,预测第k个位置的字,这种做法的缺点是对于每一个字都有唯一的embedding 表示,因此word embedding不能解决一词多义的问题。而在生物医学领域中同一个实体常常会具有不同的缩写和不同的命名方式。如果只使用word embedding作为模型的输入,这将会引起歧义,从而导致模型对于专业名词识别的效果不理想。所以本文在模型中添加了ELMo向量解决歧义问题。由于ELMo只预训练language model,word embedding 是通过输入的句子实时输出的,所以ELMo可以根据上下文单词的语义去调整单词的 word embedding 表示, 经过调整后的 word embedding 能表达单词在上下文中的具体含义, 从而缓解歧义问题,如图1所示。

图1 ELMo模型
本文在网上爬取大量的中文生物医学文本预训练中文ELMo。训练好网络后,输入的句子中的每一个字都有对应的三个embedding。
ELMo用到图1所示的双向语言模型,对于给定一个句子(t1,t2,…,tN), 前向计算方法,language model通过给定前面的k-1个位置的字序列计算第k个字的出现概率,如式(1)所示。
(1)
后向的计算方法与前向相似,如式(2)所示。
(2)
biLM训练的目标就是最大化下面的最大似然函数,如式(3)所示。
ELMo对于每个字, 通过一个L层的biLM计算出2L+1个表示,如式(4)所示。

(4)

具体应用时,将ELMo中所有层的输出R压缩为单个向量,通过一些参数来联合所有层的信息,如式(5)所示。
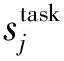
对于输入句子X,先将句子X输入预训练好的 ELMo 中,句子X中每个字在 ELMo 网络中都能获得对应的三个词嵌入,之后给予这三个词嵌入中的每一个词嵌入一个权重,这个权重可以学习得来。然后通过各自权重累加求和,将三个词嵌入整合成一个词嵌入。最后将整合后的这个词嵌入作为句子X在命名实体识别任务的网络结构中对应字的输入,本文将得到的词嵌入作为补充的新特征输入到 BiLSTM+CRF模型中。
本文的命名实体工作,首先将句子X输入预训练好的 ELMo,得到句子的向量表示,然后将其作为特征与句子的向量序列一起输入到Bi-LSTM中,用神经网络自动学习前向及后向的上下文特征,最后在BiLSTM后面增加一个条件随机场层进行句子级的序列标注。CRF层的参数是一个(k+2)×(k+2)的矩阵A(1)之所以要加2是因为要为句子首部添加一个起始转移状态以及为句子尾部添加一个终止转移状态,Aij表示的是从第i个标签到第j个标签的转移得分,进而在为一个位置进行标注的时候可以利用此前已经标注过的标签。结合了Bi-LSTM和CRF的命名实体识别,可以充分学习每个字的上下文信息及标签,从局部和全局两个层面,对词标签的分类实现更好优化,达到良好的实体识别效果。
3 生物医学关系抽取
生物医学关系抽取 (relation extraction, RE) 是指从一段生物医学文本中抽取出关系三元组 (entity1,relation,entity2)。以“利多卡因诱导的心脏停博”为例。其中“利多卡因”是实体1,实体类型为药物,“心脏停搏”是实体2,实体类型为疾病,实体之间的关系是“导致”关系,那么抽取的三元组为(利多卡因,导致,心脏停搏)。关系抽取是构建复杂知识库系统的重要步骤之一,它解决了原始文本中目标实体之间的关系分类问题。在传统方法中,大多数研究依赖一些现有的词汇资源(如WordNet)或手工提取的特征[9-10],这样的方法可能导致计算复杂度的增加,并且特征提取工作本身会耗费大量的时间和精力,特征提取质量对实验的结果也有很大的影响。由于注意力机制能够自动发现对分类起到关键作用的词, 使模型可以从每个句子中捕获最重要的语义信息,并且不依赖于任何外部的知识。因此,本文使用基于注意力机制的双向 LSTM 神经网络模型完成关系抽取任务。为了更好地提高关系抽取的结果,本文在模型的输入层添加了笔画特征,与词向量一起送入神经网络进行训练。模型结构如图2所示。

图2 关系抽取模型结构
生物医学关系抽取任务使用到的模型为长短期记忆模型(long short-term memory,LSTM)[11],LSTM是循环神经网络(recurrent neural network,RNN) 的一种。LSTM可以接受序列输入,产生对应的序列输出。不同时刻的输入之间存在着依赖关系。当前时刻的输出不仅取决于当前时刻的输入,还和上一时刻的输出有关。LSTM 具有门控机制,可以很好地解决 RNN 长距离依赖、梯度消失和梯度爆炸问题。双向长短期记忆循环模型由两个不同方向的LSTM组成,两个LSTM分别从前向和后向学习单词的上下文信息,再将二者拼接起来,作为当前时刻的输出。
本文中关系抽取的具体工作步骤如下: 首先使用预先训练好的词向量将词映射为100维词向量,随后将句子中的每个词与句子中的实体计算相对位置,从而得到位置信息。接着将句子的词向量、位置信息与笔画特征进行拼接,将得到的向量序列输入到 BiLSTM 中,用神经网络自动学习前向及后向的上下文特征。随后使用注意力机制,给重要的词较大的权重。最后在输出层使用Softmax来预测标签。
4 实验与结果分析
综上所述,本文构建了中文生物医学实体关系语料,并且人工校正2 000多条语料作为实验的测试集。数据集的统计信息如表3所示。除此之外,在中文生物医学的命名实体识别任务上,对比了几种模型的识别结果,本系统选择使用在BiLSTM+CFR模型上加 ELMo 特征作为最终的中文命名实体识别模型。

表3 中文数据集统计信息
命名实体识别模型的超参数设置如表4 所示。实验结果如表5所示。从实验结果可以看出,基于 ELMo+BiLSTM+CRF 的方法命名实体识别的F1值可以达到85.00%,在中文的数据集上,ELMo+BiLSTM+CRF 比目前最好的中文实体识别模型 Lattice LSTM[12]识别的效果要好,主要原因在于ELMo解决了一词多义的问题,使得性能提升。与加了BERT[13]的模型相比,也有一定的提升。主要原因在于BERT是使用通用语料进行训练的,缺乏生物医学领域的领域知识,而ELMo是使用大量的生物医学文本进行训练的,所以对于生物领域识别的结果会更好。从结果分析,所有的模型在疾病、化学物实体上识别的结果比DNA、RNA等实体上识别效果更好。归其原因还是由于DNA实体的特殊符号、字符和数字太多,导致模型识别的效果不佳。

表4 中文实体识别超参设置

表5 中文实体识别与关系抽取结果
在生物医学实体关系抽取任务上,本文使用目前流行的结合注意力(attention)机制的双向长短期记忆网络 (BiLSTM),并在输入层添加笔画特征,以提高关系抽取的性能。实体关系抽取模型超参设置如表6所示。最终的实验结果如表5所示。对于二分类的关系抽取,BiLSTM+ATTENTION+(特征)模型的F1值可以达到80.81%,比不加特征的模型提高了将近两个百分点。结果表明,我们设计的笔画特征确实可以提升模型在中文语料上的性能。

表6 中文实体关系抽取超参设置
本系统命名实体识别部分使用的模型为基于ELMo+BiLSTM+CRF的模型。关系抽取部分使用BiLSTM+ATTENTION+(特征)模型。图3 与图4 为生物医学文本信息抽取系统的展示,其中展示了一段医学文献的文本。因为本文使用语料的特殊性,目前无法与现有的关系抽取系统做比较,以后会进行相应的完善。本文构建的系统链接为: http://202.118.75.18:8893/precision_medicine/chinese/PM/index.html。

图3 基于中文的命名实体识别系统展示

图4 基于中文的关系抽取系统展示
5 总结与展望
本文在英文生物医学语料的基础上, 结合翻译技术与人工标注构建了中文生物医学的语料。 针对中文自然语言处理任务的特殊性, 本文搭建了基于深度学习的中文生物医学实体关系抽取系统,并且实现了流水式的命名实体识别和关系抽取。实验结果表明, 该信息抽取系统可以准确地识别实体边界和医学实体中的数字与符号, 并且可以准确地提取实体间的关系。现阶段研究中,各种预训练语言模型(如BERT[13]、RoBERTa[14]等)取得的巨大成功给NLP的发展带来了一波高潮,但是由于缺乏领域知识,导致通用领域的预训练语言模型对于特定领域的NLP任务有些乏力。在未来的研究工作中,我们希望在预训练语言模型中注入生物医学领域的领域知识,使得预训练语言模型处理生物医学领域的相关任务时,能取得更好的结果。

