许氏平鲉6个地理群体遗传多样性的微卫星分析
任建功,王青林,孙朝徽,于姗姗,王桂兴,于清海,刘 霞,宋立民,司 飞
( 1.中国水产科学研究院 北戴河中心实验站,河北 秦皇岛 066100; 2.河北科技师范学院 动物科技学院, 河北 秦皇岛 066600; 3.河北农业大学 海洋学院,河北 秦皇岛 066003; 4.天津市水产研究所,天津 300221 )
许氏平鲉(Sebastesschlegelii),属鲉形目、鲉科、平鲉属,别名黑鲪、黑头等,是广泛分布在我国渤海、黄海和东海近海海域的一种底层岩礁性鱼类[1-2]。许氏平鲉肉质新鲜细嫩、脂肪含量较少,具有生长速度快、抗逆性强、无远距离洄游优点,是北方海洋牧场和人工鱼礁区的主要经济物种,也是我国北方沿海、日本以及韩国重要的增养殖品种[3-7]。近十几年来,由于过度捕捞和环境破坏,许氏平鲉资源出现明显衰退[8]。为了恢复许氏平鲉渔业资源,我国早在20世纪90年代中期就在山东半岛沿海陆续开展小规模增殖放流工作,截至2009年累计放流许氏平鲉苗种452.4万尾。2010年以后,放流数量逐年增加,放流范围也扩大到河北、天津和江苏[9]。截至2017年,山东省累计放流许氏平鲉等岩礁鱼类2.58亿尾[10]。虽然增殖放流活动在促
进渔民增产增收和修复渔业种群资源方面起到了积极的作用[11],但大规模放流人工培育苗种对许氏平鲉的遗传多样性影响不容忽视[12-13],因此,亟需开展黄渤海许氏平鲉遗传多样性和遗传结构分析工作。
遗传多样性是生物多样性的重要组成部分,它是衡量一个种群种质资源品质的重要标准[14]。深入研究遗传结构与遗传多样性是科学制定资源保护与开发规划的基础[15]。微卫星标记,又称简单序列重复(SSR),在基因组中数量众多且分布均匀,具有多态性丰富、杂合度高和检测方便等优点,已经广泛应用于群体遗传多样性的研究[16-21]。目前利用微卫星标记对许氏平鲉遗传多样性比较的研究已有报道,王文琪等[17,22]分别利用18对和8对微卫星标记对4个山东许氏平鲉野生群体和9个中日
沿海的许氏平鲉野生群体进行了遗传多样性和群体遗传结构研究。但许氏平鲉放流群体对野生群体遗传多样性影响的相关报道较少,作为近年来增殖放流数量和范围逐渐增大的经济鱼种,其遗传多样性值得持续关注。鉴于此,笔者利用10对微卫星标记对来自辽宁丹东、营口,河北北戴河,山东长岛、威海和日照6个许氏平鲉地理群体的180尾个体进行遗传多样性和遗传结构分析,从分子遗传学角度对许氏平鲉增殖放流可能引起的遗传多样性影响进行初步评估,以期为我国许氏平鲉种质资源保护和增殖放流工作提供理论基础。
1 材料与方法
1.1 样品采集和DNA提取
2017年9月,在辽宁、山东等沿海采集许氏平鲉各30尾,6个地理群体共计180尾(图1)。剪取胸鳍平铺于采样纸上,37 ℃烘干后短期保存,待全部样本采集完毕后,使用海洋动物基因组DNA提取试剂盒(TIANGEN,DP324)提取基因组DNA。以质量浓度大于20 μg/mL、260 nm吸光度/280 nm吸光度(D260/D280)1.6~1.8为标准,用紫外分光光度计分别检测DNA的质量和纯度,未达到标准的样本重新提取。
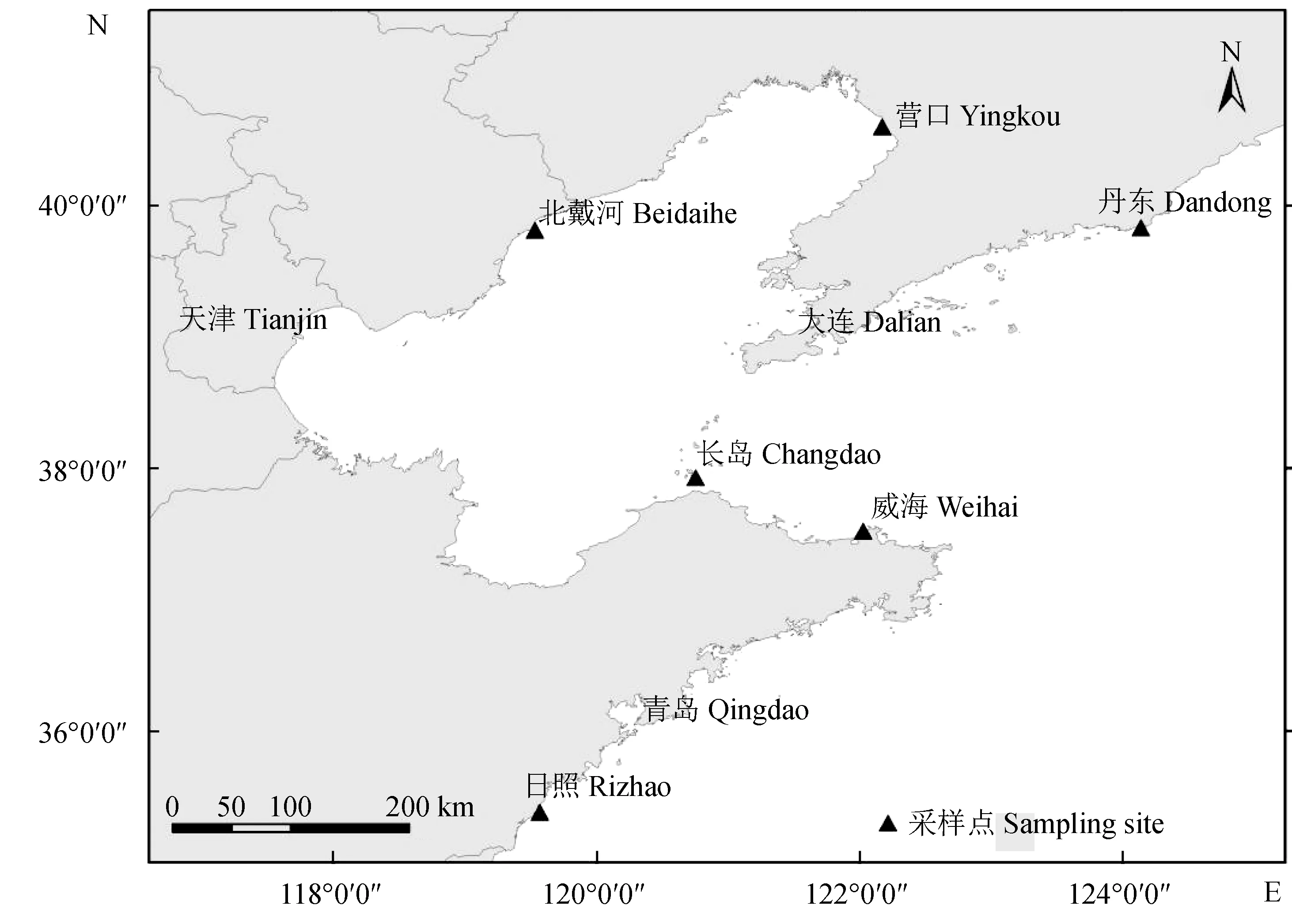
图1 许氏平鲉6个地理群体采样点(▲)Fig.1 Sampling sites (▲) of 6 geographical populations of Schlegel′s black rockfish S. schlegelii
1.2 微卫星分析
自文献[17,22]中的26对许氏平鲉微卫星标记中选取10对多态性高的标记用于本试验。各标记的名称、引物序列、退火温度等见表1,引物由生工生物工程(上海)股份有限公司合成。PCR反应体系为15 μL,包括模板DNA 1 μL,2×PCRmix 7.5 μL,上下游引物(10 μmol/L)各0.15 μL,纯水补至15 μL。PCR反应程序:94 ℃变性3 min;然后94 ℃ 20 s,54 ℃退火20 s,72 ℃延伸40 s,35个循环;最后72 ℃延伸3 min。PCR扩增在PE9700型PCR仪上进行。
将HIDI与分子量内标ROX500按20∶1的体积比混匀后,取9 μL加入上样板中,再加入1 μL稀释10倍的PCR产物。95 ℃变性3 min,立即冰水浴。然后使用ABI3730XL测序仪进行毛细管电泳,利用GeneMarker中的Fragment(Plant)片段分析软件对测序仪得到的原始数据进行分析,将各泳道内分子量内标的位置与各样品峰值的位置进行比较分析,得到片段大小。
1.3 数据分析
利用Genepop 4.0软件分析分型结果,统计各位点等位基因数、观测杂合度、期望杂合度和多态信息含量,并分析群体的哈迪—温伯格平衡状态,计算群体间各群体的基因分化系数、基因流、遗传相似性系数和Nei氏遗传距离。采用Arlequin 3.5软件对许氏平鲉6个地理群体进行分子方差分析。使用MEGA 5.0软件,采用非加权组平均法根据6个地理群体的遗传距离进行聚类,采用Structure 2.3软件进行遗传组分分析。
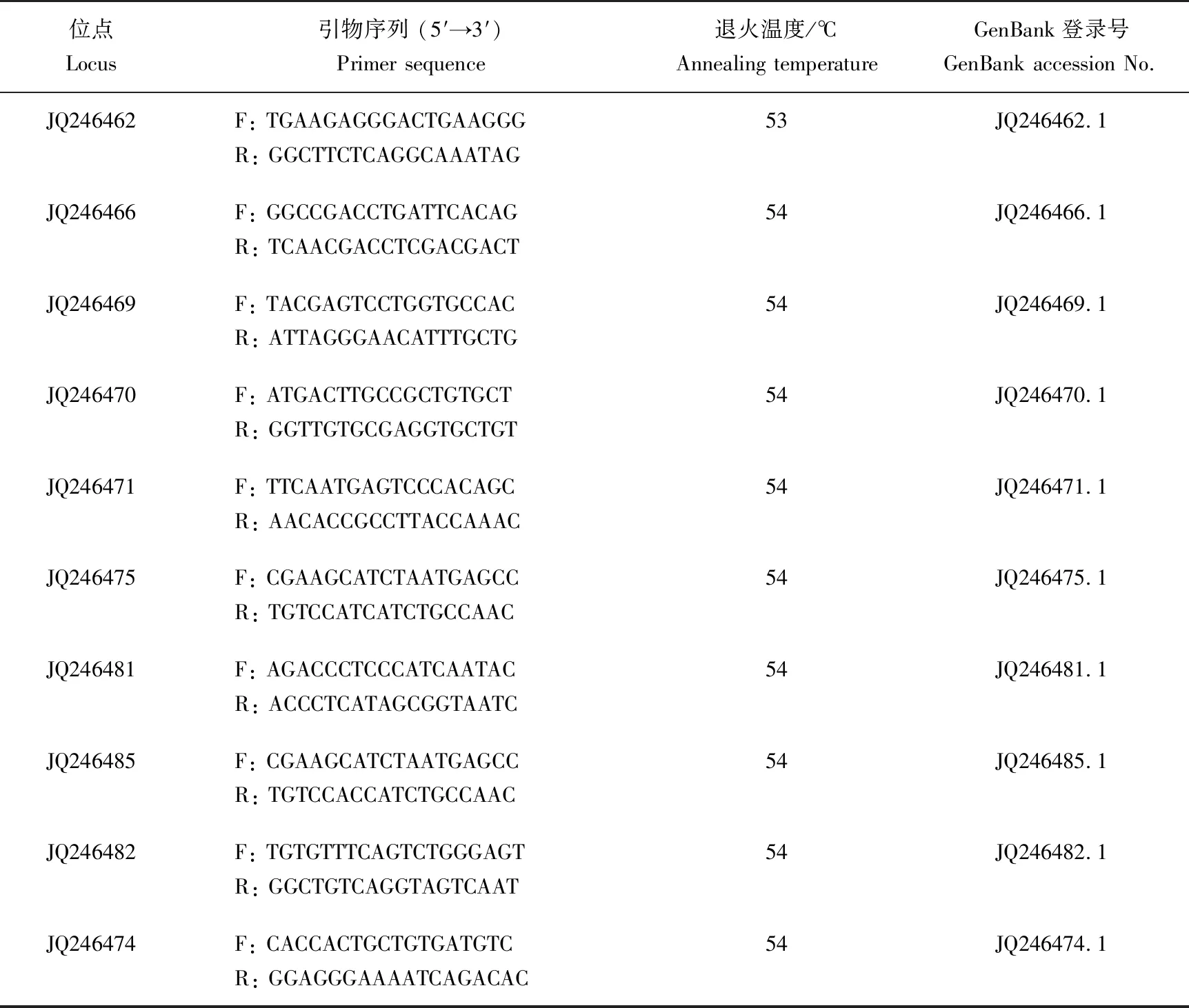
表1 10对微卫星标记的位点、引物序列、退火温度及GenBank登录号Tab.1 Locus, primer sequence, annealing temperature and GenBank accession No. of 10 microsatellitemarkers used in the experiment
2 结 果
2.1 遗传多样性参数
10对微卫星标记的遗传多样性参数等位基因数、观测杂合度、期望杂合度、多态信息含量见表2,6个地理群体的平均等位基因数为17.200~19.900个,平均观测杂合度为0.769~0.923,平均期望杂合度为0.871~0.920,平均多态信息含量为0.845~0.897。其中,北戴河群体的平均等位基因数最多,为19.900,丹东群体的平均等位基因数最少,为17.200。平均观测杂合度最大的是威海群体(0.923),最小的是北戴河群体(0.769)。平均期望杂合度最大的是长岛群体(0.920),最小的是北戴河群体(0.871)。哈迪—温伯格平衡检验结果显示,在6个群体10个多态微卫星位点中大部分未偏离平衡状态(P>0.05)。丹东群体有1个位点偏离平衡;北戴河、长岛、日照群体有3个位点偏离平衡;营口群体最多,有5个位点偏离平衡(P<0.05),威海群体没有偏离哈迪—温伯格平衡(表2)。

表2 许氏平鲉6个地理群体的哈迪—温伯格平衡检验及遗传多样性Tab.2 Genetic diversity and Hardy-Weinberg equilibrium (HWE) test in 6 geographical populations of Schlegel′s black rockfish S. schlegelii
2.2 遗传分化及聚类
6个群体各位点的基因分化系数为0.019~0.093(表3),位点JQ246469的基因分化系数最大(0.093),位点JQ246481的基因分化系数最小(0.019)。6个群体各位点的基因流为2.445~12.213(表3)。位点JQ246481的基因流最大,为12.213,位点JQ246469的基因流最小,为2.445。分子方差结果显示,主要的变异来源为群体内个体间(3.975%)和个体间(93.078%);而群体间变异所占比例较低,为2.947%(表4)。
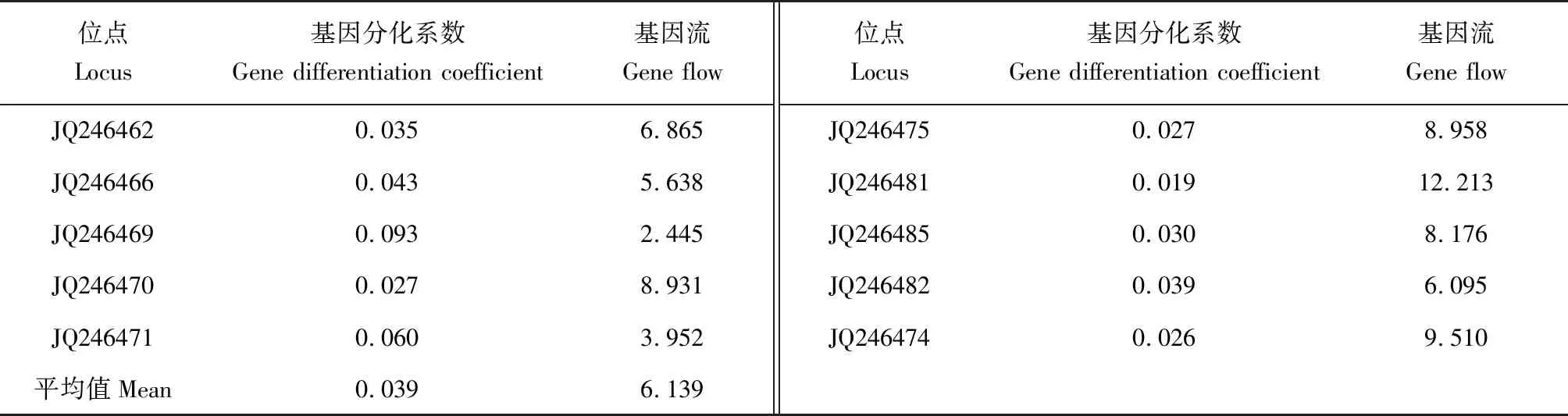
表3 许氏平鲉6个地理群体各位点的基因分化系数和基因流Tab.3 Coefficient of gene differentiation Gst and gene flow Nm among 6 geographical populations of Schlegel′s black rockfish S. schlegelii
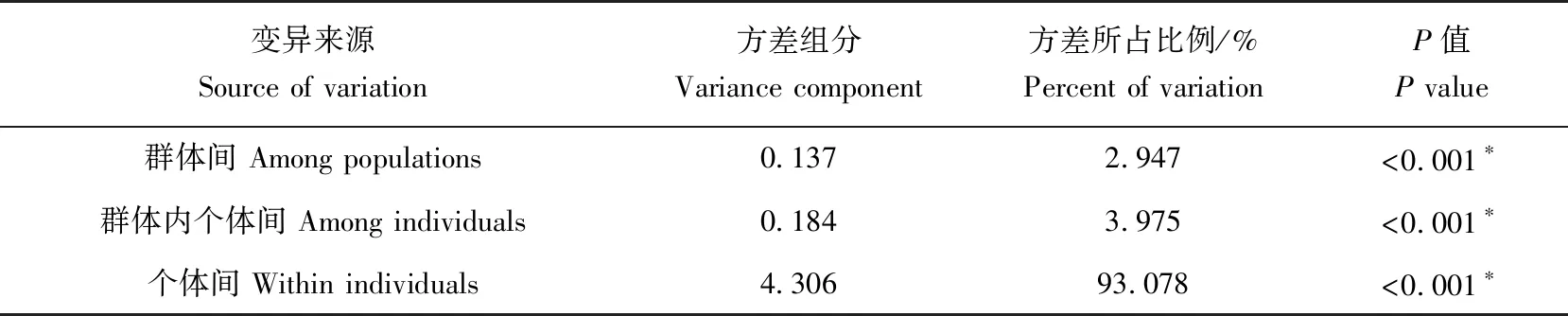
表4 许氏平鲉6个地理群体分子方差分析结果Tab.4 AMOVA among 6 geographical populations of Schlegel′s black rockfish S. schlegelii
6个群体相互间的遗传相似系数和Nei氏遗传距离见表5,分别为0.434~0.888和0.119~0.835。丹东群体和威海群体的遗传距离最近(0.119),而北戴河群体和营口群体的遗传距离最远(0.835)。根据各群体之间的遗传距离,对6个群体进行聚类(图2)。北戴河群体独立为1支;丹东群体和威海群体先聚成1支,再与长岛群体聚为1支;日照群体和营口群体聚为1支。

表5 许氏平鲉6个地理群体遗传相似性系数(对角线上方)和Nei氏遗传距离(对角线下方)Tab.5 Genetic identity (under diagonal) and Nei′s unbiased genetic distances (above diagonal) of 6 geographical populations of Schlegel′s black rockfish S. schlegelii
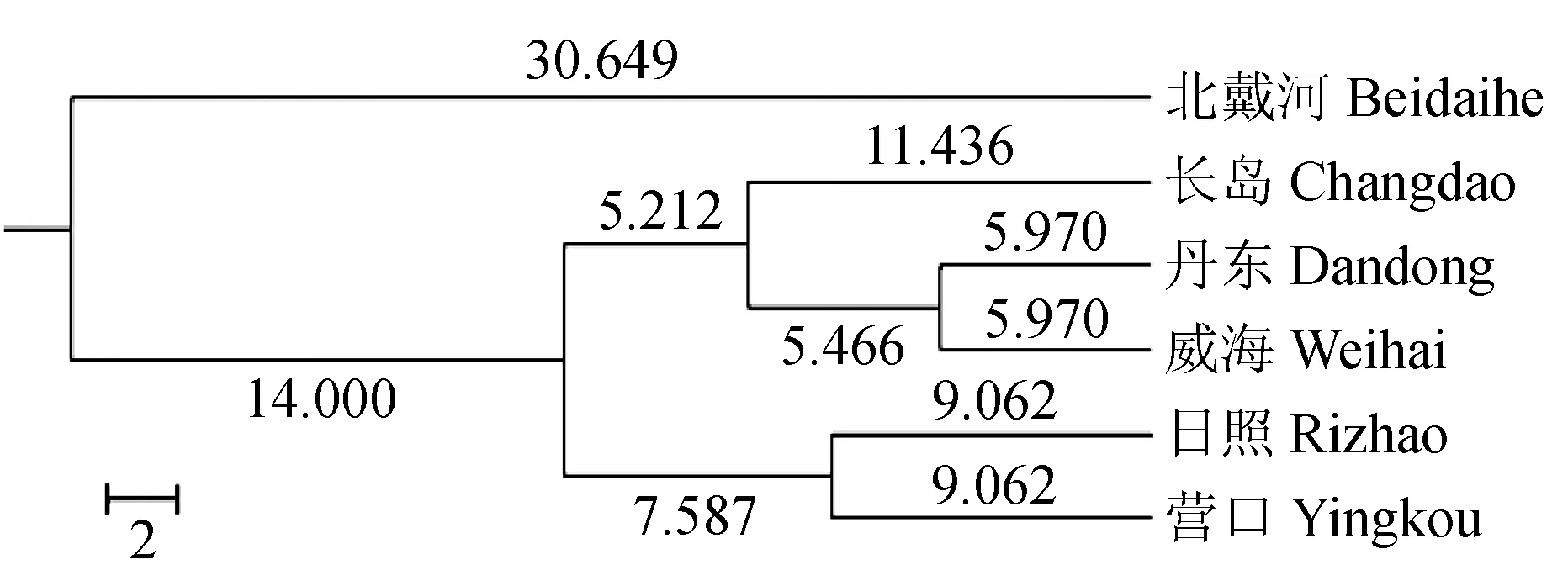
图2 根据Nei氏遗传距离用非加权组平均法构建的许氏平鲉6个 地理群体的进化树Fig.2 UPGMA dendrogram of 6 geographical populations of Schlegel′s black rockfish S. schlegelii based on Nei′s unbiased genetic distances
2.3 遗传组分分析
采用Structure 2.3软件进行遗传组分分析,6个许氏平鲉群体中共包含3种遗传组分(图3;红色、蓝色、绿色分别表示3种不同的遗传组分)。丹东群体和威海群体的遗传混杂最少,北戴河群体和长岛群体次之,营口群体和日照群体的遗传混杂较多。
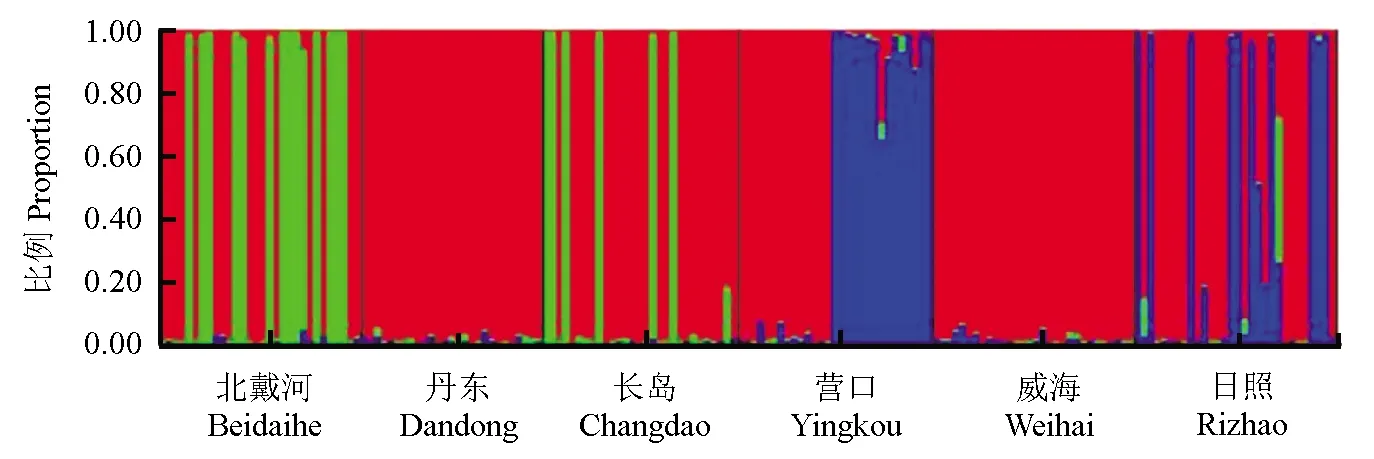
图3 6个许氏平鲉地理群体的遗传组分分析Fig.3 Genetic component among 6 geographical populations of Schlegel′s black rockfish S. schlegelii
3 讨 论
3.1 许氏平鲉不同地理群体的遗传多样性分析
多态信息含量是衡量标记遗传信息含量高低的主要参数,当多态信息含量>0.5时,表明该遗传标记具有高度的可提供遗传信息性,即高度多态[23]。本研究中,各标记的多态信息含量为0.668~0.959,表明所选用的微卫星标记具有高度多态性,可以用于许氏平鲉的遗传多样性分析。等位基因数和期望杂合度是分析群体遗传多样性的主要指标[24]。本研究中,各标记在6个群体中的平均等位基因数为17.200~19.900,各群体等位基因数排序为北戴河(19.900)>营口(19.700)>长岛(19.400)=日照(19.400)>威海(18.600)>丹东(17.200)(表2)。王文琪等[17]对荣成1、荣成2、即墨、龙口4个群体进行微卫星分析,17个微卫星位点中每个位点检测到的等位基因数为2~10个,低于本研究结果,推测可能因为使用不同微卫星标记的原因。张辉[22]研究表明,从丹东、烟台、威海和日照采集的许氏平鲉野生群体的等位基因数为11.800~13.500,结果与本研究基本相符。较高的等位基因数表明许氏平鲉各群体仍具有较高的遗传多样性丰富度。期望杂合度是反映微卫星标记在群体中遗传变异程度的最优参数,是描述遗传多样性的最适参数,杂合度越高,表明群体内遗传多样性就越高,遗传变异程度就越大,反之则群体内遗传变异程度就小[25]。本研究期望杂合度在6个群体中的变化为0.871~0.920 (表2),各群体排序为长岛(0.920)>丹东(0.905)>日照(0.904)>威海(0.903)>营口(0.882)>北戴河(0.871);王文琪等[17]分析,荣成等4个野生许氏平鲉群体的期望杂合度为0.447~0.590,张辉[22]研究表明,丹东等4个野生许氏平鲉群体的期望杂合度为0.6848~0.8643;三者结果基本一致。王文琪等[17]研究样本采集于2009年5—8月,张辉[22]研究样本采集于2008年4月—2009年4月,本研究样本采集于2017年9月,三者采样时间最长间隔9年6个月,但三者研究结果均显示,许氏平鲉的遗传多样性较高,说明近十年来大规模放流人工培育苗种尚未对许氏平鲉遗传多样性带来较为明显的影响。
3.2 许氏平鲉不同地理群体遗传结构差异分析
基因分化系数是反映群体间遗传分化程度的重要参数,基因分化系数在0~0.05之间变化,表明群体的遗传分化较弱,为轻度遗传分化[18]。本研究中,6个群体不同位点的基因分化系数为0.019~0.093,平均值为0.039,说明6个群体之间的遗传分化较弱,与张辉[22]的研究结果一致。分子方差分析结果表明,主要的变异来源为群体内个体间(3.975%)和个体间(93.078%);而群体间变异所占比例较低,仅为2.947%。基因流与基因分化系数呈负相关,即基因流越大,群体间相似性越大,遗传分化越小[14,18]。本研究各位点平均基因流为6.139,说明6个群体之间基因交流比较充分,群体间遗传分化较小,与本研究中基因分化系数相关结果相互支持。群体之间充分的基因交流可能归因于黄海暖流和中国沿岸流等海流驱动下的许氏平鲉仔稚鱼随漂流海藻团的扩散[22,26]。哈迪—温伯格平衡检验结果显示,5个群体15个位点显著偏离哈迪—温伯格平衡状态(P<0.05),这些位点多数表现出显著的杂合子缺失,与王文琪等[17]的研究结果相似。
Nei氏遗传距离构建的非加权组平均法系统进化树显示,北戴河群体独立为1支,与其他群体的遗传距离相对较大,与丹东、威海、日照分属黄海、渤海2个海区且距离较远相符;丹东群体和威海群体先聚为1支,与丹东、威海距离较近且属同一海区相符;这两群体再与地处黄渤海交界的长岛群体聚为1支,与3个群体距离较近相符;这3个群体再与日照群体聚为1支,与其距离相近或属同一海区基本相符。不同群体许氏平鲉遗传分化结果与其所处的地理位置及其所在海区具有一定的相关性。但日照和营口分属黄海、渤海2个海区,且距离较远,2个群体却聚为1支,北戴河群体与营口群体同属渤海海区且距离较近却最后聚为1支。经调查,营口群体采样所在码头存在销售外地渔获物现象,推测可能因此导致营口群体与日照群体聚为1支,但具体原因有待进一步研究。张辉[22]研究指出,分析的群体共享同一个基因库,即所有个体属于1个亚群,但是本试验中所分析群体分为3个亚群,主要是因为本研究分析的群体包含了北戴河群体和营口群体,北戴河群体和其他几个群体的遗传相似性系数相对较低,遗传距离相对较大。丁奎等[8]利用线粒体DNA控制区高变区片段对许氏平鲉野生群体和养殖群体进行了遗传多样性对比,发现养殖群体和野生群体之间以及养殖群体之间的遗传分化较大,而野生群体间遗传变异较小,其研究的野生群体组成与张辉[22]的研究结果基本相同。
4 结 论
根据本研究结果,结合历史研究数据分析,目前黄海、渤海许氏平鲉的遗传多样性仍处于较高水平,暂未受到大规模放流人工培育苗种的影响。但为了降低大规模放流可能带来的遗传多样性影响,笔者建议及时从放流地点附近海域捕捞野生许氏平鲉亲本来更新放流用亲鱼,使放流亲本保持较高的遗传多样性,进而提高放流群体遗传多样性,其对渔业资源及生态环境保护具有重要意义。

