肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析*
刘郴郴, 宋正帅, 章小平
华中科技大学同济医学院附属协和医院 1泌尿外科 2泌尿外科研究所,武汉 4300223 华中科技大学同济医学院附属武汉中心医院泌尿外科,武汉 430014
肾癌是泌尿系统最常见的恶性肿瘤之一,其中又以肾透明细胞癌(clear cell renal cell carcinoma,ccRCC)占大多数(75%)[1-2]。约30%的肾癌患者在初诊时发现转移病灶以及30%的患者在行根治性肾切除术后发现复发是造成患者不良预后的主要原因[3-4]。目前针对进展期肾癌,随机临床试验证实靶向血管内皮生长因子(VEGF)等通路的抗血管生成药物,包括舒尼替尼、帕唑帕尼等,能够有效缓解疾病进展,并已得到广泛应用[5]。然而,目前仍缺乏能有效预测ccRCC靶向药物耐药性的分子标记物,因此,寻找和确认相应的分子标记物以预测疗效,探索耐药机制,是精准治疗的关键[1]。近年来,随着高通量芯片和生物信息学技术的发展,我们可以获取和分析肿瘤、肿瘤毗邻组织和正常组织在多种基因表达水平上的差异,从而发现肿瘤发展、侵袭、转移、复发以及靶向药物耐药等表型中的潜在分子机制。本研究基于TCGA、GEO等公共数据库,利用在线生物信息学工具和软件对数据进行筛选、分析、整理,得到ccRCC对舒尼替尼获得性耐药的分子标志物,为患者预后判断及治疗选择提供依据,并对其中潜在的信号通路及分子机制进行了探讨,为后续通过实验及临床数据进行进一步验证奠定了基础。
1 材料和方法
1.1 微阵列数据
从GEO数据库中下载基因表达谱GSE76068。根据安捷伦GPL 10558平台(Illumina Humanht-12 v4.0表达芯片)提供的信息,GSE76068中共有24个样本,包括8个未处理的ccRCC样本,8个舒尼替尼敏感样本及8个舒尼替尼耐药样本。GSE76068中包含这24个样本的全基因组mRNA表达数据。
1.2 筛选差异表达基因(DEGs)
利用基于R语言的GEO数据便捷在线分析工具GEO2R(https://www.ncbi.nlm.nih.gov/geo/GEO2r/),通过将“adj.P<0.05”和“|logFC|>1”作为分界标准,得到舒尼替尼敏感样本和舒尼替尼耐药样本之间的DEGs。
1.3 蛋白-蛋白互作网络(PPI)及模块分析
基于相应DEGs蛋白之间物理和功能联系,利用相互作用基因检索工具(STRING,https://string-db.org/cgi/network.pl)构建了PPI。同时根据最大交互数量≤5个,置信度≥0.4分的标准,用STRING绘制并筛选出了10个枢纽基因。此外,利用分子复合体检测(MCODE)插件,按照节点得分值=0.2,K值≥2,最大深度=100,在Cytoscape软件中构建了各模块的PPI网络。
1.4 DEGs和枢纽基因的GO和KEGG通路分析
利用DAVID(https://david.ncifcrf.gov/summary.jsp)数据库对DEGs和枢纽基因的基因本体(GO)和京都基因与基因组百科全书(KEGG)信号途径进行分析。DAVID是一个免费开放的在线工具,为大规模的基因或蛋白质列表提供了系统全面的生物注释信息[6]。统计偏差的截止标准为P<0.05。
1.5 基因集富集分析(GSEA)
为寻找ccRCC舒尼替尼耐药的潜在生物学功能和途径,进行了GSEA分析。16个ccRCC样本基因表达信息从GSE76068下载后,按舒尼替尼敏感和舒尼替尼耐药分为2组。选择Annotated gene sets c2.cp.v6.2.symbols.gmt作为参考基因集,Illumina HumanHT-12V4.0表达芯片作为芯片平台,并以P<0.05、FDR<0.25、基因大小≥100为分界标准。
1.6 枢纽基因的诊断、预后和临床病理价值分析
从TCGA数据库中获取534例ccRCC患者总生存期(OS)和无病生存期(DFS)信息,以及患者年龄、性别、肿瘤TNM分期和G分级等临床信息(无特殊筛选标准),通过GraphPad Prism 6.0软件获得Kaplan-Meier曲线,评估枢纽基因的预后价值。并通过受试者工作特征(ROC)曲线分析,确定这些枢纽基因对ccRCC的诊断价值。综合上述结果,重点评估靶基因ISG15的临床病理价值。
1.7 统计学方法
本研究采用GraphPad Prism 6.0(GraphPad Software,Inc,USA)和SPSS 22.0(IBM SPSS,Chicago,IL)为统计分析软件,各组数据以平均值±标准差表示,以P<0.05为差异具有统计学意义。采用非配对t检验分析ccRCC与正常肾组织之间枢纽基因表达的差异;通过Mann-Whitney检验统计目标基因和肾癌患者临床病理参数之间的联系;通过Kaplan-Meier曲线和对数秩检验计算基因表达水平与患者OS、DFS的相关性,再结合OS和患者性别、年龄、TNM分期等变量做单因素Cox分析,并将筛选到的显著相关变量进行多因素Cox比例风险回归分析。
2 结果
2.1 筛选DEGs
在GSE76068中,共有8个舒尼替尼敏感和8个舒尼替尼耐药的ccRCC样本。以P<0.05、|logFC|≥1为阈值,通过GEO2R分析出DEGs,其中包括表达上调基因38个,表达下调基因57个。
2.2 枢纽基因筛选与PPI网络构建
根据连接度由高到低确定了10个枢纽基因,包括IL6、MX1、ISG15、IFIT1、OAS1、OAS2、IFIT3、IFI27、RSAD2、OASL,其连接度依次为22、17、16、15、15、15、15、15、14、14。有趣的是,我们发现一半的枢纽基因属于干扰素相关的DNA损伤抗性标志基因(IRDS)[7]。根据STRING查询的蛋白信息,我们分别构建了所有DEGs和前10位枢纽基因的PPI网络(图1A、1B);此外,Cytoscape中的MCODE插件识别出PPI网络中的前3个重要模块(图1C~1E)。

A:DEGs互作网络;B:枢纽基因互作网络;C:模块1;D:模块2;E:模块3
2.3 功能富集分析
本研究通过DAVID在线平台完成了GO功能和KEGG通路富集的分析。表1、2显示了DEGs的前5位基因本体类别。在生物过程(BP)方面,上调的DEGs主要富集在 Ⅰ 型干扰素信号通路、对病毒的防御反应;在分子功能(MF)方面,上调的DEGs主要涉及2′-5′-oligoadenylate合成酶活性、双链RNA结合、转移酶活性。此外,细胞成分(CC)分析显示,它们与胞质、线粒体有关。而KEGG途径主要富集在甲型流感、麻疹、单纯疱疹病毒感染和肠道免疫网络中产生IgA(表3)。其中图2A、2B给出了这些DEGs的GO和KEGG通路富集图。为了更全面地了解这些DEGs,我们对前10位枢纽基因进行了GO功能和KEGG通路富集。有趣的是如表4、5、6所示,这些枢纽基因的BP和KEGG通路富集在Ⅰ型干扰素信号通路、对病毒的防御反应、对病毒的反应、甲型流感、麻疹、单纯疱疹病毒感染等方面,与上调的DEGs和第1位模块基因的结果基本一致。

表1 与ccRCC舒尼替尼耐药相关上调表达基因GO分析Table 1 Gene ontology analysis of upregulated differentially expressed genes associated with sunitinib resistance

表2 与ccRCC舒尼替尼耐药相关下调表达基因GO分析Table 2 Gene ontology analysis of downregulated differentially expressed genes associated with sunitinib resistance
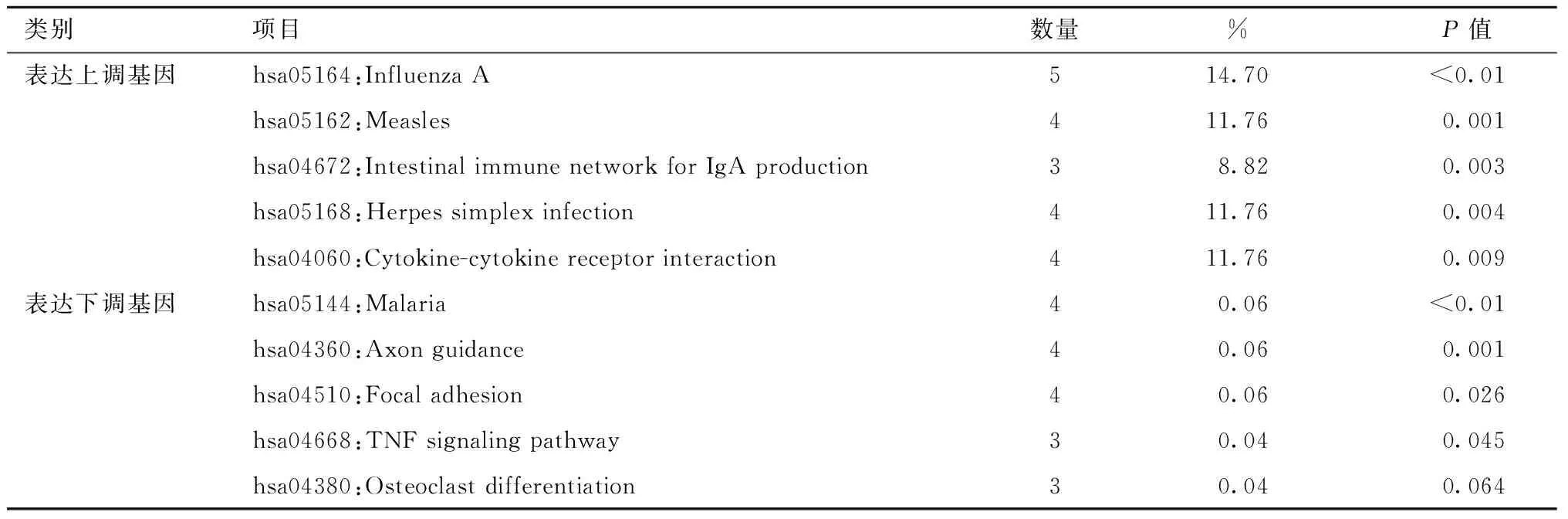
表3 与ccRCC舒尼替尼耐药相关差异表达基因KEGG分析Table 3 KEGG pathway analysis of differentially expressed genes associated with sunitinib resistance
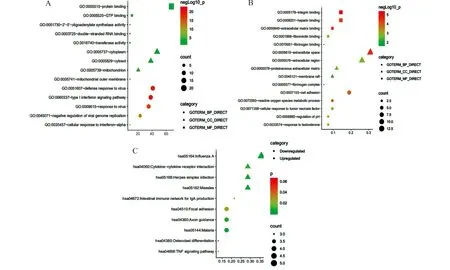
A:上调DEGs的GO分析;B:下调DEGs的GO分析;C:DEGs的KEGG通路分析

表4 与ccRCC舒尼替尼耐药相关枢纽基因GO分析Table 4 Gene ontology analysis of hub genes associated with ccRCC sunitinib resistance
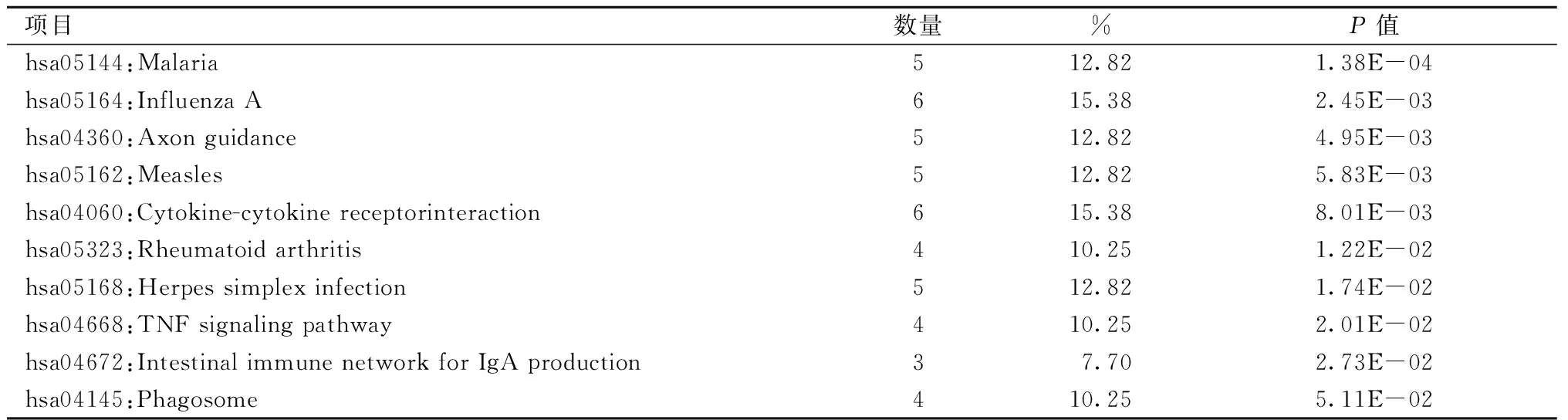
表5 与ccRCC舒尼替尼耐药相关的枢纽基因KEGG分析Table 5 KEGG pathway analysis of hub genes associated with ccRCC sunitinib resistance

表6 模块1 KEGG通路分析Table 6 KEGG pathway analysis of top1 module
2.4 基因集富集分析
为深入了解GSE76068样本中导致舒尼替尼耐药的潜在生物学途径和机制,我们进行了GSEA分析。根据截止标准FDR<0.25,P<0.05,显示了12个功能基因组(图3)。该结果表明,舒尼替尼耐药组富集在胞质DNA感知通路、Toll样受体信号通路、RIG-I受体信号通路、干扰素信号通路、激活ATR应对复制应激、G2/M检查点等。
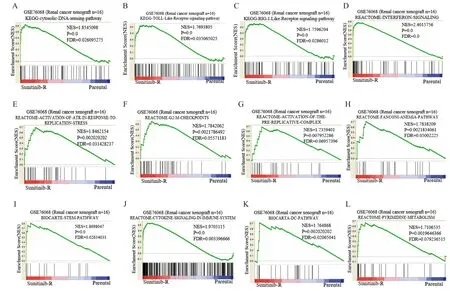
Parental:舒尼替尼敏感组;Sunitinib-R:舒尼替尼耐药组;以P<0.05且FDR<0.25为显著富集
2.5 Kaplan-Meier生存分析
在Kaplan-Meier分析的基础上,分析了枢纽基因的表达与ccRCC患者OS、DFS之间的关系。结果显示,IL6(HR=2.360,log-rankP<0.01)、ISG15(HR=1.902,log-rankP<0.01)、OASL(HR=1.842,log-rankP<0.01)的高表达预示ccRCC患者更差的OS;IL6(HR=1.387,log-rankP=0.0463)、ISG15(HR=1.74,log-rankP=0.0052)、OASL(HR=2.195,log-rankP=0.0001)的高表达预示ccRCC患者更差的DFS(图4、图5)。而IFIT1的表达和更好的OS(HR=0.5598,log-rankP=0.0001)和DFS(HR=0.6368,log-rankP=0.0112)相关。

患者总生存期信息来自TCGA数据库;A:IFIT1;B:IL6;C:ISG15;D:OASL
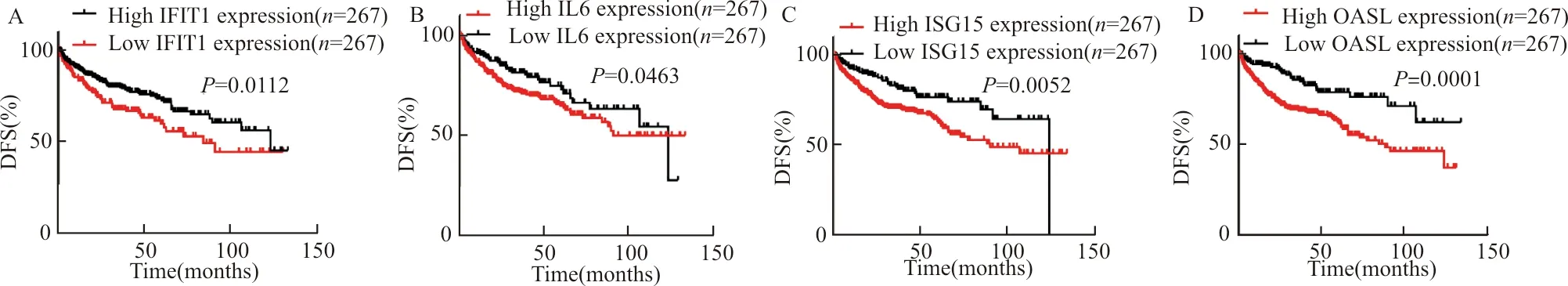
患者无病生存期信息来自TCGA数据库;A:IFIT1;B:IL6;C:ISG15;D:OASL
2.6 枢纽基因的受试者工作特征(ROC)曲线分析
我们通过ROC曲线来评估枢纽基因是否具有潜在诊断价值。如图6,所示基因可以完全区分ccRCC和配对的正常组织,曲线下面积(AUC)为0.7362~0.8914。此结果表明,这些枢纽基因可能是ccRCC患者的有效诊断生物标志物。
2.7 枢纽基因在ccRCC组织和正常肾组织中的表达
利用TCGA数据库对ccRCC和正常组织中枢纽基因的表达水平进行评估。如图6,与正常组织相比,其中部分基因如ISG15、IFIT3、OASL在ccRCC组织中的表达明显上调。基于ISG15较好的诊断和预后价值,我们进一步研究了它和ccRCC患者临床病理参数之间的相关性。
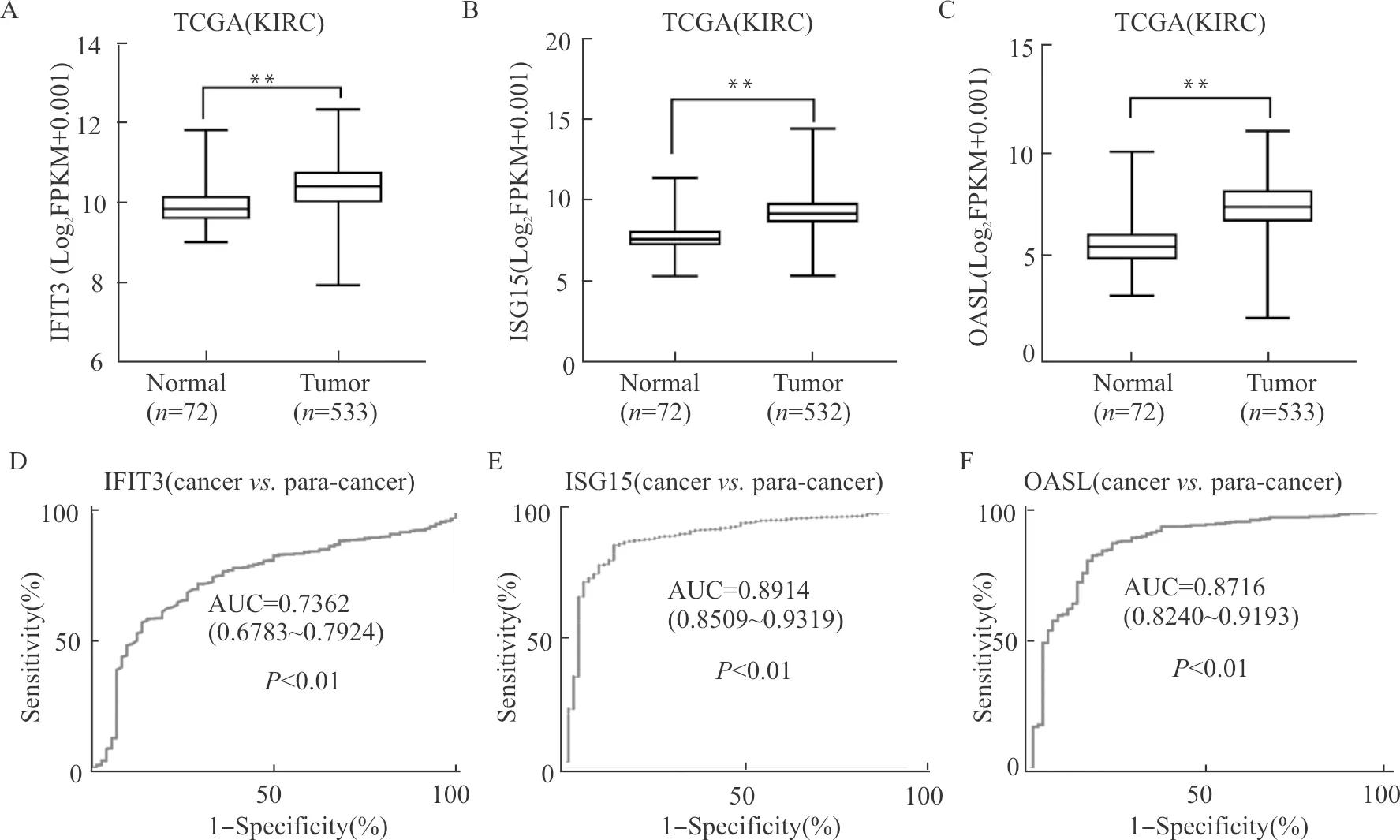
A~C:基因表达水平分析;D~F:基因表达ROC曲线分析;患者基因表达水平信息均来自TCGA数据库;**P<0.01
2.8 ISG15的表达与ccRCC的各种临床病理参数相关
对TCGA数据库中534例包含较完整信息的ccRCC样本数据(其中2例缺少T、N分期,4例缺少M分期,10例缺少G分级信息)分析显示,ISG15 mRNA表达上调与较高的病理分期分级、远处转移有显著的相关性(图7),ISG15的表达水平随着肿瘤病理分期分级升高有增高的趋势。患者临床病理资料见表7,依据ISG15表达水平降序排列,取中位均分为ISG15高表达和低表达两组。ISG15的高表达与这些临床病理参数之间有显著的相关性,与上述结果一致。
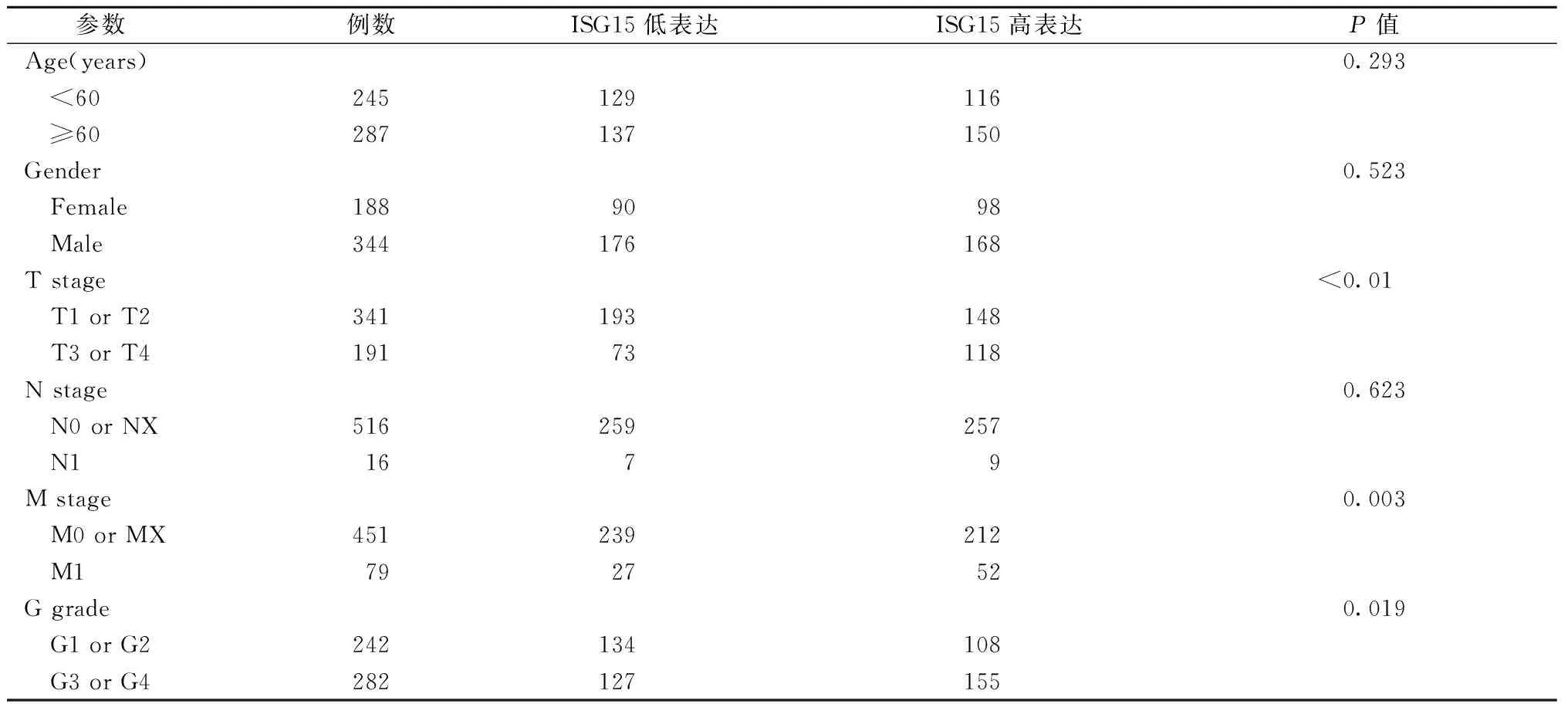
表7 ccRCC患者ISG15 mRNA表达水平与临床病理参数的关系Table 7 Association between ISG15 mRNA expression and clinicopathological parameters of patients with ccRCC

A:T分期;B:淋巴结转移;C:远处转移;D:G分级;**P<0.01
为了进一步探讨ISG15的预后价值,我们评估了其表达水平和患者各种临床病理因素与OS相关性。单因素和多因素Cox比例风险回归分析表明,ISG15高表达是ccRCC患者OS的独立风险因素,可作为独立预后指标(表8)。
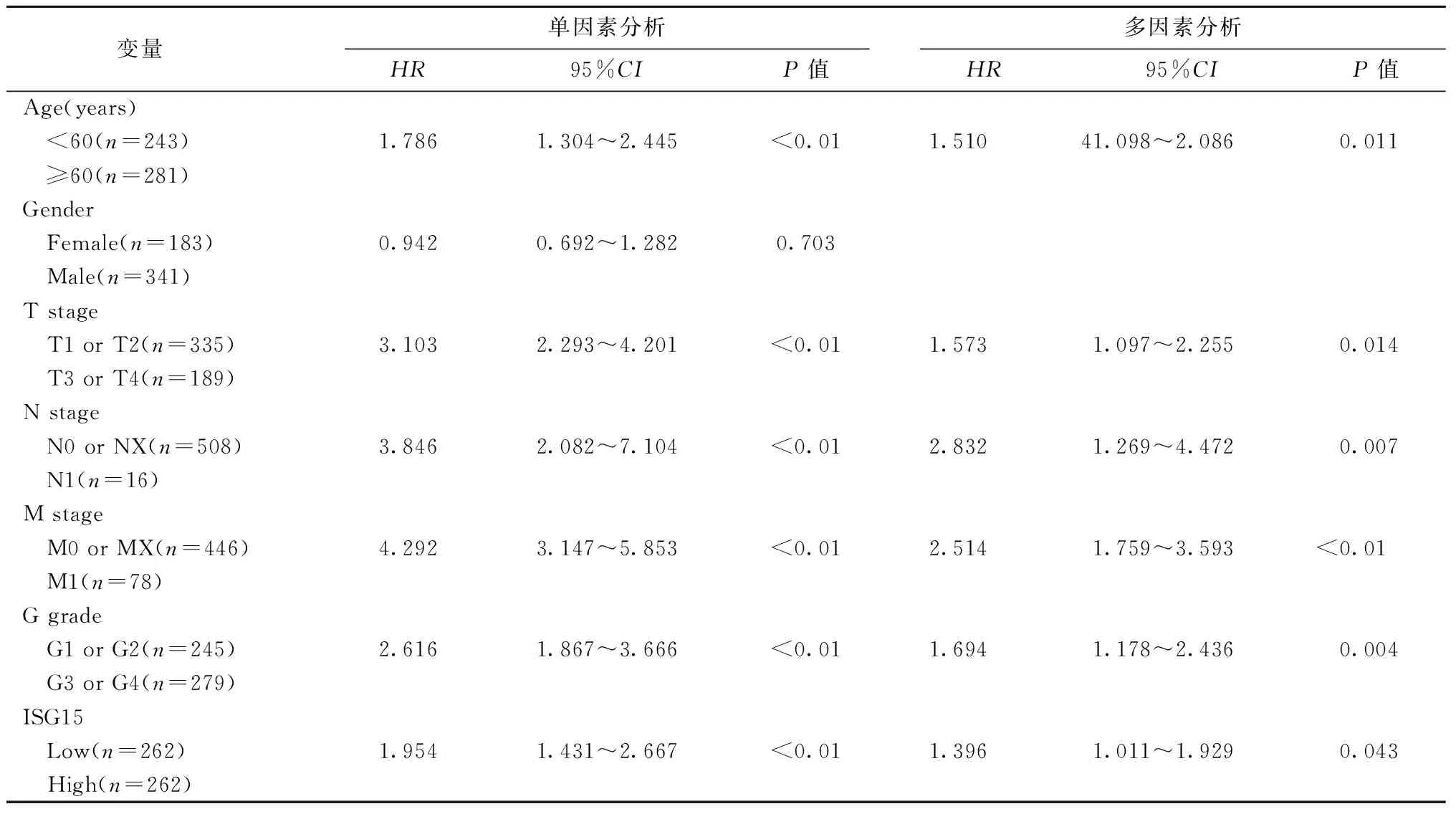
表8 ISG15 mRNA表达水平与患者总生存期的单因素和多因素回归分析Table 8 Univariate and multivariate analyses of ISG15 mRNA expression and patient overall survival
3 讨论
ccRCC是最常见的肾癌亚型,酪氨酸激酶抑制剂(tyrosine kinase inhibitor,TKI)类靶向药物,如舒尼替尼仍是进展期ccRCC治疗的基石[2,8]。事实证明,由于个体差异及肿瘤异质性等因素,不管放化疗、靶向治疗还是免疫治疗,总存在着一部分患者先天不敏感或对持续治疗耐受。所以,研究ccRCC舒尼替尼耐药的分子标志物和功能途径对ccRCC患者的治疗方式选择及生存预后评估具有重要意义。本研究利用多种生物信息学工具发现了一组具有代表意义的分子标志物,有望成为诊断甚至治疗靶点应用于临床,此外我们还分析了其中枢纽基因的诊断和预后价值,并探讨了导致ccRCC舒尼替尼耐药的潜在信号通路。
3.1 DNA感知器-Ⅰ型干扰素通路-IRDS轴可能导致ccRCC获得性耐药
到目前为止,已有大量文献报道肾癌舒尼替尼耐药相关机制,包括TKIs被溶酶体螯合、缺氧诱导因子(HIF)等关键基因的突变和修饰、血管生成替代途径(AKT/mTOR)的激活、肿瘤异质性和微环境等,但仍缺乏共识[9]。
在本研究中,GSEA分析表明DNA损伤反应相关基因集(激活ATR应对复制应激和G2/M检查点)得到显著富集。同时,根据GO/KEGG分析,我们发现生物过程(Ⅰ型干扰素信号通路、对病毒的防御反应)、信号通路(甲型流感、麻疹和单纯疱疹病毒感染)和基因集(干扰素信号传导)均得到富集(图2A、2B、3D),最重要的是,GSEA结果表明胞质DNA感知通路、Toll样受体信号通路、RIG-I受体信号通路等经典DNA感应通路均被显著富集。据此,我们认为肿瘤细胞通过靶向药物治疗,在复制应激等因素下导致DNA损伤,DNA损伤产物被DNA感应蛋白感知并激活了干扰素信号通路(如cGAS-STING信号通路)。
在过去的几十年中,研究发现Ⅰ型干扰素(IFN-Ⅰ)来源于各种免疫细胞或肿瘤细胞,作为调节数百种下游细胞因子转录的强大免疫调节因子,参与肿瘤免疫、感染调控和组织损伤等过程。一般来说,IFN-Ⅰ和Ⅰ型干扰素途径对抑制感染和杀伤肿瘤细胞尤为重要,因此在20多年前,大剂量干扰素被用于治疗转移性肾癌,但疗效不甚理想,而且由于它的副作用使其并没有被广泛应用。目前,一些临床前试验已经证明了IFN-Ⅰ定向治疗肿瘤的可取性,但更多的是需要与其他疗法,如靶向治疗、放化疗和新型免疫疗法联合使用,才能取得更好的效果。然而,近期研究结果表明,Ⅰ型干扰素通路在肿瘤进展和获得性耐药中起着至关重要的作用,尤其是在长期和持续暴露于IFN-Ⅰ的情况下,干扰素相关DNA损伤抗性标志基因(IRDS)明显上调并发挥促癌促耐药作用[10-14]。例如JAK抑制剂Ruxolitinib可以克服非小细胞肺癌对顺铂的耐药性[15]。此外,cGAS/RIG-I/TLR4等DNA感应蛋白作为明星分子,在免疫调节途径中对癌症的进展和治疗具有决定性的影响,但与Ⅰ型干扰素信号通路一样,DNA感应蛋白也具有两面性。有研究表明,DNA感应蛋白DDX41是肿瘤启动因子同时也是放疗的不良预后因子[16];另一项研究调查了在基因毒药物作用下,TLR4通路的激活和下游产物的上调保护了残余肿瘤细胞,并促进了肿瘤的转移[17]。
有趣的是本研究中我们筛选的大部分枢纽基因如ISG15、OASL、IFIT3在舒尼替尼耐药样本中上调,均属于IRDS基因,且这个独特的基因子集正是DNA感应和Ⅰ型干扰素信号通路的下游基因。综上所述,我们认为在舒尼替尼的作用下,ccRCC中细胞核酸的积累被DNA感应蛋白所感知,并引发了Ⅰ型干扰素通路的激活,继而导致IRDS的上调,促进了肿瘤获得性耐药。
3.2 IRDS是ccRCC获得性耐药的潜在分子标志
Ⅰ型干扰素启动了数百个干扰素下游基因的表达,涉及抗病毒、抗肿瘤和抗炎功能[18]。越来越多的研究表明,在抗病毒反应的第二阶段,在持续低剂量IFNβ刺激下,未磷酸化的转录因子ISGF3(U-ISGF3)高表达,该转录因子进一步介导一个特殊的干扰素刺激基因子集(ISGs)上调,发挥抵抗DNA损伤和延长抗病毒效果的作用[7,12,19-22]。耐人寻味的是,正是该基因子集中的一部分驱动了放化疗的获得性耐受,并被统称为干扰素相关DNA损伤耐药基因(IRDS),且已经作为预测乳腺癌、头颈部肿瘤等恶性肿瘤的放化疗预后因子应用于临床[7,10,12,23]。
到目前为止,已经发现了49个以上的基因属于IRDS基因。从我们富集的生物信息学结果来看,IRDS基因在舒尼替尼耐药组中显著上调。如前所述,越来越多的证据表明,细胞DNA损伤产物被cGAS/RIG-I/TLR4等感应蛋白感知,然后激活干扰素通路,促进IRDS的表达[14,24-26]。随后,该轴的激活所引发的IRDS上调通过DNA损伤修复和其他分子机制促进了肿瘤的生存[13,27]。因此我们有理由相信该过程同时促进了ccRCC的获得性耐药,但还需要更多的实验证据。我们的研究为IRDS基因ISG15、OASL、IFIT3可作为肾癌获得性耐药的有效分子标志物及预后指标提供了线索。
3.3 ISG15是ccRCC潜在的诊断和预后标志物
ISG15,即干扰素刺激基因15,1987年作为泛素样蛋白(Ubls)被发现,在各种细胞活动如稳定蛋白、调节细胞周期、应激反应和信号转导中发挥了重要作用,类似于泛素化,其与蛋白结合的方式被称为ISGylation,是未被充分开发的翻译后蛋白修饰途径(PTMs)之一。几十年来,对ISG15的研究一直集中在抗病毒复制方面。直到最近,研究人员发现ISG15在乳腺癌、前列腺癌和胰腺癌等肿瘤发病、进展和耐药中发挥了重要作用[7,13,28-30]。本研究首次探讨了ISG15在ccRCC中的表达和临床病理参数之间的联系。我们发现,与正常肾组织相比,ISG15在ccRCC组织中表达上调,并与ccRCC患者的不良预后相关;多变量回归分析表明,ISG15表达水平是ccRCC的独立预后因素。基于此,我们认为ISG15能作为一个潜在的分子标志物用于ccRCC患者的诊断和预后判断。

