基于深度学习的肺腺癌肿瘤突变负荷的预测
孙德伟 王志刚 杨啸林* 孟祥福*
1(辽宁工程技术大学电子与信息工程学院,辽宁葫芦岛 125000)
2(中国医学科学院基础医学研究所,北京协和医学院基础学院,北京 100005)
引言
肺癌已成为当今世界上最常见的死亡原因之一[1],2018年全球的肿瘤新发病例1 810 万和肿瘤死亡病例960 万,其中肺癌病例占总病例的11.6%,占癌症患者死亡总人数的18.4%[2]。5年生存率仅为18%,其中晚期非小细胞肺癌(non-small cell lung cancer,NSCLC)患者1年生存率不足40%[3]。不同类型,不同分期和分型治疗方案差别很大。在我国,由于吸烟人数众多、空气污染较为严重等情况,肺癌的发生率居高不下。
近些年,免疫检查点抑制剂(immune checkpoint inhibitors,ICPIs)的治疗发展迅猛,ICPIs 联合化疗进行癌症的治疗,极大提升了非小细胞肺癌[4]、肾细胞癌[5]和尿路上皮癌[6]等癌症患者的无进展生存期(progresion-free survival,PFS)和总生存期(overall survival,OS),逐步成为多种癌症的常规治疗方案。但是,患者对ICPIs 反应性有很大差异,研究证实,高体细胞突变数目的患者能更多地从免疫检查点抑制剂治疗中获益[7]。目前,如何有效预测患者对免疫检查点抑制剂治疗的反应性,指导快速准确制定治疗方案是该治疗方法在应用过程中的重要环节。
肿瘤突变负荷(tumor mutation burden,TMB)是衡量基因组突变数量的重要指标之一,该指标利用全外显子组测序或者靶向测序数据,计算每兆碱基测序DNA 中来源于体细胞的、编码区的非同义碱基置换和插入删除突变的数目[8]。已有研究证实,TMB 可以作为ICPI 治疗结果的独立预测因子[9]。但是,TMB 的计算依赖于新一代测序技术,整个检测的周期长且技术复杂;同时,检测的成本很高,会给患者造成较大的经济压力,限制了该方法的临床的应用。
癌症诊治过程中广泛使用的病理组织图片,为估算TMB 提供了一个另外的可能途径。2018年,Coudray 等[10]使用深度学习的方法,利用病理组织图片,实现了NSCLC 的病理学准确分类,并成功预测了肺腺癌病患是否发生STK11,EGFR,FAT1,SETBP1,KRAS 和TP53 等常见的基因突变。这说明,传统的组织病理图片能从某种程度上反映癌症基因组的变化。因此,本研究拟利用NSCLC 中肺腺癌的数字化病理全切片图像(whole slide image,WSI)对病患的TMB 水平进行预测,进而为预测免疫检查点抑制剂治疗反应性提供依据。
近年来对于医学影像的处理的研究工作,大致可分为3 个阶段,第1 阶段是利用机器学习方法对医学影像进行分类;第2 阶段是采用卷积神经网络构建医学影像分类模型;第3 阶段是借助迁移学习思想,对在某个领域训练好的深度神经网络模型进行微调,使其适用于标注数据较少的医学影像分类领域。
1)基于机器学习的预测方法
传统的医疗诊断方法都是依靠有经验的病理学家人工提取特征进行判断,这在很大程度上依赖于医生的经验和知识。在深度学习大规模应用之前,机器学习一直占据辅助治疗的主导地位。Kureshi 等[11]利用患者数据设计了一种预测模型,用于NSCLC 的个性化治疗干预,利用频发模式挖掘方法建立晚期非小细胞肺癌患者特征与肿瘤的关系,该模型使用支持向量机(support vector machine,SVM)、决策树、随机森林(random forest,RF)和分类回归树(classification and regression tree,CART)等4 种分类器进行预测。实验结果表明,分类准确率最高为76.56%。该类模型虽然得到了较为准确的分类结果,但模型不能自动提取病理学特征,而是需要把人工提取的特征作为模型输入,如果人工提取的特征是判断疾病的主要特征,则模型效果好,反之,效果较差。而实际上,人工提取的病理学特征主要靠经验,因此不能确保提取的特征质量一定有助于模型对疾病的判断,进而也就不能确保机器学习模型能够有效判断疾病。
2)基于卷积神经网络(convolutional neural networks,CNN)的预测方法
最早将CNN 引入医学图像处理的是1996年Sahiner 等[12]提出的工作。在该工作中,从乳房X线照片中提取出包含活检证实的肿块或正常组织的感兴趣区域。网络模型CNN 由一个输入层,两个隐藏层和一个输出层组成,并使用了反向传播。1993年,CNN 被应用于肺结节的检测[13]。
由于CNN 的新变体的发展以及现代GPU 运算能力的提高,深度神经网络开始应用于医学影像识别。CNN 的主要功能在于其深层次的体系结构,该体系结构能够自动从每一层网络提取不同特征,层次结构越深,提取的特征越抽象。但是,在医学领域对深层的CNN 训练是一个巨大挑战。主要原因是CNN 模型大多被使用在监督学习上,这需要大量带标签的训练数据。但在医学影像分析领域,带有完备的专家注释和标识(例如病灶区域标识)的数据稀缺,使得训练CNN 需要的数据量不足。其次,训练深层的CNN 需要大量的计算和内存资源,否则,训练过度的CNN 通常会因过度拟合和收敛问题而变得复杂,这需要对网络的体系结构或网络参数进行反复调整。基于以上原因,研究者开始考虑用迁移学习或微调的方法来解决上述问题。
3)基于迁移学习和微调的预测方法
在医学图像领域,获得如ImageNet 这类全面注释的数据集仍然是一个挑战,当没有足够的数据用于训练时,可采用迁移学习和微调这两种方法。迁移学习指的是将在自然图像数据集或不同医学领域预训练过的深度学习模型用于新的医学任务研究。首先将实验数据图像输送到预训练的模型中,这时的模型已经有了一定的识别能力,之后将模型的最后一层替换掉,变为服务于当前任务的输出层,接着只训练新加的输出层,让模型的理解力始终保持不变。这样网络前部分的庞大参数就不用再训练。例如,在Bar 等的工作中[14]用预先训练的CNN 模型作为胸部病理特征的提取器。Ginneken等[15]通过将CNN 网络模型学习到的特征与手工特征相结合,提高了结核检测系统的性能。微调指当实验数据集中等大小可用于模型训练任务时,可以将预训练的CNN 作为网络的初始化模型,然后使用当前任务的数据集对其中几个(或全部)网络层进行进一步的训练。
迁移学习和微调是在医学成像应用中使用深层CNN 的关键组成部分。Shin 等[16]和Tajbakhsh等[17]研究了这些方法的可行性,这些工作中的实验表明,使用深层预训练的CNN 进行微调不仅更容易获得较高的实验效果,而且还进一步表明了训练集尺寸越小微调的重要性就越大。
针对上述问题,以GoogLeNet[18]网络作为骨干网络,在该网络中添加注意力模块,以增强模型的学习能力,训练时将带标签的数据送入网络模型中,通过反向传播迭代训练50 次,模型训练后,将待预测的细胞病理切片数据分组送入深度卷积神经网络模型,对患者的TMB 水平进行分类预测。
1 材料和方法
1.1 材料
研究数据来源于TCGA(The Cancer Genome Atlas)数据库。该数据库是美国国家癌症研究所和国家人类基因组研究所合作建立的癌症研究项目,主体数据是39 种常见癌症患者的全基因组测序数据及其分析结果,还包括患者临床信息、病理图片和放射影像等数据。
研究涉及的基因组信息和数字化病理全切片图像来源于TCGA-LUAD 数据集(https://portal.gdc.cancer.gov/projects/TCGA-LUAD)。本研究的TMB 值利用TCGA 提供的简单核苷酸变异数据计算得到,根据计算结果进行高突变负荷和低突变负荷的分组。目前尚未对高肿瘤突变负荷有明确定义[19],参考各类文献,将TMB≥10.0 突变数/Mb 定义为高突变负荷,TMB<1.1 突变数/Mb 定义为低突变负荷。
选用该数据集中337 名受试者的肺腺癌的数字化病理全切片图像,按照上述TMB 分组,标注为高突变负荷组(High)和低突变负荷组(Low),这也将作为实验数据的标签,通过设置不同的尺寸(512×512 或299×299)将其切分成小切片,属于同一张数字化病理全切片图像的小切片数据标签与对应数字化病理全切片图像的标签相同。实验数据集采用337 张肺腺癌病理组织切片,其中高突变负荷组271 张,低突变负荷组66 张。按照裁剪尺寸不同分为两组,其中尺寸为299×299 的小切片组由539 808张高突变负荷和111 836 张低突变负荷组成;尺寸为512×512 的小切片组由185 021 张高突变负荷和38 216 张低突变负荷组成。
1.2 方法
基于深度神经网络的病理组织切片预测方法的总体解决方案如图1所示,共分3 个步骤。

图1 解决方案总体框图Fig.1 The overall framework of the solution
1)图像预处理。对原始病理图像进行切分,将病理图像数据转换成jpeg 格式存储,在将图像按照80%的训练数据20%的测试数据制作成TFRecord文件,方便网络模型读取。
2)模型训练。将训练数据集送入网络模型中,模型迭代训练50 次。
3)验证模型。将测试数据在第2 步产生的50个训练文件中分别测试,选取模型测试50 次中最优结果做为最终测试结果。
1.2.1 病理图像切分
病理切片扫描图像的像素可达上亿兆。1 张肺癌的病理染色切片,其扫描图像分辨率可为26 001像素×21 911 像素。
尽管计算机内存和计算能力都有大幅提高,但是深度网络依然不具备直接读取整张图像的能力。因此,首先对整张病理切片图像放大20 倍,然后采取无重叠的滑动方式进行切分,进而可得到若干病理小切片图像。图2 为数字化病理全切片图像切分示意,对于一个26 001 像素×21 911 像素的整张病理图像,假设以512×512 大小的窗口进行切分,可以得到2 193 个512×512 大小的小切片。小切片尺寸的大小,直接决定网络模型学习到的细胞不同分化程度的特征。小切片过小,容易造成信息的丢失,小切片过大也会影响模型区分特征的能力,且受计算机内存的限制。最后,将这些小切片(及其对应的标签,例如是否存在肿瘤变异)分批送入网络模型进行训练。
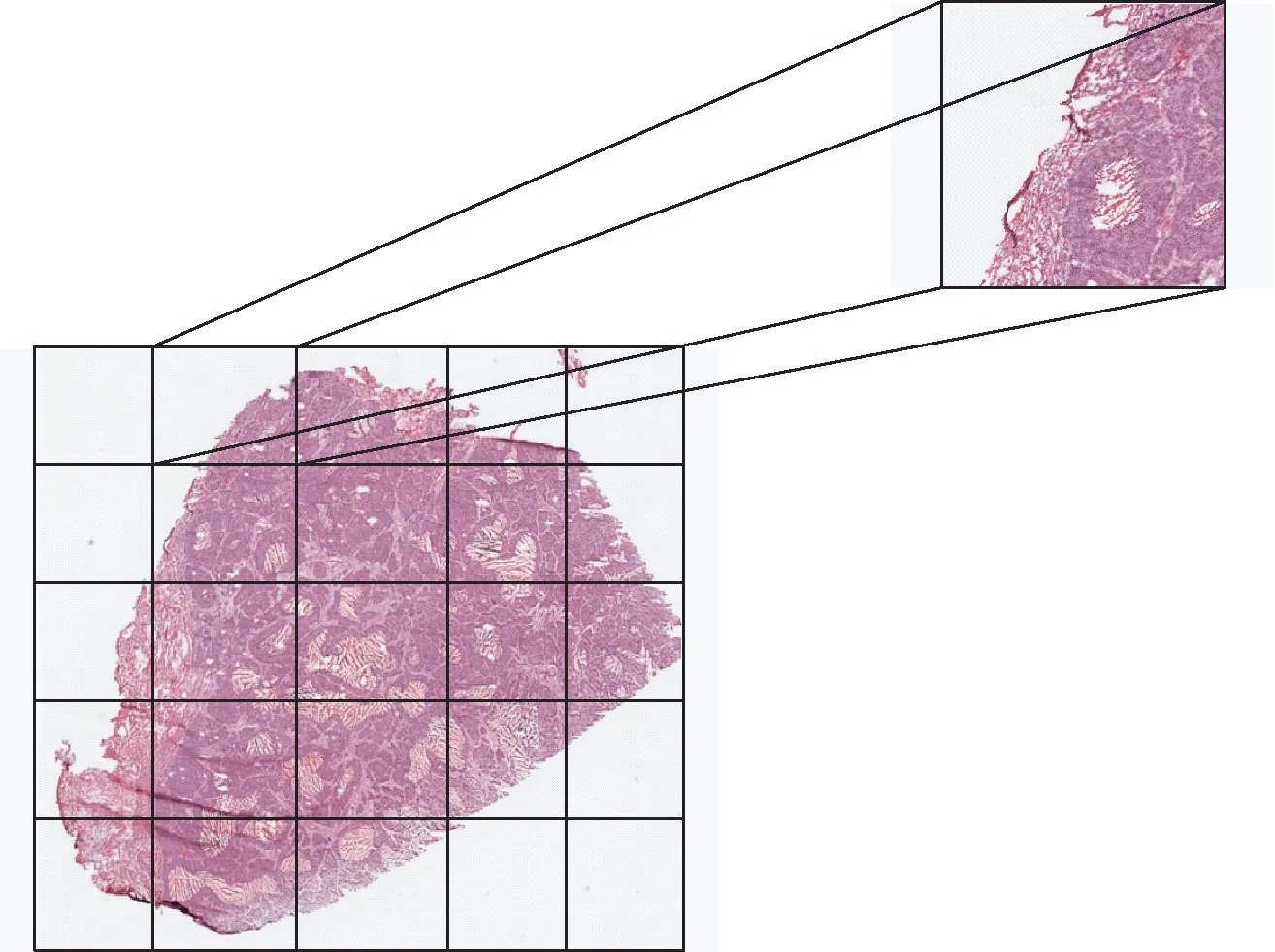
图2 数字化病理全切片图像切分示意Fig.2 The partition of whole slide image
1.2.2 强化特征学习的注意力
公共产品供给实质上是如何分配社会公共资源的问题。随着我国经济的疾速发展和一系列惠农政策的积极推行,我国农村公共产品供给已经取得明显改进。但仍然不能忽略当前我国农村公共产品供给仍然存在着诸多问题。
卷积神经网络因其强大的特征提取能力,极大地推动了计算机视觉的发展。以往的研究中通过增加网络的深度、拓宽网络的宽度来提高模型的学习能力。例如VGGNet[20]、GoogLeNet[8]。后来研究者又把目光聚焦到注意力机制上,通过合理假设选择性的关注感知空间中某一部分,而不是立即对整个信号进行加工。采用CBAM 注意力模型[21],在SENet[22]网络模型的基础上,增加空间注意力机制,使得当前的深度学习网络模型在特征的提取上有了选择,通过应用在通道和空间维度的注意力机制,有针对性的学习。而且CBAM(见图3)可嵌入到当前大多数主流网络中,在不显著增加计算量和参数的情况下,提高了网络模型的特征提取能力。

图3 CBAM 结构图Fig.3 CBAM stucture
通道注意力侧重于对输入数据的关注。计算方法为

式中,F表示输入特征图矩阵尺寸为C×H×W,其中C为特征图通道数,H为特征图的高,W为特征图的宽。Avgpool 表示在通道上做全局平均池化,MaxPool 表示在通道上做全局最大池化,σ表示Sigmoid 函数,MLP 是多层感知机,W0、W1是MLP权重,其中W0矩阵维度是C×R,W1矩阵维度是R×C,R为减少率(即MIL 隐藏层神经元个数和池化后向量长度的比值),C×R为MIL 隐藏层神经元个数,MC是输入特征图通道注意力向量。实验部分(第2.2.2 节)对减少率的不同取值进行具体对比分析。
在此基础上,通过通道注意力产生新的特征图向量X可定义为

式中,⊗表示内积。
空间注意力是对特征图的某一块区域赋予更大的权重,也是对通道注意力的补充,有
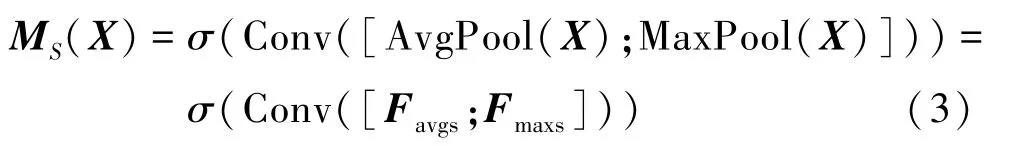
式中,AvgPool 表示对X在空间上做平局池化,MaxPool 表示X在空间上做最大池化,Conv 表示进行卷积核大小为7×7 的卷积操作,MS表示空间注意力产生的注意力向量。
在此基础上,输入特征图进行一次CBAM 产生的新特征图,可定义为

卷积操作虽然能模仿人类的视觉感应提取图像特征,但是传统卷积仅关注局部信息而通常忽略了全局信息。例如,对于3×3 的卷积,卷积核有9个像素点,目标像素的值只参照自身与周围的8 个像素点,这就意味着卷积操作只能利用局部信息来学习,而对于数字病理图像的小切片具有关联关系的组织图像,更需要从全局的角度对特征进行学习。此外,大多医学影像分类任务还停留在大类分类(如判断图像是否存在肿瘤),而本实验是在小类肺腺癌中判断TMB,这对模型的精度提出了更高的要求。所以,引入注意力机制不仅能从全局的角度学习特征,还能够增加微小特征的学习权重,从而避免在卷积过程中损失掉信息。
Inception-V3 是 GoogLeNet 的代表性版本。GoogLeNet 的新思路是拓宽了网络的宽度。通过构建Inception 块用网络自动学习来代替人工设置卷积核的大小,并且用非对称的卷积结构代替了对称的卷积结构例如7×7 大小的卷积核用7×1 和1×7的替代(见图4),提高了在处理更多、更丰富的空间特征结构以及增加特征多样性等方面的效果,同时减少了参数量。而且保留了著名的批标准化(batch normalization,BN)方法。

图4 Inception 块Fig.4 Inception block
BN 是一种非常有效的正则化方法。在对神经网络的某层使用BN 方法时,每一个mini-batch 数据的内部都会进行标准化处理。传统的深度神经网络在训练时,尽管可以用堆砌相同结构的网络来达到提升网络性能的目的,但是这些网络层的输入都是在变化的,输入输出可能分布不一致,对特征的提取带来很大的障碍,也使得基于梯度下降的训练方式显得非常困难。因此BN 算法通过规范每一层的输出,使得输入输出满足控制在统一范围内,而其他网络层得到的输入的变化就小很多。
当输入为1 个小批处理中x的值B= {x1,…,xm},需要学习的参数γ,β,BN 数据标准化过程主要包括4 个步骤。
1)求mini-batch 的均值

2)求mini-batch 的方差

3)标准化

4)转换数据,正向传播过程中通过学习γ,β的参数求出新的分布,通过反向传播更新γ,β的值。

最后,输出{yi =BNγ,β(xi)}
1.2.4 CAIM
构建的网络模型在保留了Inception-V3 网络模型优势的基础上,加入了注意力CBAM 模块。
模型的输入是一批299×299 的小切片图像,网络模型包括输入层、多层卷积层、多层池化层、Dropout 和平铺层,输出为小切片的预测结果。其中,Block 块均为由不同大小的卷积核、不同池化组,如图5所示。其中,⊕Concat 操作将4 个通道的输出数据拼接成多通道数据作为下一部分的输入。

图5 Block 块结构图Fig.5 Block structure
在实验阶段,每个batch 设置为200 个小切片,输入特征图的尺寸以及卷积操作如图6所示,在Inception-block 中,应用到的卷积核和池化的尺寸均在size 属性中标出,并通过对Padding 设置不同值进而对特征图进行缩放(例如,设置为same,经过卷积操作后输出特征图尺寸与输入特征图尺寸相同),模型所选激活函数均为Relu,以避免在深层网络模型中出现梯度消失和梯度爆炸。
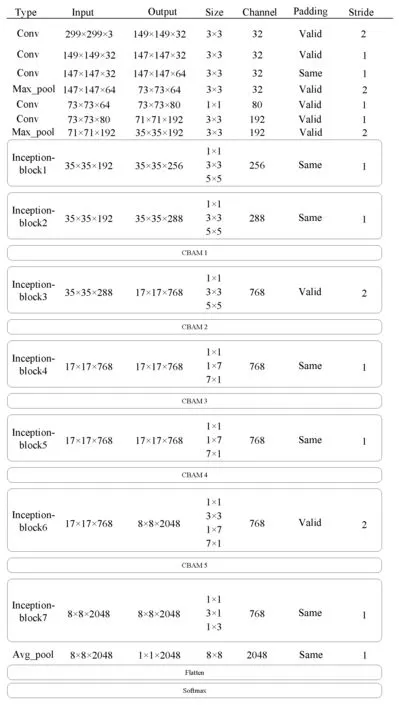
图6 CAIM 结构细节Fig.6 Details of CAIM structure
在Inception-block7 后,通过使用一个全局平均池化操作将输出为8×8×2 048 的尺寸变为1×1×2 048的矩阵,然后通过平铺(Flattern)操作将其变为1×2 048 的向量,在通过一个全连接层输出为分类的个数(在本实验中为2 分类),最后由Softmax生成最终预测概率,最终输出可定义为
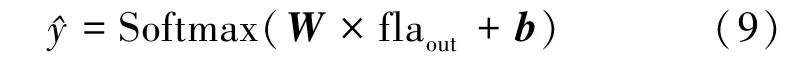
所提出的模型使用Tensorflow 框架和python 语言实现。在实验中,将数据集按照80%和20%的比例分为训练集和测试集,设置批次(batch-size)为200,学习率为0.1,使用分类交叉熵损失函数计算模型训练每次迭代的损失,采用RMSProp 算法分别对卷积核权重和偏置值进行更新,该算法对权重W的梯度使用了微分平方加权平均数

式中,Wt表示t时刻(即第t次迭代)模型的参数,dW2表示t次迭代代价函数关于W的梯度大小的平方数,St表示前t次的梯度平方的均值,β表示动量(通常设置为0.9),α表示全局初始学习率(即深度学习网络模型在反向求导过程中更新参数的步长)。ε是一个取值很小的数,从而避免分母为0。
1.2.5 评价方法
以AUC(area under curve)作为实验评价指标,AUC 被定义为ROC(receiver operating characteristic curve)曲线下与坐标轴围成的面积。所有样本被分为高肿瘤突变负荷和低肿瘤突变负荷两类,在分类后存在高肿瘤突变负荷样本分类正确(TP)、高肿瘤突变负荷样本分类错误(FP)、低肿瘤突变负荷样本分类正确(TN)及低肿瘤突变负荷样本分类错误(FN)等种情况,有

在实验时,通过选取不同的阈值,得到不同的FPR,TPR 的值,从而绘制出ROC 曲线,进而得到AUC 值。AUC 的计算方法同时考虑了分类器对于正例和负例的分类能力,在样本不平衡的情况下,依然能够对分类器做出合理评价。
实验结果由两部分组成,一部分是把属于同一张数字病理图片切分出的小切片预测的概率值求和取平均值作为整张病理切片的最终预测概率值,进而计算AUC 值;另一部分是通过聚合每个切片的阳性百分比来计算AUC 值。
1.2.6 模型验证
为验证模型的有效性,进行3 个实验。
实验一:选取小切片尺寸为299×299 的数据在Vgg 网络、Inception-V3、ResNet 以及本研究网络模型CAIM 进行对比实验。实验目的是验证不同网络模型的准确性。
实验二:选取小切片尺寸为512×512 的数据在Inception-V3 和本研究网络模型CAIM 进行对比实验。实验目的是验证不同尺寸小切片对网络预测准确性的影响。
实验三:对模型参数调优。超参数是在训练模型前,由研究者手动传入的参数,它不能通过模型自动学习,需要外部进行指定。这种外部传参的不确定因素同样影响着模型的性能,综合模型特点,选取了两个超参数学习率和减少率进行实验。
1)学习率
学习率作为监督学习以及深度学习中重要的超参数,决定着目标函数能否收敛到局部最小值以及何时收敛到最小值,并且直接体现了模型的性能表现。在本模型中,通过设置学习率衰减系数来减小学习率对实验结果的影响。实验过程中,学习率的取值分别设置为{0.1,0.01,0.001},通过在小切片尺寸为299×299,模型为CAIM 上进行对比实验,观察不同学习率下的模型效果。
2)减少率
减少率R是一个超参数,R值越大网络参数就会越多,容易导致模型收敛至局部最小值点,R值越小,通道注意力的学习能力就会减弱,导致模型的整体学习能力下降。为了使实验具有对比性,实验仍然保持小切片的尺寸为299×299,模型为CAIM上进行实验。
实验目的是选择最优超参数。
2 结果
2.1.1 对比试验
Vgg、Inception、ResNet 网络都是在ImageNet 分类任务中具有良好表现的模型,也是图像分类任务中的主流模型,因此,本实验选用以上3 种网络模型为基线模型。通过3 种网络的发展不难看出,3 种网络的分类准确度在自然图像识别领域也是逐年上升,考虑到计算成本和模型的准确度,选择在Inception 模型基础上进行改进。
实验一的结果如表1 和图7所示,通过两种计算AUC 值方式的对比,CAIM 模型的AUC 值在上述对比模型中达到了最优,进一步表明融入注意力机制的有效性。本研究模型CAIM 通过保留Inception块并结合CBAM 的模型要优于Inception-V3,在深度卷积神经网络中引入CBAM 注意力机制可以有效的让模型网络自主选择学习内容;与此同时,它可以增强特定的区域表示,在不明显增加模型的参数量和计算量的情况下提高模型的特征提取能力。

图7 切片大小为299×299 的AUC 结果。(a)Vgg分类;(b)Inception-V3 分类;(c)ResNet 分类;(d)CAIM 分类Fig.7 The AUC results for tile size of 299×299.(a)Vgg;(b)ResNet;(c)Inception-V3;(d)CAIM

表1 切片大小为299×299 的实验结果Tab.1 The AUC results for tile size 299×299
实验二的结果如表2 和图8所示,对比可见,本模型的CAIM 的AUC 值仍高于Inception-V3 的结果值,进一步表明了本模型的有效性。但通过与实验一中的Inception-V3 和CAIM 两种模型在不同尺寸切片下实验的比较可见,在切片尺寸为299×299 的情况下,模型的AUC 值高于在切片大小为512×512的AUC 值,切片大小对实验结果有一定影响,在实验数据上,把整张病理图像切分成299×299 的小切片更为合适。
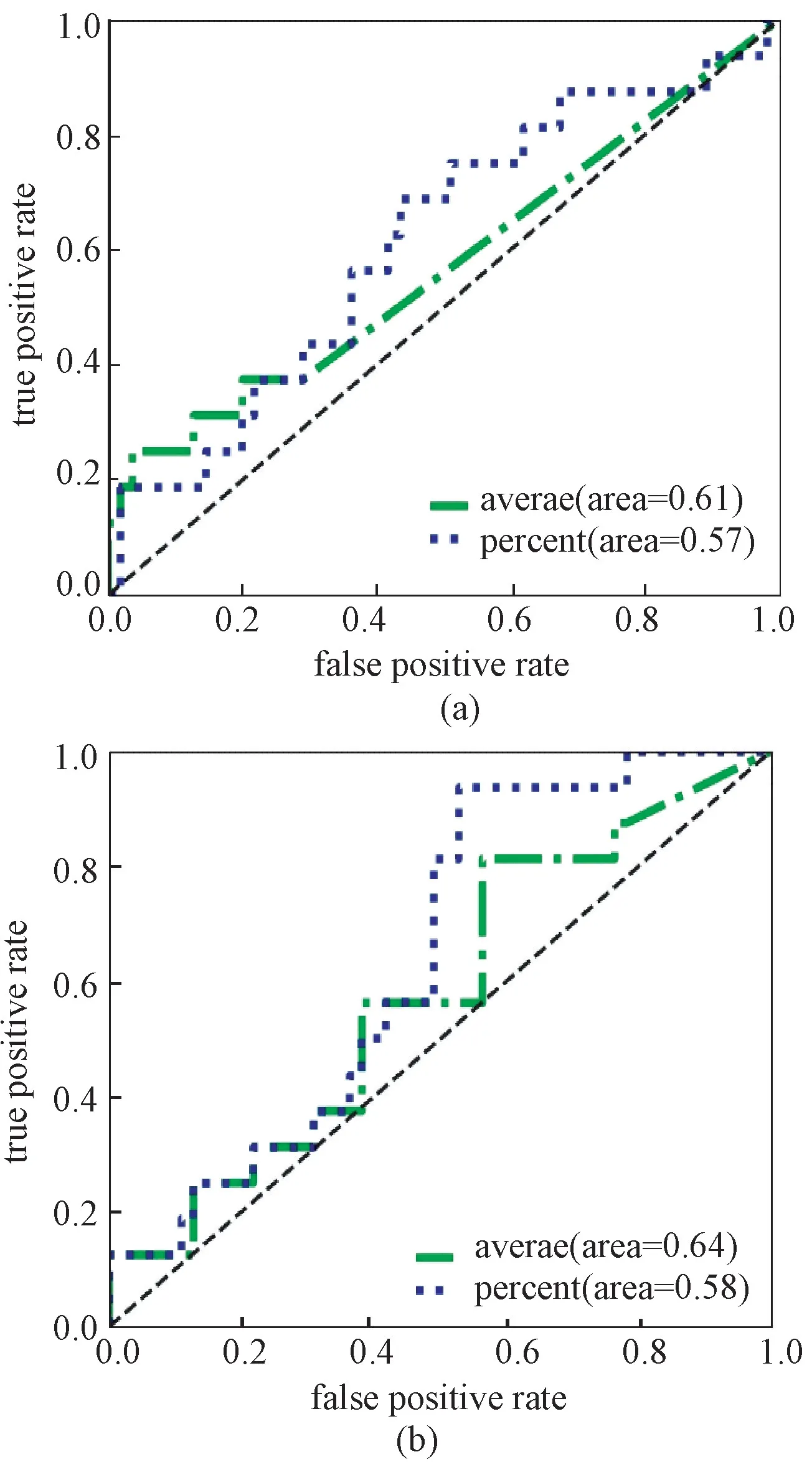
图8 切片大小为512×512 的AUC 结果。(a)Inception-v3 结果(b)CAIM 结果Fig.8 The AUC results for tile size of 512×512.(a)Inception-v3;(b)CAIM
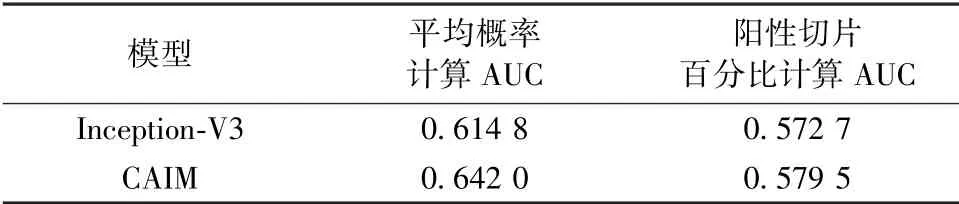
表2 切片大小为512×512 实验结果Tab.2 The AUC results for the tile size of 512×512
2.2.2 超参数学习
实验三中超参数学习率的实验结果如表3 所示。从表中可以观察到3 种学习率对实验结果并无显著影响,说明在实验中加入学习率衰减因子是有效的,极大避免了学习率对模型准确度的影响。

表3 学习率对比实验结果Tab.3 Learning rate comparison experiment results
实验三中超参数减少率的实验结果如表4所示。实验结果表明,通过选取不同的减少率的值,影响着模型的性能,在本模型中,减少率值为0.5时,模型效果最优。

表4 减少率对比实验结果Tab.4 Reduction rate comparison experiment results
3 讨论
TMB 是近年来发现的可用于预测免疫治疗疗效的独立生物标记物,在免疫治疗中的预测能力不仅限于非小细胞肺癌、黑色素瘤等“热点肿瘤”,还能作为其它癌症的生物标志物。然而,对于TMB 的检测主要通过全外显子组测序(whole exome sequencing,WES)等新一代测序技术获得,过高的检查成本和漫长的检测周期限制了TMB 在临床试验和临床实践中的应用[23],另外由于不同的测序平台的检测标准不统一,导致对TMB 的估计存在不同程度的偏差。本研究显示,利用深度学习方法,可以通过大量训练数据学习到存在于病理组织切片中的特征,进而估测患者的TMB 水平。与可能需要两到三周完成的测序相比,有效缩短了检测时间[24]。
将深度学习模型迁移至医疗学习任务,主要有两大关键影响因素。一种是数据集本身的影响,如对于病理组织图像,病变区域和正常组织区域是否有详细的标识。就TCGA 数据库中LUAD 的样本制作过程而言,制作方式就有多种,这就会造成不同的制作过程产生的数据样本的质量参差不齐;另外一种是深度学习模型中超参数的影响,这类参数在梯度下降的过程中控制着模型的收敛速度以及模型如何求得最优解[25]。通过表3 的实验结果可见,加入学习率衰减因子能有效避免超参数学习率的影响,在实验过程中,通过BN 操作规范数据,使模型在反向传播中更容易收敛,加快模型学习速度。
表1 和表2 的结果显示,裁剪成不同尺寸的小切片,对实验效果有着很大的影响,是因为在小切片中包含细胞组织的信息量不同。而本研究的网络模型,通过Inception 块实现了卷积核的并行计算,采用多个不同的卷积核提取特征,根据病理组织小切片的特点,融合注意力机制,通过全连接层利用Softmax 函数,提升了网络模型的预测效果。
随着卷积神经网络的发展,越来越多的大型网络得以开发。起初,人们把焦点集中在自然图像上,逐渐迁移到医学图像领域[26]。本研究尽管没有明确地预测出TMB 数值,但通过较高的AUC 值进一步表明了深度学习算法的可行性和高效性。随着深度学习技术的进一步发展和更大数据集的产生,提出的方法可以进一步推广和验证,以增加CAIM 的鲁棒性。
本研究结果表明,组织病理学图像包含以前未被探索但在临床上有用的特征,这些特征可以使用深度学习技术来检测。而且,对比于其他医学影像数据集[27],数字病理切片的图像特征更加微小,也对网络模型提出了更高的要求。此外,与其他诊断工具(WES 测序)不同,CAIM 预测的是图像每个区域的TMB,这可能体现出了高TMB 相关组织特征之间的异形性,或者是TMB 自身的异形性。CAIM 也是应用深度学习将组织病理图像与全基因组的特征联系起来的第一步,将有助于研究癌症基因型和表现型之间的关系。因此,CAIM 是一种从大量的组织病理图像中快速提取高效信息的一种方式,从而辅助减少患者样本的检测时间。
本研究工作也存在一定的局限性。在数据预处理阶段,采取滑动窗口对整张数字病理图像进行切片的方式,会导致切片之间的关联信息丢失,而实际上,TMB 分组是对整张病理图片的标注,而切成小片后,所有切片(无论是同一切片中的肿瘤组织区域还是正常组织区域)都具有相同的标签,从而为网络的自动特征提取带来了不确定性。在未来工作中,一方面要收集病理切片级别的标注,另一方面构建病理切片级别的网络[28],从而避免切片之间上下文信息的缺失,进一步提升预测的准确性。另外,由于病理切片及其对应的TMB 值样本数量不够充分,导致当前的训练数据还存在不平衡和不全面等问题,使得本模型只能预测病理切片的TMB 是高于阈值还是低于阈值(即二分类),如果能够收集到更全面的高质量样本,经过适当调整还可预测更多更具体的TMB 值区间(即多分类问题),因此数据样本的收集也是未来的一项工作。
4 结论
通过在Inception-V3 的基础上融合了CBAM 注意力机制进行研究和对比,验证了提出的在联合肿瘤突变负荷在肺腺癌的预测中是有效的。该方法通过使用深度学习方法学习数字化病理全切片图像特征,针对肺腺癌病患的TMB 水平进行预测,从而为医疗人员进一步实施精准治疗提供辅助支持。

