常绿木质藤本植物黑老虎基因组SSR特征分析及引物开发
(湖南省森林植物园,湖南 长沙 410116)
黑老虎Kadsura coccinea(Lem.) A.C.Smith 亦称冷饭团,为五味子科Schisandraceae 南五味子属常绿木质藤本,天然分布于东南亚以及我国南方地区。黑老虎用途广泛,在药材、食品和园林观赏方面具有广阔应用前景。其果实富含人体所需的各种氨基酸和维生素C 等营养物质[1],且香甜可口,风味独特,作为一种新奇水果,在我国水果市场颇受青睐[2]。黑老虎根和茎富含木脂素和三萜类活性成分[3-4],其提取物具有显著的美白护肤功效[5];其根亦为一种传统中药材,具有抗氧化、降血脂、活血化瘀、行气止痛等功效,可治疗风湿性关节炎、跌打肿痛等病症[6-7];黑老虎还具有抗肿瘤、抗病毒、抗炎、抗HIV 和抗肝纤维化等药理活性[4]。黑老虎挂果时间长,鲜果颜色多样、色泽艳丽,果形独特,亦为优良的观赏植物。
近年来,由于利益驱动,黑老虎野生资源分布地出现掠夺式采果和挖根现象,生境遭受严重破坏,黑老虎野生资源急剧减少,导致其种质资源破坏殆尽,对于黑老虎遗传改良将造成不可挽回的损失[8],开展黑老虎种质资源保护迫在眉睫。另一方面,目前黑老虎在湖南、贵州和广西等省区迅速推广种植,湖南通道县种植尤为普遍,该县黑老虎已成为国家地理标志产品和支柱产业[9]。然而生产上缺乏良种,目前普遍采用挖取野生苗或随意采种育苗,良种选育研究才开始起步。简单重复序列(SSR)标记具有信息含量高、重复性好、操作简单、成本较低,而且为共显性,为学者们广泛应用[10-12]。本研究首次开发黑老虎SSR标记,旨在为其群体遗传学研究、种质资源收集、保存与评价以及育种计划制定等奠定基础。
1 材料和方法
1.1 植物材料
在湖南共收集到黑老虎及其近缘种91 个植株的嫩叶(表1),包括会同、城步和桂东天然群体的66 个黑老虎植株,南岳树木园10 个南五味子K.longipedunculata植株,南岳林场4 个异形南五味子K.heteroclita、4 个翼梗五味子Schisandra henryi、5 个华中五味子S.sphenanthera和2 个狭叶五味子S.lancifolia等15 个植株。采样植株间的距离在30 m 以上。嫩叶采用硅胶干燥,应用改良的CTAB 法提取其DNA[13]。
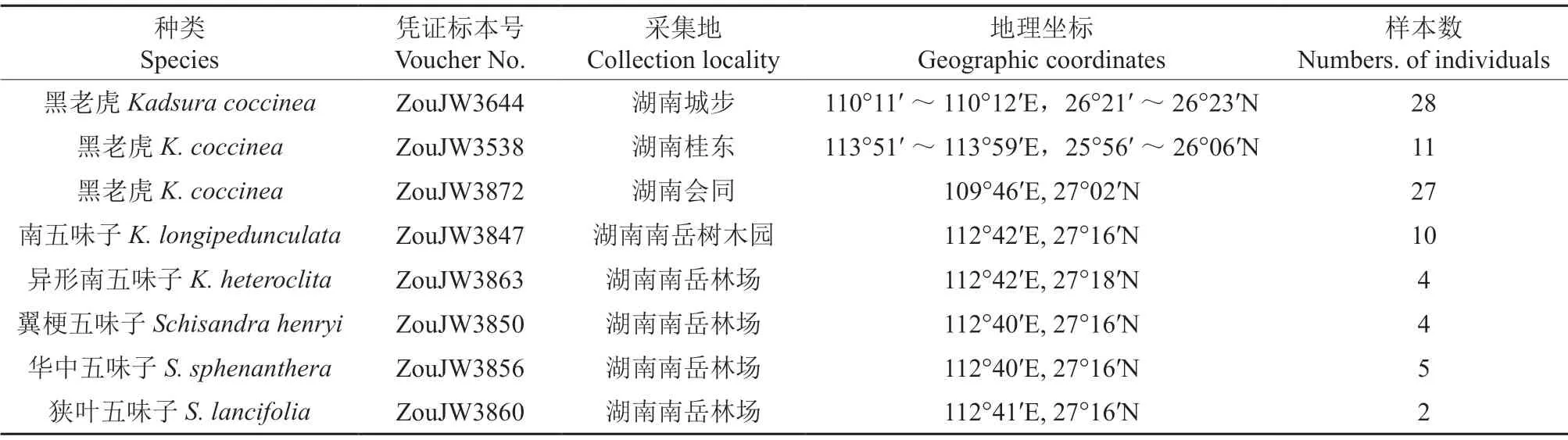
表1 用于SSR 标记开发的南五味子属和五味子属样品信息†Table 1 Information of all Kadsura and Schisandra samples for SSR markers development
1.2 高通量测序
从湖南省城步群体选取1 个黑老虎植株的总DNA 用于SSR 位点搜索。利用DNA 片段化试剂盒AIRTMDNA Fragmentation Kit (Bioo Scientific Corp.,Austin,USA)将其总DNA 打断。经切胶纯化,选取其中300~500 bp 的片段,应用高通量快速建库试剂盒NEXTflex Rapid DNA-Seq Kit(Bioo Scientific Corp.,Austin,USA) 建库。利用Illumina HiSeq 2000 PE 150 平台(Illumina,San Diego,USA)进行高通量测序。获得下机序列后,应用Trimmomatic 软件[14]对其进行质量控制,删除其中Q20<90%和正反向序列不匹配比率高于10%的序列。应用SPAdes 3.9.0 软件[15]对序列进行基因组拼接,参数选择默认值。
1.3 SSR 挖掘与引物设计
应用软件MISA[16](http://pjrc.ipk-gatersleben.de/misa/)对拼接好的序列进行SSR 位点搜索,其参数设置如下:重复单元样式为单、二、三、四、五和六核苷酸重复,其最小重复次数分别为10、5、4、3、3 和3;复合位点的重复单元间序列碱基数不超过20。获得含有SSR 位点的序列后,应用软件Primer 3[17](http://fokker.wi.mit.edu/primer3/)设计引物,参数设置为默认值。
1.4 引物筛选与位点多态性检测
从会同、城步和桂东群体各选取1 个DNA样品对设计的引物进行PCR 扩增,确定实验体系并检测引物的可用性。为了方便后续位点多态性检测,选择单元重复数不小于7 的SSR 位点合成160 对引物,并在其正向引物的5′端增加一段M13 序列(5′-CACGACGTTGTAAAACGAC-3′)。PCR 扩增反应采用10 μL 体系,具体包括:50 ng DNA 模板、150 μM dNTPs、2.0 μM MgCl2、0.5 μM M13 标记正向引物、0.5 μM 反向引物、1×PCR 缓冲液(天根生化科技有限公司,北京)和0.04 U/μL Taq DNA 聚合酶(天根生化科技有限公司,北京)。扩增反应在PCR 扩增仪Master cycler Gradient Thermal Cycler (Eppendorf)上 进行,其程序为:94℃预变性 4 min;94℃变性30 s,60℃退火30 s,72℃延伸30 s,30 个循环;72℃延伸10 min;最终产物保存于4℃冰箱。扩增产物经1%琼脂糖凝胶电泳检测,选择给定片段长度位置具有清晰且单一条带的引物,用于后续位点多态性检测。
选取会同、城步、桂东群体的66 个DNA 样品,应用M13-tailed 引物法[18]进行SSR 位点多态性检测。其PCR 扩增反应采用10 μL 体系,具体包括:50 ng DNA 模板、150 μM dNTPs、2.0 μM MgCl2、0.5 μM 荧光标记的M13 引物、0.5 μM M13 标记的正向引物、0.5 μM 反向引物、1×PCR 缓冲液(天根生化科技有限公司,北京)和0.04 U/μL Taq DNA 聚合酶(天根生化科技有限公司,北京)。扩增反应程序与前面部分相同。
采用3730 XL自动测序仪(Applied Biosystems,USA 检测PCR 产物,选用内标为GeneScan 500 LIZ Size Standard(Applied Biosystems,USA)。使用软件GeneMarker V2.2.0[19]判读基因型。进一步选取5 个近缘种的25 个DNA 样品(表1)开展引物通用性检测。
1.5 遗传多样性分析
应用MICRO-CHECKER 2.2.3 软件[20]检测和修正哑等位基因;应用POPGENE version 1.31 软件[21]计算每位点等位基因数(A)、实际杂合度(Ho)和期望杂合度(He),检测位点是否偏离Hardy-Weinberg 平衡(HWE)以及是否存在连锁不平衡现象(LD)。
2 结果与分析
2.1 基因组SSR 位点的分布特征
通过序列质量控制和拼接组装获取687 805条unigene,其中,82 454 条含有SSR;61 032 条可用于设计PCR 引物,其预期产物片段大小为100~280 bp。SSR 重复长度为10~100 bp,其平均长度为14 bp。共获得152 207 个候选SSR 位点,其中,二核苷酸重复比率最大(57.68%),其次为单核苷酸重复(17.30%)和四核苷酸重复(10.6%),三核苷酸重复、五核苷酸重复和六核苷酸重复的比率低得多,3 者总和为14.32%(表2)。在检测到的序列中,单、二、三、四、五和六核苷酸重复的SSR 位点间平均距离分别7.67、2.30、17.12、12.43、41.41 和39.48 kb。
分析SSR 重复单元类型可以看出,单核苷酸重复单元(motif)中,A/T 最为普遍,占98.06%,C/G 仅为1.94%(图1a);二核苷酸重复单元以AC/GT(57.30%)最为普遍,其它依次为AG/CT(36.57%)、AT/AT(6.04%) 和CG/CG(0.09%)(图1b);三核苷酸重复单元有10个类型,以AAG/CTT 最普遍(41.91%),ACG/CGT 最少(0.34%)(图1c);AAAT/ATTT、AAAAT/ATTTT 和ACACAT/ATGTGT 分别是四核苷酸、五核苷酸和六核苷酸重复单元中最普遍的,其比率为29.55%、29.18%和40.31%(图1d—f)。
2.2 SSR 位点的多态性分析和种间应用
引物筛选结果表明,参与测试的160对引物中,有102 对能够清晰扩增出预期大小的特异片段(clear specific amplicons);其中40 个位点具多态性。而40 个位点中,有12 个位点出现多个片段,难以判读,因此,成功筛选出28 个多态性位点(表3)。应用3 个天然群体对28 个位点进行遗传多样性分析可知,每位点等位基因数(A)为2~14,共计168 个等位基因,实际杂合度(Ho)和期望杂合度(He)分别为 0.000~0.900 和0.000~0.860(表4)。其中9 个位点可能含有哑基因(95%置信区间);对哑基因进行修订(correction)后发现,城步、桂东和会同群体分别有13、7 和9 个位点显著偏离Hardy-Weinberg 平衡(P<0.05)。28 个位点的所有成对分析中,均未发现显著连锁不平衡现象(P≥0.05)。
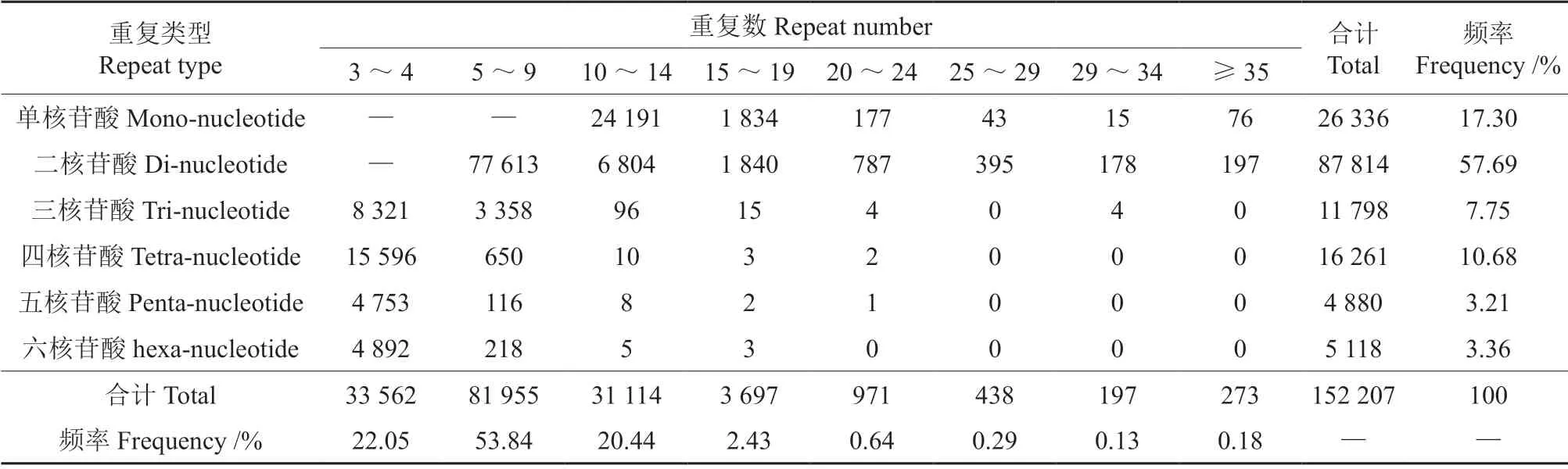
表2 黑老虎基因组SSR 的分布特征Table 2 Distribution of SSRs in the genome of Kadsura coccinea
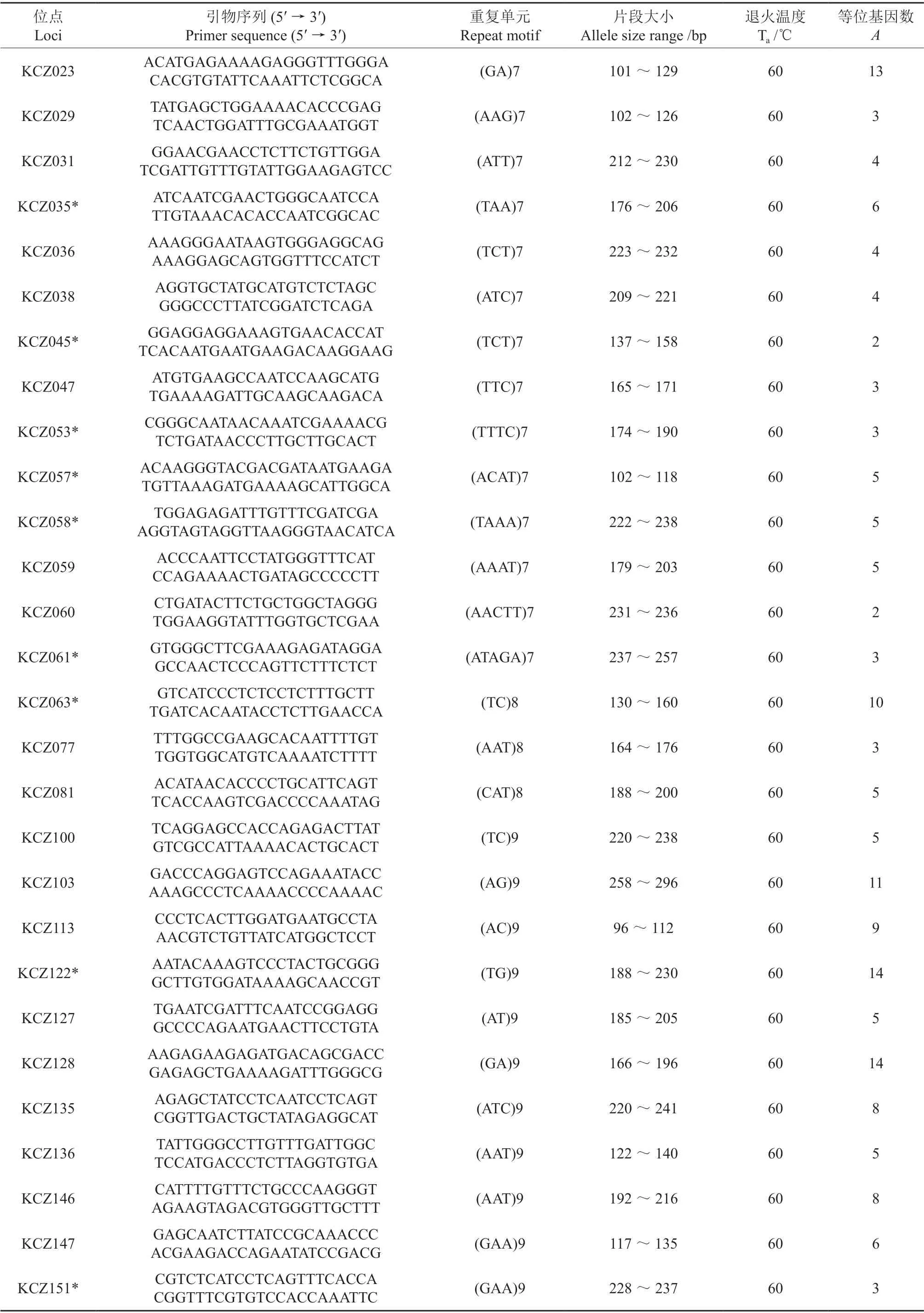
表3 黑老虎28 个SSR 多态位点的特性†Table 3 Characterization of 28 polymorphic SSR loci for Kadsura coccinea.
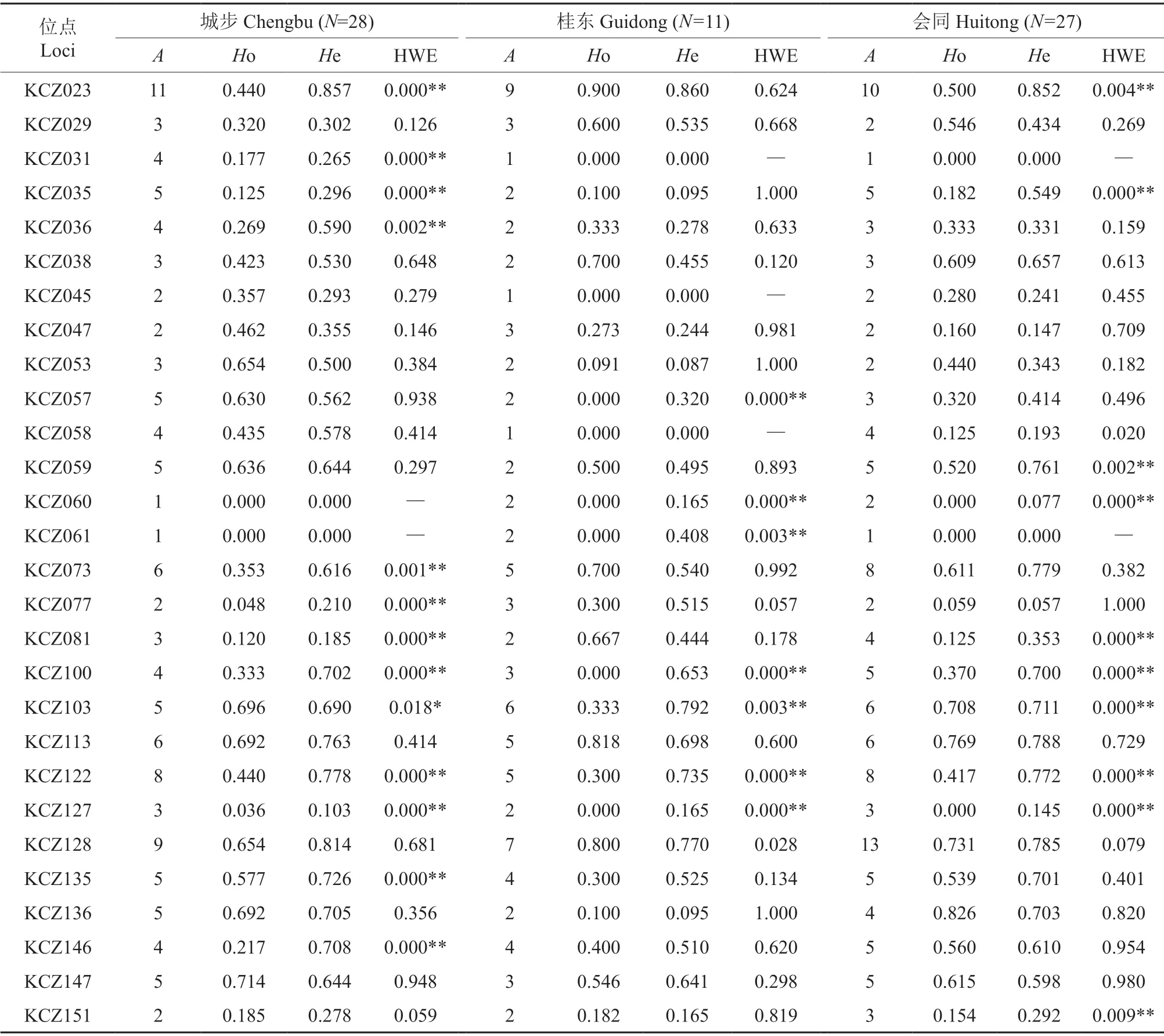
表4 28 个SSR 位点在3 个黑老虎群体的遗传多样性数据†Table 4 Genetic diversity data of 28 polymorphic SSR loci in three Kadsura coccinea populations
28 对引物的通用性因植物种类而异。对于南五味子属,有24 对引物可应用于南五味子,14 对引物用于异形南五味子,分别占85.71%和50%;相比而言,28 对引物在五味子属中的通用性低得多,仅6、9、7 对引物分别在翼梗五味子、华中五味子和狭叶五味子获得成功扩增,分别占21.43%、32.14%和25.00%(表5)。从表5还可以看出,有KCZ031、KCZ036、KCZ045、KCZ053、KCZ058、KCZ073、KCZ100、KCZ103、KCZ122、KCZ127、KCZ128、KCZ136、KCZ146、KCZ147 等14 对引物在南五味子属的2 个种均能成功扩增;仅引物KCZ147能成功应用于南五味子属和五味子属的5 个种,说明该引物的通用性最佳。
3 结论和讨论
3.1 黑老虎基因组SSR 的特征
本研究通过Illumina高通量测序检索到152 207个候选SSR 位点,以二核苷酸重复的比例最大,占57.69%,AC/GT 基元占二核苷酸重复总数的57.30%;单核苷酸和和四核苷酸重复的比例分别为17.30% 和10.68%,以A/T 和AAAT/ATTT 基元为主,占比为98.06%和29.55%。杨梅Myrica rubar基因组中,二核苷酸重复的占比高达82.99%,其中AG/CT 基元出现频率高达55.70%,本研究与之基本一致;所不同的是,杨梅基因组中二至六核苷酸重复的占比呈明显下降趋势[22],而在本研究中其占比则出现波动。王家等[23]亦采用Illumina 高通量测序开发山白树Sinowilsonia henryi的SSR 标记,从7 614 条序列中搜索到694个SSR 位点,以单核苷酸重复最多,占51.78%,重复基元以A/T 最多;其次为二核苷酸重复,占34.01%,以AG/CT 为主;从单核苷酸至六核苷酸重复,其所占比例整体上越来越少,本研究与之差异较大。而椭圆叶花锚Halenia ellipitica则以三核苷酸重复最为丰富,其比例为43.11%,以基元AAT 为主;其次为四、二核苷酸重复,比例分别为29.31%和21.55%,以重复基元AAAT 和AT 为主[24],本研究亦与之明显不同。说明SSR 在基因组的特征因植物种类不同而存在明显差异。
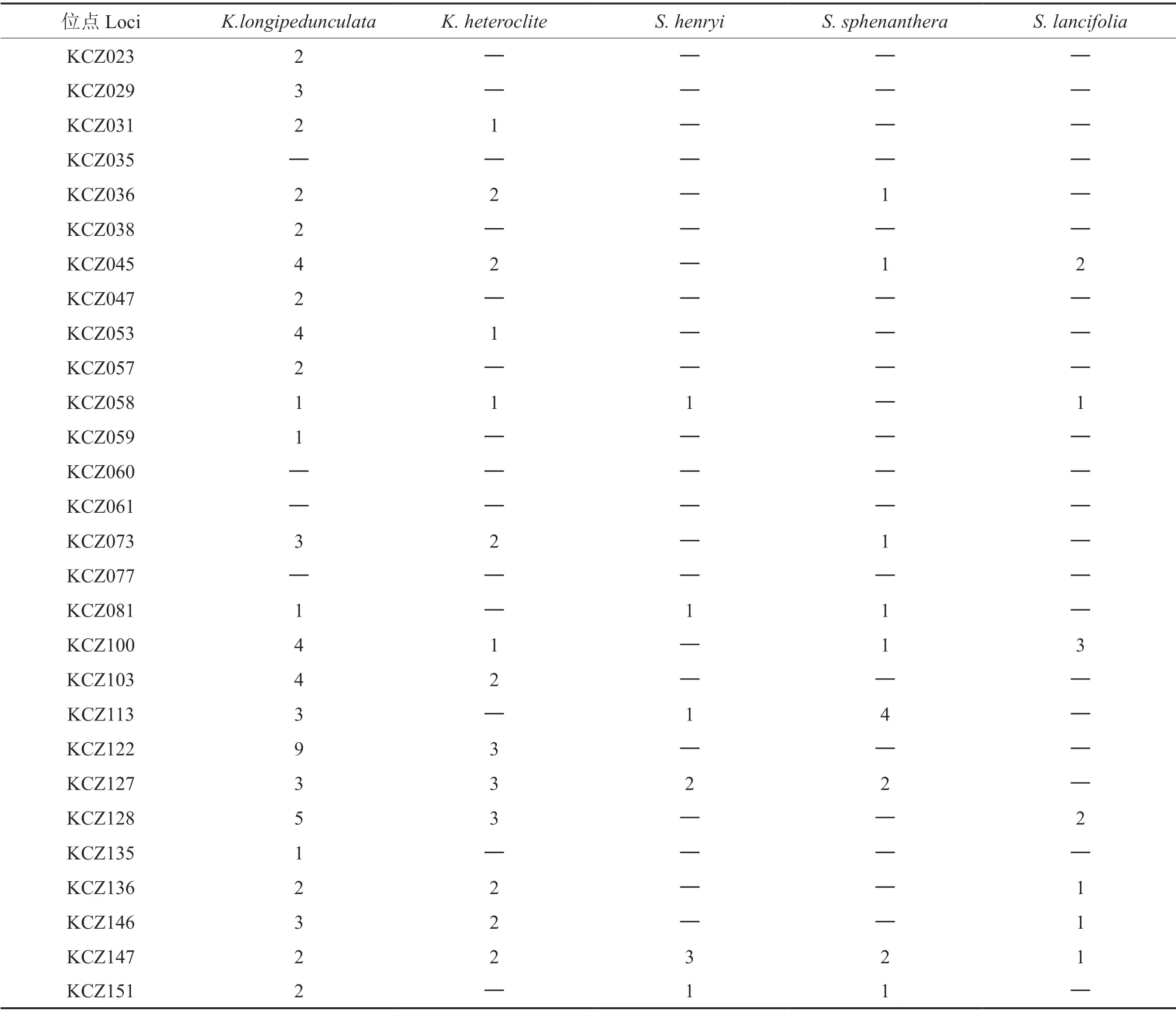
表5 黑老虎28 个SSR 位点在近缘种的应用†Table 5 Allele numbers obtained in cross-amplification trials of 28 SSR loci for relatives of Kadsura coccinea
3.2 黑老虎SSR 标记开发
开发SSR 引物有很多种方法,利用高通量测序获得SSR 位点数量远远高于探针杂交富集等传统方法。如在五味子科中,Yan 等[25]基于华中五味子的AFLP 片段序列构建富集SSR 基因组文库,经SSR 探针杂交,获得61 条含有SSR 的序列,设计50 对引物开发出9 个多态性SSR 标记;Sun等[26]基于SSR 探针杂交富集SSR,获得五味子234 条含SSR 的序列,设计137 对引物开发出14个多态性SSR 标记;而在本研究中,应用Illumina高通量测序获得82 454 条含有SSR 的序列,设计160 对引物,获得28 个多态性SSR 标记。在本研究中,我们为了探讨重复单元对引物筛选效率的影响,设计的160 对引物中,二、三、四、五核苷酸重复各40 对,由表3可以看出,4 种类型分别开发出14、8、4、2 对引物,其等位基因数依次为5~14、2~8、3~5、2~3,开发的引物数和多态性均呈下降趋势。由此可见,引物筛选效率与检测引物有关,宜选择二、三核苷酸重复的SSR 位点设计并开发引物。
本研究开发出的28 对SSR 引物,分别有24 和14 对可应用于同属的南五味子和异形南五味子,成功率分别为85.71%和50.00%;而仅有6~9 对引物在五味子属的翼梗五味子、华中五味子和狭叶五味子得到成功扩增,成功率为21.43%~32.14%。Sun 等[26]亦发现14 对五味子SSR 引物中有11 对可用于同属的华中五味子,成功率近80%。石晓蒙等[27]从同属的无梗花栎Quercus petraea、蒙古栎Q.mongolica、夏栎Q.robur、麻栎Q.acutissima、辽东栎Q.liaotungensis5 个种的62 对基因组SSR 和EST-SSR 引物中筛选出41 对可用于栓皮栎Q.variabilis,成功率达66.13%。说明亲缘关系明显影响引物的种间应用,同属植物间SSR 引物的通用性要远高于不同属。因此,应用别种植物的SSR 引物进行筛选时,尽量选择亲缘关系近的种。一般而言,宜选择同属植物,以提高引物筛选效率以及SSR 标记的多态性。
综上所述,本研究开发出28 个多态位点;通过3 个天然群体的多样性分析可知,每位点等位基因数为2~14,实际杂合度和期望杂合度分别为0.000~0.900 和0.000~0.860,说明这些标记具有较高的多态性,可用于揭示黑老虎群体间和群体内的遗传多样性,为我们深入开展黑老虎群体遗传学研究、种质资源收集、保存和评价以及分子标记辅助育种研究提供了技术支撑。本研究开发的引物数量对于基因组关联分析等研究而言尚偏少,然而大量SSR 候选位点序列也为我们将来大规模标记开发进而为黑老虎基因组关联分析、基因组选择等奠定了基础。

