薄壳山核桃SCAR标记开发及其在品种间的多态性
,,,,
(1.浙江省林业科学研究院,浙江 杭州 310023;2.森林食品资源利用与质量控制国家林业局重点实验室,浙江 杭州 310023)
薄壳山核桃Carya illinoinensis(Wangenh.) K.Koch是胡桃科山核桃属植物,因其性状优良和核仁营养价值高,在世界五大洲被广泛引种栽培,是重要的世界性干果和油料作物[1-2]。薄壳山核桃是雌雄同株的风媒花异交植物,按照雌雄花成熟期的先后,大致可分为雄先型和雌先型2 种类型[3]。不同品种在不同气候条件下会存在花期不遇的问题,这是影响薄壳山核桃稳产和高产的重要原因之一。美国是薄壳山核桃的原产地之一,目前也是薄壳山核桃的中心产区,其产量占全球产量的85%以上。美国现有1 000 多品种,在建园时往往可以根据当地的气候特点进行有效的杂交配置,壮年林干果产量可达1 500 kg/hm2以上。虽然我国引种薄壳山核桃已有100 多年的历史,但是目前生产上常用的品种仅几十个。多年的生产实践结果证明我国不少地区是薄壳山核桃的适生区,但是较多薄壳山核桃产区面临产量低下和产量不稳定的问题,尤其是广大亚热带地区。造成这一现象的原因有多个方面,比如实际使用的品种较为有限,再加上引种时存在错误以及一些杂交后代定名混乱,难以进行有效的亲本选择和亲缘关系分析,更难以根据当地的气候特点进行有效的杂交配置。
目前,用于薄壳山核桃品种鉴定的分子标记方法主要有RAPD[4-5]、SSR[6-9]、SRAP[10]、ISSR[11-12]和SNP[13]等。一般来说,SSR 和SNP 等标记须进行大量基因组或转录组的测序和分析[13-14],得到稳定性和多态性较好的标记的成本较高。在利用SSR引物对薄壳山核桃进行品种鉴定时,重复性检测中也存在一些问题[7],这主要是因为:一方面薄壳山核桃高度杂合,基因组大且有较多重复,导致SSR 引物存在一定的非特异性;另一方面,SSR 和SNP 这类标记对检测的要求较高,且检测识别时存在一定的人为干扰。随着测序技术的进步以及越来越多的薄壳山核桃基因组数据的公布,利用品种间基因组数据的差异挖掘序列特异性标记(sequence characterized amplified region,SCAR)是一个提高分子标记的稳定性和特异性的办法。本研究中通过对不同薄壳山核桃品种叶片DNA 进行简单基因组测序,寻找品种特异性的SCAR 标记,旨在针对生产上常用品种从分子水平上开发出稳定、特异的DNA 指纹标记,为辅助实现薄壳山核桃品种准确快速鉴定和亲缘关系分析提供参考。
1 材料与方法
1.1 试验材料
薄壳山核桃样品采集于建德薄壳山核桃基地,地理位置为119°32.86′E、29°33.76′N,海拔97.5 m。5月上旬,采摘植株新鲜嫩叶,冰浴运输,当天保存于-20 ℃冰箱中备用。所涉及的品种见表1。其中,Ind23 和Ind24 为不同来源的同一品种,Ind23 为20 世纪80年代从美国德克萨斯州美国山核桃试验站引进,Ind24 为1999年从美国国家无性系种质资源库(National Clonal Germplasm Repository)引进。“雄先”和“雌先”的判断是根据不同性别花刚开始成熟的时间,雄花是指花粉开始散粉,雌花是指柱头出现可授粉状态[3]。

表1 薄壳山核桃试验品种及其生理特点Table 1 Experimental varieties and physiological characteristics of C.illinoensis
1.2 试验方法
1.2.1 DNA 提取
叶片DNA 的提取采用新型快速植物基因组DNA 提取试剂盒(BioTeke,北京)。取各品种叶片样品0.02 ~0.03 g,剪碎后放入2.0 mL 离心管内,加入直径3 mm 碳化钨研磨珠2 颗,用液氮冰冻5 ~10 min,再用MM400 研磨仪(莱驰,德国)30 Hz 研磨2 min,然后按照试剂盒说明书操作。提取好的DNA 经1.0%的琼脂糖凝胶电泳检测完整性,然后用NanoDrop2000 超微量分光光度计检测其质量浓度,置于-20 ℃冰箱备用,使用前将DNA 质量浓度稀释标定至20 ng/μL。
1.2.2 简化基因组测序和分析
简化基因组测序是基于RAD(restriction-siteassociated DNA sequencing)方法进行的。具体步骤:提取高质量DNA,使用多种限制性内切酶对DNA样品分别进行酶切,根据酶切试验的结果选择合适的酶,构建长度为300 ~500 bp 的pair-end 文库,用Illumina HiSeq PE150 测序文库,获得每种样品的简化基因组原始数据。
后续分析方法:1)对每种样品中的Reads1(双端PE 测序后前端的数据,即含有EcoRI 酶切位点AATTC 的片段)进行比对聚类;2)对每种样品的序列聚类结果进行内部比对,得到样品内部各序列位点信息;3)将不同样品的序列互相进行比对,寻找个体之间碱基差异信息,并根据样品信息寻找亚群特异的序列位点;4)根据特异序列信息获得双端数据进行组装,得到亚群特异的基因组片段。
具体组装方法:1)使用cap3 软件(http://seq.cs.iastate.edu/cap3.html)将Reads1 与Reads2 进 行局部组装;将特异Taq(unique Tag)与最长的组装结果进行连接(中间加入5 个“N”),获得初步组装结果;2)将某一样品获得的组装Tag 与其他样品的序列进行blastn 比对,过滤掉相似序列,比对使用的软件为blastn,参数e-value 为1×10-5。另外,在设计引物前,为了增加引物扩增的有效率,使 用MISA(http://pgrc.ipk-gatersleben.de/misa/)对组装结果进行SSR 预测,剔除含有SSR 的组装结果。
使用Vector NTI 9.0 软件设计引物,各样品特异片段的引物设计原则:引物长度18 ~22 bp,解链温度56 ~62 ℃,产物长度150 ~300 bp。
1.2.3 SCAR 标记开发
对使用上述引物预测的片段进一步开展blast分析,挑选出与核桃(Juglans regia,现有全基因组数据中与薄壳山核桃最近物种)同源性最高的DNA 序列片段,作为后续SCAR 标记开发的候选序列。对这些序列进行PCR 筛选,挑选出23 个品种中存在多态性的DNA 条带,切胶回收纯化这些条带,并利用PCR 引物进行两端测序,测序结果经Vector NTI 9.0 软件中的ContigExpress 拼接,将重测序得到的序列与之前的基因组测序数据进行比对,补充这些序列中间的未知碱基(这些碱基之前以“NNN”表示),将得到的完整序列作为新的SCAR 标记。
1.2.4 PCR 筛选
常规PCR 试验所用试剂为BioTeke PCR MasterMix(BioTeke,北京),整个反应体系除引物和模板外,均按照试剂手册要求操作。在15 μL体系中,引物用量为上游和下游引物(10 μmol/L)各0.6 μL,模板为2 μL。用于直接测序前,反应体系扩大到60 μL,模板为前面15 μL 产物稀释1 000 倍。混合多对引物进行PCR 反应时,适当调整每对引物的量,至最佳扩增效果,其他条件不变。所有PCR 反应在Genepro Thermal Cycle(Bioer,杭州)PCR 仪上进行,程序:94 ℃ 300 s,95 ℃ 10 s,56 ℃ 50 s,72 ℃ 40 s,共35 轮循环;72 ℃,300 s。扩增产物经1.5%琼脂糖凝胶电泳检测,排枪上样各5 μL,泳道顺序对应样品分别是Ind01,Ind13,Ind02,Ind14……。Marker为DL2000(Takara),条带大小依次为2 000、1 000、750、500、250、100 bp,上样量3 μL。EB染色和ChemiDoc(Biorad,北京)拍照。
1.2.5 亲缘关系分析
在各品种中,所筛选较为稳定的SCAR 标记的有无,分别以“1”“0”进行记录,然后使用NTSYS 2.2 软件建立聚类分支树状图。首先用similarity 程序组中的SimQual 计算形似系数矩阵,然后使用Clustering 程序组中的SHAN 进行计算,聚类方法选用UPGMA,其他参数按默认设置。
2 结果与分析
2.1 RAD 测序和差异分析
选取性状差异较大的5 个品种的样品进行Illumina HiSeq PE150 测序,分别是Moore、Nacono、Van Deman、Davis 和Pawnee,测序结果见表2。一般情况下A 与T 数量相等,C 与G 数量相等,(A+T)/(C+G)的值则具有物种特异性。从表2可知,由于该简化基因组文库是对酶切片段附近的序列进行测序,所以碱基并不平衡,且头部5 个碱基为固定的“AATTC”序列,薄壳山核桃(A+T)/(C+G)的值约为1.63。从碱基长度质量指标Q20%和Q30%来看,该文库碱基质量良好,可用于后续分析。
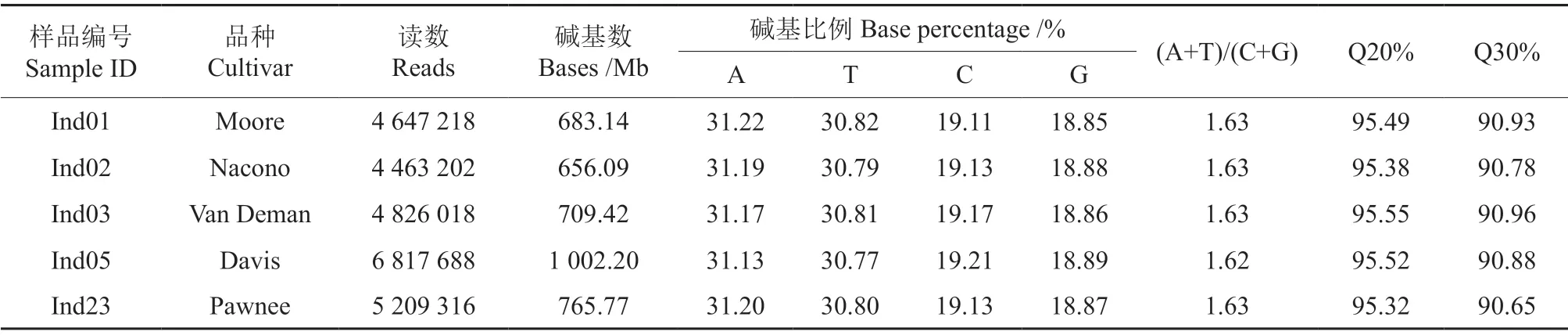
表2 性状差异较大的5 个薄壳山核桃品种样品的高通量测序结果Table 2 Deep sequencing results of five C.illinoensis cultivars with different characteristics
测得的数据经初步组装、blast 分析、精确组装和比较,得到各品种的特异序列。其中Ind23 特异序列数量最多,达1 080 条,而Ind03 最少,仅为553 条。分析这些片段的二级结构、二聚体形成和碱基分布等,设计了1 133 对引物(表3)。再结合扩增产物与核桃基因组序列的同源性,挑选其中473 对作为最终候选引物。

表3 性状差异较大的5 个薄壳山核桃品种特征序列和引物搜索Table 3 Searching for characteristic sequences and primers of five C.illinoensis cultivars with different characteristics
2.2 PCR 引物筛选结果
PCR 筛选结果表明,绝大部分条带在各品种间无差异,仅86 对引物在23 个品种中产生多态性,约占引物数量的1/5,其中80 对单一条带可用于后续分析(图1)。

图1 80 对SCAR 引物在23 个薄壳山核桃品种中的多态性分布Fig.1 The polymorphism distribution of 80 SCAR markers among 23 cultivars
2.3 用于品种鉴定的SCAR 引物及其组合
所开发SCAR 标记,大多数在多个品种中仅产生1 个条带,其中有极少数可以特异性指示单一品种(图2)。但是,也有少量引物在各品种中产生的条带数量存在差异(图3),这些较为复杂的多态性有待深入研究。将产生多态性的这些单一条带进行组合,获得一系列可用于品种鉴定的引物组合(表4),这些引物的具体信息见表5。

图2 薄壳山核桃品种特异性SCAR 标记引物对及单一条带Fig.2 Cultivar-specific primer pairs and their unique bands of SCAR markers

图3 薄壳山核桃某些SCAR 标记引物的多态性Fig.3 Polymorphism of some scar markers in C.illinoensis
2.4 SCAR 标记用于品种亲缘关系的分析
利用80 对引物在各品种间的多态性,进行24个薄壳山核桃品种的聚类分析,结果如图4所示。由图4可见,聚类结果与实际的亲缘关系有部分一致性,但也存在不一致的地方。例如,来自不同产地的Ind23 和Ind24 同为Pawnee 品种,聚在一起;Ind07(Choctaw)是Success×Mahan 的后代,Ind13(Dependable)是Jewett×Success 的后代,二者聚在一起,可能与其有较近的亲缘关系有关;Ind10(Barton)是Moore×Success 的后代,与Ind01(Moore)聚在一起。但是,Ind04(Forkert)是Success×Schley 的杂交后代,Ind07(Choctaw)是Success×Mahan 的杂交后代,同为Success 的杂交后代,二者在聚类关系上却相差较远,这可能是其遗传组合的不同造成的,也可能是因为所筛选SCAR 标记较为有限,在分析亲缘关系方面尚存在一定的局限性。
3 结论与讨论
因为分子标记能够从DNA 水平反映品种遗传的本质,成为品种鉴定最为重要的依据之一。然而,因为品种之间的遗传差异较小,挖掘其多态性位点较为困难,特别是获得稳定的序列特异性标记更为不易。利用简单基因组测序可以获得大量的差异位点。薄壳山核桃品种繁多,本研究中通过对大量基因组测序数据的分析,再加上PCR的反复验证,获得了一批可用于薄壳山核桃品种鉴定的SCAR 标记。虽然后续PCR 筛选的工作量较大,但SCAR 标记有其明显的优点,比如可以通过普通的PCR 检测条带的有无,使用成本较低。研究结果也表明,400 多对引物扩增率虽然较高,但多态性比例并不高,因为这种简单基因组测序是通过酶切后进行的,测序的信息主要来源于酶切位点附近,如果品种的差异点正好出现在酶切位点上,那么测序就会出现差异条带,但事实上由此设计的引物能在所有品种中扩增出相似的条带,这也是该方法的局限性造成的。同时,也可以利用简单基因组测序获得大量的SNP 位点,研究初期由于参考了核桃基因组数据,未能得到可靠的SNP 数据。因此,现有这些测序数据有进一步挖掘的潜力,比如利用薄壳山核桃品种87MX3-2.11 作为参考基因组[15],后续将进一步分析SNP 位点。

表4 用于薄壳山核桃品种鉴定的特异SCAR 标记组合举例Table 4 The combination of specific SCAR markers for identification of pecan cultivars

表5 用于薄壳山核桃品种鉴定的特异SCAR 标记引物的具体信息Table 5 Detailed information of primers with specific SCAR markers for identification of pecan cultivars

续表5Continuation of Table 5
但是,PCR 扩增有时会出现一定的假阳性,SCAR 引物也不例外,有时条带较弱,经多次重复才能扩增。其关键是提取DNA 的质量以及DNA的分装等问题。DNA 提取方式是影响DNA 质量的重要因素,SSR 标记的扩增也是如此[16]。DNA的分装也会影响标记扩增的稳定性。通常情况下,较高浓度的DNA 须稀释后用于后续的扩增,而稀释后经常会面临分装的问题,由于DNA 是大分子,分装会带来分配不均的问题,有时会影响到试验的重复性。而这一问题经常被研究人员忽视。通过定量PCR 试验证实,如果未较好地进行分装,那么即使是同一初始模板和同一引物分装在不同PCR 管中也会有一定的扩增差异。因此在分装DNA 样品时,应进行必要的均质化,如超声波震荡等,对标记扩增的一致性有重要的作用。另外,扩增片段的大小也有重要影响,小片段更容易获得具有一致性的扩增结果。就鉴定的方便程度来说,可以通过大小片段的组合进行混合引物PCR,但为了取得较为稳定的检测结果,应测试各引物的不同浓度组合[17]。另外,在本研究中有些引物能产生多条特异性片段,其在品种鉴定中可能具有独特的价值,但是鉴于其多态性较为复杂,本研究中暂未考虑,这些位点有待深入研究。

图4 基于80 对引物的24 个薄壳山核桃样品的聚类结果Fig.4 Cluster analysis of 24 C.illinoensis samples based on 80 pairs of primers
利用分子标记进行聚类分析,有助于分析品种间的亲缘关系,对于后续的杂交亲本选育具有重要意义。本研究中利用分离到的80 个SCAR 标记对现有24 种样品进行分析,因为标记数量不够多等原因,其结果存在与现有亲缘关系不一致的问题,而来自薄壳山核桃品种园的杂交品种较多,后续将采集更多的品种进行类似的遗传分析。

