互操作性本体:智能精准医学的基础*
何勇群,谢江安,万 灵,杨啸林,朱 彦,周 伟,李元放,陆伟胜,吴健民,刘开永,王海河0,刘清平,余 红*
(1 University of Michigan Medical School,Ann Arbor,MI 48109,yongqunh@med.umich.edu; 2 重庆邮电大学生物信息学院,重庆 400065;3 南京昂吉网智网络技术有限公司,江苏 南京 211199; 4 中国医学科学院基础医学研究所,北京 100005;5 中国中医科学院中医药信息研究所,北京 100700;6 国家人口健康科学数据中心,北京 100700;7 Faculty of Information Technology,Monash University,Clayton,Vic 3800;8 北京大学肿瘤医院癌症生物信息学中心,北京 100142;9 安徽医科大学公共卫生学院,安徽 合肥 230032; 10 哈尔滨医科大学大庆分校,黑龙江 大庆 163319;11 广州中医药大学,广东 广州 510006; 12 贵州大学医学院/贵州省人民医院/国家卫生健康委员会肺脏免疫性疾病诊治重点实验室,贵州 贵阳 550002)
2002年诺贝尔生理与医学奖获得者Sydney Brenner教授曾说:“We are drowning in a sea of data and thirsting for knowledge.Most biology today is low input,high throughput,no output biology.”(我们渴望从数据中获得知识,但却被淹没在数据的海洋中。当今大多数生物学领域能用很低的投入获得高通量的数据,却无法得到有用的生物学知识)。如何解决从海量数据中获取高价知识仍然是当前生物医学大数据研究领域面临的最大挑战。基于此,我们结合实际研究案例提出:在生物医学,尤其是在大数据支撑的精准医学研究中,具备异构数据标准化与智能分析功能的互操作性本体可以有效应对上述挑战。
1 生物医学大数据及其面临的语义标准化挑战
2012年5月,联合国发布了《大数据与人类发展:挑战与机遇》白皮书,指出大数据对人类发展是一个历史性机遇,我们可以使用极为丰富的数据资源对社会经济进行前所未有的实时分析,帮助政府更好地响应社会和经济运行。其中,生物医学大数据表现最为突出,其促成因素主要有:①生命的整体性和疾病的复杂性。例如,严重威胁人类健康的各种慢性疾病多为复杂性疾病,其发生发展的分子遗传机制受到基因与环境交互作用的影响,因而其病因学研究将产生大量的数据;②得益于高通量技术的发展,基因测序成本急速下降。当前高通量技术可以完成数百万个DNA的同时测序任务,这使从物种的基因组和转录组水平进行全面细致的分析成为可能。从一滴血中我们可以得到大量基因转录与翻译的数据用于快速Omics分析;③医疗信息化和IT业的高速发展,越来越多的人体数据能够获得储存和利用。如,仅在2015年,美国平均每家医院需要管理665T的数据量,中国各大医院的电子健康记录也收录了海量的个人健康数据(病历、心电图、医疗影像等)。
大数据不仅仅是数据量大,而现实中“大数据”的概念常被滥用且应用成效很低。大数据“4V”模型指出其具有数据容量大(volume)、多样化(variety)、高速(velocity)和真实性(veracity)的特点[1]。其中,数据真实性不仅指大数据本身的质量,还包括数据源、数据类型及其处理的可信度。做好数据真实性,我们必须要消除偏差、异常或不一致,保证数据可重复利用。生物医学的任何领域都极其复杂,无法融合共享的异构数据是事实上的无效数据或数据“垃圾”,而未经过标准化处理的生物医学大数据即使再强的人工智能也都会无功而返。现实中“大数据”除了各种BI(business intelligence,商业智能)可视化界面外,真正的大数据应用成果寥若晨星。在数据可视化逐渐有点审美疲劳的当下,如何打破“信息孤岛”,实现优质数据的无歧义融合,保证各类科学研究和临床实践活动所创建的数据内容能被其创建者、维护者以及外部用户群体同等有效地共享和使用成为生物医学大数据领域研究关注的焦点。
目前,大多数临床数据缺乏系统的语义标准化整合。以缺铁性贫血为例(见图1):医院1和医院2采用了不同的疾病分类编码体系,导致了同为缺铁性贫血在医院1的疾病代码为d16758,疾病名称为Iron-deficiency anemia(英文)或缺铁性贫血(中文),而在医院2中却为1015674、Sideropenic anemia和缺铁贫血症。此类数据依靠临床医生或科研人员的经验判断可以得出正确结论。然而对于机器(如计算机)来说,如果没有事先定义或标准化映射,是无法准确识别其中的有效信息的(如患者12和患者78均为缺铁性贫血患者)。同时,从图1中我们也可以看到有5人患有贫血症,但如果要问计算机哪些患者有贫血,这就是一个更为复杂的问题了。我们可以用自然语言处理的方法去查询,但自然语言处理有它自身的缺陷,如可能不能识别“缺铁”与“缺铁性”“贫血”与“贫血症”之间的区别;给同义词不同的代码也会造成混乱;单纯的代码也不能告诉计算机哪些贫血是缺铁性的,哪些是恶性贫血,哪些是慢性病贫血。因此,自然语言处理对于基于计算机的智能查询没有太大帮助。此时数据的语义化显得尤为重要,而不同来源的生物医学大数据之间的共享、整合和再利用的基础任务即为实现数据的语义标准化。

图1 生物医学大数据分析中的语义标准化问题——以缺铁性贫血为例
人工智能(artificial intelligence,AI)的一个核心是让机器理解语言,因而对语义的标准化也有着极高的要求。例如,我们与机器对话:“我是人,我得肺结核病,有什么药可用?”(图2A)。要回答这些问题,机器需要知道:什么是人?什么是肺结核?有哪些药物可以用来治疗肺结核?而患者(我)适合采用哪些药物治疗?在此过程中,让机器知道这些术语词汇的本质及其相互之间的逻辑关系是关键性的环节。
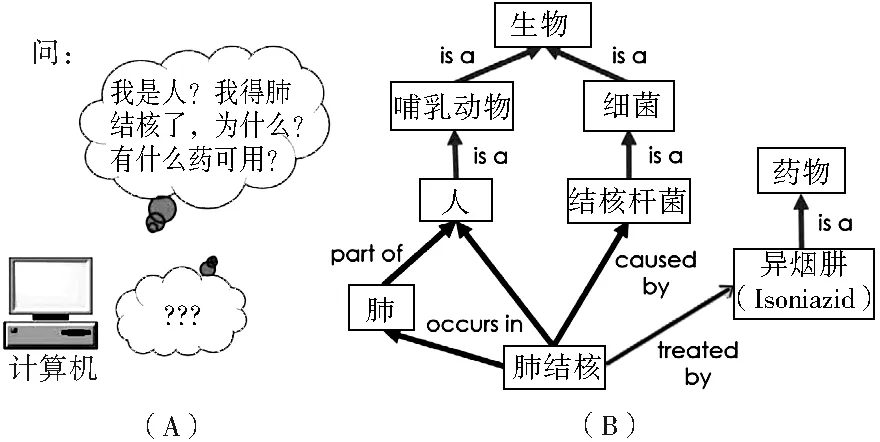
(A)人工智能的例子 (B)基于本体方法的对这个问题的回答
针对图2A举的有关怎样让机器理解语言的人工智能问题,图2B给出了一个基于本体学方法的答案。人是一种哺乳动物,肺结核病是由肺结核杆菌(一种细菌)引起的;人与结核杆菌都是生物体;肺结核病发生部位在肺,肺是人的一部分;肺结核病可以用抗结核药治疗(如异烟肼、利福平、吡嗪酰胺等)治疗;每个术语和关系都有唯一识别代码表示,同一个代码还可以表示不同的同义词。当机器理解这个本体所表述的内容时,就可以从本质上掌握其词汇术语之间的语义关系,并有针对性地回答相关问题了。当然,治疗方案的制定需要结合患者的实际情况,而这是一个精准医学层面的问题。图2表明本体可以用来实现数据的语义标准化,进而促进医学人工智能的发展。
2 本体的定义、功能及发展简介
本体论(ontology)原本是哲学的一门分支,且被亚里士多德认为是第一哲学。“onto”表示being(是)和reality(存在),本体学是用来研究事物的本源和存在问题。在计算机与人工智能领域,本体是用人和计算机都可以理解的术语(terms)及关系(relations)来描述某一领域内的实体(entity)及实体之间的相互关系,从而提供一个对此领域事物本质的统一认识。因此,本体可以描述概念和事物之间的关系以及事物的类别。笔者先前发表的论文给出了详细的本体基本定义和分类的介绍[2]。以下从功能、发展史及语义复杂度方面给出关于本体更新的解读。
基因本体(gene ontology,GO)[3]被认为是第一个现象级的成功本体。GO最初于1998年被构建用来注释三种已经完成的模式生物的基因组,即酿酒酵母(saccharomyces cerevisiae)、秀丽隐杆线虫(caenorhabditis elegans)和黑腹果蝇(drosophila melanogaster)[3]。此后,许多生物如人和小家鼠的基因组项目也陆续加入了GO。GO逐步发展成一种系统的注释物种基因组及其表达产物属性的方法。目前,GO主要包括三个分支:细胞组件、分子功能和生物过程。除了用来注释基因组,GO还被用来做各种应用,如对实验数据进行基因富集组分分析[4]和文献检索[5]等。迄今,GO的原始文献[3]被引用超过25,000次,GO已成为基因组及其相关表达产物分析研究的常规工具。
受GO启发,研究人员认为可以开发出不同领域的本体发挥专业化、精细化的用途。图3总结了一些常见的本体的应用。首先,本体可以用来命名。例如,NCBITaxon物种分类本体(https://github.com/obophenotype/ncbitaxon)包含了约一百万个词条,命名了各种各样的物种并给出了它们的分类。本体可以提供一个复杂的知识网络体系用来表达各种知识,如疫苗本体(vaccine ontology,VO)[6-7]涵盖并分类了几千种人和动物用疫苗,同时给出了这些疫苗的组成成分及其接种的对象和抵抗的疾病。因此,GO和VO等本体被广泛用来做自然语言处理[8-9](见图3)。本体对数据的标准化、整合、共享和分析有着重要作用(见图3)。BioPAX本体已经被用来作为分子通路数据共享的标准[10]。除了上面提到的GO基因富集组分分析[4],本体可以用来做许多其他的数据分析工作[11-12]。
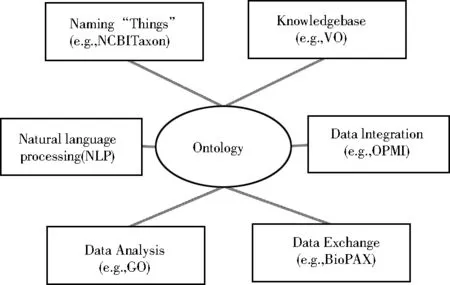
图3 常见本体的应用与举例
本体也可作为语义网(semantic web)、链接数据(linked data)和知识图谱(knowledge graph)的基础。语义网的最终目的是使机器能够理解互联网上的数据并使来自各种资源的数据语义互联互通。RDF(资源描述框架)和OWL(Web本体语言)技术可以对数据进行语义编码。链接数据是一种互联网数据语义关联的方法,由互联网之父Tim Berners-Lee于2006年提出。链接数据建立在HTTP、RDF和URI等标准Web技术的基础上,不仅为读者提供网页链接,而且使计算机能够自动读取与链接有关的信息。因此,链接数据使得语义查询变得更加便捷高效。链接数据一般以RDF三元组的图数据库模式存储,体现了数据治理、语义连接的思想,有利于大规模数据的整合与利用。本体主要表示的是类别层面的数据与关系,链接数据主要转达的是个体之间的数据,各自的链接数据系统也都需要本体在数据类别框架上去把不同的数据关系打通。Google的知识图谱就是利用语义关系把各种实体关联起来并以图谱形式呈现出来的知识库,其本质是语义网和链接数据技术在Google知识体系中的一种应用。
本体的建模方法和表达方式是基于已有方法演化而来,但较已有方法区别也很明显(见图4)。就建模方法而言,条目(glossary)或叙词表(thesaurus)没有权威认定与赋予编码的限制;然而,控制术语集(controlled terminology)、通用数据模型(common data models,CDM)、分类表(taxonomies)和本体等都有权威认定与赋予编码的限制。很多控制术语集,如MeSH (medical subject headings)和MedDRA (medical dictionary for regulatory activities)在医学界广泛应用;OMOP (observational medical outcomes partnership)的CDM对于数据的标准化也有着广泛的应用。但是,就语义精确度而言,本体精确度最高。CDM一般是基于关系型数据库,术语之间的语义关系较弱,分类表只有is_a关系,而本体还具有其他重要的关系,如part_of、occurs_in和has_participant等。此外,语义的表达方式区别显著,包括Word/HTML、XML和XSLT、XML和UML、关系型数据库、RDF和OWL(见图4)。作为一种基本的标记语言,XML定义了一组用于人机共识的格式文档编码规则。RDF和OWL可用XML的编写,RDF是用于描述Web资源的框架,OWL是用于编写本体的知识表达语言。XML、RDF和OWL都是W3C推荐的标准[13]。与其他方法比较,OWL和本体构建的时间成本是最高的,但同时得到的语义精确度也是最高级的(见图4)。基于本体的知识网络体系可以自动被计算机理解与应用,有利于做复杂数据的储存、查询和知识推导。本体可以用SPARQL语言做查询[14],很多基于本体的算法或软件也已被开发出来。
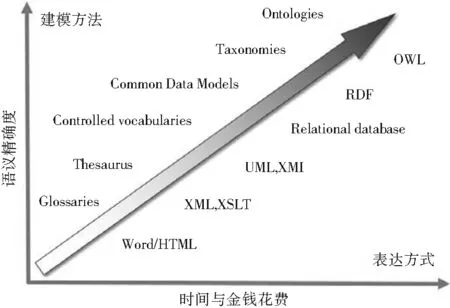
图4 语义复杂度(complexity of semantics)
3 人工智能的兴起和本体的推动作用
人工智能(artificial intelligence,AI)是计算机科学中研究、设计和应用智能机器的一个分支。某些方面像人类一样,AI可以“看到”和“听到”,并且作出判断和行动,从而实现某种目标。AI已经被大量用作包括语音识别、图像识别、自然语言处理、深度学习、人机交互系统的工作。目前,医疗领域人工智能主要应用在医学影像与诊断、医学生物研究、医疗风险分析和药物疫苗挖掘四个方面。
这里我们讨论AI的两个与知识处理有关的分支,即机器学习(machine learning,ML)与知识表达和推理(knowledge representation and reasoning,KRR)。近年来,ML方法已成为人工智能领域中最炙手可热的研究方向,尤其以深度学习为代表,很多以往对计算机非常有挑战性的问题都能够被机器学习有效的解决。KRR是人工智能的一个传统分支,是本体语言的理论基础。KRR以逻辑方法为主,在此基础上设计了很多不同的本体语言,开发了很多本体推理算法和推理机。现代本体语言的一大特点是它语义信息的准确性,这样的语义信息可以被机器处理,用来判断本体的正确性及推理出本体里隐含的知识。
虽然ML与KRR都属于AI分支,然而二者却有很多不同。ML以统计概率或神经网络为基础,KRR以数理逻辑为基础,它们传统上独立发展,鲜有交叉。但怎样让这两个分支共同促进、协调发展已经成为近年来的研究热点。
用ML做数据整合与分析存在着很多缺陷和挑战。ML非常依赖大量高质量的标注数据。但生物医学和临床的数据通常是复杂异构而难以整合和处理的。生物医学大数据标准化处理也需要进行数据建模,使得计算机能够模拟明确的推理过程,而深度学习无法做到。目前,训练AI的技术在模拟人脑思考过程上并没有取得实质性的突破,总体而言,只是停留在模拟大脑的阶段,并不能进行真正的语义推理。另外,在生物医学领域存在大量的先验知识(研究文献、成果数据库等),但ML对先验知识仍然不能有效利用。深度学习能给出答案,但无法解释背后的逻辑关系并将其结果整合到现有知识中去。换言之,深度学习并不是在真正的扩展我们的知识,而只是解决“黑匣子”问题。
基于本体的KRR技术有望帮助ML和深度学习。人工智能的核心是数据,而大部分数据是非结构化的,要更好地实现人工智能,我们需要把数据做归一化和结构化。当前,将所有先验集成数据进行人工或计算机自动标注已经具备现实可行性。本体技术的出现恰逢其时,利用本体技术对数据语义标准化被越来越多的专业人士接受和使用。基于本体的生物医学等相关领域的先验知识可以被计算机和AI自动识别,这给ML的大范围的自动利用提供了基础。此外,本体是基于计算机可以理解的逻辑规则产生的。我们可以把这个特点加到ML算法中并开发出更加强大的机器学习功能。可以预测,基于本体的KRR将在人工智能上发挥越来越重要的作用。
ML也可对KRR和本体开发发挥正向促进作用。现阶段本体的构建基本是人工完成的,如何利用机器学习和自然语言处理等人工智能的方法自动从文本等非结构化或半结构化数据上自动构建、更新和融合复杂本体会对现代生物医学大数据与精准医疗带来积极帮助。同时,更好的ML和深度学习方法也可以推动更好的本体知识的查询和KRR方法的建立。
4 智能精准医疗的兴起与挑战
精准医学(precision medicine),又称精准医疗,是针对患者的个性化医疗保健、医疗决策与治疗。精准医学模型通常会根据患者的遗传成分、分子或细胞分析、病理影像及临床健康数据进行综合分析,找出最佳的治疗策略。精准医学研究自2015年美国政府率先启动以来,在全世界引发研究热潮,国内也是成为热门课题。
精准医学起源于这样一个科学认知,即实验动物做出来的结果经常对人的医疗没有指导作用。这是因为一方面实验动物与人有着各种各样的基因差别;另一方面,试验用老鼠一般是inbred老鼠且实验条件是特别控制的,所以需要关注的变量较少(图5A)。然而人是更加复杂的动物,而每个人又存在个体差异性(图5B)。美国国立卫生研究院(NIH)资助的GUDMAP项目专注于老鼠模型的肾研究,然而,多年研究却发现基于老鼠模型得到临床结果基本不适用于人类。因此,现在NIH资助的大型肾精准医学项目(kidney precision medicine project,KPMP)已经用人作为直接对象研究了(详情见后)。
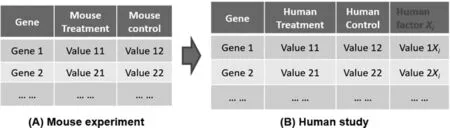
(A)老鼠实验变量少 (B)人的临床研究影响变量多
当今医学人工智能技术与互联网和物联网技术息息相关。物联网医学是将物联网技术应用于医院信息化、健康辨识与管理、诊断和治疗等人口健康领域而形成的一个交叉学科。近年来,兴起的“智慧医疗”即是物联网医学应用的典型案例。智慧医疗是通过建立一个有机的健康档案信息平台,利用先进的物联网技术,实现患者与医务人员、机构及设备之间的互联互动,以达到信息化智能化的医疗[15]。最新的5G技术对于智慧医疗的发展将会起到极大的推动作用。
综合以上各种模式,我们提出智能精准医学(intelligent precision medicine,IPM)的概念。在互联网和更进一步的物联网的基础上,智能精准医学的基本核心是把人工智能与精准医学相结合,更好地服务于精准医疗。怎样通过人工智能的手段来加强精准医疗的应用效果是个巨大挑战。在此过程中,关键问题还是要精确处理好高速涌现的大量的异构化数据。智能精准医学需要把这些大数据完整处理并发现它们之间的相互关系,这并不是一项容易完成的任务。下文我们将论证构建具有互操作性的本体是完成智能精准医学挑战的关键。
5 “河马假设”:互操作性本体对智能精准医疗的关键作用
2016年ScientificData杂志发表了一篇题为“FAIRGuidingPrinciplesforscientificdatamanagementandstewardship”的文章,正式提出了FAIR原则,即数据可查找(findable)、可访问(accessible)、可互操作(interoperable)和可重复使用(reusable)[16]。现在FAIR原则已被国内外广泛采纳。2018年6月发布的美国NIH数据科学战略计划明确承诺确保该机构支持的所有数据科学活动和产品遵守FAIR原则。
在FAIR原则中可互操作是个关键。可互操作原则要求数据之间高度集成,数据与应用程序或工作流进行互操作,以便进行分析、存储和处理。可互操作原则包括三个具体要求:①(元)数据使用正式的、可访问的、共享的和广泛适用的语言来表示知识;②(元)数据使用符合FAIR原则的词汇;③(元)数据包括对其他(元)数据的合理引用。本体内容本身是一种特殊的数据,所以本体之间也需要互操作。
可互操作本体可以支持数据的可互操作性,同时支持其他三个FAIR原则。比如,可互操作本体代码的应用支持数据的可查找、可访问和可重复使用。因为本体对数据的标准化与集成起到关键作用,我们把本体的互操作性做好就能够把数据之间的互操作性做好。但是,一方面,很多生物医学领域的词汇没有被构建为本体,只有少量的数据资源采用本体指导的策略,这限制了数据的互操作性和分析能力;另一方面,当前生物医学领域已有数百种本体,但是它们的互操作性尚显不足。已有本体之间的术语常常冗余且无法相互识别,导致数据无法互操作。随着生物医学本体的深入开发,确保本体的互操作性以及使用可互操作的本体进行标准化数据表示和集成变得至关重要。
这里,我们提出一个“河马假设”(HIPPO hypothesis),或称“智能精准医学-互操作性本体假设”。“河马假设”可以表示为“Hypothesis of Intelligent Precision medicine and its Positive correlation with interoperable Ontologies”:未来智能精准医学的发展进程与具有互操作性的本体体系构建成正相关并相互影响。
“河马假设”至少包括两方面的内容。首先,当智能精准医学继续发展时,我们需要更加具有互操作性的本体体系。现在的精准医学使用的可以跨学科的互操作性本体数量和范围远远不够,这与人工智能在精准医学领域的低水平应用相符合。但随着人工智能在生物医学领域各方面的扩展及技术的提高,我们认为互操作性本体的需求会更多;其次,更具有互操作性的本体体系也会对智能精准医学的发展起到促进作用。我们认为互操作性的本体数量、质量及覆盖面会越来越好,同时基于互操作性本体研发的优秀算法与软件会越来越多,这样对智能精准医学的发展和应用也会有极大的推动作用。虽然现在刚刚开始,但是我们预计智能精准医学的应用与具有互操作性的本体体系的开发使用这两者之间有个正相关的关系并且相互影响,共同提高。
目前,互操作性本体的发展还处于初级阶段。全世界生物医学领域有数千种常用的数据库和知识库,但是它们大部分没有本体化,更不用说互操作性的本体化。互联网与物联网的产生使得我们可以把各种各样的事物联系起来,但实际操作上我们无法把所有事物整合成单个数据库或单个本体,这意味着我们需要许多本体,并且为了使所有本体相互理解,我们需要这些本体可互操作。
前面提到各种基于语义网的链接数据系统也是一个数据库,也需要本体。尽管链接数据系统使用各种各样的本体对数据进行标准化,但是链接数据所基于的本体通常是不可互操作的,从而使链接数据系统成为孤岛并且难以集成。为了打通不同的链接数据系统,我们需要使用具有互操作性的本体体系。当前,互操作性本体体系构建选择开放性生物信息本体集(the open biomedical ontologies,OBO)。开放性生物医学本体铸造工厂(OBO Foundry)是众多生物医学本体开发者合作的一个具有重大影响的国际联盟(http://obofoundry.org/)[17]。OBO Foundry共同开发了一系列原则,如开放、合作和使用通用格式[17],目的是开发一类具有互操作性并可以用于大数据标准化和应用的本体体系。目前OBO本体库已有180多个本体,如BFO[18]、 HPO[19]、GO[3]、OBI[20]等。
6 构建基于互操作性本体的智能精准医学体系
怎样才能建立一个好的具有可互操作性的生物医学本体呢?我们提出了一系列可扩展的本体开发原则(eXtensible ontology development,XOD)[21]。XOD原则包括四项(图6):XOD1-本体的再利用原则(term reuse);XOD2-本体的语义对齐原则(semantic alignment);XOD3-基于本体设计模式原则(ontology design pattern);XOD4-本体的社区合作开发原则(community extensibility)。其中,XOD1本质上就是“拿来主义”,强调重用现有可靠本体中的术语;XOD2强调有机整合;XOD3侧重于快速有效添加新术语与注释;XOD4体现整体原则。
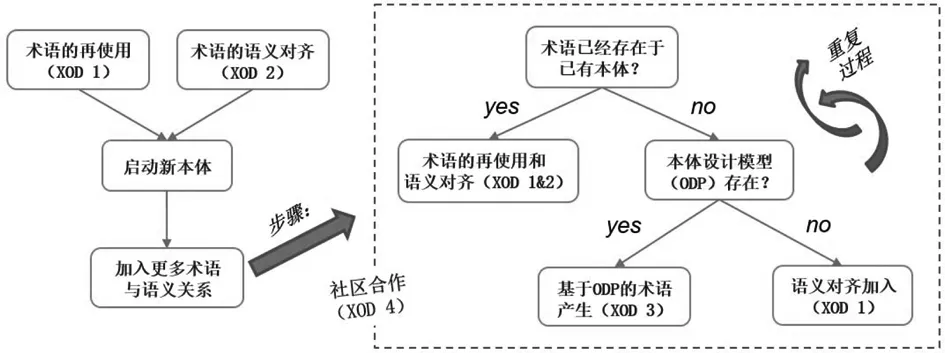
图6 基于XOD原则开发互操作性本体
很多适用于构建互操作性本体的工具已被开发。例如,ontoanimal(本体动物)工具箱包括各种用于支持本体开发的在线工具,如ontofox(本体狐狸)[22]、ontodog(本体狗)[23]和ontorat(本体鼠)[24]。ontofox和ontodog可用于支持提取并再利用其他本体的术语。这两个工具可以提取选定的类、属性、注释及其相关术语并以OWL格式保存结果,生成的OWL输出文件可以使用owl:imports的功能导入到新开发的本体中。本体的语义对齐可以通过预先设计的方法加入到ontofox输入程序中[22]。基于特定的本体设计模式,我们可以设计Excel制表并用它来收集和储存数据,然后用ontorat自动把Excel文件里的信息转化为OWL本体格式文件。例如,我们用Ontorat自动生成了超过1,000个日本RIKEN研究院收集的细胞系信息到细胞系本体(cell line ontology,CLO)[25]。这些Ontoanimal在线工具被广泛应用在生物医学本体开发社区中,尤其是对于那些没有或只有有限的软件编程技能的本体开发者来说非常实用。ROBOT是一个命令性Java工具,支持多种XOD原则,可以用来提取本体术语和子集,并且还具有许多其他功能[26]。
近年来,基于社区的互操作性本体在国内也在逐步展开。例如,刘清平等应用XOD的方法开发了风湿病中医本体(ontology of chinese medicine for rheumatism,OCMR),并应用OCMR系统分析了26种抗风湿中药。风湿病代表任何以关节、肌肉或结缔组织发炎和疼痛为特征的疾病。长期以来,中药已被用于治疗风湿病。已知抗炎和抗恶性增殖作用对于抗风湿病药物很重要。但是,具体的中药抗风湿病机制仍不清楚。这项研究首先系统地收集了有关26种传统中药饮片药物的信息,基于ontofox软件,采用NCBITaxon物种分类本体、不良反应本体(ontology of adverse events,OAE)和ChEBI[27]化学成分本体分别表示这26种物种及其相关的184种不良反应和555种化学成分以及与这些术语有关的上层结构和关系,这些关系再被有机组合在一起形成OCMR(见图7)。
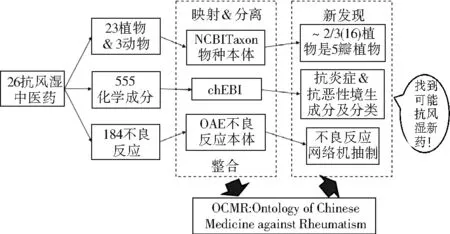
图7 抗风湿中药的互操作性本体的构建过程与结果分析
通过OCMR本体的分析得出了一些新的科学认知。例如,26种有抗风湿药效的物种中(3种动物和23种植物)有16种(约2/3)植物是五瓣植物;对来自抗风湿中药中555种化学成分的ChEBI本体分析确定了18种抗炎的化学成分,33种抗肿瘤成分和9种同时具有抗炎和抗恶性增生作用的化学物质(包括3种二萜成分和3种三萜成分);此外,22种二萜和23种三萜,包括16种可能对风湿病具有生物活性的五环三萜,被预测为可能的新的抗风湿病的化学药物。验证这些预测的生化以及动物模型实验正在设计与实施。
OCMR把从NCBITaxon、ChEBI和OAE等本体提取出来的术语及关系有机地整合在一起(XOD2原则)。这种整合并不只是简单的信息叠加,而且还包括新加入的术语之间的语义连接。例如,通过一个语义连接关系has_part,我们可以把一个抗风湿中药与它的化学成分连接起来。有了这样的整合,我们能够做更好的数据分析与查询,如我们可以很快地找到哪些抗风湿病中药有五环三萜的化学成分。同时,OCMR也是一个永久的抗风湿病中医本体,它可供以后拓展和进一步研究,这种新颖的本体方法也可以应用于其他中药的系统表示和分析。
另一个例子是万灵等[28]开发的ICDO (international classification of diseases ontology)本体。ICD是在全球范围内识别健康趋势和相关健康问题统计的基础。常用的包括ICD-9、ICD-10和目前国内还没普及的ICD-11,每个版本包含2万多条目术语。很多国家已采用ICD标准并开发了自己的修改版本,如美国版的ICD-10-CM和德国版的ICD-10-GM。在国内外医疗管理部门中,ICD被广泛用作各类系统中疾病分类的受控术语,如HIS(健康信息系统)、LIS(实验室信息系统)、PACS(图片存档和通信系统)和EMR(电子病历)。ICD代码和疾病诊断相关分组(DRG)是医疗保险控制的主要方法,DRG依赖于ICD的正确性。
国内的ICD系统有很多问题。临床医生给出的疾病名称是使用中文自然语言的,使用现有的IT工具缺乏语义理解能力,无法获得语义层面的一致性。同时,更为严重的是疾病名称存在多种地域性表达类型,在实践中,我国在ICD10的标准应用上存在多达10种以上的版本(包括国家标准V.1.1、GB/T14396-2016和国家临床1.1版),卫生行政管理部门逐级上报采集的数据由于各级信息平台所采用的数据标准的版本差异,导致数据非标准化现象极为严重,语义上的错配使最终结果错误率增加,数据有效利用率大减。不同版本之间的巨大差异可能会导致许多问题,例如,出现具有不同值但代码相同,或具有不同代码的相同值的大量数据。又如,DRG收付费改革试点工作的开展,标志着我国医疗服务机构收费制度改革首次上升到国家战略层面。然而,DRG收费模式的成功必须依赖于医疗服务相关信息的标准化,即治疗效果必须采用统一的信息标准表示,如同一个病情应该用统一的疾病编码。这也会影响基于ICD的DRG分组的准确性、Medicare支付的准确性以及死亡原因的统计准确性。
本体技术是解决不同ICD版本之间语义映射问题非常有效的工具。ICDO将每个疾病分解成不同的成分,并根据我们的疾病设计模式标记成分(见图8)。目前,OBO工场内的DO[29]和MONDO疾病本体都把疾病描述为一种倾向(disposition),指的是还没有发生的事。但是,ICD中的疾病应该更好地表示为疾病过程(disease process,DP),因为它指的是已经发生的事。这个疾病过程实现(realizes)疾病的倾向。ICDO的疾病模式同时将每个疾病定义为:caused by某个病因,occurs in某个解剖部位,has quality某个性质,has disease output某个病理解剖体。比如,作为疾病过程的一个子类,急性中耳炎DP发生在中耳,有炎症病灶产生,是一个急性的过程。ICDO开发的优势是多样的。ICD只有疾病术语,但ICDO除了疾病术语还包括解剖体(anatomic entity)、性质(quality)和病原(etiology)等,并通过语义关系把这些类型的术语串联起来(见图8)。这样做首先使得语义明晰化(见图2),可以让人工智能和计算机真正理解每个疾病的内涵和意义。据此我们也可以做各种应用,如我们可以通过各种不同的解剖部位查询所有在这些部位发生的相关疾病,我们也可以通过性质给不同疾病精确分类,这些以前只能通过自然语言处理或机器学习来大概知道。ICDO也可以对不同的ICD版本有比映射更优越的整合功能,这是因为ICDO本质上不是通过人为的规定产生的,而是通过对疾病过程的精确语义定义产生的。虽然代码不同,但不同ICD版本的具体疾病在语义上应该是一样的。所以通过基于语义的定义我们可以把不同的ICD版本整合在一起。我们还可以使用本体推理器(reasoner)对不同疾病进行有效的语义分析与工具开发。ICDO将会改善各种ICD系统之间的可用性和互操作性。ICDO还可用于数据标准化和分析不同国家不同语言之间的国际多中心临床试验、DRG分组、数据标准化和医院内部信息系统的数据规范化处理,以及区域卫生信息平台的数据标准化。
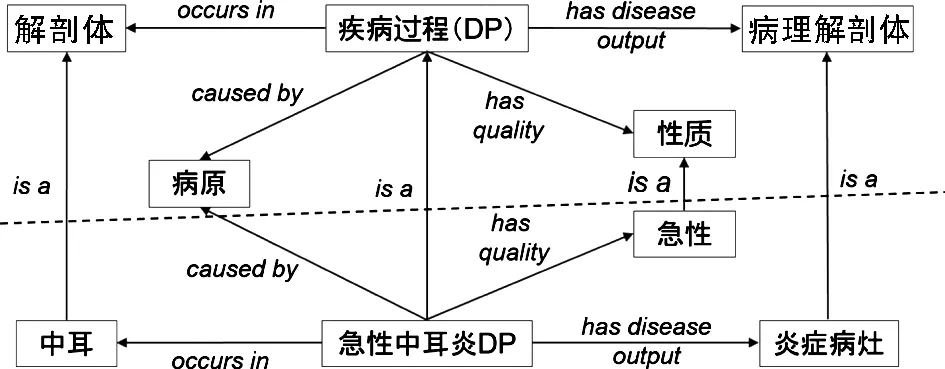
图8 互操作性的ICDO本体的设计模型及其举例虚线以上是顶层设计,虚线以下是以急性中耳炎为例进行的说明演示。
我们一般讲的疾病过程是由自然因素引起的,而疫苗与药物不良反应是一种在疫苗或药物使用之后发生的非预期的有害过程。药物不良反应是目前人口死亡的第四到第六大杀手[30]。不良反应本体OAE是一个基于社区驱动、针对医疗干预后发生的不良反应进行数据标准化和术语逻辑关系分类的生物医学本体。在顶层设计上,OAE区别“不良反应”(adverse event,AE)和“因果不良反应”(causal adverse event,CAE)。AE与医疗干预之间有时间先后关系,但没有因果关系;相反,CAE有这样的因果关系。OAE这样的定义与美国FDA的定义一致并有助于不良反应的真实报道与因果关系的分析。OAE严格按照开放生物医学本体OBO规定的开源、协作、使用通用写作语法的构建原则,逻辑化地定义了医疗干预与不良反应之间的关系及个体差异对不良反应后果的影响。
肾脏精确医学项目(kidney precision medicine project,KPMP)是目前美国NIH资助的第一个大型的以患者为研究对象的精准医学合作项目,旨在寻找人类肾脏疾病的新疗法[31-32]。KPMP主要关注的是AKI和CKD两大类肾脏疾病。急性肾损伤(Acute Kidney injury,AKI)是急性的肾功能损害,可能发展为不可逆肾功能丧失。慢性肾脏病(chronical kidney diseases,CKD)表现为肾功能逐渐下降,并最终导致终末期肾病。AKI和CKD在人群中的患病率都很高,导致严重公共卫生问题。这些肾脏疾病有复杂的致病机理和环境因素。肾脏的起源和发展尚未完全了解,妨碍了针对许多肾病的有效预防和的治疗。美国NIH以前资助十多年的针对肾病的老鼠模型实验,但转化效果不好。KPMP联盟一共包括20多个大学院所,涉及招募、临床数据采集、肾组织活检、病理检查和Omics分析。所有数据最后汇总、处理、可视化并做系统分析,各种人工智能手段也正在被开发。
KPMP同时资助了两个开源互操作性本体的开发[31]。精准医学及研究本体(ontology of precision medicine and investigation,OPMI)是一种针对精准医学领域的参考本体,它表示用于描述并整合精确医学研究领域的各种数据与元数据,帮助数据的标准化与语义分析。美国的KPMP项目包括大约30个临床报告表格(clinical report forms,CRFs)。这些CRF包括超过2,000个的临床问题和大量临床术语。OPMI被用来对这些临床元数据进行标准化,从而显著改善了不同机构之间基于本体的数据集成[33]。KPMP也启动了肾脏组织图谱本体论(kidney tissue atlas ontology,KTAO),旨在用本体的方式表示和链接肾脏组织图谱的各部分,包括肾脏组织结构、细胞、基因标记物、肾病表型及各部位的关系[34]。
OPMI的另一个用例是其对通用数据模型(CDM)的本体表示及应用。CDM是临床数据处理的一个常用手段。OMOP (observational medical outcomes partnership) CDM是由OHDSI(observational health data sciences and informatics)组织开发并在全世界广泛应用的CDM[35]。OMOP CDM本质上是一个关系型数据库的模式,但OMOP CDM在数据模型层面不能区分由于医疗干预(例如手术或药物治疗)造成的不良反应或自然疾病造成的症状表型,而OPMI本体框架可提供了这种区分。在各种心脏手术后,经常会观察到符合急性肾损伤(acute kidney injury,AKI)症状的不良事件,其发生率高达30%~50%[36]。上述OPMI本体框架被用来分析一个大型的与OMOP CDM兼容的数据集,并发现了许多与心脏手术后急性肾损伤不良事件相关的特定模式[33]。
越来越多的基于本体的与精准医学有关的研究正在国内外涌现。例如,中国自然基金项目资助了谢江安等开展的利用OAE研究中国上市甲乙肝疫苗相关的不良反应分类图谱。谢江安等利用美国公开的VAERS不良反应数据库的数据和基于OAE的研究方法,研究发现美国上市的甲肝疫苗和乙肝疫苗伴随多种不良反应,并且甲肝疫苗和乙肝疫苗的同时注射会导致13种有协同增效作用的不良反应[37]。密西根大学-北京大学医学院联合研究院最近资助了一个研究胃癌与微生态相互作用的项目。每年全球近一半的胃癌新发病例来自中国,更是中国第二大癌症死亡原因。慢性幽门螺旋杆菌感染是胃癌发生的首要危险因素,但并非所有幽门螺旋杆菌感染者都会发生胃癌。胃癌的发生可能是宿主遗传易感因素、环境、幽门螺旋杆菌以及其他肠道菌群之间复杂的相互作用导致的结果。本研究应用系统生物学的方法,整合基因组测序、转录组测序、类器官建模和生物信息学的手段来深入研究这些相互作用。宿主-微生态本体(ontology of host-microbiome interactions,OHMI)[38]和其他本体将被用于这个项目中产生的临床与组学数据的整合与分析并帮助产生可验证的科学假设。
互操作性本体的应用也是刚刚起步。除了以上给出的实例,很多基于互操作性本体的算法与软件已被开发和应用。例如,Althubaiti等[39]开发了融合不同互操作性本体的识别癌症驱动基因的新方法;王丽伟等[40]开发了基于互操作性OAE本体计算药物类效应算法;Groza等[19]利用HPO和DO[29]对常见病和罕见病的语义统一等。2020年10月德勤(四大国际会计师事务所之一)的一份分析报告指出,真正具有互操作性的数据是实现以患者为中心,以预防为导向的医疗保健服务的核心;并且对具有可互操作性和安全性的数据进行人工智能分析将成为洞察力和决策流程背后的关键引擎[41]。各种各样的基于本体统计与ML相关算法也被开发出来[42-43]。
7 本体中国(OntoChina)及国内互操作本体的合作开发与应用
目前,我国已经成为生物医学科学原始数据的生产大国,但是具有国际声誉的数据产品甚少,在数据管理方面与国际先进水平相比,始终处于追赶地位。主要表现在:生物医学数据标准化和规范化建设滞后,导致数据整合和再利用困难;数据孤岛现象严重,技术、文化和管理等多方面原因导致数据公开共享程度不高,整合使用程度不高;数据质量参差不齐,数据质量管理从技术上难以落实,原始数据的再加工程度低,影响后续的分析、整合与再利用;生物医学和信息科学的复合型人才缺乏。
标准化数据及其语义化的智能处理是生物医学大数据分析的前提,只有实现了原始数据的标准化和语义化才有可能达到有用数据的人机共识,进而为实施精准医疗提供必要的数据支撑。我国现有的大数据标准化工作通常关注IT技术层面的数据归一化,对于生物医学大数据的语义标准化较少关注,既缺乏相关标准术语集,更缺乏语义标准化技术支撑系统。生物医学大数据的语义标准化指的是在统一规范的标准术语集指导下,通过技术手段对现存储于各类生物医学数据库中的海量信息点实现语义层面的内涵一致性工作。我国现有的绝大部分的生物医学数据库(集)尚未达到“科研数据库(集)”或“临床试验数据库(集)”的标准,在未实现生物医学数据语义标准化的状态下,既缺乏相关标准术语集,更缺乏语义标准化技术支撑系统,极大制约着生物大数据的研究、分析、发掘和利用。
语义标准化的核心是本体化。同时,为了各种各样的数据之间能够打通,我们不只需要基于本体的语义标准化,而且需要具有互操作性的本体构建及其基于互操作性本体的语义标准化。为了加速国内生物医学信息本体的研究,通过提高本体共享和应用促进产业健康发展,2017年国家人口与健康科学数据共享服务平台(现改名为国家人口与健康科学数据中心)成立了“中国生物医学信息本体联合工作组”(China Biomedical Ontology Consortium),简称本体中国或OntoChina(http://ontochina.org)[44-45]。本体中国宗旨是:致力于通过生物医学领域的广泛协作,引入先进本体建设理念和模式,建设为国内生物医学信息系统和相关领域科学研究服务的本体资源;促进生物医学本体在信息化建设和科学研究中的使用。本体中国面对全社会的组织及个人开放。
在过去三年中,本体中国系统引入并翻译basic formal ontology (BFO)[18]、OBI、relation ontology (RO)[46]、ontology for general medical science (OGMS)、human phenotype ontology (HPO)[19]和cell line ontology (CLO)[25]等OBO Foundry本体,并整合了LOINC、ICD-10和ICD-11等中文术语本体资源;利用NCBO BioPortal框架,建设了 MedPortal 本体资源库,提供整合的本体服务;此外,还在国内建立了Ontobee和Ontofox的工具服务(http://ontoanimals.bmicc.cn/),为开发标准化、规范化的本体提供软件支持。新冠肺炎疫情发生后,OntoChina成员也参与共同开发了coronavirus infectious disease ontology (CIDO)本体[47]。我们也正在开发基于互操作性本体的OntoChina本体元数据的体系。
基本形式化本体(basic formal ontology,BFO)[18]已经被200多种本体用作上层本体。OBO工场现在有可与BFO顶层本体相切合的100多个生物医学本体。BFO包含两个分支,“常体”(continuant)和“行体”(occurrent)。常体表示与时间无关实体(如物质实体),行体表示与时间相关的实体(如过程)。使用BFO作为上层本体,能实现与其他100多个符合BFO的本体的无缝集成。目前遵循BFO生物医学本体大多侧重于基础医学方面,临床医学有关的本体还较欠缺。
朱彦等专家也翻译了MIT出版社出版的Barry Smith等撰写的BFO本体著作“BuildingOntologieswithBasicFormalOntology”。本书已由人民卫生出版社在2020年出版。本译著将第一次系统性地向国内读者介绍BFO及基于BFO构建本体的理论、方法和技术,是一本不可多得的入门教程和参考书籍。
杨啸林等把国内的细胞系基于国际通用细胞系本体(cell line ontology,CLO)格式开发出CLO的中文版。该本体将中国国家实验细胞资源共享平台(Chinese National Infrastructure of Cell Line,http://cellresource.cn/)中的2704种细胞系信息整合入国际版CLO细胞系本体中,建立了国内细胞系与国际细胞系信息学上的对应,对国内细胞特征的详细描述设计了新的语义表达模式,并以符合OBO规范的双语言表示呈现。该版本CLO的构建,对于帮助实现国际范围内细胞系信息整合具有支撑作用。
中医药领域也正借鉴OBO Foundry原则理念和可扩展互操作性本体开发的策略方法,使用BFO作为上层本体来构建中医药领域本体,搭建与现代生物医学知识体系互联互通的桥梁,这将是中医药的现代化与国际化工作的一个重要环节。
本体中国将进一步推进本体在国内的研究与规范化应用,促进国内本体研究社群的发展与合作。第一,提供中英文本体资源服务平台,提供更多的中文特色服务;第二,吸收国际经验,推进规范化本体资源建设,将国内数据资源与本体相结合;第三,推进本体在生物医学数据管理和建设方面的应用;第四,建立广泛的交流合作平台,促进国内国际间关于本体的交流合作。
8 倡议、前景与展望
在健康医疗领域随着信息技术与医疗的深度融合,大数据时代亦随之到来。大数据标准体系框架尚处于顶层设计阶段,缺乏实际应用支撑。上述框架中,数据类标准指生物医学大数据采集、表达、处理、传输和交换等过程中涉及的相关数据标准,是保证语义层无歧义的重要基础。包括数据元标准、分类与编码标准、数据库(集)标准和共享文档规范等。本体可以在数据的结构化、共享和智慧分析中起到关键作用。
在本体技术的使用方面我国与国际先进水平有差距,但也在迎头赶上。OntoChina的组织与推广行动也必将使本体技术在中国的推广和应用获得更大的空间。为了更好地促进中国的最广泛的本体合作开发及其在生物大数据与精准医学上的应用与推广,我们在此提出以下倡议:
①鼓励加入、共商共建、合作开发,促进互操作性本体的开发、应用与推广。
②轮值主持,开放、透明、公正、公平的运行机制。
③参与国际本体领域的合作交流、优秀本体的翻译和引用。
④促进有中国特色的互操作性本体(如与中医药有关的本体)的开发与推广。
⑤基于互操作性本体的数据整合以及人工智能算法与软件的研究与开发。
⑥产学研共同发展,积极响应产业需求,形成产业与研究良性互动。
我们鼓励和欢迎商业应用与投入。工业本体工场(industrial ontologies foundry,IOF)在欧美已经成立(https://www.industrialontologies.org/),其进展值得我们时刻关注。我们可以基于OBO和未来的IOF本体进行算法与软件开发并应用于临床实践解决具体问题。在解决具体问题的过程中我们又会产生新的灵感开发更好的工具与功能。这些不只是在国家科学数据管理层面有所帮助,而且在医疗卫生信息业务数据标准化服务、公共卫生数据标准化与分析定制化服务、文献挖掘与数据分析、生物医学知识本体化管理等方面都意义重大。我们也需要基础生物医学研究与临床医学数据融合及精准医学产品开发服务,如前所述的ICDO与中医药本体的开发与应用。
医学人工智能的发展伴随着更高的医学伦理要求。医学人工智能可以极大地提高我们的医疗服务质量,但如果没有事先预判其可能引发的医学伦理问题将会带来各种潜在危险。例如,医学人工智能实施过程中使用的数据标准、储存、安全和共享,必然涉及个人隐私和知识产权,进而关联其背后的伦理、法制等一系列问题。另外,当前人工智能的不可解释性在医疗领域可能面临更大的伦理挑战。例如,假设医疗AI在医疗活动中犯了致命错误,那么谁来承担这个责任。是医生吗?是写程序的程序员?这些涉及医学人工智能的伦理问题必须仔细分析、广泛探讨,找出合理有效的解决方法。只有这样,我们的医学人工智能才有可能真正走向临床实践。
(致谢:感谢中国医学科学院关健教授邀请何勇群参加在温州举行的“第四届健康医学〈大〉数据共享与合作高峰论坛”并作主题报告,本文基于该主题报告内容展开。感谢关健教授对本论文在起草、撰写、发表等过程中的指导和帮助。)

