应用环境DNA 技术对邵伯湖浮游动物物种检测的初步研究
唐晟凯,钱胜峰,沈冬冬,张彤晴,刘燕山,许飞,王华,李大命
(1.江苏省淡水水产研究所,江苏省内陆水域渔业资源重点实验室,江苏 南京 210017;2.江苏省高宝邵伯湖渔业管理委员会办公室,江苏 扬州 225009)
引言
在水体生态环境中,水生生物通过新陈代谢向水体中释放DNA。“环境DNA 技术”是环境DNA 宏条形码技术的简称,是通过从环境介质中提取DNA,对样本片段进行生物测序,进而了解环境中的生物群落分布情况[1-3]。环境DNA 技术最早应用于微生物学研究,近年来逐步被应用于水生生物的监测[4-13],但目前该技术在湖泊浮游动物物种检测方面的应用仍然较少。
邵伯湖位于江苏省扬州市,与高邮湖和宝应湖共同组成高宝邵伯湖,属淮河入江水道上的湖泊之一,在渔业生产、蓄洪、农业灌溉等方面具有重要作用。在2016 年的调查中,邵伯湖的浮游动物共计有12 科16 属20 种[14]。该研究运用环境DNA 技术对邵伯湖浮游动物进行资源监测,一方面为邵伯湖渔业资源与水生生物资源保护提供了基础数据,同时也探索了在浅水湖泊进行浮游动物监测的新方法。
1 材料与方法
1.1 采样点位设置
在全湖设置了15 个采样点,其中北部敞水区、中部敞水区和南部河道区各设置了4 个、5 个和6个采样点。采样点位置分布见图1。
1.2 采样及样品前处理
样品采集于2018 年11 月7 日。在每个采样点,用无菌聚丙烯瓶(Thermo;US)采集2 L 表层水(在水面以下约20 cm 处采集),现场用0.8 μm 孔径的混合纤维滤膜(易基诺;南京)过滤。过滤后的滤膜置于-20 ℃冰箱,冷冻保存,直到DNA 提取。
1.3 DNA 提取
使用DNeasy Power Water Kit(QIGEN;Germany)提取滤膜DNA,具体操作如下:将滤膜置于带有研磨珠的5 mL 离心管内,加入1 mL 的55 ℃PW1,均质5 min,全部吸取并离心1min;取上清加入200 μL IRS,涡旋混匀后于4 ℃静置5 min,离心1 min;取上清加入650 μL PW3,混匀后过MB Spin Column富集DNA;分别加650 μL PW4 和650 μL 乙醇清洗,用100 μL EB 洗脱。使用Nano Drop 2000 测定DNA浓度和纯度,并于-20 ℃冷冻存储。
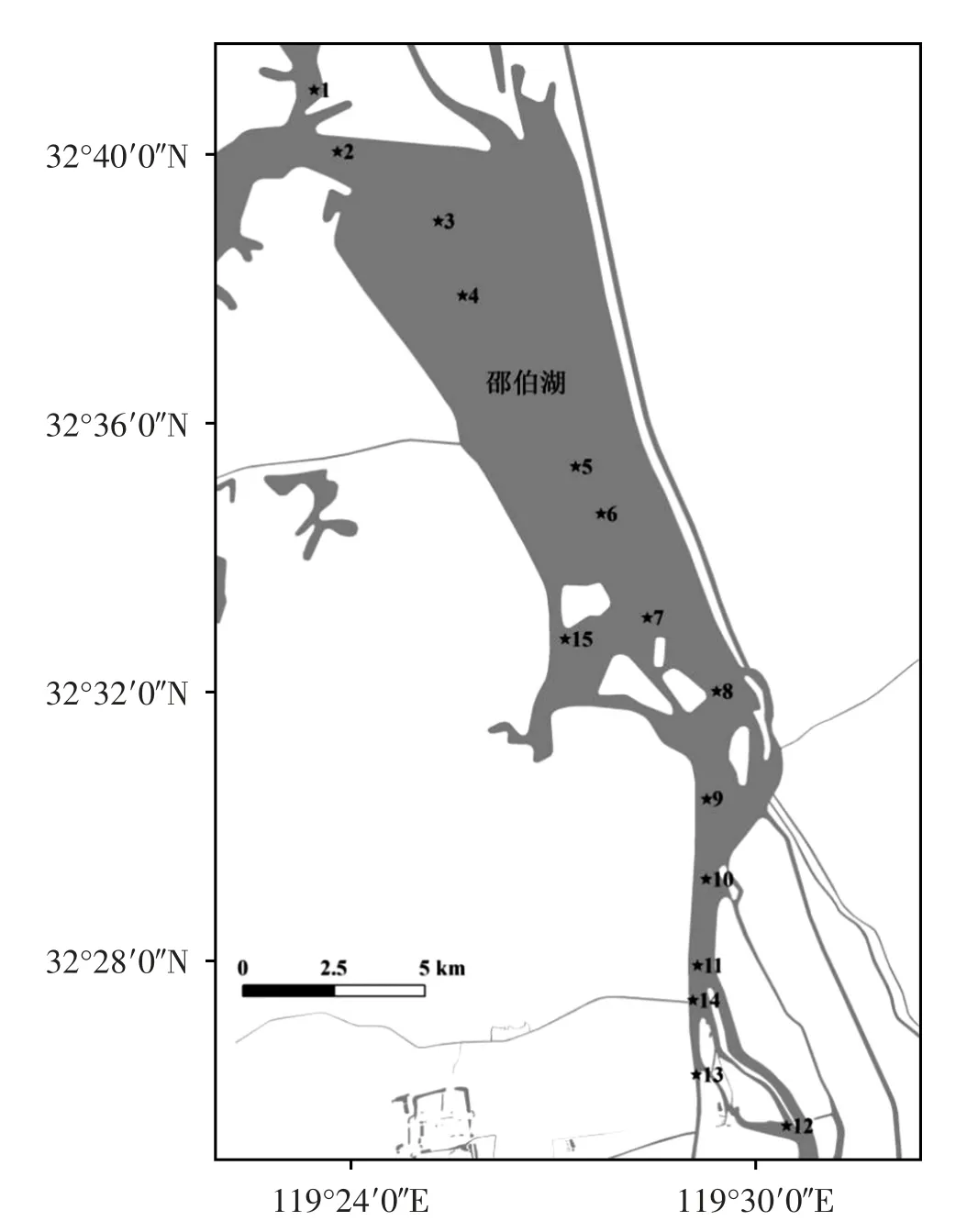
图1 采样点位置
1.4 PCR 扩增
利用线粒体DNA 12S rRNA 引物进行PCR 扩增,PCR 扩增体系:总体系50 μL,包含2X Phusion Master Mix with HF Buffer 25 μL(Thermo;US),上下游引物各2 μL,DNA 模板2 μL,ddH2O19 μL;扩增条件:98 ℃预变性30 s,98 ℃变性10 s,66 ℃退火20 s,72 ℃延伸20 s。以ddH2O 为模板设置PCR 阴性对照。PCR 产物使用2%琼脂糖凝胶电泳进行条带检测,PCR 阴性对照扩增结果为阴性。使用AMPure XP(Beckman Coulter; US)磁珠纯化PCR产物。
1.5 高通量测序及数据分析
使用NEBNext Fast DNA Library Prep Set for Ion Torrent 试剂盒(NEB;US)构建二代测序文库后,Agilent 2100 检测DNA 文库质量,稀释DNA 文库至100 pmol/L,利用Ion Proton 测序仪进行样品的二代测序。二代测序数据下机后,数据分析全部基于ubuntu 14.04 版本下的EcoView 软件。
2 结果与分析
2.1 环境DNA 浓度与PCR
环境DNA 浓度介于17.3 至179.3 ng/μL 之间,平均浓度54.25 ng/μL;DNA 样本的260/280 介于1.74 至1.93 之间,平均值1.83;DNA 样本的260/230介于0.98 至2.13 之间,平均值1.54。其中,82 个样本(94.44%)的DNA 浓度>20ng/μL;87 个样本(100.00%)的260/280 介于1.8 至2.0 之间,87 个样本(94.44%)的260/230 介于1.8 至2.0 之间(图2)。
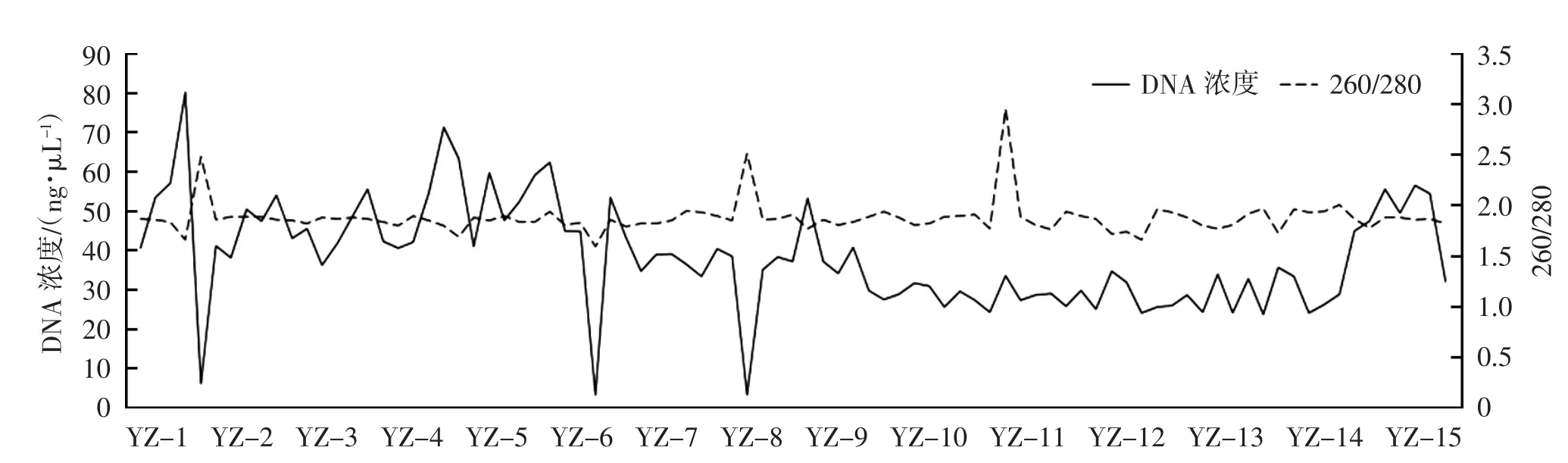
图2 样本DNA 浓度及260/280
2.2 物种检测
该次测序共获得邵伯湖1 114 482 条DNA 序列(Reads 数),DNA 片段长度约为380 bp。15 个采样点均获得DNA 序列信息测序数据共聚类形成646 个OTUs,注释出的浮游动物共有68 个OTUs,隶属于5 目14 科18 属22 种(图3),其中鳃足纲共有26 个OTUs ,包括1 目6 科7 属8 种,象鼻溞科、溞科、粗毛溞科、裸腹溞科和仙达溞科各1 种,薄皮溞科2 种;甲壳纲共有17 个OTUs,包括2 目2 科3 属3 种;单巢纲共有25 个OTUs,包括2 目6科8属11 种,聚花轮科、三肢轮科、晶囊轮科、疣毛轮科和鼠轮科各1 种,臂尾轮科6 种。在国内外研究工作者的基础上,根据浮游动物的大小、摄食习性以及浮游动物之间的相互作用,将淡水生态系统浮游动物划分为原生动物滤食者PF、原生动物捕食者PC、轮虫滤食者RF、轮虫捕食者RC、小型浮游动物滤食者SCF、小型浮游动物捕食者SCC、中型浮游动物滤食者MCF、中型浮游动物捕食者MCC、大型浮游动物滤食者LCF 和大型浮游动物捕食者LCC 10 个浮游功能群[15](表1)。根据淡水浮游动物功能群分类将邵伯湖22 种浮游动物分为RF(9 种)、RC(1 种)、SCF(5 种)、SCC(1 种)、MCF(1种)、MCC(2 种)、LCF(1 种)和LCC(2 种),分别占总种类数的40.91%、4.55%、22.73%、4.55%、4.55%、9.09%、4.55%和9.09%。Reads 数占比较高的物种为脆弱象鼻溞(47.79%),透明薄皮溞(20.24%),球状许水蚤(16.10%)等(图4)。邵伯湖浮游动物种类组成见表2。

图3 邵伯湖浮游动物OUTS 数量图

表1 淡水浮游动物功能群分类
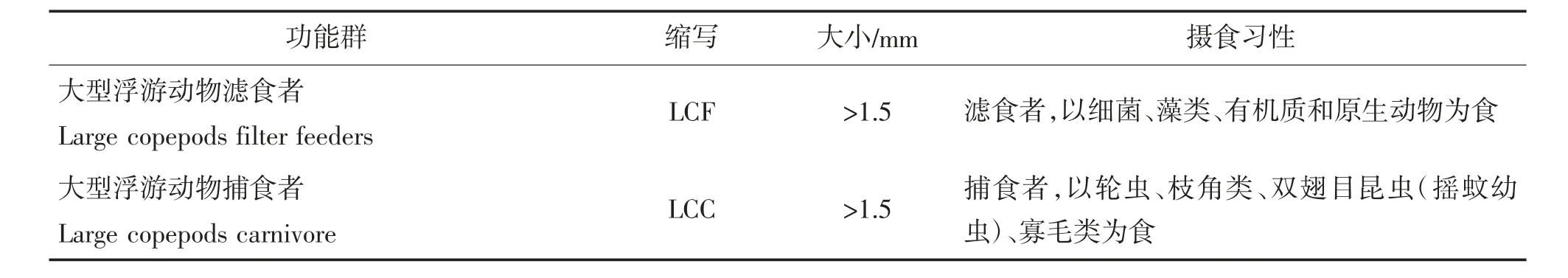
功能群 缩写 大小/mm 摄食习性大型浮游动物滤食者Large copepods filter feeders LCF >1.5 滤食者,以细菌、藻类、有机质和原生动物为食Large copepods carnivore LCC >1.5 捕食者,以轮虫、枝角类、双翅目昆虫(摇蚊幼虫)、寡毛类为食大型浮游动物捕食者
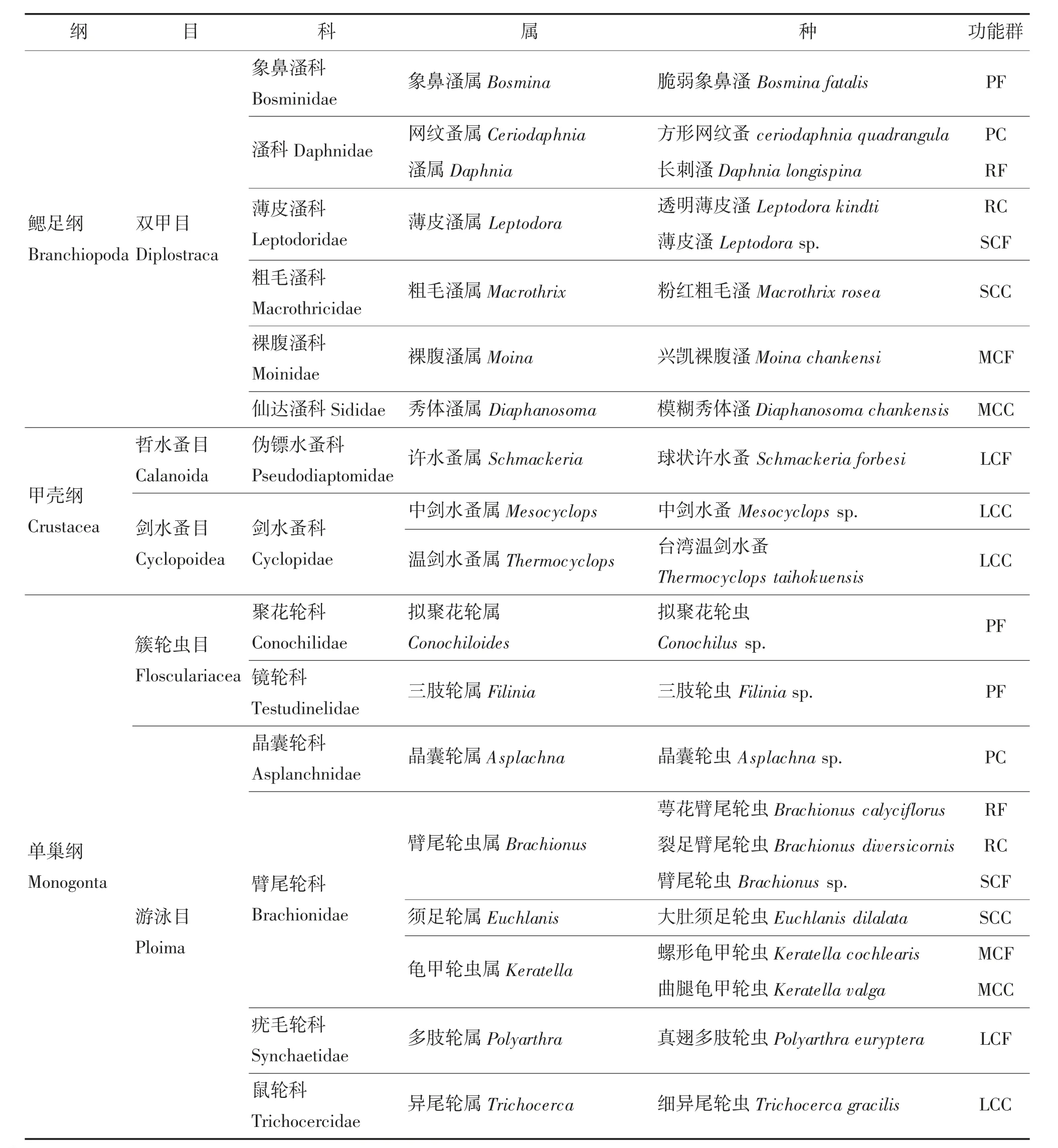
表2 邵伯湖浮游动物种类组成

图4 邵伯湖浮游动物Reads 数占比组成图
2.3 α 多样性
如表3 所示,各样点shannon 指数变化范围为1.9433~4.8113,最小值出现在YZ-8,最大值出现在YZ-9。YZ-8 的各项指数均普遍低于其他点位,表明其浮游动物多样性程度较低;YZ-9 的各项指数均普遍高于其他点位,表明其浮游动物多样性程度较高。

表3 邵伯湖各采样点位浮游动物α 多样性指数表
2.4 β 多样性
如表4 所示,各样点Bray-Curtis、Hellinger、Unweighted unifrac、Weighted unifrac 的最大值普遍出现在YZ-9 和YZ-11,由于不同群落或某环境梯度上不同点之间的浮游动物共有种越少,β 多样性越大[15]。YZ-9 和YZ-11 的各项指数均显著高于其他点位,与其他点位共有物种较少。
3 讨论
3.1 浮游动物物种检测
该研究监测到浮游动物14 科18 属22 种,其中有16 种注释到“种”,6 种注释到“属”。在2016 年的调查中,邵伯湖浮游动物共计12 科16 属20种[14]。与该次调查结果相比,粗毛溞、秀体溞属、中剑水蚤属、三肢轮属、晶囊轮属、臂尾轮属、龟甲轮属、多肢轮属和异尾轮属相同,两次调查均为单巢纲种类最多,其次为鳃足纲,甲壳纲最少;与该次调查相同种类仅有1 种,为萼花臂尾轮虫;该次调查较2016 年的调查多2 科2 属2 种。
与2016 年调查种类上有所差异可能是由于:①调查点位的设置不同。该次调查设置的点位较均匀地分布于全湖且点位较多,而2016 年调查设置的点位多位于岸边。②调查的频次与季节不同。该次调查为1 个频次,2016 年调查为1 月、5 月和9月共3 个频次。③调查方法的差异。与传统调查方法相比,利用环境DNA 技术对浮游动物进行鉴定更具客观性,而运用传统的镜鉴方法进行物种鉴定(如2016 年的调查)则主观性更强。当然,现阶段的环境DNA 技术在某些方面仍然存在一些不足[16-22],因此在现阶段的浮游动物物种检测中,环境DNA 技术尚难以完全取代传统方法。

表4 邵伯湖各采样点位浮游动物β 多样性指数
该研究中,薄皮溞sp.,中剑水蚤sp.,拟聚花轮虫sp.,三肢轮虫sp.,晶囊轮虫sp.及臂尾轮虫sp. 等6 种浮游动物的DNA 序列未注释到“种”的水平,原因可能在于选择的12S rRNA 标志基因保守性较大,对近缘物种的辨识度不够。因此将来运用环境DNA 技术对浮游动物进行物种检测时,扩增引物还需进一步优化。
3.2 邵伯湖浮游动物功能群分布分析
该研究发现邵伯湖浮游动物功能群以轮虫滤食者RF 的轮虫为主,大型浮游动物几乎缺失,浮游动物显著小型化,这可能主要是由于近年来水体鱼类资源量下降,大型鱼类尤其是肉食性鱼类持续减少,主要以小型鱼类为主,而小型鱼类对枝角类和桡足类等浮游动物摄食压力显著[23]。影响浮游动物功能群分布的因素有很多,温度、营养盐、浮游植物的上行效应、鱼类摄食的下行效应、种间竞争等是影响浮游动物生长的主要因素[24-25]。因此,浮游动物的动能群的分布,也可以反映出生态环境的整体状况,利用环境DNA 技术对浮游动物进行调查,能为水体生态环境评价提供参考。
3.3 邵伯湖浮游动物多样性分析
各样点Shannon 指数变化范围为1.9433-4.8113,最小值出现在YZ-8,最大值出现在YZ-9。YZ-8 的α 多样性各项指数均显著低于其他点位的原因较多,可能是因其处在邵伯湖多个支流汇集处而导致流速过大有关,也可能与该点位的水质状况有关。其他样点α 多样性的各项指数较为均匀,表明邵伯湖浮游动物生态结构较为均衡和稳定。
邵伯湖的β 多样性的各项指数显示最大值均出现在YZ-9 和YZ-11,与其他样点共有物种较少,YZ-9 和YZ-11 共有物种较少可能与样点的生态环境有关,温度、营养盐、浮游植物的上行效应、鱼类摄食的下行效应、种间竞争等是影响浮游动物生长的主要因素[24-25]。各样点共有物种较为集中,表明邵伯湖浮游动物的生态结构较为均衡,空间分布上较为均匀。
3.4 环境DNA 技术在湖泊浮游动物资源监测中的应用前景
环境DNA 技术为淡水浮游动物资源监测、物种多样性评价等提供了新的方法,具有广阔的应用前景。相比传统的监测方法,运用环境DNA 技术监测浮游动物具有以下主要优势:不依赖物种鉴定专家,减少了物种鉴定中的主观差异;灵敏度高,对较为稀有种类的监测效果较好;可大批量检测样品,比传统的物种鉴定节约时间与人力。但运用环境DNA 技术进行浮游动物资源监测仍存在一些局限,如缺乏技术规范、少数近缘物种因序列相似而难以区分以及无法反映浮游动物的生理状态、生长发育阶段等生物学特征等。
因此,在现阶段的淡水浮游动物资源监测中,可根据监测任务的需要,将环境DNA 技术与传统的监测方法结合使用。
4 结论
在应用环境DNA 技术对邵伯湖浮游动物资源的监测中,共检出浮游动物22 种,邵伯湖浮游动物功能群以轮虫滤食者RF 的轮虫为主,大型浮游动物几乎缺失,浮游动物显著小型化,预示了邵伯湖鱼类可能正趋于小型化;各样点浮游动物的α 多样性各项指数和β 多样性各项指数差异较小,表明邵伯湖浮游动物的生态结构较为稳定。环境DNA 技术适用于浅水湖泊浮游动物群落监测,在现阶段的淡水浮游动物资源监测中,该技术宜与传统的监测方法结合使用。


