1株基因重组猪繁殖与呼吸综合征病毒的新型变异和遗传进化分析
胡 栋,朱迎春,赵 情,庞 恒,李传刚,王亭亭,常维山,彭 军*,吴家强
(1.山东农业大学 动物科技学院 山东省动物生物工程与疾病防治重点实验室/农业部动物疫病病原生物学华东科学观测实验站,山东 泰安271018;2.山东省农业科学院 畜牧兽医研究所 山东省畜禽疫病防治与繁育重点实验室,山东 济南 250100)
猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrom,PRRS)是由猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus,PRRSV)引起的一种抑制机体免疫系统及主要引起母猪流产的传染病[1]。该病在临床上主要导致怀孕母猪流产、早产、死胎、弱胎、木乃伊胎及病猪部分神经性症状[2]。PRRSV于1987年首次在美国北卡罗来纳州被发现[3],后来经分离确认PRRSV传播至欧洲。我国于1996年首次发现并确认该病,并迅速蔓延至全国,后来又经2006年暴发“高热病”以来,该病一直不断发生,现已成为影响全球养猪业健康发展的重大疫病之一。PRRSV按基因的变异类型可划分为以VR2332株为代表的经典美洲型和以Lelystad Virus株为代表的经典欧洲型和近来确认的NADC30美洲型这三大基因型。PRRSV不同毒株主要是依据基因组的变异程度大小来进行区分,其中以Nsp2和ORF5基因的变异程度最大,ORF3次之。对PRRSV经典美洲型毒株和经典欧洲型毒株的基因组进行比较,以Nsp2所编码的氨基酸的同源性约为40%,以ORF3所编码的氨基酸的同源性约为55% ,而以ORF5所编码的氨基酸的同源性约为53%。因此,对比不同毒株Nsp2、ORF3及ORF5基因的变异程度基本上可以反映出PRRSV整个基因组序列的变异情况[4]。
本试验所采集病料猪场的病猪在临床表现出不同程度的精神沉郁、食欲减少、发热、呼吸困难;怀孕母猪流产,部分猪舍母猪可达到60%左右的流产率,早产、死胎、木乃伊胎现象严重;部分新生仔猪出现呼吸困难、运动失调及偏瘫等症状且生长速度较其他无症状仔猪慢;少数仔猪耳部及体表皮肤有紫色点状出血。剖检病猪可见心肺部有大小不一的出血点、肾脏呈点状出血、肠系膜淋巴结出血肿大、腹股沟淋巴结高度肿大且出血坏死。
通过采集山东泰安某疑似发病猪场发病猪的肺脏、淋巴结和血液等病料,经临床诊断及RT-PCR鉴定后,扩增Nsp2、ORF3及ORF5基因并克隆测序,利用相关分子生物学软件进行对比并作遗传进化分析,以了解山东地区猪场猪群感染PRRSV基因遗传变异的情况,为PRRSV基因的变异情况、流行病学研究和疫苗的研制等方面提供科学依据。
1 材料与方法
1.1 样品处理与病毒分离无菌采取山东省泰安市某猪场疑似PRRS发病猪的肺脏、肝脏、脾脏等病料组织,标记编号后于-20℃冰箱保存。取少许疑似发病猪的肺脏、脾脏、淋巴结等组织置于研磨器中,加入1 ml灭菌PBS进行研磨。将研磨充分的组织悬液反复冻融3次,4 000 r/min离心15 min,取上清液在0.22 μm的无菌滤膜中过滤除菌后,加入培养有MARC-145细胞的细胞培养瓶中,37℃、5%CO2细胞培养箱中培养。每隔一段时间观察MARC-145细胞的状态是否出现皱缩、聚集和脱落等状态。
1.2 主要试剂MiniBest Viral RNA/DNA Extraction Kit 5.0、Agarose Gel DNA Purification Kit、MiniBEST Plasmid Purification Kit Ver.4.0、PrimeSTAR Max DNA Polymerase、PrimeScriptTMⅡ 1st Strand cDNA Synthesis Kit、DL2000 DNA Marker、E.coliDH5α Competent Cells等均购自TaKaRa公司。
1.3 引物设计参考GenBank已收录的PRRSV JXA1、VR2332、CH-1a等毒株核苷酸序列,通过DNAStar软件分析对比,采用Primer 6.0设计1对可以区分经典毒株与高致病性毒株Nsp2基因的引物R1:5′-CAACACCCAGGCGACTTCA-3′,R2:5′-TGCGTAGCAGGATCACCAAG-3′,经典毒株扩增产物大小930 bp,高致病性毒株扩增产物大小840 bp;扩增ORF3全基因引物P1:5′-GTACTTTGGATCAGGTGTTTGC-3′,P2:5′-GTGACATTGGCAGTGATGGT-3′;扩增ORF5全基因引物P3:5′-ATGAGGTGGGCAACCGTTT-3′,P4:5′-ACTGGCGTGTAGGTAATGGAA-3′。病毒全基因组扩增引物序列如表1。引物送生工生物工程(上海)有限公司合成。

表1 扩增PRRSV全基因组扩增引物序列
1.4 PRRSV全基因组cDNA片段的RT-PCR扩增以出现CPE的第2代MARC-145细胞的培养物为模板用于扩增PRRSV全基因组cDNA。以2 μL RNA作为模板进行反转录,反转录程序按照试剂盒说明书进行获得cDNA。PCR采用50 μL体系,其中取cDNA 4 μL作为模板,2×Taq Master Mix 24 μL,每个体系分别应用9对引物(表1)的上、下游引物各1 μL,补ddH2O至50 μL。反应参数为预变性95℃ 5 min;95℃ 30 s,55℃ 30 s,72℃ 30 s,共32个循环;72℃再延伸7 min。取部分PCR产物进行1%琼脂糖凝胶电泳观察,其余置4℃冰箱保存。
1.5 PRRSV Nsp2、ORF3和ORF5基因的PCR扩增采用50 μL反应体系,其中取cDNA 4 μL作为模板,2×Taq Master Mix 24 μL,分别用扩增Nsp2、ORF3和ORF5基因的上、下游引物各1 μL,补ddH2O至50 μL。反应参数为预变性95℃ 5 min;95℃ 30 s,55℃ 30s,72℃ 30s,共32个循环;72℃再延伸7 min。取10 μL PCR产物进行1%琼脂糖凝胶电泳观察,并置4℃冰箱保存。
1.6 目的基因的克隆测序对目的片段作胶回收并连接pMD18-T载体,反应体系为pMD18-T载体1 μL、胶回收DNA 1 μL、缓冲液5 μL、ddH2O 2 μL。16℃反应30 min后将其加入100 μL的JM109感受态细胞中冰浴30 min,经42℃加热45 s后迅速放入冰中1 min,加入890 μL的SOC培养基并放入恒温摇床37℃振荡培养60 min,之后将培养液均匀涂布在含有Amp的琼脂板上培养过夜,挑取白斑接种于LB液体培养基,按照质粒小提试剂盒提取质粒并测序(上海生工)。
1.7 Nsp2、ORF3、ORF5基因的序列比对和分析先将测序结果通过NCBI的Blast进行比对分析,找出部分同源性较高的毒株及国内外录入的代表毒株,再利用DNAStar、DNAman、MEGA等软件对比分析。
1.8 细胞传代后病毒Nsp2基因检测将经过传代出现CPE的第5代和第10代分离毒株分别提取病毒RNA,扩增Nsp2、ORF3和ORF5基因后送测序。
1.9 重组分析根据GenBank中登录的参考序列,利用RDP4软件对分离的SD18毒株进行基因重组分析,筛选出亲本毒株和提供重组片段的病毒株。根据RDP4软件分析结果,利用SimPlot对SD18毒株进行全基因序列重组分析。
1.10 GP5蛋白信号肽预测利用在线软件SignalP4.1中的神经网络法对SD18毒株的GP5蛋白进行信号肽预测。
2 结果
2.1 病毒的分离阳性病料组织上清接种初代MARC-145细胞即出现CPE,病变细胞呈现圆缩、聚堆、拉网等状态,而对照细胞无明显变化(图1),然后对分离毒株继续传代培养。

A.正常MARC-145细胞;B.病变MARC-145细胞
2.2 PRRSV分离株Nsp2基因扩增结果经RT-PCR扩增鉴定,得到840 bp大小的条带,符合高致病性PRRSV条带大小(图2)。
2.3 Nsp2、ORF3、ORF5基因的PCR扩增经RT-PCR分别扩增Nsp2、ORF3、ORF5基因片段后,得到约1 066,872,737 bp的特异性条带,与预期条带大小相符(图3)。
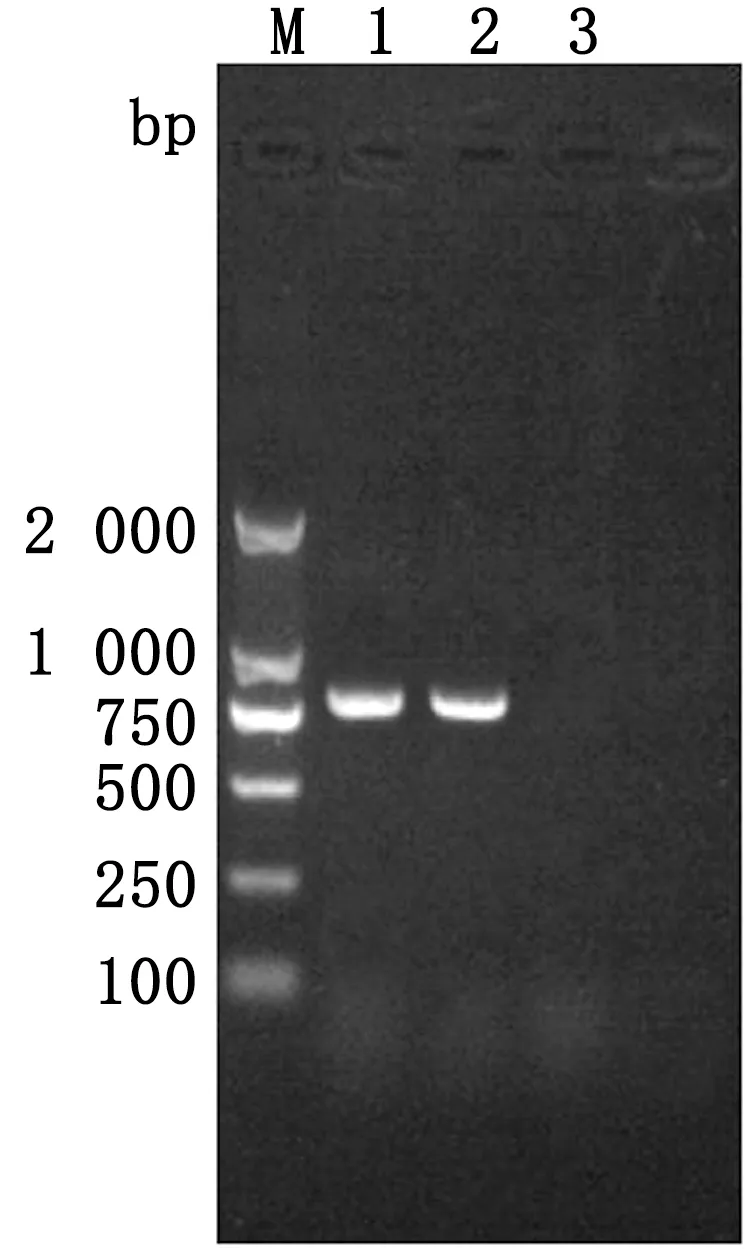
M.DL2000 DNA Marker;1.阳性对照;2.SD18毒株样品;3.阴性对照
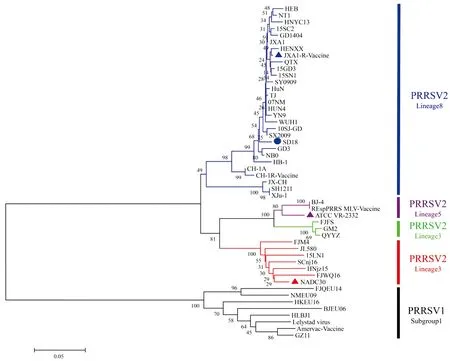
2.4 SD18毒株全基因组序列遗传进化树将SD18毒株全基因组序列与国内外参考毒株进行比较并绘制遗传进化树,结果显示PRRSV可分为美洲型毒株与欧洲型毒株2大类,且美洲型毒株又具有多个不同的亚种;SD18毒株为高致病性美洲毒株中的一个亚群,与早期国内分离株CH-1a等经典毒株相距甚远,PRRSV仍在不断变异。该分离毒株已明显区别于以JXA1、HUN4、TJ等为代表的高致病性中国分离株,形成一个新的独立分支,这些变异提示该分离毒株很可能在病毒的编码蛋白及其免疫原性等方面出现了较大变化(图4)。
2.5 SD18毒株部分氨基酸序列分析
2.5.1Nsp2基因核苷酸及编码氨基酸序列分析 运用DNAStar软件对SD18毒株Nsp2基因核苷酸及编码氨基酸序列进行同源性分析表明,SD18毒株的Nsp2基因核苷酸序列与近几年国内常见高致病性毒株的同源性可达87.0%~91.2%(85.3%~90.2%),与经典美洲毒株VR2332同源性为72.0%(67.4%),与国内的HPBEDV毒株同源性为88.0%(81.2%),与美国变异毒株NADC30同源性为74.7%(69.7%),而与经典欧洲毒株Lelystad Virus同源性为50.5%(30.2%)。SD18毒株的Nsp2基因编码氨基酸序列分别在对应VR2332毒株的830位和864位分别出现2个氨基酸的缺失,920~948位出现了29个氨基酸的连续缺失,其中第830位氨基酸位点是最新发现的缺失氨基酸。同时,Nsp2在690~894位也出现了多个氨基酸位点的变异(图5,表2)。

M.DL2000 DNA Marker;A1.SD18 Nsp2基因扩增条带; B1.SD18 ORF3基因扩增条带;C1.SD18 ORF5基因扩增条带;A2,B2,C2.阴性对照
●表示本研究的分离毒株;▲表示重组毒株

图5 SD18毒株与PRRSV参考株Nsp2基因编码氨基酸序列比对结果
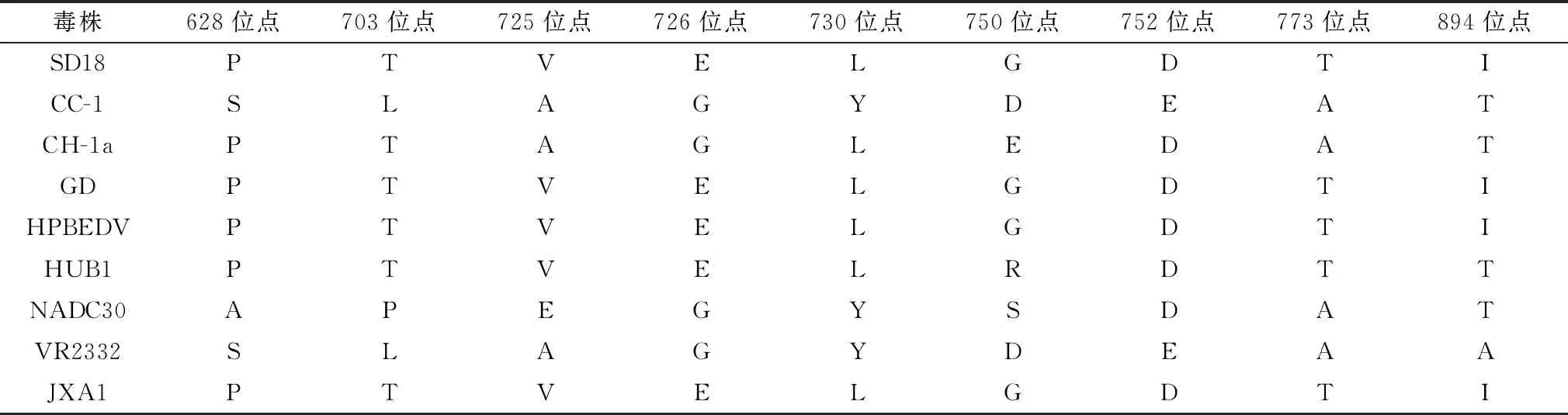
表2 SD18毒株Nsp2基因编码氨基酸突变位点
2.5.2ORF3基因核苷酸与编码氨基酸序列分析 与国内外的参考毒株进行对比分析发现,SD18毒株ORF3全基因与近几年国内高致病性毒株同源性可达87.6%~95.7%,与代表性高致病毒株CH-1a同源性为92.3%,与美国新型变异株NADC30同源性为82.5%,与欧洲经典株Lelystad Virus同源性为64.8%。而在其编码氨基酸方面,SD18毒株的ORF3在13,71,151,225,260等位点出现了与参考毒株相比不同程度的变异,在245,246和250~259位点出现了与其他高致病性毒株相同的缺失位点,主要变异情况见表3。
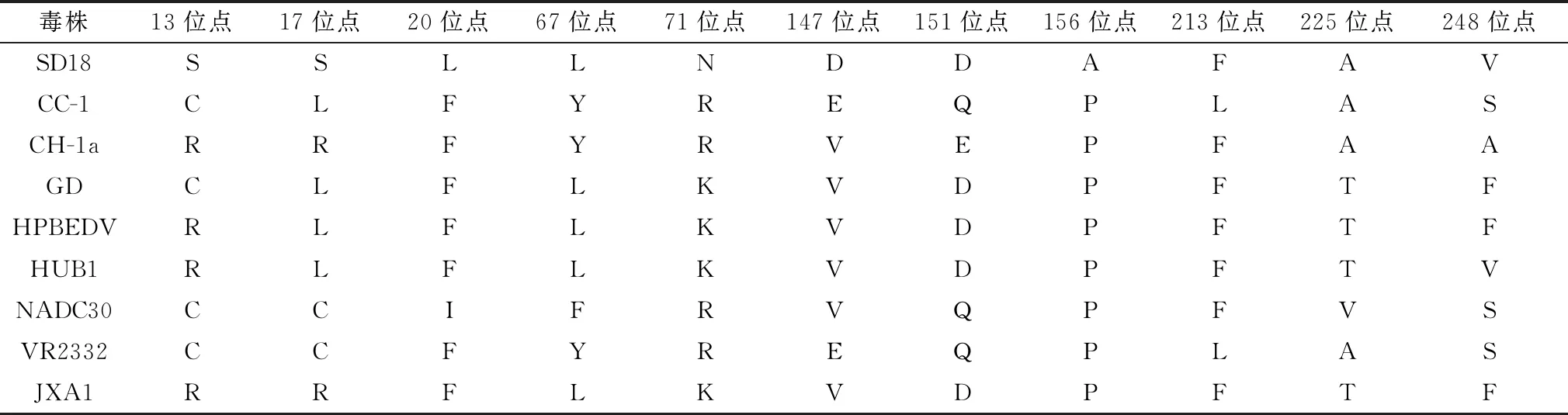
表3 SD18毒株ORF3基因编码氨基酸突变位点
2.5.3ORF5基因核苷酸与编码氨基酸序列分析 ORF5编码氨基酸序列变异性分析显示在18,30,40,44,63,100,107位点出现与其他毒株不同程度的变异(表4)。

表4 SD18毒株OR5基因编码氨基酸突变位点
2.6 ORF5基因编码产物抗原性分析与参考株ORF5基因抗原对比分析发现,SD18毒株ORF5编码产物抗原表位主要集中在6~16,30~39,50~60,128~132,136~141,146~155,161~183 aa与WUH1毒株具有相似的抗原性特征,但与VR2332毒株差异性较大,主要差异在30~39 aa区域,相较于VR2332毒株,SD18毒株抗原区域明显变窄,而在6~16,50~60,136~141 aa等区域,SD18毒株抗原性却明显高于VR2332毒株(图6)。
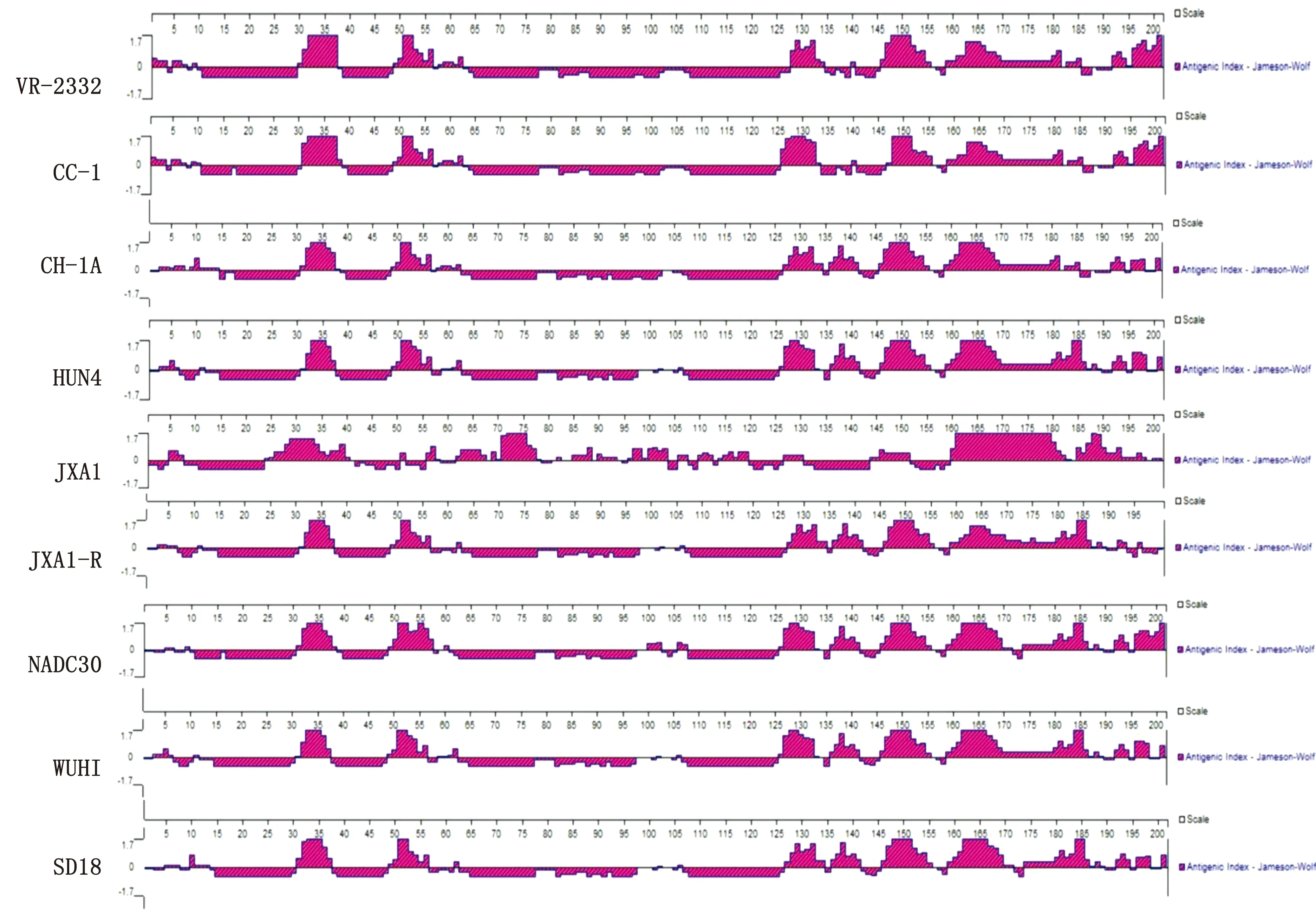
图6 PRRSV SD18毒株ORF5基因编码产物抗原性预测分析
2.7 分离株传代后Nsp2基因检测扩增出现CPE的第5代和第10代细胞SD18毒株的Nsp2、ORF3和ORF5基因并测序,结果显示其基因序列并未发生改变,条带大小也未见变化(图7,8)。
M.DL2000 DNA Marker;A1.SD18 Nsp2基因扩增条带;B1.SD18 ORF3基因扩增条带;C1.SD18 ORF5基因扩增条带;A2,B2,C2.阴性对照
M.DL2000 DNA Marker;1.SD18 Nsp2基因扩增条带;B1.SD18 ORF3基因扩增条带;C1.SD18 ORF5基因扩增条带;A2,B2,C2.阴性对照
2.8 基因重组分析利用RDP4和SimPlot对SD18分离株和参考株的全基因组序列进行重组分析,结果显示SD18毒株在多个位置发生基因重组现象(表5,图9)。SD18毒株的重组亲本毒株为VR2332株,提供重组片段的病毒株为JXA1-R及NADC30-like毒株。

表5 以VR2332为亲本的SD18分离株的重组信息分析结果
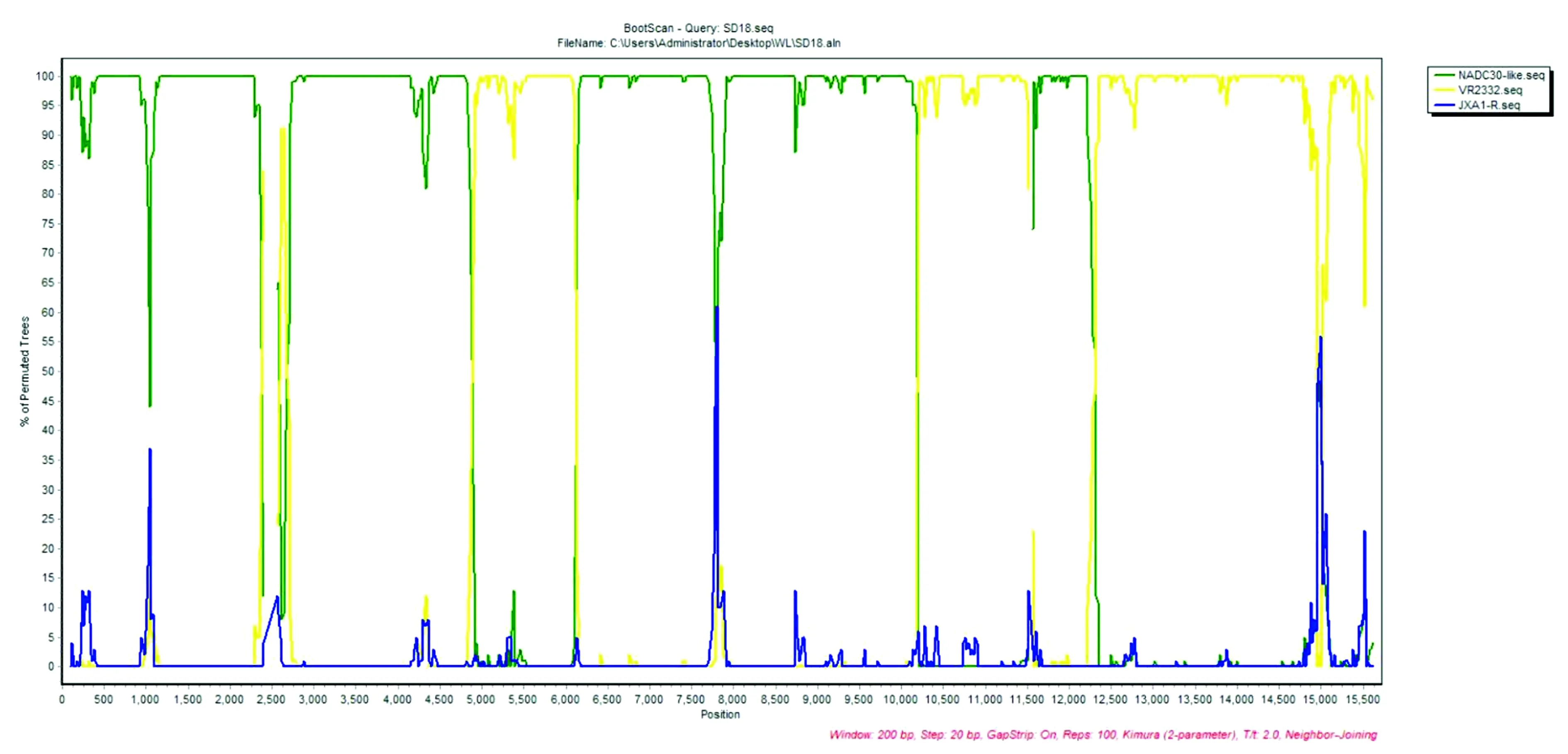
图9 PRRSV SD18毒株的基因重组分析
2.10 GP5蛋白信号肽预测利用软件SignalP4.1在线分析功能,采用神经网络法预测SD18毒株GP5蛋白的信号肽,该预测方法主要有3个参数,即S、C和Y值对应每个氨基酸。预测结果中一条起伏较大的曲线表示S值的变化趋势,在信号肽区域内S值较高。C值为剪切位点值,在剪切位点处该值最高。Y值为综合考虑S值和C值之后的参数,要比单独考虑C值更精确。SD18毒株GP5蛋白的信号肽预测结果如图10所示,1~15 aa区域的S值较高且在12 aa处达到最高0.971,在32 aa处C值和Y值分别达到最高0.571和0.643,由此可知,SD18毒株GP5蛋白存在信号肽,其信号肽区域为1~31 aa,其剪切位点为31~32 aa。
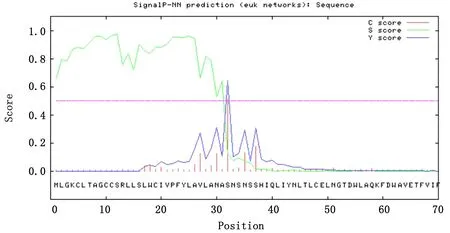
图10 SD18毒株 GP5蛋白信号肽预测结果
3 讨论
PRRSV自20世纪末传入我国以来,给我国养猪行业造成了巨大的经济损失,尤其是2006年出现的高致病性变异毒株更对该病的防控带来了严峻的挑战[5]。该病威胁巨大的原因之一是病毒基因变异迅速,可以通过不断的缺失、突变、重组从而极大地增加了病毒致病力的变异几率[6],导致原有疫苗对猪体产生的免疫保护力不足。研究调查发现,我国目前流行的PRRSV仍是以高致病性美洲毒株为主[7-8]。随着近几年生猪贸易进一步的扩大以及各种弱毒疫苗的不合理使用,使得PRRSV毒株的遗传变异更加趋于复杂化和广泛化,其中Nsp2、ORF3和ORF5基因在影响PRRSV毒株的变异规律、对宿主感染前后的致病力和病原本身的免疫原性方面有重要的作用,具有较大的研究价值。因此,加强对PRRSV基因遗传变化规律的监测及分析,特别是对动物机体和相关免疫程序的影响,对防控PRRSV具有非常重要的理论和现实意义。
Nsp2作为PRRSV整个基因组中变异性最大的基因之一,其碱基序列的缺失、突变导致的某些氨基酸位点的变异可以作为监视PRRSV基因组遗传变化的一个重要标志[9]。Nsp2基因的变化不仅表现在某些碱基位点的突变与缺失,更有不同长度的连续性缺失,如我国最早发现的Nsp2基因存在12个连续缺失的HB-2株[10]。另外由于2006年我国各地大规模的暴发PRRS,一种Nsp2基因出现了90个不连续碱基位点缺失的PRRSV被作为新型美洲高致病性PRRSV进入了人们的视线[11]。根据不同的报道发现,新型变异的PRRSV毒株Nsp2基因编码氨基酸缺失位点数量正在呈不断增加的趋势[12]。由此可见,PRRSV的Nsp2基因变异程度高、缺失数目大,以及不同类型毒株之间的遗传特性和抗原性的差异,使得新型突变病毒与原流行毒株之间的免疫交叉保护力较低,这或许是PRRSV难以有效防控的重要原因之一。相关研究显示[13],PRRSV Nsp2基因上存在与毒株致病力相关的氨基酸位点,即668位与952位氨基酸。本研究的SD18毒株Nsp2基因第668位氨基酸为R,与国内分离的早期高致病性毒株JXA1、HUN4相同。同时SD18毒株在628,703,725,752等多个位点的突变均与高致病性毒株JXA1、HUB1相同,表明SD18毒株同样为一株具有高致病性的强毒株。而在某些Nsp2基因氨基酸位点上,SD18毒株又与JXA1、HUB1表现出不同的变化,如在749(A→V)、780(M→V)、783(H→L)、844(V→A)等出现了突变,表明SD18毒株呈现向新型毒株变异的趋势。
ORF3同样为PRRSV变异性最大的基因之一,其遗传变异也在一定程度上反映了PRRSV整个毒株的变异程度和流行趋势[14]。有研究表明,由ORF3基因编码的GP3蛋白具有较好的免疫原性,能够刺激机体免疫系统产生一定水平的抗体,但与GP5的抗体水平相比较低[15],这或许是由于GP3蛋白的抗原性主要诱导机体的细胞免疫有关。相关研究证明,病毒蛋白中含有大量的糖基化蛋白,它们在识别宿主特定的靶细胞、诱导机体产生免疫应答反应与免疫逃避等方面具有重要作用[16-17]。GP3蛋白上存在许多与GP5蛋白具有相似功能的糖基化位点,而这些位点均在整个病毒的免疫逃避中发挥着重要的作用。GP3蛋白上的糖基化位点主要有N30、N43、N51、N132、N153、N161和N196,而SD18毒株GP3蛋白的这7个糖基化位点均未发生氨基酸的变异,但在别的位点却发生了多个氨基酸的变异。与国内的高致病性代表毒株JXA1和HUB1比较,在20(F→L)、69(P→S)、71(K→N)、96(P→S)、147(V→D)、153(A→T)等氨基酸位点出现了变异,而无论与美洲经典株或高致病性株相比,SD18毒株第17位氨基酸插入了1个氨基酸S。相关试验表明[18],GP3蛋白在PRRSV感染受体细胞与病毒粒子装配等方面发挥重要作用,而SD18毒株的ORF3基因这些氨基酸位点变异,是否会影响PRRSV的病原性与致病性还有待进一步研究。
据报道,PRRSV疫苗株的GP5蛋白第137位为丙氨酸,而野毒株在该位点则是丝氨酸[19]。另外,可以通过GP5的13和151位的氨基酸变异情况来判断毒株毒力的强弱[20]。通过对本试验SD18毒株ORF5所编码的氨基酸分析表明,其137位为丝氨酸,符合强毒株特性之一,排除SD18毒株为疫苗株。对比SD18毒株与JXA1株等强毒株发现,在第13位与第151位氨基酸均为精氨酸,表明该试验分离的SD18毒株为野毒株。各项研究说明,GP5蛋白在病毒入侵机体时表现出较强的抗原性[21],表明其在破坏机体细胞免疫的过程中发挥重要作用。此外,大部分PRRSV的中和位点均位于GP5蛋白上[22],包括27~30,37~45,180~197 aa,而37~45 aa被认为是发挥主要作用的位点。在上述位点中,起到识别中和抗体的位点分别为38,42,43,44 aa,而发挥结合中和抗体的位点则有39,40,41 aa[23],以及剩余的2个诱骗位点,即当机体感染PRRSV时,这2个诱骗位点会首先刺激机体的免疫系统产生大量针对其表位的非中和抗体,从而延缓了免疫系统针对主要抗原表位产生的中和抗体,使得有效的特异性抗体和病毒之间存在一定的时空差异。而与VR2332等经典美洲高致病性毒株相比,SD18毒株的许多结合位点均发生了突变,首先第39位结合位点的氨基酸(L→I)突变,其次第185位(V→A)也发生了突变,这2个中和位点的突变或许会影响PRRSV刺激机体生成中和抗体的敏感性,使得机体对病毒粒子的免疫清除能力受到影响,这也许是该分离株引起本次疾病暴发的原因之一。此外,GP5蛋白作为存在多个糖基化位点的囊膜蛋白, 分别包括第30,33,34,35,44,51位氨基酸位点,而糖基化则会阻碍附近的中和抗原表位和中和抗体进行结合。本试验分离的SD18分离株与经典高致病性毒株VR2332相比则分别在34(D→N)、35(S→N)2个氨基酸位点发生了突变,且突变的氨基酸种类一致,该糖基化位点的变化是否会影响病毒粒子中和表位的暴露以及其免疫逃避机制与致病力等都有待于进一步的调查研究。抗原性指数(antigenic index,AI)是将蛋白质片段的氨基酸序列、结构、构想与活动性等多方面因素综合考虑而建立的一种大致预测蛋白质不同区域抗原性的分析方法,AI值的高低在一定程度上反映了相关蛋白抗原性的高低,并与组成其抗原表位的可能性大小直接相关[24]。通过分析SD18毒株ORF5的抗原性表明,其部分抗原表位与HPBED具有较为相似的特征,而与VR2332有较大的差异,主要差异表现为SD18毒株在30~39 aa抗原区域变窄,而在50~60 aa及136~141 aa等区域则明显高于VR2332株,这些AI的改变能否干扰毒株的致病力或影响不同PRRSV疫苗的免疫保护能力等都有待进一步试验研究。基因重组一直是人们关注的焦点之一,揭示PRRSV的基因重组对了解PRRSV的流行性有较大的必要性。参照已有的方法,本研究利用RDP4和SimPlot对SD18毒株进行重组分析,发现该株病毒在多个基因位点存在基因重组现象。SD18毒株以VR2332为亲本,以疫苗株JXA1-R和国内最新出现的NADC30-like毒株提供重组片段进行基因重组,而重组的部位和数量不尽相同,重组大多发生在非结构蛋白和次要结构蛋白的区域。该PRRSV分离株由疫苗毒株提供的重组片段,提示弱毒疫苗株基因片段参与了病毒流行株的基因重组,这种现象应该进一步引起业界警惕而制定更合理的免疫计划,避免PRRSV的进一步变异。
综上所述,本研究分离的SD18毒株与经典毒株和高致病性毒株相比,其在Nsp2、ORF3和ORF5基因均出现了多处新的变异或缺失,包括Nsp2基因出现了不同于早期国内分离的高致病性毒株新的缺失部位、GP3蛋白的多处糖基化位点发生了改变,而GP5表面抗体中和位点同样也出现了多个突变,表明该病毒蛋白的免疫原性可能发生了改变,从而可能影响了病毒的致病性,引起疫苗免疫接种保护效果降低,这或许是该分离株引起本次PRRSV流行的最主要原因。本研究通过对SD18病毒的Nsp2、ORF3以及ORF5基因的遗传变异分析,丰富了PRRSV的基因信息数据库,同时也为PRRSV的突变趋势、致病机理及对研制新型疫苗提供了科学参考资料。

