大麻GRAS转录因子全基因组研究
张苗苗,于江珊,施 江,杨 雨,孟 雪,孙 伟,万会花*,薛建平*
1.淮北师范大学安徽省特色资源植物利用工程实验室,安徽 淮北 235000
2.中国中医科学院中药研究所,北京 100700
转录因子的研究是功能基因组学的重要组成部分。自从第一个转录因子在玉米中被发现[1],至今大量的转录因子已经被证明参与高等植物的各种生理过程和调控网络。GRAS 蛋白是一个重要的植物特异性蛋白家族,是以该家族中最早鉴定的3个成员GAI(gibberellic-acid insensitive)、RGA(repressor of GAI)和SCR(scarecrow)[2-3]命名。GRAS 转录因子作为植物所特有的因子,参与植物的信号转导、生长发育、逆境胁迫等过程。该家族蛋白具有保守的羧基末端,其中包括LRI、VHIID、LRII、PFYRE 和SAW 5 个基序,在N 端各不相同的序列是基因功能特异性的主要原因。到目前为止,GRAS基因家族已经在多种植物中进行了全基因组研究,包括杨树、拟南芥、水稻、大白菜、梅花、松树等[4-7]。在模式植物拟南芥和水稻基因组中分别有34、60 个GRAS基因家族成员。以拟南芥GRAS家族成员的分类为依据,将大麻GRAS基因家族分为10 类。GRAS基因家族的代表基因是DELLA基因,它是环境与激素,激素与激素信号途径之间的联系枢纽,如负调节赤霉素(gibberellin,GA)信号通路、脱落酸信号传导和光信号传导[8]等,同时还参与植物次生代谢产物的合成调控,例如在缺氮条件下,DELLA 蛋白促进花青素的合成和积累[9]。王立儒[10]研究发现丹参SmDELLA1 蛋白可能是丹参总酚酸和总黄酮生物合成途径中的正调控因子。
大麻Cannabis sativaL.又名火麻,是大麻科(Cannabinaceae)大麻属CannabisL.一年生草本植物[11],具有很高的药用价值和商业价值。我国是最早利用大麻的国家,也是大麻原产地之一[12]。大麻以合成大麻素而闻名,大麻素是具有多种药理活性的酚萜类物质,主要积累在雌花的腺毛中[13]。大麻二酚(cannabidiol,CBD)具有阻断某些多酚对人体神经系统的影响,并且有止痛抗炎、抗癫痫、抗焦虑、抗类风湿关节炎和抗失眠等一系列生理活性功能,对治疗多发性硬化症具有良好的效果[14]。随着高纯度大麻二酚液体制剂Epidiolex®药物在美国上市,并且成为治疗小儿癫痫的“孤儿药”,大麻素的市场需求量迅速增加。具有精神活性的四氢大麻酚(tetrahydrocannabinol,THC)还具有抗癌、抗菌、抗呕吐、利尿等作用,但四氢大麻酚也会导致焦虑、认知功能障碍和降低免疫力,是被严格管控的物质[15]。大麻环萜酚(cannabichromen,CBC)具有消炎等作用,是大麻幼苗期主要的大麻素成分,但随着植株的逐渐成熟大麻环萜酚的含量迅速降低,以至于可以忽略不计[16]。李秋实等[12]明确了大麻种质资源管理三级分类体系,根据THC 的含量将大麻分为工业大麻、药用大麻和医用大麻3 类。THC<0.3%时为药用大麻和工业大麻,其中CBD 含量高为药用大麻,CBD 含量低为工业大麻,用于获取纤维和种子;THC>0.3%时为医用大麻,严禁种植和使用。
大麻素的前体物质来源于甲基赤藓糖醇-4-磷酸(methylerythritol 4-phosphate,MEP)和脂肪酸2个代谢途径。首先,在MEP 途径,顺式异戊烯己基二磷酸(cis-isopentenyl dephosphate,IPP)在IPP异构酶以及GPP 合成酶作用下形成了香叶基二磷酸(geranyl diphosphate,GPP)。同时,脂肪酸类合成途径中丙二酰辅酶A(malonyl-CoA)在橄榄酸环化酶(olivetolic acid cyclase,OAC)和橄榄醇合成酶(olivetol synthase,OLS)的作用下形成2,4-二羟基-6-戊基苯甲酸(olivetolic acid,OA)。然后,底物OA 和GPP 在大麻醇酸合成酶(cannabigerolic acid synthase,CBGAS)的作用下合成了大麻素生物合成的主要前体物质大麻醇酸(cannabigerolic acid,CBGA),CBGA 在四氢大麻酸合成酶(tetrahydrocannabinolic acid synthase,THCAS)、大麻二酸合成酶(cannabidiolic acid synthase,CBDAS)和大麻黄酸合成酶(cannabichromenic acid synthase,CBCAS)的催化作用下分别形成了四氢大麻酚酸(tetrahydrocannabinolic acid,THCA)、大麻二酚酸(cannabidiolic acid,CBDA)和大麻色烯酸(cannabichromenic acid,CBCA)。最后在光和热的物理作用下发生脱羧基反应分别生成了四氢大麻酚、大麻二酚和大麻环萜酚。
随着大麻全基因组序列的公布,对整个基因家族的综合分析变得便捷。由于GRAS 蛋白在调控植物次生代谢和逆境胁迫方面起重要作用,但是该基因家族在大麻中的信息缺乏,因此,本研究首次对大麻中GRAS基因家族全序列进行了研究。本研究鉴定了44 个CsGRAS基因,并对它们的基因分类、染色体定位、系统发育和外显子-内含子分布进行了分析。此外,采用数据库及本课题组完成的转录组数据,分析大麻GRAS基因家族的表达模式,推测参与大麻素生物合成的相关基因。本研究提供了GRAS基因家族的详细信息,为进一步研究GRAS基因在大麻中的功能奠定了基础。
1 材料与方法
1.1 大麻GRAS 家族成员的获取和染色体定位
从NCBI(https://www.ncbi.nlm.nih.gov) 网站上获取大麻(GCA_900626175.1)、拟南芥(GCA_000005425.2)和水稻(GCA_000005425.2)的全基因组及注释文件。采用的大麻品种为CRBRx,性别为雌株。应用TBtools 软件[17]提取的大麻蛋白序列,放入PlantTFDB 网站进行转录因子预测,预测得到的所有GRAS家族成员去除重复转录本数据,得到大麻GRAS家族的gene ID,利用TBtools 软件获得大麻GRAS家族的gene ID 所对应的蛋白序列。使用在线生物信息学工具ExPASy-ProSite(http://web.expasy.org/protparam/)对大麻GRAS 蛋白的分子量和等电点进行预测。利用 Cell-PLoc (http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)网站进行亚细胞定位预测。使用TBtools 软件解析大麻的注释文件,确定每个GRAS基因的位置信息并绘制其所对应的染色体物理位置图。
1.2 大麻、水稻和拟南芥GRAS 家族共线性分析
使用从NCBI 网站上下载的大麻、拟南芥及水稻全基因组及注释文件,利用MEGA-X 软件,采取邻接法(neighbor-joining,NJ)法,对拟南芥和大麻GRAS 蛋白构建无根系统发育树。使用TBtools软件对其3 者共线性关系进行可视化,并找到3 者GRAS基因家族的同源基因对。
1.3 大麻GRAS 蛋白系统发育树的构建及motif发掘与分析
使用MEGA-X 通过NJ 方法构建了基于比对的系统发育树,并进行了重复1000 次的Bootstrap 检验,所有的设定均采用默认值。大麻GRAS 蛋白质motif 的发掘与位置分布均采用 MEME(http://meme-suite.org/tools/meme) 蛋白质序列分析在线程序,并设定目标motif 数量为10。通过使用TBtools 软件对大麻的GRAS 注释文件的解析,对大麻GRAS基因家族进行了外显子-内含子、保守结构域和motif 3 者可视化处理,并使用DNAMAN 软件进行保守结构域蛋白序列的比对分析。
1.4 大麻GRAS 基因家族顺式作用元件分析
使用PlantCare(http://www.plantcare.co.uk/)网站对从TBtools 软件提取的大麻GRAS基因上游2000 bp 序列进行顺式作用元件预测,并使用TBtools 软件对其可视化构图。
1.5 大麻GRAS 基因的表达模式分析
利用本课题组ZYS 品种(由Purple Kush 和Dinamed Autoflowering CBD 杂交获得的高CBD、低THC 品种)大麻花、茎和叶的转录组数据,使用TBtools 软件绘制大麻GRAS家族基因表达模式热图,GRAS成员基因表达量(FPKM)经row scale 均一化处理,并进行聚类和差异表达分析。
2 结果与分析
2.1 大麻GRAS 基因的鉴定与染色体分布
从NCBI(https://www.ncbi.nlm.nih.gov) 网站上获取CRBRx 品种的大麻全基因组及注释文件,将 TBtools 软件提取的大麻蛋白序列,放入PlantTFDB 网站进行转录因子的预测,预测得到54个GRAS,去除10 个重复转录本数据后,最终得到44 个GRAS家族成员,命名为CsGRAS1~CsGRAS44。利用大麻GRAS家族的gene ID,使用TBtools 软件获得大麻GRAS家族的gene ID 所对应的蛋白序列。大麻 GRAS 蛋白长度最小的是CsGRAS8,由436 个氨基酸残基组成,长度最大的蛋白质为CsGRAS21,包含757 个氨基酸残基。大麻GRAS 蛋白质等电点介于4.77~7.24,相对分子质量介于49 164.54~85 748.52(表1)。
GRAS基因在染色体上的位置分布如图1所示,该家族44 个基因不均匀地分布在大麻的10 条染色体上( NC_044370.1、NC_044371.1、NC_044372.1、NC_044373.1、NC_044374.1、NC_044375.1、NC_044376.1、NC_044377.1、NC_044378.1、NC_044379.1 分别分布了3、6、3、6、5、7、5、4、1、4 个GRAS基因),其中NC_044375.1 染色体上GRAS基因家族数目最多,含有 7 个基因,其次是NC_044371.1 和NC_044373.1 都含有6 个基因,最少的是NC_044378.1 只含有1 个基因。最长的CsGRAS33基因在NC_044374.1 染色体下臂,最短的CsGRAS7在NC_044376.1 染色体上臂。NC_044371.1 染色体上的CsGRAS34和CsGRAS35,CsGRAS43和CsGRAS44;NC_044372.1 染色体上的CsGRAS2和CsGRAS3;NC_044373.1 染色体上的CsGRAS38和CsGRAS40;NC_044374.1 染色体上的CsGRAS12 和CsGRAS13;NC_044375.1 染色体上的CsGRAS20、CsGRAS21、CsGRAS22、CsGRAS23、CsGRAS24、CsGRAS25和CsGRAS26;NC_044376.1 染色体上的CsGRAS6、CsGRAS7、CsGRAS8和CsGRAS9成簇排列,序列比对分析发现这些成簇的基因之间存在串联重复现象。
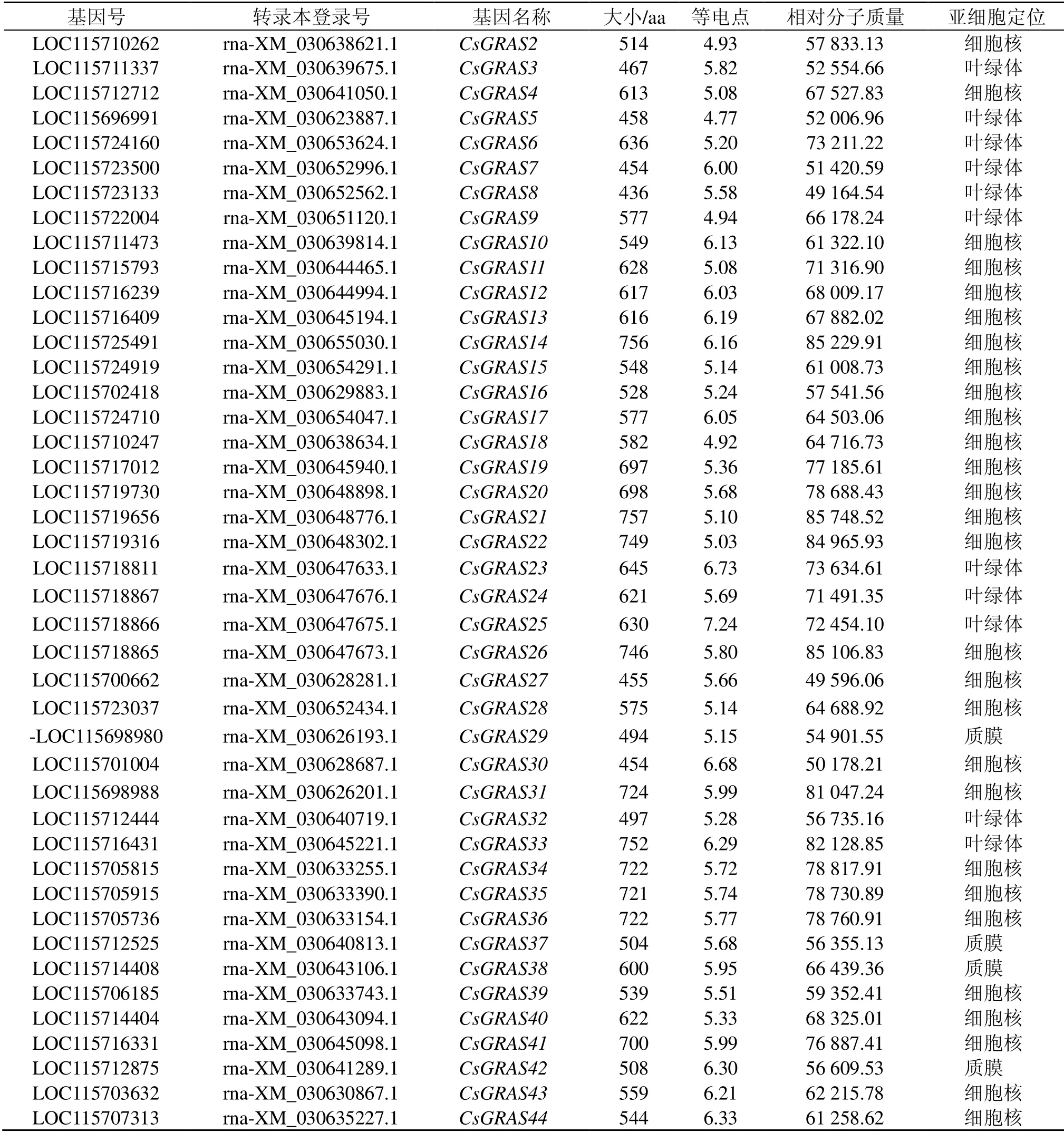
表1 大麻GRAS 基因家族基本信息及特征Table 1 Information and characteristics of CsGRAS genes
2.2 大麻GRAS 家族进化的的特性
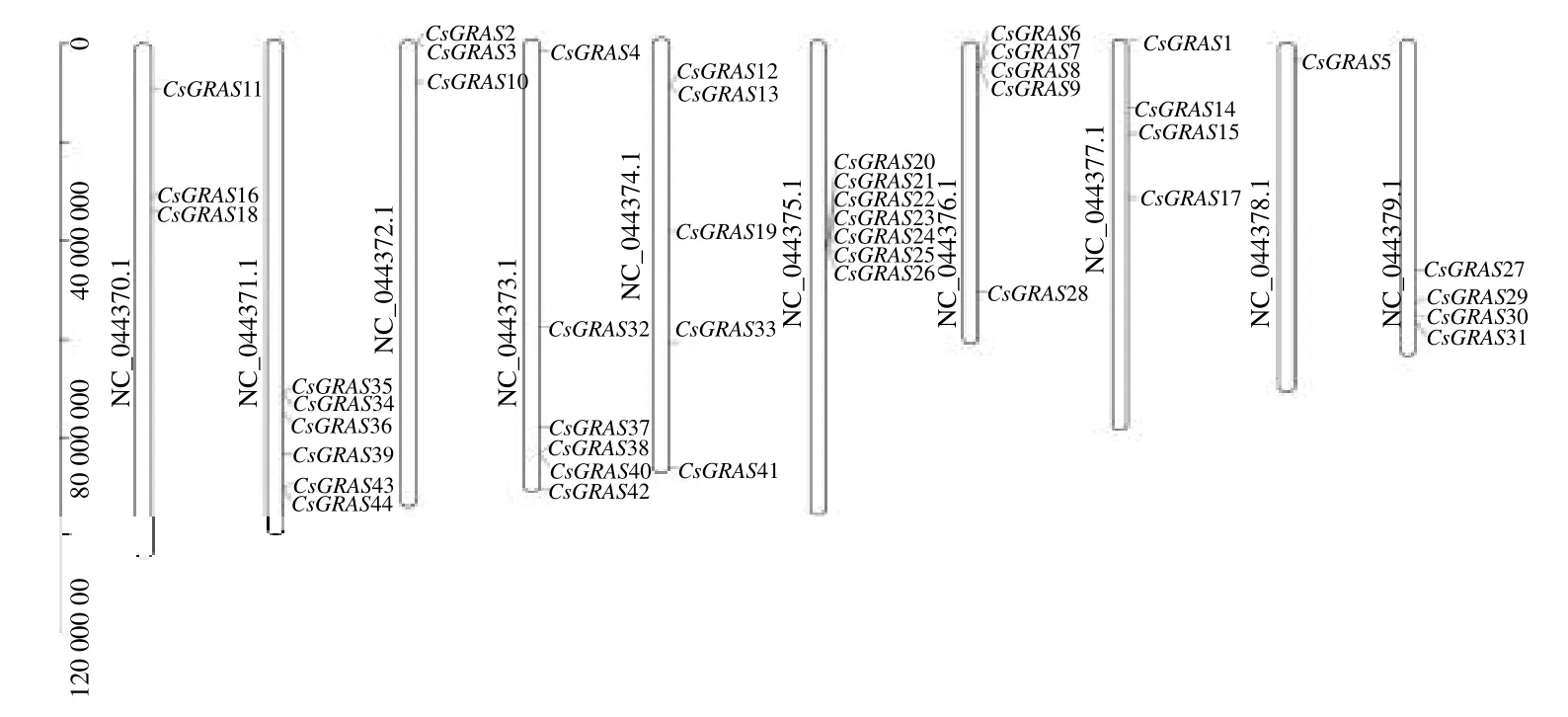
图1 大麻44 个GRAS 基因在10 条染色体的分布Fig.1 Distribution of 44 GRAS genes on 10 chromosomes in C.sativa
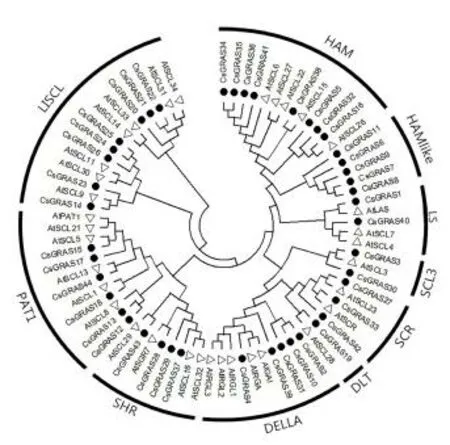
图2 大麻和拟南芥 GRAS 转录因子系统发育树Fig.2 Phylogenetic trees of GRAS transcription factors in C.sativa and Arabidopsis thaliana
利用MEGA-X(图2)软件,采取NJ 法,对拟南芥34 个和大麻44 个GRAS 蛋白进行分析,构建无根系统发育树。根据系统发育树,GRAS 蛋白可分为10 个亚家族:HAM、LS、DLT、SCR、DELLA、SCL3、LISCL、SHR、PAT1 和HAMlike。HAMlike亚家族包含5 个CsGRAS 蛋白(CsGRAS6、CsGRAS7、CsGRAS8、CsGRAS9、CsGRAS11),与HAM属于同一分支却不包含拟南芥GRAS蛋白。HAM 亚家族有8 个CsGRAS 蛋白(CsGRAS5、CsGRAS16、CsGRAS32、CsGRAS34、CsGRAS35、CsGRAS36、CsGRAS38、CsGRAS41)。有 5个CsGRAS 蛋白(CsGRAS2、CsGRAS4、CsGRAS10、CsGRAS31、CsGRAS39)属于DELLA 亚家族。PAT1 亚家族包括来自工业大麻的 6 个成员(CsGRAS12、CsGRAS13、CsGRAS15、CsGRAS17、CsGRAS18、CsGRAS44)。SHR 和SCR 亚家族分别拥有4 个蛋白(CsGRAS28、CsGRAS29、CsGRAS37、CsGRAS43)和3 个蛋白(CsGRAS27、CsGRAS33、CsGRAS42),推测这些蛋白可能与植物的发育有关[18]。SCL3 亚家族包含2 个蛋白(CsGRAS3、CsGRAS30),在拟南芥中同家族AtSCL3 蛋白通过整合多种信号来调节根细胞的伸长[19]。CsGRAS19 是DLT 亚家族中唯一的成员,它通过调节油菜素甾体信号参与控制株高[20]。LS 有2 个成员CsGRAS1 和CsGRAS40。其余8 个蛋白(CsGRAS14、CsGRAS20、CsGRAS21、CsGRAS22、CsGRAS23、CsGRAS24、CsGRAS25、CsGRAS26)属于LISCL 家族。
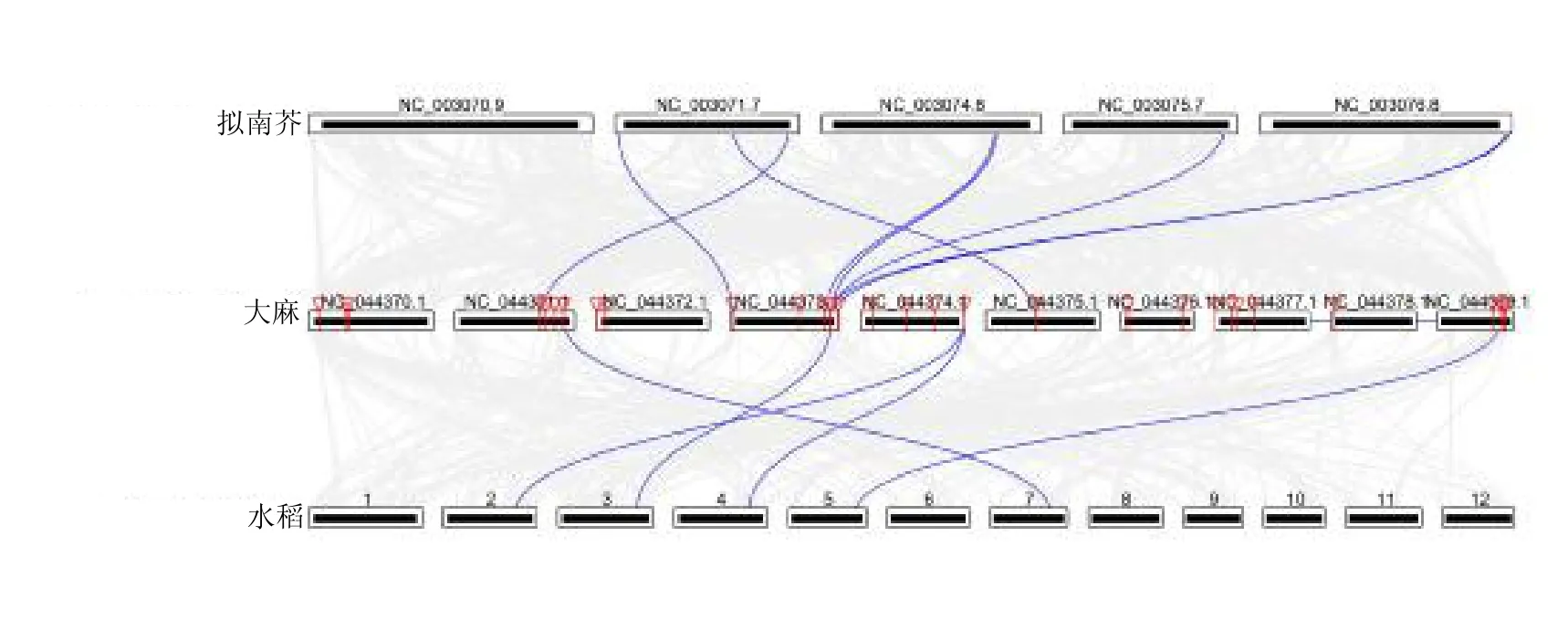
图3 拟南芥、大麻和水稻共线性关系Fig.3 Collinear relationship between A.thaliana,C.sativa,and Oryza sativa
利用水稻、拟南芥和大麻的同源关系,运用比较基因组学的方法找到大麻基因组、拟南芥基因组和水稻基因组之间的共线性关系,图3中一共有12 条亮线,代表有12 个GRAS同源基因对,大麻和拟南芥中,一共发现7 对同源基因,分别为CsGRAS34/AtSCL23、CsGRAS4/AtRGA、CsGRAS37/AtSCL22、CsGRAS37/AtSCL27、CsGRAS38/AtSCL16、CsGRAS40/AtSCL2和CsGRAS40/AtSCL3,与大麻和拟南芥构建的系统发育树结果一致;在大麻和水稻中,一共发现了5 对同源基因,分别为CsGRAS44/Os07t0583600-01、CsGRAS40/Os03t0723000-02、CsGRAS41/Os02t0662700-01、CsGRAS41/Os04t 0555000-01 和CsGRAS29/Os05t0500600-01,根据大麻与拟南芥和水稻的同源基因可以推测其可能具有相似的生物学功能。
2.3 大麻GRAS 家族基因结构和蛋白保守基序分析
为了了解大麻GRAS 转录因子的保守性及相关性,对大麻GRAS 蛋白构建进化树,并整合了domain 和motif 信息,如图4所示。基因家族成员是由同一个基因通过复制重组不断演化而来。因此,基因家族成员在序列上具有高度的保守性,包含了一些高度保守性的motif。大麻GRAS家族中共鉴定出10 个保守基序,CsGRAS8 包含的保守基序种类最少,为8 个。结合图4分析发现大麻GRAS家族蛋白的domain 和motif 的分布相对保守。大麻GRAS每个亚家族所包含的基序数量也有所差别,保守基序在一定程度上反映了大麻GRAS基因家族在进化过程中的结构特点,为研究基因家族的功能提供了理论基础和研究依据。大麻GRAS家族基因结构相对比较简单,内含子数量在0~2,其中 81.8% 基因不含有内含子,15.9% 基因(CsGRAS5、CsGRAS6、CsGRAS8、CsGRAS19、CsGRAS26、CsGRAS37、CsGRAS39)含有1 个内含子,只有2.3%基因(CsGRAS2)含有2 个内含子。
通过MEME 网站的分析,大麻GRAS家族一共发现了10 个motif(图5),其中有5 个最保守的motif 分别为VHIID、PFYRE、LRI、SAW、LRII。经过分析发现motif 1包含了GRAS家族中最保守的VHIID 结构域,其在靠近N 端的部位出现5 个保守的位点。motif 3 为PFYRE 区;motif 4 为LRI 区;motif 5 为SAW 区,在C 端末尾处存在保守的3 个氨基酸残基;motif 10 为LRII 区。
使用Jalview 软件对44 个大麻GRAS 蛋白保守结构域进行多重序列比对,在图6中,比对结果显示,所有蛋白序列中VHIID 基序中的H 位点绝对保守;不同序列中的LRI 和PFYRE 基序存在较大的差异,但所有的LRI 基序均呈现亮氨酸富集状态,PFYRE 基序保守性不如VHIID 基序,但也表现出高度的相似性;LRII 基序的保守性比LRI 基序高;在SAW 基序内检测出了S-A-W 3 个保守氨基酸残基。以上结果表明,大麻GRAS 蛋白在C 端存在高度的保守性。
2.4 大麻GRAS 基因家族顺式元件分析
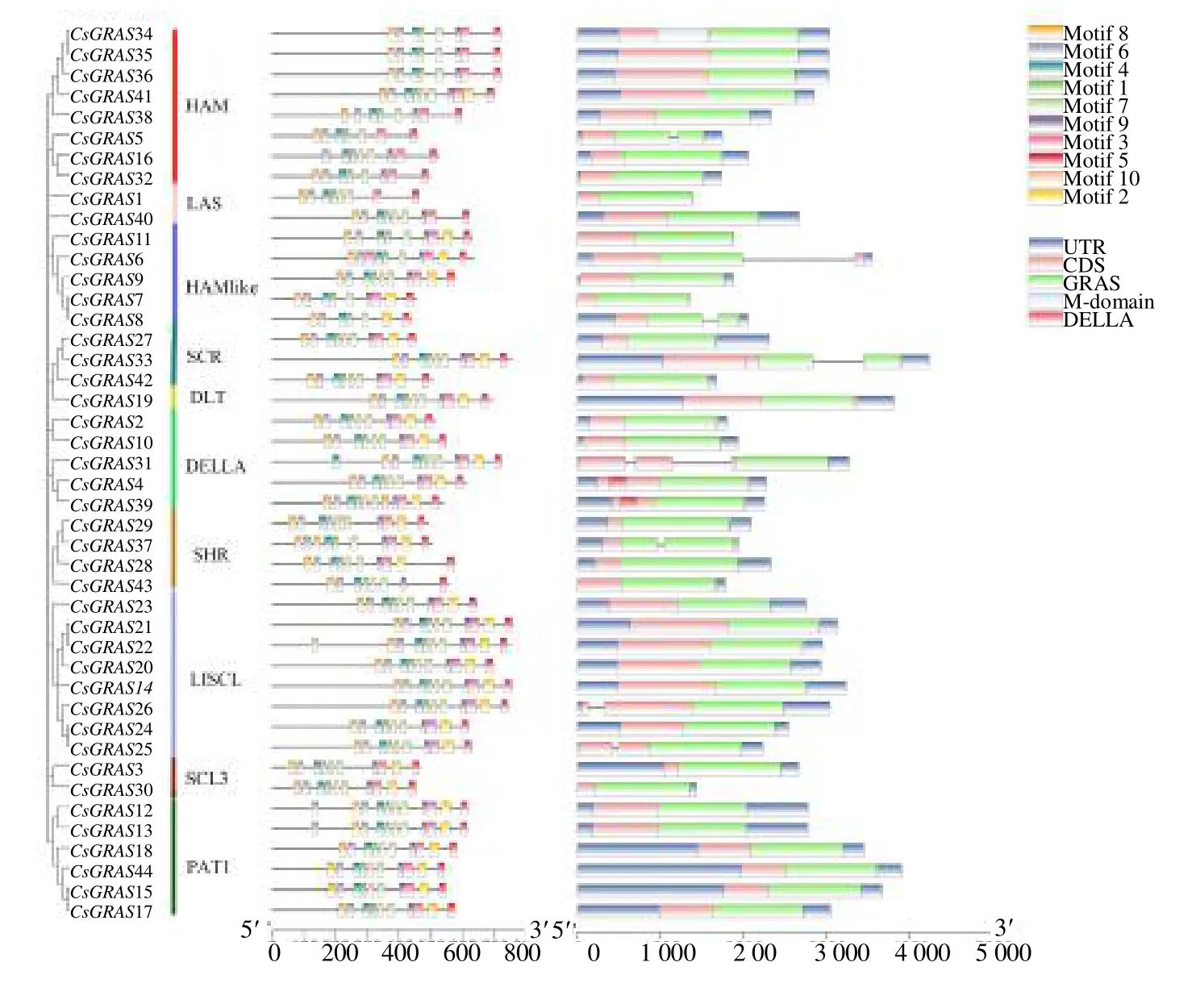
图4 大麻GRAS 家族进化树、保守基序及基因结构Fig.4 Phylogenetic,conserved motifs,and gene structure analyses of GRAS transcription factor family of C.sativa
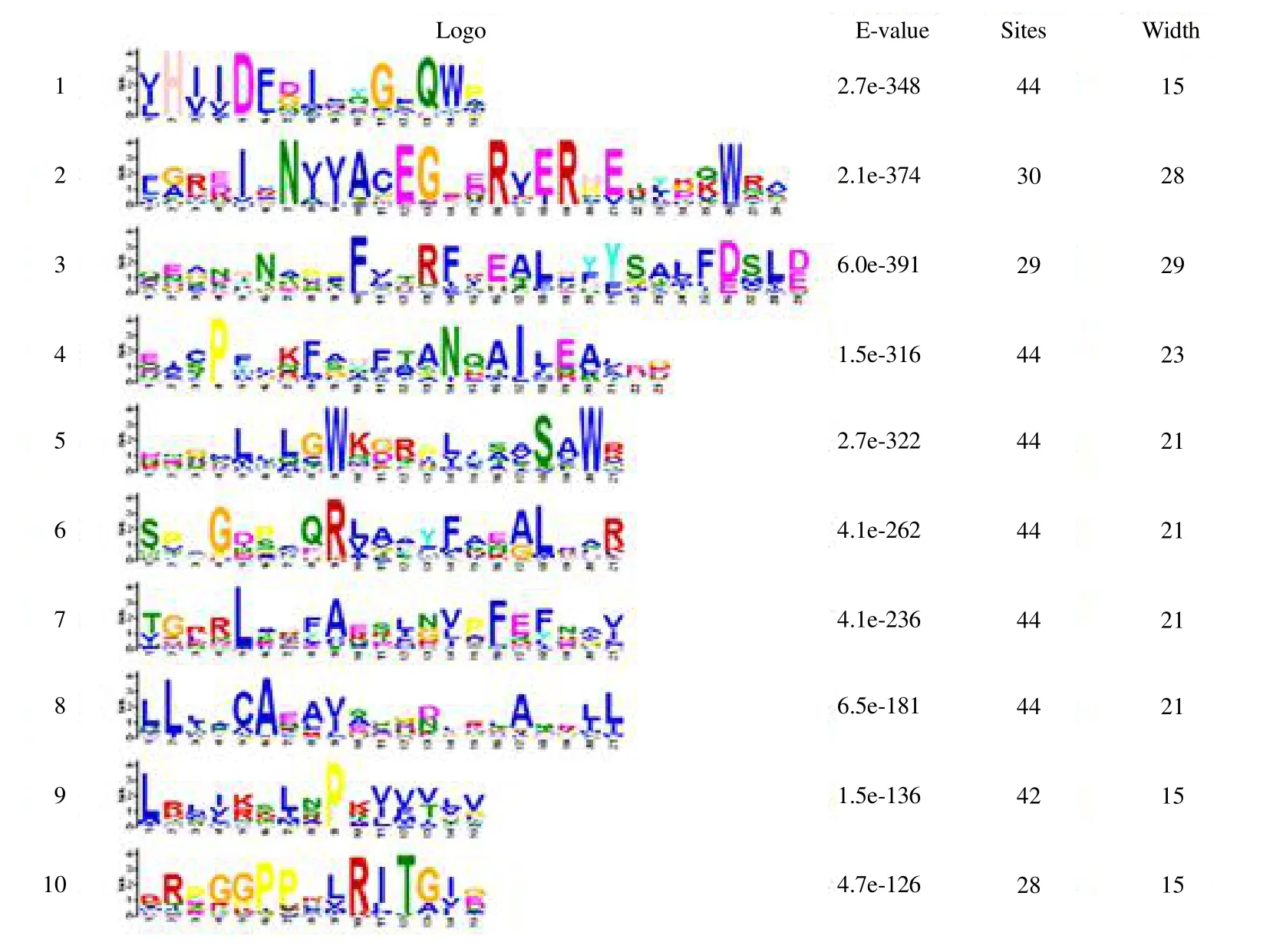
图5 MEME 预测的10 个motifs LOGOFig.5 Logos of ten motifs predicted by MEME online software

图6 大麻GRAS 家族保守结构域序列比对Fig.6 Sequence alignment of conserved domains of GRAS family proteins in C.sativa
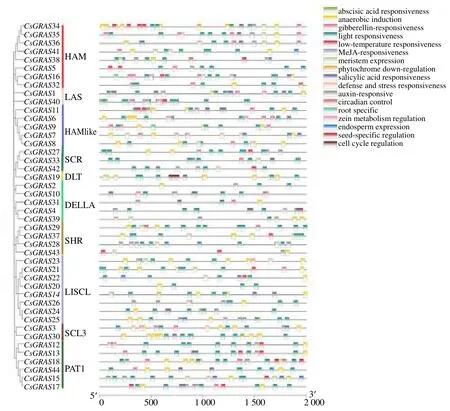
图7 大麻GRAS 基因家族顺式作用元件预测图Fig.7 Prediction of cis-elements of hemp GRAS genes
对大麻GRAS 基因家族成员顺式作用元件进行预测。从图7可以看出,大麻GRAS 家族主要包含光响应元件、厌氧诱导元件、脱落酸响应元件、赤霉素响应元件和茉莉酸甲酯响应元件等。光响应元件涵盖了所有CsGRAS 家族成员,元件数量也是最多,共有284 个。其次,预测到厌氧诱导的顺式作用元件为104 个,响应脱落酸的顺式作用元件为88 个,响应茉莉酸甲酯的顺式作用元件为72 个,含量最少的是光敏色素下调表达的顺式作用元件和根特异性顺式作用调控元件,都只含有2 个,分别存在于CsGRAS14、CsGRAS34和CsGRAS9、CsGRAS16启动子区。
2.5 大麻GRAS 家族基因表达模式分析
为探究大麻GRAS基因的表达情况,利用课题组的1 个大麻品种花、茎和叶的转录组数据,绘制大麻GRAS基因表达热图(图8),并将表达模式进行聚类。GRAS基因在大麻ZYS 品种花、茎和叶中的表达谱分析表明,在花中,除CsGRAS42不表达外,其他基因均有不同程度表达,其中CsGRAS4、CsGRAS13、CsGRAS15、CsGRAS17和CsGRAS44相对其他38个基因的表达量更高,其中最高的基因是CsGRAS17,CsGRAS2、CsGRAS9、CsGRAS11、CsGRAS23和CsGRAS31基因表达量极低;在叶中,除CsGRAS8、CsGRAS9、CsGRAS18、CsGRAS23和CsGRAS24不表达,其他基因均有不同程度表达,其中CsGRAS4和CsGRAS39相对其他37 个基因的表达量更高,其中最高的基因是CsGRAS4,CsGRAS1、CsGRAS5、CsGRAS11、CsGRAS31、CsGRAS32和CsGRAS42基因表达量极低;在茎中,除CsGRAS9、CsGRAS18和CsGRAS24不表达,其他基因均有不同程度表达,其中CsGRAS4、CsGRAS39、CsGRAS40和CsGRAS44相对其他37 个基因的表达量更高,其中最高的基因是CsGRAS40,CsGRAS8、CsGRAS23、CsGRAS29、CsGRAS31和CsGRAS37基因表达量极低。GRAS基因在ZYS 品种不同器官中的表达量存在显著差异,其中CsGRAS17在花中显著高表达。
3 讨论
本研究从大麻基因组中筛选到44 个GRAS基因家族成员,GRAS 转录因子在杨树、拟南芥、水稻、番茄、蓖麻、百脉根、大麻黄等作物中分别有106、34、60、53、48、18、11 个家族成员[4,21-24],GRAS基因家族在不同物种中数量差异较大。
大麻与拟南芥GRAS家族成员进行系统进化分析,根据结果可分成10 个亚族,与Tian 等[25-27]的系统发育树一致,基因结构域分析发现大麻GRAS基因家族在C 端出现多个保守结构域。大麻GRAS基因家族成员81.8%都不含有内含子,在梅花、番茄、拟南芥、水稻和杨树中无内含子GRAS 蛋白的比例分别占82.2%、77.4%、67.6%、55.0%、54.7%[4-5,28-30]。说明了大麻GRAS家族基因结构相对简单,植物中无内含子的GRAS基因所占比例较高。
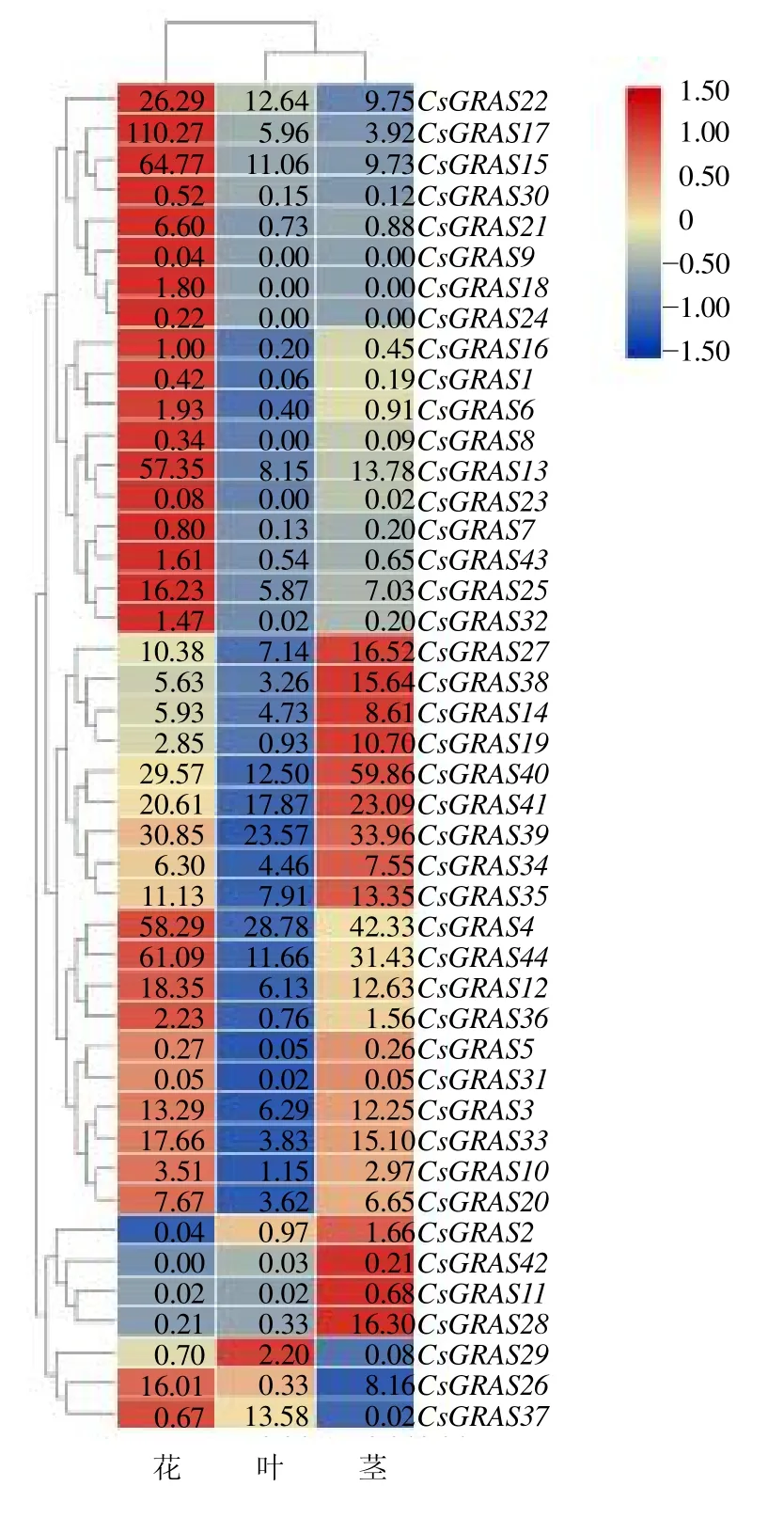
图8 ZYS 品种不同部位的CsGRAS 基因表达模式Fig.8 CsGRAS gene expression pattern of different organs of ZYS
很多GRAS 转录因子的功能已在模式植物中得到鉴定,但该家族在药用植物中的功能仍有待进一步研究。在本研究中,通过系统进化树的划分,根据己报道基因的功能[31],能帮助了解和预测分在同一支中大麻GRAS基因的功能。如CsGRAS15、CsGRAS17与AtPAT1基因分在一支,通过查询AtPAT1的功能,推测CsGRAS15和CsGRAS17可能参与了光敏色素phyA 信号转导[31]。大麻素是一种具有精神活性的酚萜类物质。除了种子,大麻植物其他部位都含有大麻素,它们主要在花中合成和积累[13],其生物合成涉及酚类和萜类化合物。花青素属于酚类化合物,与大麻素具有相同的上游代谢途径。在拟南芥中RGA、RGL1 和RGL2 这3 种DELLA蛋白对拟南芥的花瓣、花蕊及花药的发育具有重要的调控作用[32],GA 调控成花的作用部位为叶片和茎尖,而DELLA 蛋白在长日照下可沉默叶片中的miR172基因和茎尖中的MADS基因,参与GA 信号转导途径抑制开花[33],PAP1 蛋白是参与花青素合成的转录因子,GAI 和RGA 蛋白能与PAP1 蛋白相互作用,从而促进花青素合成[34-35]。而在缺氮条件下,DELLA 蛋白又能通过与PAP1 蛋白相互作用,实现花青素的积累[36],而DELLA 蛋白又可能是酚酸和黄酮生物合成途径中的正调控因子[10]。根据以上信息,推测大麻中的5 个DELLA 蛋白CsGRAS2、CsGRAS4、CsGRAS10、CsGRAS32和CsGRAS40 可能调控大麻的花期,参与大麻素的合成。在系统发育树中,CsGRAS4 蛋白与AtGAI和AtRGA 蛋白聚为一支,在共线性关系分析中也显示为同源基因对,CsGRAS15 和CsGRAS17 与AtPAT1 聚为一支,其中CsGRAS17 在花中显著高表达,推测CsGRAS4 蛋白可能降低大麻素生物合成相关的蛋白表达水平,抑制大麻素合成,而CsGRAS15 和CsGRAS17 蛋白可能与CsGRAS4 蛋白相互作用,从而促进大麻素合成[34]。CsGRAS3与AtSCL3 蛋白是同源基因,推测CsGRAS3 蛋白可能正向调控GA 的信号通路[37],促进大麻素合成。
在大麻GRAS基因家族中发现很多参与光响应的顺式作用元件、根特异性顺式作用调控元件和光敏色素下调表达的顺式作用元件,表明该基因家族广泛地参与了大麻的生长发育过程,在种子萌发和腋芽及根的发育[38]等方面发挥重要的调控作用;而赤霉素、脱落酸、茉莉酸甲酯等激素诱导类顺式作用元件的发现,可以推测出大麻GRAS基因受到多种激素信号分子的诱导[39]。对大麻GRAS成员的鉴定、系统发育和表达分析将有助于全面研究GRAS基因家族的功能,更好地理解它们在大麻的生长发育、大麻素的生物合成、生物和非生物胁迫中的作用。
利益冲突所有作者均声明不存在利益冲突

