基于线粒体DNA控制区序列的珠江和长江水系光倒刺鲃群体遗传变异分析
李文俊 李强 钟良明 桂林

基于線粒体DNA控制区序列的珠江和长江
水系光倒刺鲃群体遗传变异分析
李文俊1,李 强1*,钟良明2,桂 林1
(1广州大学生命科学学院,广州 510006;2韶关市金粤水产科技有限公司,广东韶关 512335)
摘要:【目的】明确珠江和长江水系不同光倒刺鲃地理群体的遗传变异及进化关系,为其种质资源保护和可持续开发利用提供科学依据。【方法】从珠江水系(增江、流溪河、北江、连江、漓江、柳江和郁江)和长江水系(阊江、赣江和湘江)的10个支流(群体)采集347尾光倒刺鲃样本,扩增并测定其线粒体DNA(mtDNA)控制区序列;通过MEGA 6.0、DnaSP 6.10和Arlequin 3.5等在线软件统计序列碱基组成、变异位点、遗传距离、核苷酸多样性(π)、单倍型多样性(Hd)及遗传分化系数(Fst),并进行分子变异分析(AMOVA)、核苷酸错配分布分析、中性检验,以及构建单倍型的系统发育进化树和网络结构图。【结果】光倒刺鲃mtDNA控制区序列长度为519 bp,其中T、C、A、G占比平均值分别为32.57%、19.16%、32.16%和16.11%。在所有mtDNA控制区序列中共检测到62个变异位点,34个单倍型;10个光倒刺鲃群体的Hd平均为0.917,π平均为0.0359,遗传距离为0.0018~0.0790,Fst为-0.0007~0.9978。光倒刺鲃mtDNA控制区序列63.46%的分子遗传变异来自各地理分组间;单倍型网络结构图和系统发育进化树均显示,10个光倒刺鲃群体聚类为三大支系,除了有个别交集外,东江水系、西江水系、赣江水系+湘江水系的光倒刺鲃群体分布在3个不同的独立分支上,而北江+流溪河水系群体分别与东江水系群体和西江水系群体有较多交集。10个光倒刺鲃群体的Fu’s Fs为-1.339~23.759,平均为9.857,但P均大于0.05;Tajima’s D为-2.613~2.824,仅有3个群体的Tajima’s D为显著性负值(P<0.05);其核苷酸错配分布图谱呈多峰形式,即珠江和长江水系光倒刺鲃群体尚未发生过种群扩张。【结论】珠江和长江水系光倒刺鲃群体遗传多样性总体上偏低,应加强其种质资源保护,尤其是北江水系群体野生资源相对较丰富宜作为重点保护单元。复杂的地形地貌导致珠江和长江水系光倒刺鲃群体形成明显的地理隔离,群体间已发生明显分化,且受各种人为活动干扰导致其种群收缩,因此亟待采取有效防护措施以保证光倒刺鲃种质资源的可持续利用。
关键词: 光倒刺鲃;mtDNA控制区;遗传多样性;遗传变异;地理隔离
中图分类号: S965.199 文献标志码: A 文章编号:2095-1191(2021)11-3121-09
Analysis of genetic variation among Spinibarbus hollandi in the Pearl River and the Yangtze River based on mitochondrial DNA control region sequences
LI Wen-jun1, LI Qiang1*, ZHONG Liang-ming2, GUI Lin1
(1College of Life Science,Guangzhou University, Guangzhou 510006, China; 2Jinyue Aquatic Products
Technology Co., Ltd., Shaoguan, Guangdong 512335, China)
Abstract:【Objective】 To characterize the genetic structure and phylogeographic pattern of different populations of Spinibarbus hollandi in the Pearl River and the Yangtze River, in order to provide the scientific basis for the conservation and sustainable utilization of S. hollandi populations resources. 【Method】 In this study,347 S. hollandi samples were collected from 10 populations in the Pearl River (the Zengjiang, Liuxi, Beijiang, Lianjiang, Lijiang, Liujiang and Yujiang rivers) and the Yangtze River (the Changjiang, Ganjiang and Xiangjiang rivers). The mitochondrial DNA (mtDNA) control region sequences from each individual were identified and analyzed. According to the MEGA 6.0, DnaSP 6.10 and Arlequin 3.5 software, the base composition, the locus of variation, genetic distance, nucleotide diversity(π), haplotype diversity (Hd) and genetic variation value (Fst) were calculated. In addition, molecular variation analysis(AMOVA), nucleotide mismatch distribution analysis, neutral test, the construction of a haplotype phylogenetic evolutionary tree and network diagram were also carried out. 【Result】 The sequence length of the mtDNA control region was 519 bp, in which T, C, A and G accounted for 32.57%, 19.16%, 32.16% and 16.11%, respectively. A total of 62 mutated loci and 34 haplotypes were detected in the sequences. The average haplotype diversity, nucleotide diversity, genetic distance and Fst were 0.917, 0.0359, 0.0018-0.0790 and -0.0007-0.9978, respectively. 63.46% of the molecular genetic variation of the mtDNA CR was observed in the distinct geographical groups. According to the haplotype phylogenetic evolutionary tree and network diagram, ten S. hollandi populations were divided into three groups, with the populations in the Dongjiang River, Xijiang River and Ganjiang River plus Xiangjiang River distributed in three independent branches. However, the Beijiang River and Liuxi River populations had more overlaps with the Dongjiang River and Xijiang River populations. Among the ten populations, the Fuʼ S Fs ranged from -1.339 to 23.759, with an average of 9.857, but all P values were greater than 0.05. Tajimaʼs D ranged from -2.613 to 2.824 and was significantly negative in only three populations(P<0.05). The map of nucleotide mismatch distribution showed a multi-peak pattern, indicating that population expansions did not occur in the Pearl River and Yangtze River. 【Conclusion】The genetic diversity of S. hollandi population is generally low. Therefore, the protection of germplasm resources needs improvement, and populations from the Beijiang River should be taken as a key protection unit. The complex landform results in the obvious geographical isolation of S. hollandi populations in the Pearl River and the Yangtze River, and the obvious differentiation among the populations, and the population shrinkage caused by anthropogenic activities had being formatted. Therefore, it is urgent to take effective protective measures to ensure the S. hollandi resources.
Key words: Spinibarbus hollandi; mitochondrial DNA control region; genetic diversity; genetic differentiation;geographical isolation
Foundation item: National Natural Science Foundation of China(41673110); Science and Technology Planning Project of Guangzhou(201804010486)
0 引言
【研究意义】光倒刺鲃(Spinibarbus hollandi)隶属于鲤形目(Cypriniformes)鲤科(Cyprinidae)鲃亚科(Barbinae)倒刺鲃属(Spinibarbus),主要分布在我国长江以南各大水系(潘烔华等,1991;乐佩琦,2000)。光倒刺鲃具有较高的营养和药用价值,是一种重要的名优经济鱼类(蔡子德等,2007)。随着光倒刺鲃人工驯养和繁育技术的推广,现已发展成为华东及华南地区重要的水产养殖品种之一。自20世纪80年代以来,由于过度捕捞、环境污染及水利工程建设等因素影响,光倒刺鲃自然种群数量急剧下降,为此农业农村部先后在广东、广西、江西、福建、湖南及安徽等地建立了10个光倒刺鲃国家级水产种质资源保护区(罗刚等,2017)。线粒体DNA(mitochondrial DNA,mtDNA)是一种细胞质DNA,结构简单,遵循母系遗传,进化速度较快,是研究动物群体遗传学和分子系统学的理想分子标记(董志国等,2013)。mtDNA控制区不参与编码蛋白,具有遗传变异位点多及进化速率快等特点,特别适合种群遗传多样性分析,已广泛应用于亲缘关系近的物种或种群间遗传多样性研究(Xiao et al.,2009;Vilà et al.,2010;Sha et al.,2019;韩浩园等,2020)。因此,基于mtDNA控制区序列分析珠江和长江水系光倒刺鲃的群体遗传特点,可为了解光倒刺鲃遗传结构及开展种质资源保护提供参考依据。【前人研究进展】目前,mtDNA已广泛应用于动物起源进化和群体遗传特征研究。Chen等(2015)将怒江裂腹鱼(Schizothorax nukiangensis)mtDNA的COI基因、Cyt b基因与控制区序列组合进行分析,发现不同怒江区域的裂腹鱼群体存在明显差异,其地理隔离分化较明显。Munian和Bhassu(2015)基于mtDNA控制区局部序列对马来西亚半岛河流的4个黑肋纹唇鱼(Osteochilus melanopleurus)群体进行分析,结果表明黑肋纹唇鱼的遗传多样性较低,可能是近期经历了种群扩张或瓶颈效应,4个群体可整体作为一个保护单元进行管理。Brahimi等(2016)基于线粒体Cyt b基因和控制区序列对阿尔及利亚吉尔盆地亮鲃属物种Luciobarbus pallaryi进行群体遗传学和形态学进行分析,结果发现在第四纪中期该物种上下游群体间开始形成长期的地理隔离,最终衍化成2个完全不同的谱系。Guo等(2016)基于线粒体Cyt b基因及其控制区序列对青藏高原地区特有物种异齿裂腹鱼(S. o’connori)的7个群体进行种群动态分析,结果表明青藏高原隆升和第四纪气候振荡对异齿裂腹鱼群体遗传状况产生重要影响。李潮等(2018)对珠江流域6个赤眼鳟(Squaliobarbus curriculus)群体的mtDNA控制区序列进行分析,结果发现珠江流域赤眼鳟未出现明显分化,其种群历史上曾发生过扩张事件,西江是珠江流域赤眼鳟的扩散中心。Chen等(2019)基于线粒体Cyt b基因和控制区序列对长江中游地区增殖放流前后的鲢鱼(Hypophthalmichthys molitrix)群体进行比较,结果发现增殖放流对长江中游野生鲢鱼群体未产生显著的遗传效应。此外,有研究通过分析mtDNA以揭示鱼类的遗传学关系,并探讨线粒体遗传变异与鱼类种群结构的相关性,涉及的鱼类有滇池金线鲃(Sinocyclocheilus grahami)、高肩金线鲃(S. altishoulderus)(Wu et al.,2009)、北江光唇鱼(Acrossocheilus beijiangensis)(Lin et al.,2015)、黑尾近红鲌(Ancherythroculter nigrocauda)(Yan et al.,2020)及多种鮈亚科鱼类等(Zhao et al.,2016),其研究结果均表明mtDNA与种属遗传多样性具有非常密切的联系。【本研究切入点】至今,有关光倒刺鲃的研究主要集中在营养与繁殖(岳磊,2016;李成等,2018)及功能基因(周惠强等,2019;Yang et al.,2019)等方面,而针对光倒刺鲃群体遗传多样性、遗传结构及亲缘地理等方面的研究(陈国生,2011;蓝昭军等,2016)鲜见报道,且涉及的种群也较少。【拟解决的关键问题】通过测定珠江和长江水系10个光倒刺鲃地理群体的mtDNA控制区序列,分析光倒刺鲃群体的遗传变异及进化关系,旨在了解其遗传背景,为光倒刺鲃种质资源保护和可持续开发利用提供科学依据。
1 材料与方法
1. 1 样品采集
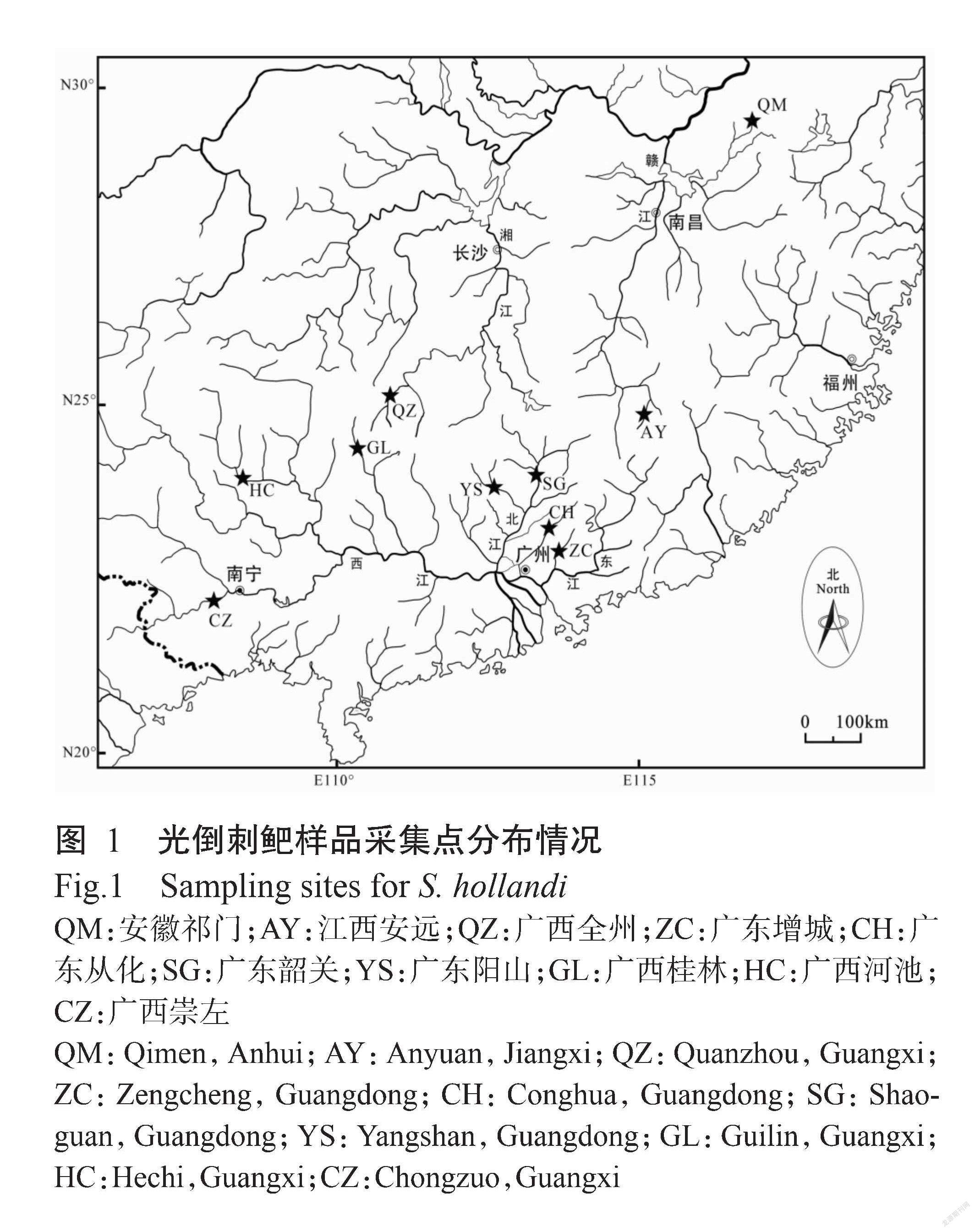
光倒刺鲃样品于2018—2019年采自安徽祁门(QM)、江西安远(AY)、广西全州(QZ)、广东增城(ZC)、广东从化(CH)、广东韶关(SG)、广东阳山(YS)、广西桂林(GL)、广西河池(HC)和广西崇左(CZ),共计10个群体,所在水系分别为阊江、赣江、湘江、增江、流溪河、北江、连江、漓江、柳江和郁江。每个群体样品均采集30尾以上,以采样点缩写为群体编号。各群体采样点分布情况见图1。
1. 2 DNA提取及测序
采用DNA抽提试剂盒[生工生物工程(上海)股份有限公司]提取光倒刺鲃样品基因组DNA;设计扩增引物(F:5'-CTCCCTAGCGCCCAGAAAA-3';R:5'-CCTTGATTAACTTTTGAGCCTGC-3')用于PCR扩增。PCR反应体系25.0 μL:2×Taq PCR MasterMix[0.1 U/mL Taq DNA聚合酶,500 mmol/L dNTP,50 mmol/L Tris-HCl(pH 8.7),20 mmol/L KCl,4 mmol/L MgCl2]12.5 μL,正、反向引物(10 mmol/L)各1.0 μL,DNA模板3.0 μL,蒸餾水7.5 μL。PCR扩增反应在伯乐T100型PCR仪(美国)上进行,扩增程序:94 ℃预变性3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 90 s,进行35个循环;72 ℃延伸10 min。PCR扩增产物以1.0%琼脂糖凝胶电泳进行检测,目的片段送至生工生物工程(上海)股份有限公司进行纯化并测序。
1. 3 数据分析
使用Muscle v5对测序获得的序列进行比对分析,并辅以人工校对。利用MEGA 6.0计算碱基组成、变异位点、转换与颠换比及遗传距离(Tamura et al.,2011),并基于Kimura’s-2-parameter模型构建单倍型系统发育进化树,Bootstrap设为1000次(Felsenstein,1985);通过DnaSP 6.10(邹辉等,2020)和Arlequin 3.5程序包(Excoffier et al.,2007)统计各群体的核苷酸多样性(π)、单倍型多样性(Hd)及遗传分化系数(Fst);并进行分子变异分析(Analysis of molecular variance,AMOVA)、核苷酸错配分布分析(Mismatch-distribution)及中性检验(Neutrality tests);使用Network 10.1以Median-joining法构建各单倍型间的网络结构图(Bandelt et al.,1999)。
2 结果与分析
2. 1 光倒刺鲃mtDNA序列特征及其遗传多样性
在10个光倒刺鲃群体中有347尾光倒刺鲃扩增获得mtDNA控制区序列,序列长度为519 bp,其中T、C、A、G占比平均值分别为32.57%、19.16%、32.16%和16.11%;在所有mtDNA控制区序列中共检测到62个变异位点,包含60个简约信息位点和2个单变异位点。在347个mtDNA控制区序列中,共定义出34个单倍型(表1)。其中,10个单倍型为2个或2个以上群体共享的单倍型,12个单倍型为单一群体内多个个体所共享,其余12个单倍型仅存在于单一个体中。遗传多样性分析结果表明,10个光倒刺鲃群体的Hd平均为0.917,π平均为0.0359。以SG群体的Hd最高(0.827),CH群体(0.757)和YS群体(0.745)次之,ZC群体的Hd最低(0.057);π的范围为0.0001~0.0422,以YS群体的最高(0.0422),SG群体(0.0377)和CH群体(0.0310)次之,ZC群体的最低(0.0001)。
2. 2 光倒刺鲃群体的遗传距离及遗传分化情况
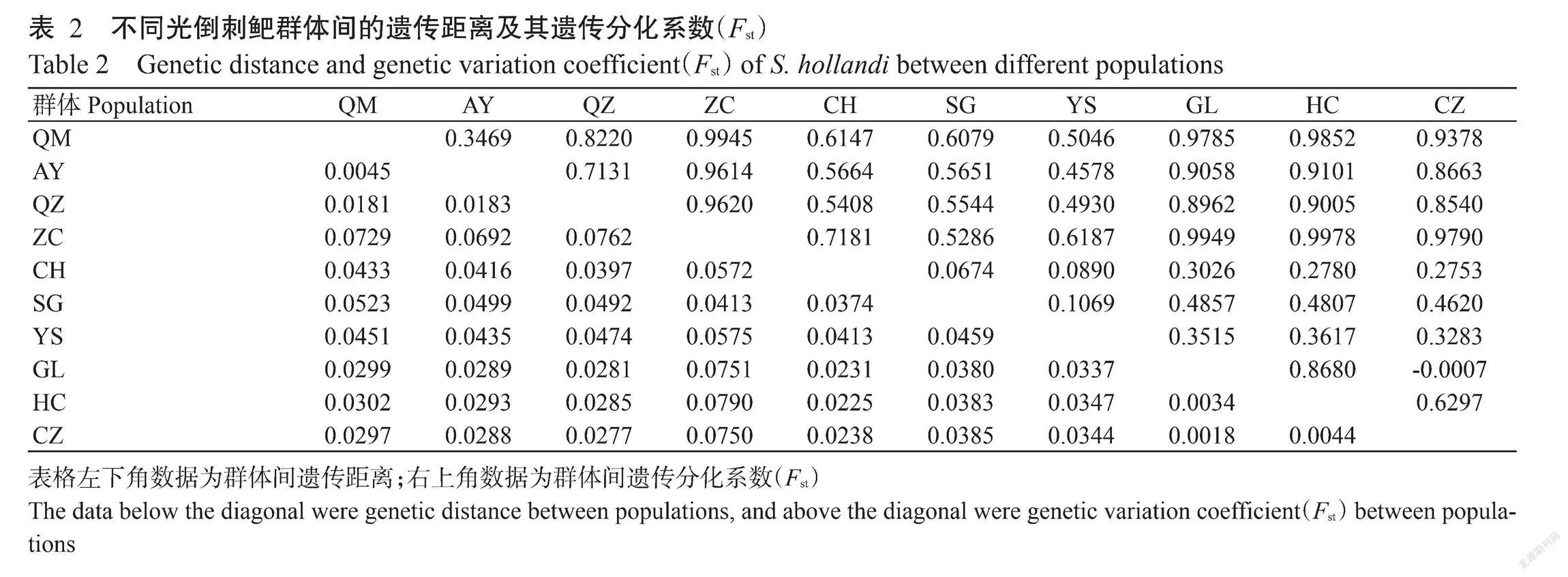
基于mtDNA控制区序列分析光倒刺鲃群体的遗传距离和Fst,结果(表2)显示,10个光倒刺鲃群体间的遗传距离为0.0018~0.0790。其中,GL群体与CZ群体间的遗传距离最小;ZC群体与HC群体间的遗传距离最大,且与其他群体间的遗传距离普遍较大(0.0413~0.0751)。10个光倒刺鲃群体间的Fst为 -0.0007~0.9978。其中,CH群体与2個北江水系群体(SG和YS)的Fst均较低(0.0674和0.0890),表明CH群体与北江水系群体(SG和YS)间的遗传分化水平较低;ZC群体间与其余群体的Fst普遍较高(0.5286<Fst<0.9978),即ZC群体与其余群体间的遗传分化水平较高。
2. 3 AMOVA分析结果
为了解光倒刺鲃群体分子变异的地理分布模式,将10个光倒刺鲃群体划分为5个地理组群[长江的赣江水系(QM和AY群体)、湘江水系(QZ群体)、珠江的东江水系(ZC群体)、北江(SG和YS群体)+流溪河水系(CH群体)及西江水系(GL、HC和CZ群体)]进行AMOVA分析,结果(表3)表明,光倒刺鲃群体分子遗传变异来自各区域分组间的占63.46%,来自各区域分组内的仅占6.14%,而各采样群体内变异占30.40%,说明光倒刺鲃mtDNA控制区序列的遗传分化主要来自各地理分组间。
2. 4 单倍型网络结构分析结果
对10个光倒刺鲃群体进行单倍型网络结构分析,结果(图2)表明,不同单倍型的关系网络可分成3个子网络(Subnet I~Subnet III)。其中,Subnet I包含所有赣江水系群体(QM和AY)和1个西江水系的CZ个体;Subnet II包含东江水系群体(ZC)、北江(SG和YS群体)和流溪河水系(CH群体)的部分个体和1个赣江水系的AY个体;Subnet III包含北江(SG和YS群体)和流溪河水系(CH群体)的部分个体、西江水系群体(GL、HC和CZ)和1个湘江水系的QZ个体。大多数单倍型间只有1~2步的变异步数;Subnet I与Subnet II间的变异步数在33步以上,Subnet I与Subnet III间的变异步数在13步以上,而Subnet II与Subnet III间的变异步数在51步以上,是网络结构图中变异步数最大的单倍型。
2. 5 单倍型系统发育分析结果
为了更好地了解光倒刺鲃群体间的亲缘关系,利用邻接法(Neighbor-joining,NJ)对34个单倍型进行系统发育进化分析,由构建的系统发育进化树(图3)可知不同单倍型聚类成两大支系。其中,支系I又分为2个小分支(A和B),分支A包含北江+流溪河水系(CH、SG和YS群体)的部分个体、西江水系群体(GL、HC和CZ)和1个湘江水系的QZ个体,分支B则包含长江水系群体(QM、AY和QZ)和1个西江水系的CZ个体;支系II包含东江水系(ZC群体)所有个体、北江+流溪河水系(CH、SG和YS群体)的部分个体及1个赣江水系的AY个体,群体分布与单倍型网络结构图(图2)基本一致。
2. 6 光倒刺鲃种群动态分析结果
对10个光倒刺鲃群体的mtDNA控制区序列进行中性检验,结果(表4)显示,AY、QZ和CZ群体的Tajima’s D为显著性负值(P<0.05),对应的Fu’s Fs分别为1.532、4.190和-1.021,均不显著(P>0.05,下同);其余群体的Tajima’s D为-1.136~2.824,Fu’s Fs为-1.339~23.759,但统计均不显著。此外,对10个光倒刺鲃群体进行核苷酸错配分布分析,结果显示各群体的核苷酸错配分布图谱呈多峰形式。
3 讨论
3. 1 光倒刺鲃遗传多样性
遗传多样性是指生物种内与种间的遗传差异程度,由生物与环境长期进化而演变形成,种群遗传多样性越高,表明种群适应环境的能力越强,其生存和进化的能力也越强(杨金权等,2008;张鹤千等,2015;董丁健和戴习林,2020)。其中,Hd和π是影响遗传多样性的2个重要参数(韩浩园等,2020)。本研究结果表明,10个光倒刺鲃群体的Hd平均为0.917,π平均为0.0359。其中,北江+流溪河水系3个群体(CH、SG和YS)具有較高的遗传多样性;而东江水系群体(ZC)和西江水系群体(GL、HC和CZ)的遗传多样性相对较低。依据Grant和Bowen(1998)对鱼类mtDNA控制区序列Hd和π的划分可知,珠江和长江水系光倒刺鲃种群的遗传多样性总体上偏低。近年来,由于光倒刺鲃人工养殖的兴起,个别养殖户仍以捕捞江河光倒刺鲃的鱼苗作为养殖苗种,导致江河群体补充数量不足,光倒刺鲃群体遗传多样性逐渐降低。另一方面,河流水质污染也是影响鱼类多样性的重要因素。刘乾甫等(2019)对西江干流水质的调查发现,采样区域氮源污染超标率超过90%,尤其是人口较多、生产活动较密集的区域,部分区域氨态氮等污染物大量存在,对幼鱼和鱼卵等产生毒性效应,进而威胁鱼类生存。在本研究中,西江水系光倒刺鲃群体遗传多样性水平较低,也可能是受到水质污染的影响。此外,各流域中建设大量水利工程则造成鱼类的栖息地严重萎缩和片段化分布,阻隔各水系间的基因交流,导致鱼类资源下降(李德旺,2012;李跃飞等,2015),也可能导致光倒刺鲃群体遗传多样性进一步降低。遗传多样性降低会导致物种对复杂环境的适应能力减弱和优良性状衰退,不利于种质资源的保护及利用(Crandall et al.,1999)。因此,亟需采取有效保护措施以实现对光倒刺鲃种质资源的可持续利用。
3. 2 光倒刺鲃群体遗传分化
由于生存环境的变更及长期的地理隔离,导致同一物种不同地理种群间产生遗传分化(Duncan et al.,2016)。本研究中,10个光倒刺鲃群体间的Fst为 -0.0007~0.9978,其中,ZC群体与其余群体间的Fst普遍较高(0.5286<Fst<0.9978),即ZC群体与其余群体间的分化水平较高。将10个群体划分成赣江水系、湘江水系、东江水系、北江+流溪河水系和西江水系等5个地理组群进行分析,结果发现东江水系群体与其他水系群体间均具有较高的遗传分化,尤其是与赣江水系群体、湘江水系群体和西江水系群体间的Fst均超过0.9500;西江水系群体与赣江水系群体和湘江水系群体间的Fst也较高,均在0.8500以上;群体间的遗传距离也表明不同水系光倒刺鲃群体间存在较明显的遗传分化。此外,AMOVA分析结果显示,63.46%的光倒刺鲃mtDNA控制区序列分子遗传变异来自各地理分组间,进一步证实各区域分组间的光倒刺鲃群体已发生明显分化。单倍型系统发育进化树及其网络结构图均显示,10个光倒刺鲃群体聚类为三大支系,除了有个别交集外,东江水系、西江水系、赣江水系+湘江水系的光倒刺鲃群体分布在3个不同的独立分支上,而北江+流溪河水系群体分别与东江水系群体和西江水系群体有较多交集。可见,长江水系与珠江水系光倒刺鲃群体间的亲缘关系较远;在珠江水系内部,东江水系与西江水系光倒刺鲃群体间的亲缘关系也较远,而北江+流溪河水系与东江水系和西江水系光倒刺鲃群体间的亲缘关系较近。南岭山脉的形成可能是长江水系与珠江水系光倒刺鲃群体分化的重要原因之一。从地理位置来看,西江与北江干流通过思贤滘联通,北江水系光倒刺鲃群体与西江水系光倒刺鲃群体发生基因交流,致使其亲缘关系较接近;而流溪河下游多处与北江联通,光倒刺鲃群体发生频繁的基因交流,导致其亲缘关系非常接近。狮子洋将东江与北江和西江连接、并与西江隔离开来,间冰期海平面上升时,东江将西江和北江形成一定地理隔离(Wang et al.,1999;Liu et al.,2003;梁晓旭等,2010),而后期东江水系光倒刺鲃群体又可在潮汐交替时通过狮子洋与北江水系光倒刺鲃群体接触,使得东江水系与北江水系的光倒刺鲃群体发生基因交流。即复杂的地形地貌导致珠江和长江水系光倒刺鲃群体形成较明显的地理隔离。
3. 3 光倒刺鲃种群动态分析
了解物种的种群遗传结构及其历史动态,可用于预测物种进化潜能,为保护各水系鱼类的遗传多样性提供依据。中性检验常用于判断种群是否经历过扩张事件,若Tajimaʼs D和Fuʼs Fs均呈负值,且在统计学上具有显著性,说明基因序列中含有较中性进化模型更多的核苷酸位点变异,即该种群可能发生过扩张事件(Tajima,1989)。本研究结果表明,10个光倒刺鲃群体的Fuʼs Fs为-1.339~23.759,平均为9.857,但P均大于0.05;Tajimaʼs D为-2.613~2.824,其中AY、QZ和CZ群体的Tajima’s D为显著性负值;核苷酸错配分布分析结果显示各群体的核苷酸错配分布图谱呈多峰形式。可见,珠江和长江水系光倒刺鲃群体尚未发生过种群扩张,与蓝昭军等(2016)基于Cyt b基因对部分珠江和长江光倒刺鲃群体的分析结论相似。
4 结论
珠江和长江水系光倒刺鲃群体遗传多样性总体上偏低,应加强其种质资源保护,尤其是北江水系群体野生资源相对较丰富宜作为重点保护单元。复杂的地形地貌导致珠江和长江水系光倒刺鲃群体形成明显的地理隔离,群体间已发生明显分化,且受各种人为活动干扰导致其种群收缩,因此亟待采取有效防护措施以保证光倒刺鲃种质资源的可持续利用。
参考文献:
蔡子德,林岗,倪家延,叶钊,唐志斌,张盛. 2007. 光倒刺鲃的繁殖生物学研究[J]. 广西农业科学,38(2):200-204. [Cai Z D,Lin G,Ni J Y,Ye Z,Tang Z B,Zhang S. 2007. Study on the reproductive biology of Spinibabus calddwellis[J]. Guangxi Agricultural Sciences,38(2):200-204.] doi:10.3969/j.issn.2095-1191.2007.02.025.
陈国生. 2011. 应用RAPD技术分析闽江中上游黑脊倒刺鲃和黄颡鱼遗传多样性[J]. 现代渔业信息,26(5):9-11. [Chen G S. 2011. Application of RAPD technology on analysis of genetic diversity of Spinibarbus caldwelli & Pelteobagrus eupogon in Minjiang River[J]. Modern Fisheries Information,26(5):9-11.] doi:10.3969/j.issn.1004-8340. 2011.05.003.
董丁健,戴習林. 2020. 罗氏沼虾不同群体世代遗传多样性的SSR分析[J]. 南方农业学报,51(2):421-428. [Dong D J,Dai X L. 2020. SSR analysis on genetic diversity in different populations and generations of Macrobrachium rosenbergii[J]. Journal of Southern Agriculture,51(2):421-428.] doi:10.3969/j.issn.2095-1191.2020.02.023.
董志国,李晓英,王普力,王文进,张庆起,阎斌伦,孙效文. 2013. 基于线粒体D-loop基因的中国海三疣梭子蟹遗传多样性与遗传分化研究[J]. 水产学报,37(9):1304-1312. [Dong Z G,Li X Y,Wang P L,Wang W J,Zhang Q Q,Yan B L,Sun X W. 2013. Genetic diversity and genetic differentiation of swimming crab (Portunus trituberculatus) from six geographical populations of China Sea based on mitochondrial D-loop gene[J]. Journal of Fisheries of China,37(9):1304-1312.] doi:10.3724/SP.J. 1231.2013.38630.
韩浩园,权凯,闫志浩,高慧军,刘德奇,王先宁,尹慧茹,赵金艳,贾万里,李红霞,余世锋,王拥庆,李君. 2020. 河南奶山羊线粒体D-loop区遗传多态性和遗传分化研究[J]. 河南农业大学学报,54(4):643-649. [Han H Y,Quan K,Yan Z H,Gao H J,Liu D Q,Wang X N,Yin H R,Zhao J Y,Jia W L,Li H X,Yu S F,Wang Y Q,Li J. 2020. Genetic diversity and genetic differentiation analysis of mitochondrion D-loop region in Henan dairy goats[J]. Journal of Henan Agricultural University,54(4):643-649.] doi:10.16445/j.cnki.1000-2340.2020.04.012.
蓝昭军,李强,舒琥,谷平华,王杭军,吴土金. 2016. 基于线粒体细胞色素b基因的光倒刺鲃遗传多样性与遗传结构研究[J]. 海洋与湖沼,47(6):1158-1165. [Lan Z J,Li Q,Shu H,Gu P H,Wang H J,Wu T J. 2016. Genetic diversity and structure of Spinibarbus hollandi based on nucleo-tide sequences of mtDNA cytochrome b gene[J]. Oceanologia et Limnologia Sinica,47(6):1158-1165.] doi:10. 11693/hyhz20160300064.
乐佩琦. 2000. 中国动物志 硬骨鱼纲 鲤形目(下卷)[M]. 北京:科学出版社. [Le P Q. 2000. Chinese zoology,Osteichthyes,Cypriniformes(Volume II)[M]. Beijing:Science Press.]
李潮,蓝昭军,赵俊. 2018. 基于线粒体控制区(D-loop)序列的珠江赤眼鳟遗传多样性与遗传分化[J]. 应用与环境生物学报,24(3):615-622. [Li C,Lan Z J,Zhao J. 2018. Genetic diversity and genetic differentiation of Squaliobarbus curriculus from the Pearl River based on mitochondrial D-loop sequences[J]. Chinese Journal of Applied & Environmental Biology,24(3):615-622.] doi:10.19675/j.cnki.1006-687x.2017.07013.
李成,秦溱,李金龙,何志刚,伍远安,黄华伟,向劲,程小飞. 2018. 不同蛋白水平饲料对光倒刺鲃幼鱼生长、消化酶及体成分的影响[J]. 饲料工业,39(24):34-39. [Li C,Qin Q,Li J L,He Z G,Wu Y A,Huang H W,Xiang J,Cheng X F. 2018. Effect of dietary with different protein levels on the growth,digestive enzyme activities and body composition of the juvenile Spinibarbus hollandi Oshima[J]. Feed Industry,39(24):34-39.] doi:10.13302/j.cnki.fi.2018.24.007.
李德旺. 2012. 长江上游生态敏感度与水电开发生态制约研究[D]. 武汉:武汉大学. [Li D W. 2012. Study of the eco-environmental sensitivity of upper Changjiang River and ecological restriction on hydropower development[D]. Wuhan:Wuhan University.]
李跃飞,李新辉,杨计平,李捷,帅方敏. 2015. 珠江干流长洲水利枢纽蓄水后珠江鳡鱼(Elopichthys bambusa)早期资源现状[J]. 湖泊科学,27(5):917-924.[Li Y F,Li X H,Yang J P,Li J,Shuai F M. 2015. Status of Elopichthys bambusa recruitment stock after the impoundment of Changzhou hydro-junction in Pearl River[J]. Journal of Lake Sciences,27(5):917-924.] doi:10.18307/2015.0519.
梁晓旭,庆宁,杨柯林,万彩霞,赵俊,陈湘粦. 2010. 广东地区宽鳍鱲种群遗传变异和亲缘地理[J]. 水生生物学报,34(4):806-814. [Liang X X,Qing N,Yang K L,Wan C X,Zhao J,Chen X L. 2010. Population genetic variation and phylogeography of Zacco platypus in Guangdong region[J]. Acta Hydrobiologica Sinica,34(4):806-814.] doi:10.3724/SP.J.1035.2010.00806.
劉乾甫,杜浩,赖子尼,帅方敏,郭霞. 2019. 珠江中上游水环境状况分析与评价[J]. 中国渔业质量与标准,9(4):36-47. [Liu Q F,Du H,Lai Z N,Shuai F M,Guo X. 2019. Analysis and assessment of water environmental quality of the upper and middle reaches of the Pearl River[J]. Chinese Fishery Quality and Standards,9(4):36-47.] doi:10.3969/j.issn.2095-1833.2019.04.006.
罗刚,张胜茂,邹国华. 2017. 国家重点保护经济水生动植物图谱[M]. 北京:中国农业出版社. [Luo G,Zhang S M,Zou G H. 2017. Graph of economic aquatic animals and plants under special state protection[M]. Beijing:China Agriculture Press.]
潘烔华,钟麟,郑慈英,伍汉霖,刘家照. 1991. 广东淡水鱼类志[M]. 广州:广东科技出版社. [Pan D H,Zhong L,Zheng C Y,Wu H L,Liu J Z. 1991. The freshwater fi-shes of Guangdong Province[M]. Guangzhou:Guangdong Science and Technology Press.]
杨金权,胡雪莲,唐文乔,林弘都. 2008. 长江口邻近水域刀鲚的线粒体控制区序列变异与遗传多样性[J]. 动物学杂志,43(1):8-15. [Yang J Q,Hu X L,Tang W Q,Lin H D. 2008. mtDNA control region sequence variation and genetic diversity of Coilia nasus in Yangtze River estuary and its adjacent waters[J]. Chinese Journal of Zoology,43(1):8-15.] doi:10.3969/j.issn.0250-3263.2008.01.002.
岳磊. 2016. 光倒刺鲃(Spinibarbus hollandi)杂交育种及遗传多样性研究[D]. 广州:广州大学. [Yue L. 2016. Cross bree-ding and genetic diversity for Spinibarbus hollandi populations[D]. Guangzhou:Guangzhou University.]
张鹤千,杨子拓,李桂峰,肖诗斌,孙际佳,杨慧荣,赵会宏,谢堂晖,韩兴鹏,刘丽. 2015. 珠江流域野生黄颡鱼Pelteobagrus fulvidraco的Cyt b基因序列分析[J]. 中山大学学报(自然科学版),54(5):102-108. [Zhang H Q,Yang Z T,Li G F,Xiao S B,Sun J J,Yang H R,Zhao H H,Xie T H,Han X P,Liu L. 2015. Genetic diversity of mitochondrial Cyt b sequences in Pelteobagrus fulvidraco from Pearl River[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni,54(5):102-108.] doi:10.13471/j.cnki.acta. snus.2015.05.020.
周惠強,陈凯棱,舒琥,杨扬,钟东明,张明清. 2019. 光倒刺鲃mc5r基因的克隆及对饥饿的响应[J]. 南方水产科学,15(4):99-106. [Zhou H Q,Chen K L,Shu H,Yang Y,Zhong D M,Zhang M Q. 2019. Cloning and starvation response of mc5r gene in Spinibarbus hollandi[J]. South China Fisheries Science,15(4):99-106.] doi:10.12131/20190027.
邹辉,韦玲静,黄杰,甘宝江,莫洁琳,杨著山,滕忠作,刘康,叶香尘. 2020. 基于线粒体CO I基因和D-Loop区序列的7个鲤群体遗传差异分析[J]. 南方农业学报,51(5):1209-1216. [Zou H,Wei L J,Huang J,Gan B J,Mo J L,Yang Z S,Teng Z Z,Liu K,Ye X C. 2020. Genetic differences among seven carp populations based on mitochondrial CO I gene and D-Loop region sequences[J]. Journal of Southern Agriculture,51(5):1209-1216.] doi:10.3969/j.issn.2095-1191.2020.05.028.
Bandelt H J,Forster P,Röhl A. 1999. Median-joining networks for inferring intraspecific phylogenies[J]. Molecular Biology and Evolution,16(1):37-48. doi:10.1093/oxfordjournals.molbev.a026036.
Brahimi A,Tarai N,Benhassane A,Henrard A,Libois R. 2016. Genetic and morphological consequences of Quaternary glaciations:A relic barbel lineage(Luciobarbus pallaryi,Cyprinidae) of Guir Basin(Algeria)[J]. Comptes Rendus Biologies,339(2):83-98. doi:10.1016/j.crvi.2015. 12.003.
Chen H J,Wang D Q,Guo J,Duan X B,Liu S P,Chen D Q,Li Y. 2019. Monitoring the genetic effects of broodstock enhancement of silver carp(Hypophthalmichthys molitrix) in middle Yangtze River based on cytb gene and D-loop sequences[J]. Journal of Freshwater Ecology,34(1):323-332. doi:10.1080/02705060.2019.1588795.
Chen W T,Du K,He S P. 2015. Genetic structure and historical demography of Schizothorax nukiangensis(Cyprinidae) in continuous habitat[J]. Ecology and Evolution,5(4):984-995. doi:10.1002/ece3.1413.
Crandall K A,Posada D,Vasco D. 1999. Effective population sizes:Missing measures and missing concepts[J]. Animal Conservation,2(4):317-319. doi:10.1111/j.1469-1795.1999.tb00078.x.
Duncan C J,Worth J R P,Jordan G J,Jones R C,Vaillancourt R E. 2016. Genetic differentiation in spite of high gene flow in the dominant rainforest tree of southeastern Australia,Nothofagus cunninghamii[J]. Heredity,116(1):99-106. doi:10.1038/hdy.2015.77.
Excoffier L,Laval G,Schneider S. 2007. Arlequin (version 3.0):An integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics,1:47-50. doi:10.1177/117693430500100003.
Felsenstein J. 1985. Confidence limits on phylogenies:An approach using the bootstrap[J]. Evolution,39(4):783-791. doi:10.1111/j.1558-5646.1985.tb00420.x.
Grant WA S,Bowen B W. 1998. Shallow population histories in deep evolutionary lineages of marine fishes:Insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity,89(5):415-426. doi:10.1093/jhered/89.5.415.
Guo X Z,Zhang G R,Wei K J,Yan R J,Ji W,Yang R B,Wei Q W,Gardner J P A. 2016. Phylogeography and popu-lation genetics of Schizothorax o'connori:Strong subdivision in the Yarlung Tsangpo River inferred from mtDNA and microsatellite markers[J]. Scientific Reports,6:29821. doi:10.1038/srep29821.
Lin T T,Li Q,Shu H,Yue L,Lin J F,Hou L P. 2015. The complete mitochondrial genome of Acrossocheilus bei-jiangensis(Cyprinidae,Barbinae)[J]. Mitochondrial DNA. Part A:DNA Mapping,Sequencing,and Analysis,27(6):4540-4542. doi:10.3109/19401736.2015.1101553.
Liu Z F,Trentesaux A,Clemens S C,Colin C,Wang P X,Huang B Q,Boulay S. 2003. Clay mineral assemblages in the northern South China Sea:Implications for East Asian monsoon evolution over the past 2 million years[J]. Marine Geology,201(1-3):133-146. doi:10.1016/S0025-3227(03)00213-5.
Munian K,Bhassu S. 2015. Genetic structure of locally threa-tened cyprinid,Osteochilus melanopleurus,in Peninsular Malaysia River systems inferred from mitochondrial DNA control region[J]. Biochemical Systematics and Ecology,61:336-343. doi:10.1016/j.bse.2015.06.034.
Sha Y Z,Gao C X,Liu M M,Zhao S G. 2019. Evaluation of the genetic diversity of six Chinese indigenous chickens[J]. Asian-Australasian Journal of Animal Sciences,33(10):1566-1572. doi:10.5713/ajas.19.0606.
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics,123(3):585-595. doi:10.1093/genetics/123.3.585.
Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S. 2011. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology & Evolution,28(10):27-31. doi:10.1093/molbev/msr121.
Vilà C,Amorim I R,Leonard J A,Posada D,Castroviejo J,Petrucci-Fonseca F,Crandall K A,Ellegren H,Wayne R K. 2010. Mitochondrial DNA phylogeography and population history of the grey wolf Canis lupus[J]. Molecular Ecology,8(12):2089-2103. doi:10.1046/j.1365-294x. 1999.00825.x
Wang L,Sarnthein M,Erlenkeuser H,Grimalt J,Grootes P,Heilig S,Ivanova E,Kienast M,Pelejero C,Pflaumann U. 1999. East Asian monsoon climate during the Late Pleistocene:High-resolution sediment records from the South China Sea[J]. Marine Geology,156(1-4):245-284. doi:10.1016/S0025-3227(98)00182-0.
Wu X Y,Wang L,Chen S Y,Zan R G,Xiao H,Zhang Y P. 2009. The complete mitochondrial genomes of two species from Sinocyclocheilus(Cypriniformes:Cyprinidae) and a phylogenetic analysis within Cyprininae[J]. Mole-cular Biology Reports,37:2163-2171. doi:10.1007/s11033- 009-9689-x.
Xiao Y S,Zhang Y,Gao T X,Yanagimoto T,Yabe M,Sakurai Y. 2009. Genetic diversity in the mtDNA control region and population structure in the small yellow croaker Larimichthys polyactis[J]. Environmental Biology of Fi-shes,85(4):303-314. doi:10.1007/s10641-009-9497-0.
Yan T M,Wang X Y,Li S,He Z D,Luo J,Zhang Q,Yang D Y,He Z. 2020. Genetic analysis of wild Ancherythrocul-ter nigrocauda in tributaries and the main stream of the upper Yangtze River basin of China[J]. Mitochondrial DNA. Part A:DNA Mapping,Sequencing,and Analysis,31(1):17-24. doi:10.1080/24701394.2019.1698558.
Yang Y,Zhou H Q,Hou L P,Xing K,Shu H. 2019. Transcriptional profiling of skeletal muscle reveals starvation response and compensatory growth in Spinibarbus hollandi[J]. BMC Genomics,20(1):938. doi:10.1186/s12864-019-6345-2.
Zhao J,Xu D,Zhao K,Diogo R,Yang J,Peng Z. 2016. The origin and divergence of Gobioninae fishes(Teleostei:Cyprinidae) based on complete mitochondrial genome sequences[J]. Journal of Applied Ichthyology,32(1):32-39. doi:10.1111/jai.12920.
收稿日期:2020-11-19
基金項目:国家自然科学基金项目(41673110);广州市科技计划项目(201804010486)
通讯作者:李强(1983-),https://orcid.org/0000-0002-5899-3776,博士,主要从事淡水鱼类生态与亲缘地理研究工作,E-mail:LQ512328 @163.com
第一作者:李文俊(1991-),https://orcid.org/0000-0001-8020-3534,研究方向为鱼类生态学,E-mail:leewenjun@163.com
1898501186284

