1株塞内卡病毒A型的基因组序列与演化分析
李 宁,郭慧芳,王白玉,乔麒龙,黄 庆,李永涛,王 增,赵 军
(河南农业大学牧医工程学院,郑州 450046)
塞内卡病毒A(Senecavirus A, SVA)是小RNA病毒科(Picornaviridae)塞内卡病毒属(Senecavirus)唯一成员,为无囊膜单股正链线性RNA病毒[1-3]。SVA基因组大小约为7.3 kb,由5′端非编码区(5′-UTR)、1个编码多聚蛋白的开放阅读框(open reading frame, ORF)和3′端非编码区(3′-UTR)组成[4-6]。SVA早期被称作塞内卡谷病毒(Seneca Valley virus,SVV),原型为SVV-001[1,7]。塞内卡病毒病(Senecavirus disease,SVD)最先于2007年在加拿大被报道,其特征是鼻吻和蹄冠部产生明显水疱,发病母猪伴随发热和厌食,新生仔猪表现为腹泻、脱水、神经症状和急性死亡,尤其是1~4日龄的新生仔猪发病率可达70%,病死率为15%~30%[3]。随后在美国、加拿大、哥伦比亚、巴西、泰国、越南等国家的猪群中均报道有SVA的广泛分布[8-16]。
在中国,SVD于2015年在广东首次被报道,随后在广西、福建、湖北、河南、山东和黑龙江等省(区)陆续发生SVD疫情[17-34]。SVA引起的临床症状与口蹄疫、猪水疱病、水疱性口炎等疫病的临床症状相似,临床上难以区分。SVA在我国的广泛流行严重危害我国养猪业的健康发展。当前,我国尚没有商品化疫苗控制SVA的感染,加强SVA流行毒株的分子流行病学监测对于筛选未来用于制备疫苗的候选毒株,研发行之有效的疫苗和检测试剂至关重要。
2019年11月,笔者从河南省新乡市长垣县某育肥猪场送检的临床样品中分离到1株SVA,本研究对其进行了全基因序列测定和分析,以期为全面了解SVA在我国的流行状况和制定合理的防控措施提供参考。
1 材料与方法
1.1 病毒
SVA CH-HNCY-2019株由河南农业大学传染病教研室自河南新乡长垣县某发生典型的水疱病的育肥猪猪场分离并保存,病毒滴度为10-7TCID50· 0.1 mL-1。
1.2 引物设计和SVA基因扩增
参照SVA原型毒株SVV-001株全基因序列(GenBank登录号:NC011349),设计用于分段扩增SVA基因组序列的引物(表1)。提取SVA CH-HNCY-2019 病毒RNA并反转录成cDNA。以cDNA为模板进行PCR扩增SVA CH-HNCY-2019基因组片段。
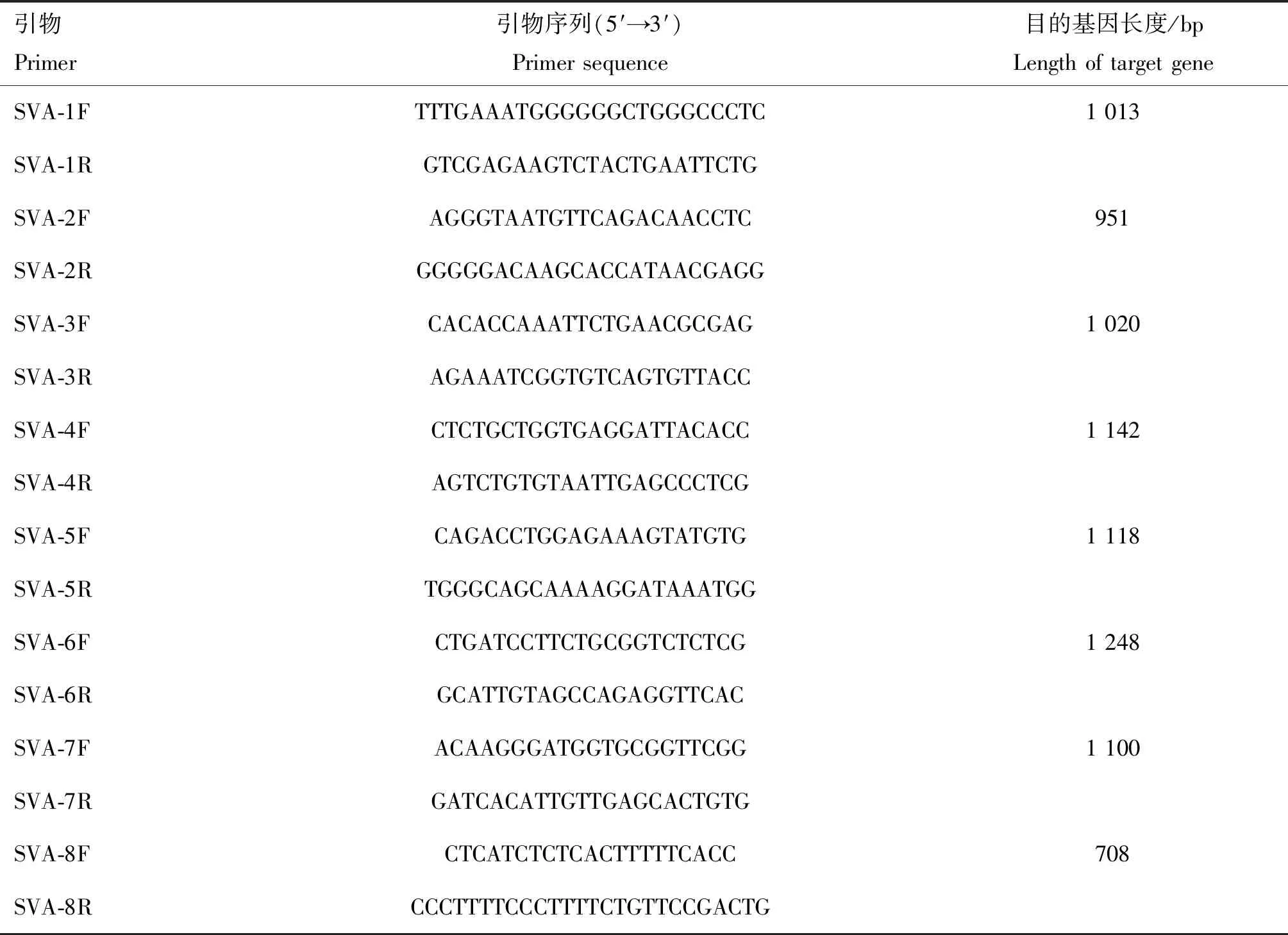
表1 SVA全基因序列分段扩增引物
1.3 SVA CH-HNCY-2019全基因序列测定与比较分析
将扩增的SVA基因各片段纯化后,送生工生物工程(上海)股份有限公司测序。将所得序列利用DNA Star Lasergene v7.0软件包拼接,获得完整病毒基因组序列并上传到GenBank数据库,并与GenBank上已发表的中国SVA流行毒株和国外SVA代表株序列进行同源性比对分析;应用MEGA6软件中的Neighbor-joining统计方法绘制系统进化树。
2 结 果
2.1 目的基因扩增
利用设计的SVA特异性引物将CH-HNCY-2019基因组进行分段扩增,扩增出8个片段,与预期的目的片段大小相符(图1)。

M.DL2000 DNA分子质量标准; F1~F8.8个覆盖SVA基因组的重叠片段(1 018、951、1 020、1 142、1 118, 1 248、1 100和705 bp)
2.2 SVA CH-HNCY-2019株全基因序列测定和比较分析
CH-HNCY-2019基因组全长为7 294 bp,包括671 bp的5′UTR,1个编码2 182个氨基酸的前体多聚蛋白开放阅读框以及76 bp具有Poly(A)特性的3′UTR。将CH-HNCY-2019 全基因组序列上传到GenBank,获得登录号为MT713137。
将CH-HNCY-2019全基因组与GenBank中公布的国内外不同年代的SVA代表株的全基因序列进行遗传进化分析显示(图2),包括CH-HNCY-2019在内的所有中国毒株与美国2015年及其以后的毒株,以及哥伦比亚、巴西、越南的毒株都属于一个大群,提示这些毒株具有相同的遗传来源。所有中国毒株又分属4个独立的分支,其中,CH-HNCY-2019和另一个广东2019年分离株CH-GDZS-2019位于大多数2017—2018年中国毒株所在的中国Ⅰ这一独立分支中,CH-HNCY-2019与GD03/2017、CHhb17、GD01/2017、SVA/CHN/01/2017和SVV-SC-0102018属于中国Ⅰ独立分支的同一小分支。另外3个广东2019年分离株CH-GDHZ01-2019、CH-GDHZ02-2019、CH-GDMZ-2019则与部分2017—2018年中国毒株属于另一个中国Ⅱ独立分支,中国Ⅱ分支毒株与美国2015年及其以后的毒株进化关系较近。由分离自广东、福建和辽宁的部分2017—2018年的毒株组成的中国Ⅲ分支与哥伦比亚的毒株进化关系较近;由2015—2016年分离株组成的中国Ⅳ分支与最近报道的越南分离株和2015年的巴西分离株亲缘关系较近。
全基因组序列同源性分析显示,SVA CH-HNCY-2019毒株与其他SVA毒株的核苷酸相似性为93.2%~99.0%,与SVA原型毒株SVV-001的相似性为93.2%,与其他美国毒株的相似性为93.3%~98.1%,与加拿大毒株的相似性为95.9%~96.3%,与泰国毒株的相似性为94.4%~94.8%,与越南毒株的相似性为96.4%,与巴西毒株的相似性为97%,与哥伦比亚毒株的相似性为97.8%,与中国分离株的相似性为95.9%~99.0%。
将包括CH-HNCY-2019在内的2019年中国SVA分离株的结构基因氨基酸序列与中国其他年代的分离株以及美国经典毒株SVV-001和强毒代表株SD15-26进行比较分析发现,CH-HNCY-2019的VP1蛋白的722位氨基酸由L突变成了Q,而其他2019年中国SVA分离株CH-GDZS-2019、CH-GDMZ-2019、CH-GDHZ01-2019和CH-GDHZ02-2019,中国其他年代的分离株,以及SVV-001和SD15-26毒株的722位氨基酸均为L;VP3蛋白的499位氨基酸由A突变为V,528位氨基酸由P突变位S,582位氨基酸由E突变位K;3A蛋白的1 430位 氨基酸由A突变位V。所有2019年中国分离株的3C蛋白1 685位氨基酸均为M,而其他国内外SVA毒株3C蛋白1 685位氨基酸均为L。此外,与SVV-001相比,所有中国毒株和SD15-26毒株的VP1均存在多处相同的氨基酸替换(Q735A、E736T、A766V、G770D、F834Y和I912V);VP2蛋白也存在3处相同的氨基酸替换(T165I、N368S和A428T);VP3蛋白存在多处氨基酸替换(L452I、E491G、V497E、T502A和V603I)。
3 讨 论
SVA在我国的广泛流行严重危害我国养猪业的健康发展,本研究获得SVA分离株CH-HNCY-2019毒株与其他SVA毒株的核苷酸相似性为93.2%~99.0%,与中国分离株的相似性为95.9%~99%,而与SVA原型毒株SVV-001的相似性最低,只有93.2%。中国自2015年以来的所有毒株在全基因组进化树上分属同一大群的4个不同分支,包括CH-HNCY-2019在内的5株2019年中国SVA分离株分属两个不同的分支。这一结果提示,我国SVA的流行株具有相同的起源,但存在多样性,且在不断演变。研究这些处于进化树上不同分支的SVA中国流行株在免疫学上的交叉反应性对于研发防控SVA感染的疫苗具有重要意义。
对中国的SVA分离株的结构蛋白氨基酸序列进行比对分析结果显示,CH-HNCY-2019的VP1、VP3蛋白均存在多位点突变。这些位点氨基酸的突变对于病毒的毒力、免疫原性和在体外细胞培养中的滴度、蚀斑大小等特性的影响值得进一步深入研究。本团队目前正在通过反向遗传技术进行这方面的研究。
4 结 论
成功获得1株SVA CH-HNCY-2019全基因组序列,研究结果提示,SVA中国流行株具有多样性,而且在不断地演变,应加强SVA的分子流行病学调查、生物安全措施和疫苗研发以防止SVA在我国猪群中的广泛传播。

