梅花鹿与欧洲马鹿染色体水平基因组的比较研究
唐丽昕,王天骄,刘华淼,张然然,邢秀梅
(中国农业科学院特产研究所 特种经济动物分子生物重点实验室,长春 130112)
梅花鹿(Cervusnippon)和马鹿(Cervuselaphus)是哺乳纲、偶蹄目、鹿科、鹿属的两个物种,其种间分化时间很短,一般很难得到确切的演化关系[1]。早在20世纪80年代,王宗仁和杜若甫[2]就从细胞遗传学角度,通过染色体核型来解释梅花鹿和马鹿的起源演化关系,指出鹿科动物染色体的进化方式主要是罗伯逊断裂[3],主张马鹿(2n=68)是从梅花鹿(2n=66)演化而来的。而Frank等[4]就线粒体全基因组的研究指出,天山马鹿、东北马鹿和梅花鹿亚种之间的分化时间小于0.01 MYA(millions of years ago,距今百万年),推测它们可能不是两个物种,可能属于梅花鹿的变种。现有的细胞遗传学和线粒体全基因组方面的研究,并没有很好的解释梅花鹿和马鹿演化关系的问题。随着生物技术的飞速发展,越来越多的学者从全基因组角度出发来分析物种进化关系[5-7],这为梅花鹿和欧洲马鹿的演化关系研究提供了新的思路。
比较基因组学(comparative genomics)是在基因组图谱和测序基础上对已知的基因和基因组结构进行比较,来了解基因的功能、表达机理和物种进化的学科,其涉及物种内及物种间的基因组同源性、组成和功能的评估,并且已用于揭示从基因组进化到疾病调控等多方面的相关过程[8]。比较基因组学分为种内基因组比较和种间基因组比较两大类。种内基因组比较通常用于进行与物种适应性等特性有关的功能基因及特异性位点的研究[9-13];种间基因组比较可以揭示基因潜在的功能、为阐明物种进化关系及基因组的内在结构提供理论基础[14-20]。
本研究对梅花鹿和欧洲马鹿染色体水平基因组进行比较基因组学分析,通过两个物种基因组染色体共线性及染色体上基因共线性的分析以及对染色体倒位涉及的基因进行功能富集,揭示了两个物种基因组的同源性,将染色体倒位与嗅觉表型进行相关分析,以期为梅花鹿与欧洲马鹿起源进化关系的研究提供新的理论基础。
1 材料与方法
1.1 梅花鹿与欧洲马鹿基因组序列数据
梅花鹿的基因组序列来自本实验室采用Pacbio三代测序技术结合Hi-C(high-throughput/resolution chromosome conformation capture)辅助组装技术组装得到的基因组序列(暂未发表)。另外,从NCBI数据库下载得到欧洲马鹿的基因组序列Cerla1.0(GCA_002197005.1)[21]。梅花鹿(♀)基因组组装得到33条染色体序列,包括32条常染色体序列以及1条X性染色体序列,基因组大小为2 481 763 803 bp,其中Scaffold N50的长度为78 786 809 bp;下载的欧洲马鹿(♂)基因组得到34条染色体序列,包含33条常染色体序列以及X、Y两条性染色体序列,基因组大小为3 438 623 608 bp,其中Scaffold N50的长度为107 358 006 bp。两个基因组序列均已组装至染色体水平(表1)。

表1 梅花鹿和欧洲马鹿基因组的组装概况
1.2 梅花鹿与欧洲马鹿比较基因组学分析
1.2.1 染色体共线性分析 为了初步获得梅花鹿与欧洲马鹿两个基因组序列的共线性,本研究使用MUMmer v3.1(http://mummer.sourceforge.net/)软件[22],以梅花鹿基因组为参考,对梅花鹿和欧洲马鹿的基因组序列进行比对,参数采用默认,从而获得两个物种间的正、反向最大精确匹配,然后借用show-coords实用工具获得比对的坐标位置、百分比等标识,以获得各条染色体上基因组序列的比对情况。
同时,使用标准参数运行Mummerplot程序,得到gnuplot脚本,用于生成绘图命令,完成基因组序列染色体水平共线性图谱的绘制。
1.2.2 基因功能富集分析 将梅花鹿与欧洲马鹿基因组比对得到的基因组结构变异(染色体倒位)涉及的基因进行功能富集分析。使用KOBAS 3.0数据库[23]以鹿科动物的近源物种牛作为背景数据集进行GO[24]以及KEGG[25]功能富集分析。以明确与这些基因高度相关的功能和生物学通路。
1.3 梅花鹿与欧洲马鹿基因共线性分析
利用共线性分析工具MCScanX,获得梅花鹿和欧洲马鹿两个基因组间基因序列的共线性同源区。首先使用BLASTP将梅花鹿基因组的蛋白编码序列建库,与欧洲马鹿基因组全部蛋白质序列进行all-vs-all比对[26],为了避免由于串联阵列而产生大量的局部共线基因对,BLASTP的E值默认为10-5[27]。全基因组BLASTP结果用于计算所有可能的染色体和scaffolds对的共线性区块。接着使用MCScanX程序,将GFF格式文件和BLASTP表格文件作为输入,根据所有可能的染色体和scaffold对的位置,在两个转录方向上对基因之间的匹配进行分类。相邻的共线基因连接成共线性区块,将成对的共线块和串联基因对分别输出。
同时,对基因共线性数据统计得到两个基因组间共线性区域的数量、共线区域内包含的基因对数量以及梅花鹿与欧洲马鹿基因组间共线性基因占各自基因组的百分比,利用TBtools[28]软件输入两个物种同源基因对的CDS序列,计算出同源基因的同义突变率(Ks)以及Ka/Ks的值。
2 结 果
2.1 梅花鹿与欧洲马鹿染色体共线性分析
2.1.1 基因组序列的同源性 为了获得梅花鹿和欧洲马鹿染色体序列的共线性,将两个物种进行基因组序列比对,结果显示,梅花鹿与欧洲马鹿两个基因组序列表现出同源性(图1)。梅花鹿基因组序列长度为2 481 763 803 bp,欧洲马鹿基因组序列长度为3 438 623 608 bp,梅花鹿与欧洲马鹿同源基因序列长度为1 865 425 381 bp,占梅花鹿基因组序列的75.17%,占欧洲马鹿基因组序列的55.10%。
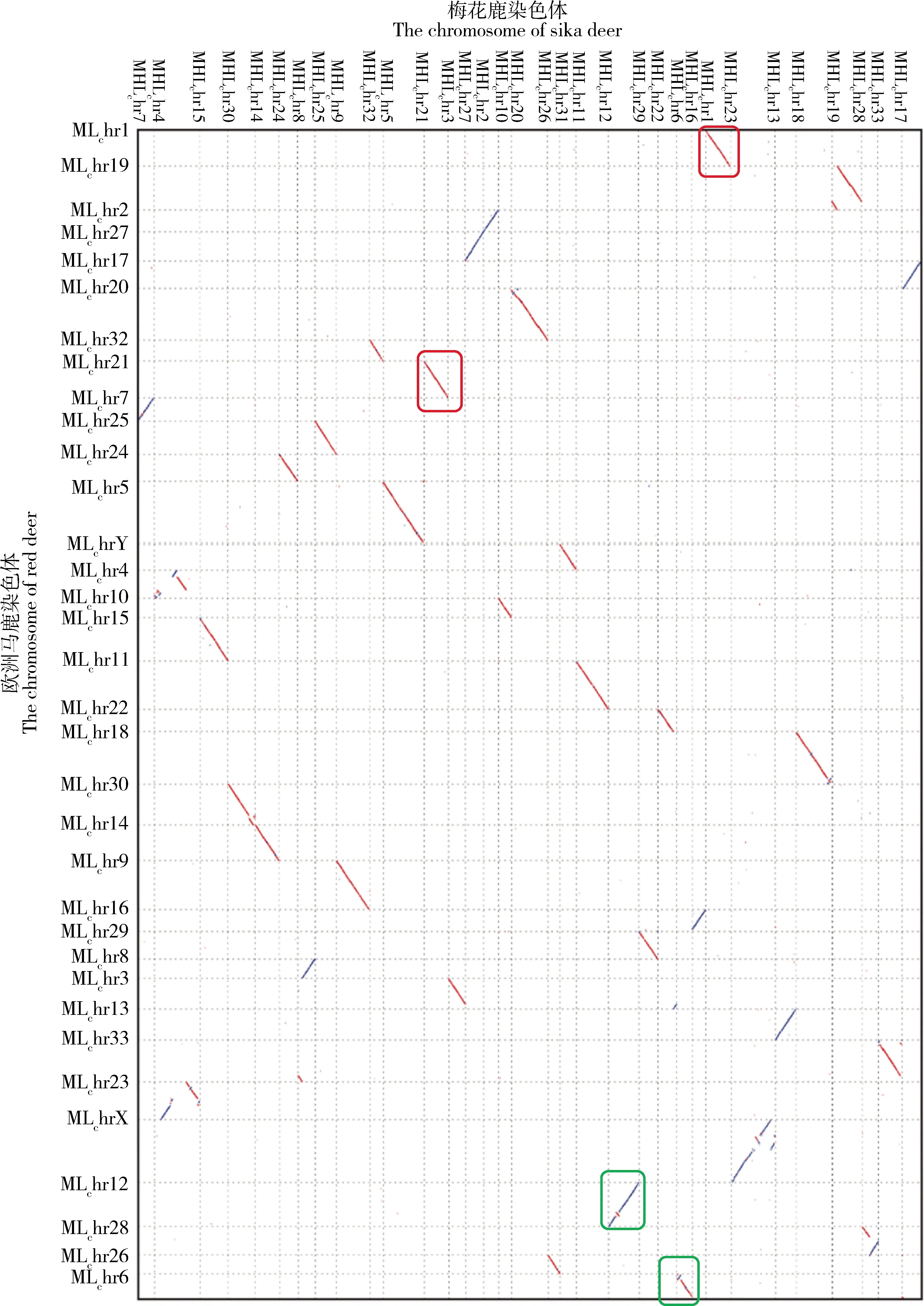
染色体共线性图谱显示了两种鹿基因组染色体的同源性。红色线代表两条染色体之间是正向比对,蓝色线代表两条染色体之间是反向比对。图中横坐标代表梅花鹿染色体,纵坐标代表欧洲马鹿染色体
本研究以梅花鹿的基因组作为参考基因组,将梅花鹿与欧洲马鹿染色体序列的比对结果进行统计分析,筛选染色体间同源区相似性为95%及以上,且对同源区长度大于1 kb的数据进行统计。以梅花鹿基因组为标准,统计欧洲马鹿对应染色体的比对情况发现,梅花鹿的33条染色体与欧洲马鹿对应的染色体均有较好的同源性,且有27条染色体的同源性在95%以上,其中5、10、11、20、21、25、26、27号染色体比对率在99%以上,表明梅花鹿与欧洲马鹿对应染色体有较高的同源性(表2)。
2.1.2 梅花鹿基因组X染色体的确定 本试验在梅花鹿基因组33条染色体中,一直没有找到与X染色体对应的染色体。结合两个物种的基因组序列对比结果发现,梅花鹿的23号染色体与欧洲马鹿的23号染色体比对率极低,仅有0.4%(表2)。为了进一步验证梅花鹿的23号染色体是否对应欧洲马鹿X染色体,将梅花鹿33条染色体与欧洲马鹿的两条性染色体(X和Y染色体)分别进行比对的统计结果显示,梅花鹿的23号染色体与欧洲马鹿的X染色体比对同源性最高,为81 051 312 bp(表3)。基于以上两方面数据支持认为,梅花鹿23号染色体对应欧洲马鹿基因组中的X染色体。
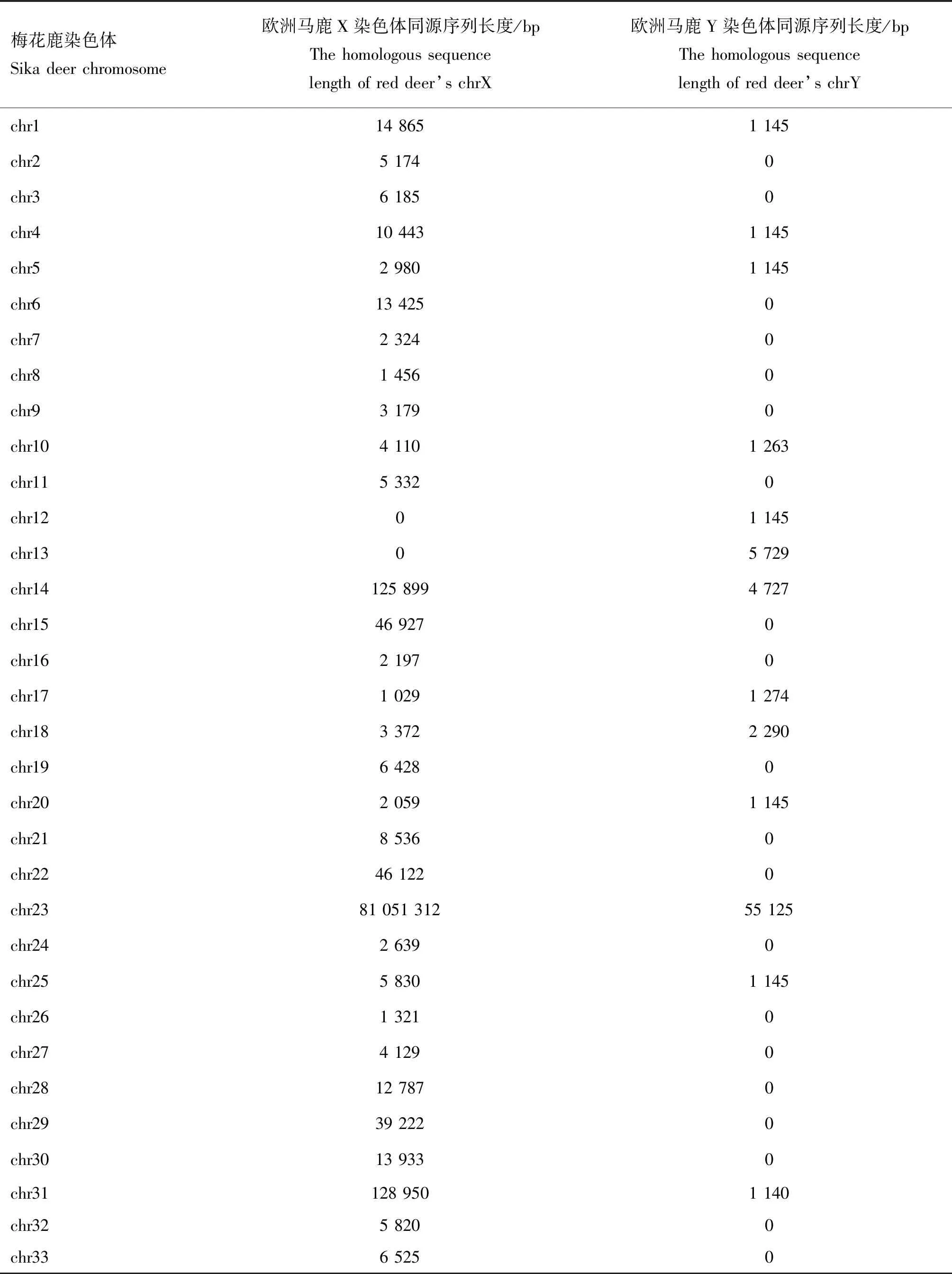
表3 梅花鹿基因组与欧洲马鹿X、Y染色体序列比对统计
2.1.3 基因组的结构变异 从梅花鹿与欧洲马鹿的基因组序列进行比对得到的染色体共线性图中发现了梅花鹿与欧洲马鹿各条染色体间序列的同源关系,除了像1号、21号染色体有很好的同源性,在图中呈现平滑连续的红色斜线和蓝色斜线外(图1红框);还有像6号、12号染色体同源性并不好,在图中表现出有一段断开的反向序列,即连续红色斜线和连续蓝线中出现一段断开的蓝色斜线或者红色斜线(图1绿框)。这种染色体间序列中断开的反向序列是基因组比对过程中染色体发生的一种重排现象,被称为染色体倒位,属于基因组结构变异的一种[29]。
将梅花鹿与欧洲马鹿基因组序列的比对结果中发现的基因组结构变异进行统计发现,梅花鹿的33条 染色体上均有一定数量的倒位,共计倒位数目为37 847个。4、23、28号染色体上的倒位数目较多,超过了4 000个,其中23号染色体上倒位最多,为5 238个;而3、10、16、17、19、21、25号染色体上的倒位数目较少,低于100个(表4)。37 847个倒位片段中,片段长度在1~5 kb的倒位数目最多,共有25 281个;倒位片段长度>50 kb的大片段倒位共有15个(图2)。
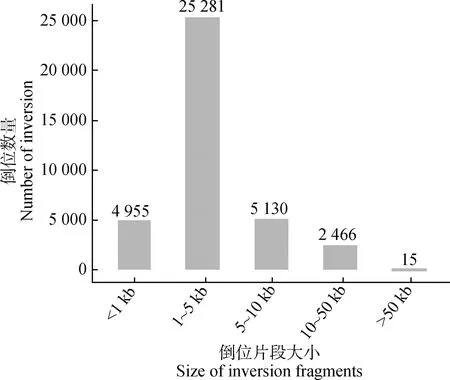
图2 梅花鹿基因组倒位片段大小统计
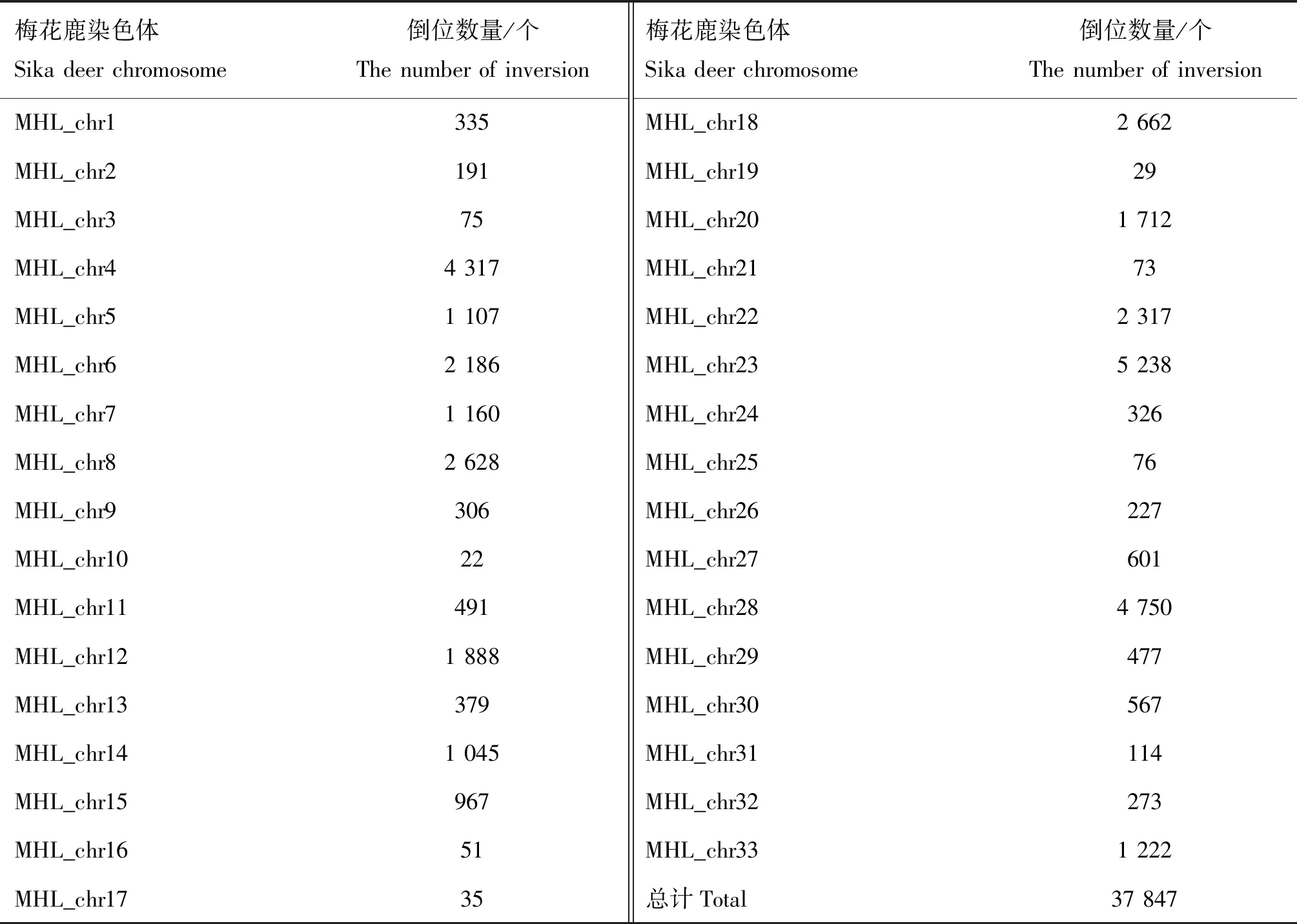
表4 梅花鹿各条染色体倒位数量
2.2 梅花鹿与欧洲马鹿染色体倒位涉及基因的功能富集分析
通过将梅花鹿与欧洲马鹿的全基因组序列进行比对,得到两物种染色体间序列的共线性及对应染色体上基因组的结构变异,这些基因组结构变异主要以染色体上序列片段倒位为主。将因大片段倒位而受到影响的基因大致分为两类:被倒位断点截断的基因和在倒位内部的基因。对这两类基因分别进行了功能富集分析。被倒位截断的基因GO功能富集分析主要涉及的生物学过程及分子功能主要有:嗅觉中化学刺激的检测(GO:0050911);嗅觉感觉(GO:0007608);嗅觉感受器活动(GO:0004984);信号传感器活动(GO:0004871);信号传导(GO:0007165)。KEGG功能富集分析主要涉及的信号通路是嗅觉传导(hsa04740)(图3,表5)。未被倒位断点截断的基因GO功能富集分析主要涉及的生物学过程及分子功能主要有: G蛋白偶联受体信号通路(GO:0007186);嗅觉中化学刺激的检测(GO:0050911;嗅觉感觉(GO:0007608);G蛋白偶联受体活性(GO:0004930);嗅觉感受器活动(GO:0004984)。KEGG功能富集涉及的主要信号通路是嗅觉传导(hsa04740)(图4,表6)。富集在通路的基因主要是嗅觉受体家族的成员,其中O10 J1除了与嗅觉信号通路有关,还可能在受精过程中参与趋化[30]。
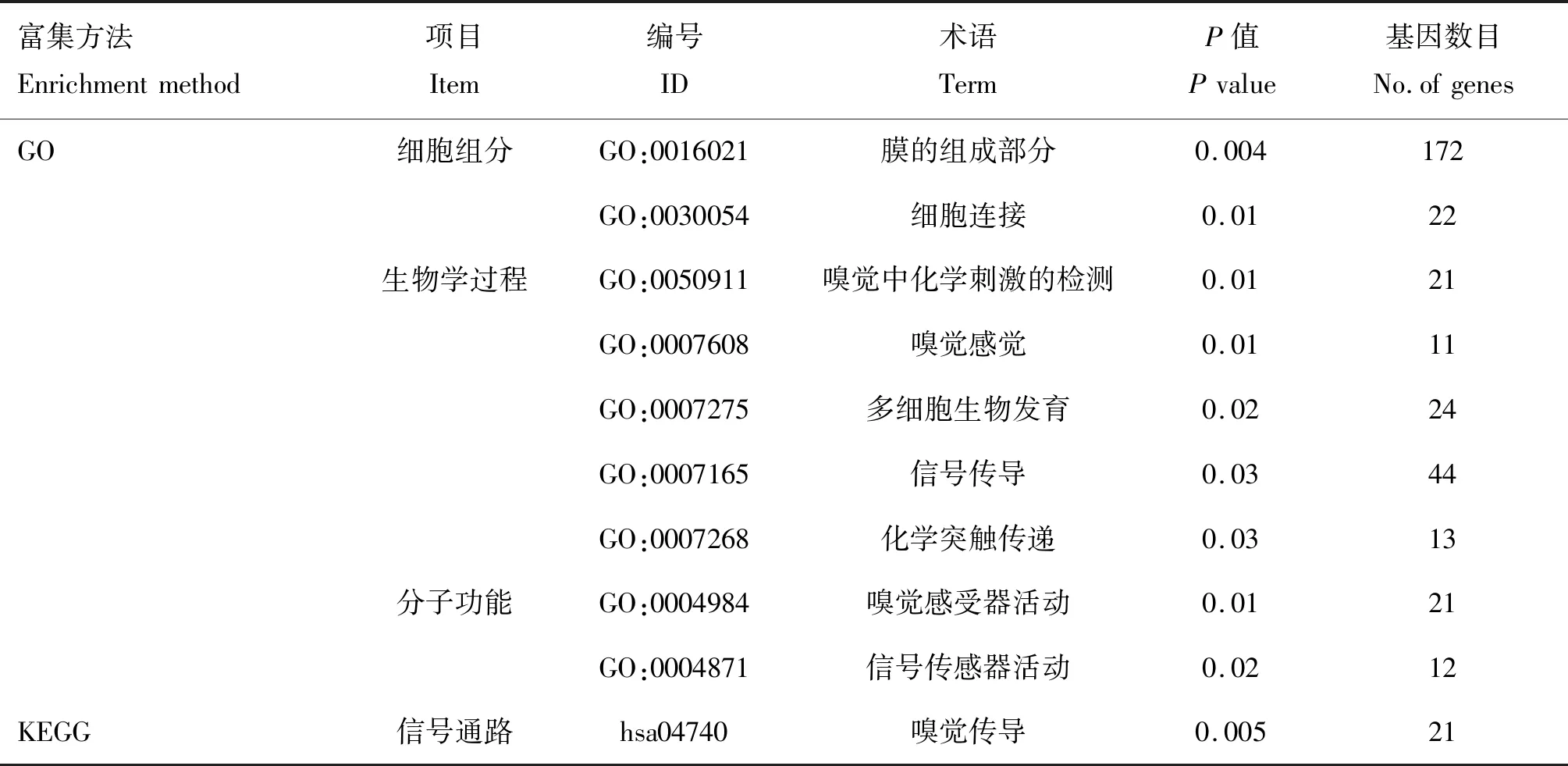
表5 被倒位截断的基因GO和KEGG主要的富集统计
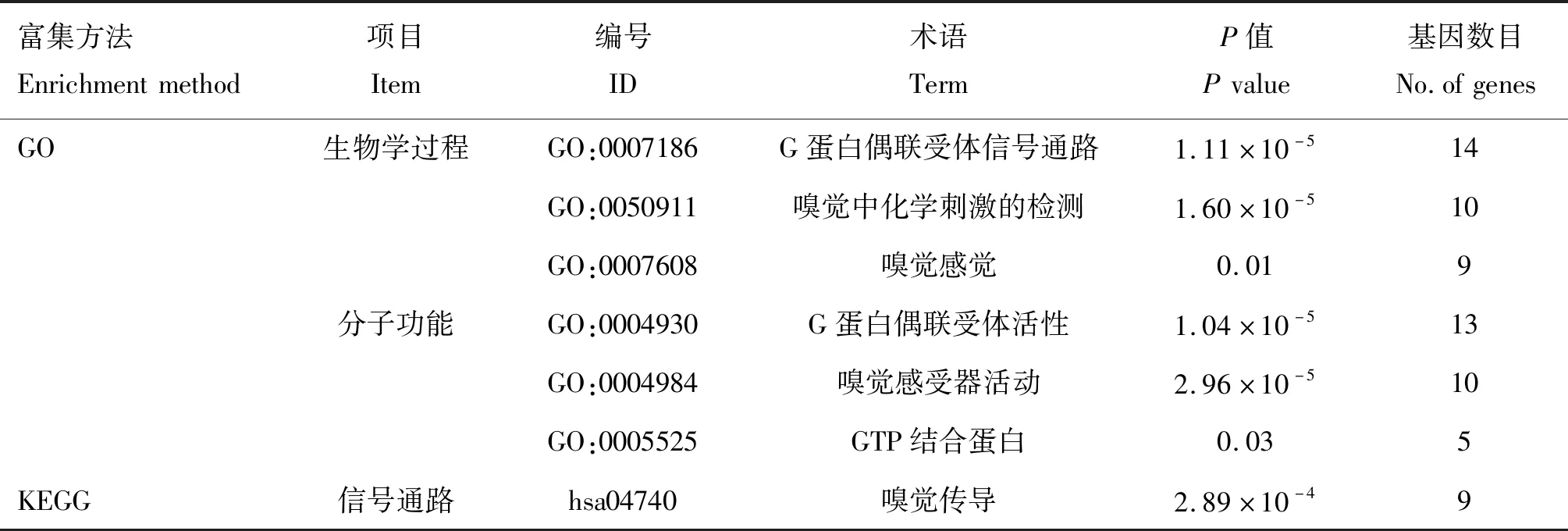
表6 倒位内部未被截断的基因GO和KEGG主要的富集统计
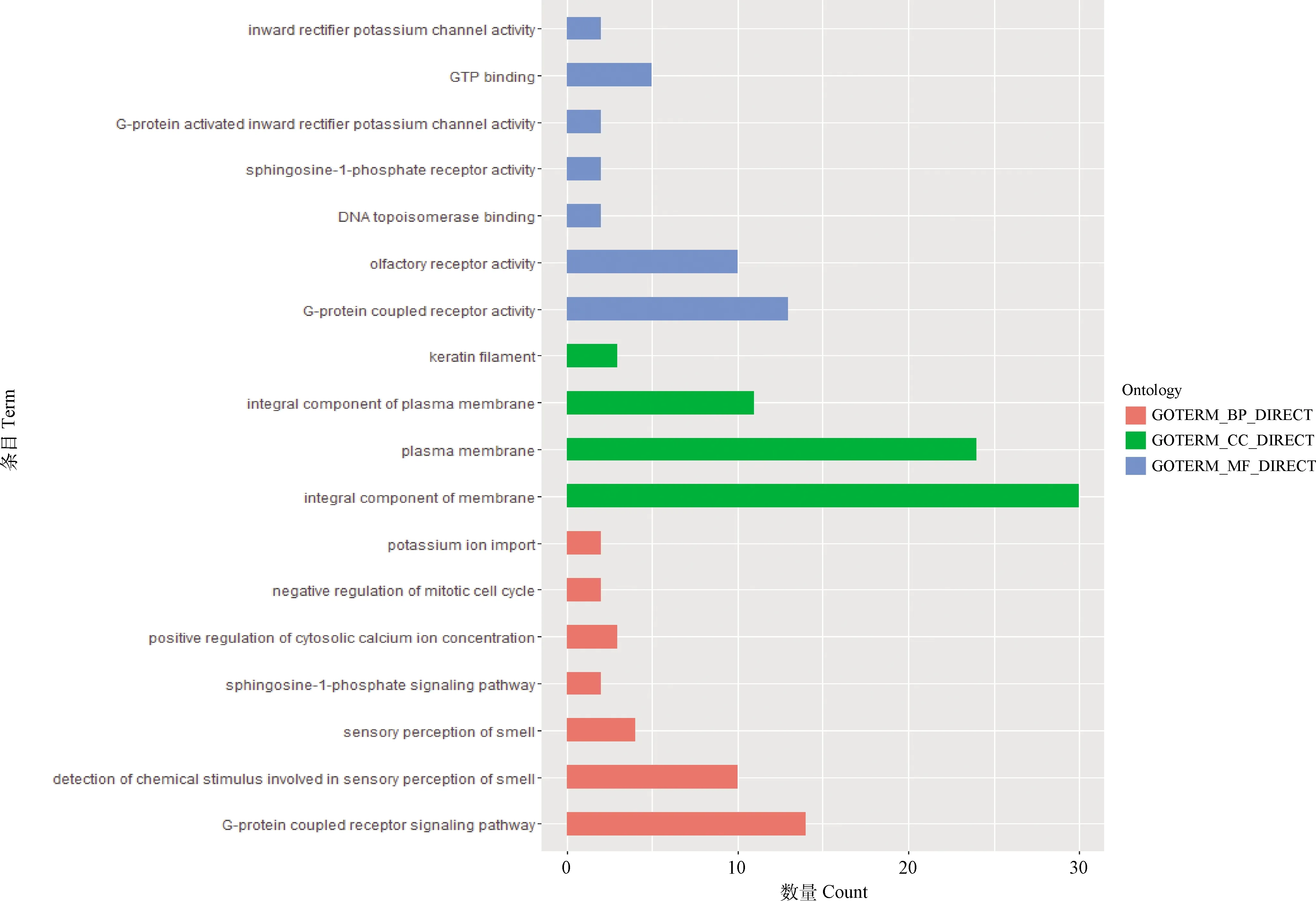
图4 在倒位内部未被截断的基因功能富集
值得注意的是,被倒位断点截断的基因和未被倒位断点截断的两类基因主要的功能富集分析基本一致,均包括:嗅觉中化学刺激的检测(GO:0050911);嗅觉感觉(GO:0007608);嗅觉感受器活动(GO:0004984);信号传感器活动(GO:0004871)以及嗅觉传导(hsa04740)信号通路。
2.3 梅花鹿与欧洲马鹿基因共线性的结果
2.3.1 梅花鹿与欧洲马鹿直系同源区鉴别 为了获得梅花鹿和欧洲马鹿两个基因组间的直系同源区,将两个物种的全部基因序列进行共线性分析,在比对结果中共检测到梅花鹿和欧洲马鹿基因组间79个同源区段,占梅花鹿基因组序列长度的95.54%。由于组装的欧洲马鹿基因组空位共有1.5 Gb,这些同源区段只占欧洲马鹿基因组长度的41.89%;平均每个同源区段含有161个基因对,单个同源区段最多涵盖777个基因对,覆盖基因组长度104 886 123 bp。单个同源区段最少涵盖14个基因对,覆盖基因组长度3 494 220 bp。同源区段平均长度30 063 070 bp,最大的同源共线性区域在5号染色体上,共线性区段内同源基因的平均Ks值0.096(表7)。

表7 梅花鹿与欧洲马鹿直系同源区统计
2.3.2 梅花鹿与欧洲马鹿进化分析 在基因共线性分析中,共检测到12 629个直系同源基因(表7),为了估计梅花鹿和欧洲马鹿两个物种的分化时间,计算了12 629个直系同源基因的同义突变率Ks为0.007,每位点每年中性突变率为1.1×10-8,根据公式:t=Ks/2 μ(其中,t为物种分化时间,Ks为同义突变率,μ为中性突变率)计算得出梅花鹿和欧洲马鹿分化时间大概是在0.318 MYA。
基于两个物种基因共线性结果分析,计算出检测的同源基因对的Ka/Ks值,可以发现,同源基因对的Ka/Ks值主要分布在0~1之间,且最大分布是在0.5~0.75这个范围内(图5)。表明梅花鹿与欧洲马鹿的大部分基因在进化过程中受到纯化选择。

图5 同源基因对Ka/Ks值的分布
3 讨 论
目前,有关梅花鹿与马鹿演化关系的研究报道仅限于细胞遗传学和系统发生学的研究,并没有比较基因组学的相关研究。Wang等[5]通过对芥属的黑芥和芥菜的B亚基因组进行基因共线性的分析,找到了两个物种间染色体上的结构变异,为芥属植物的起源进化提供了理论基础。而本研究以梅花鹿和欧洲马鹿两个鹿属动物的基因组为基础,通过对两个物种基因组的同源性以及基因序列的共线性分析,同样也发现了两个物种基因组的结构变异以及染色体上序列的共线性关系,证明从基因组层面研究梅花鹿与欧洲马鹿起源进化关系是可行的,而且类似的研究在鸟类进化起源研究中也有涉及[31]。
本研究选取两个不同性别物种基因组进行分析,就不同性染色体带来的基因差异是否会对研究结果产生影响进行分析,首先,在研究前期已经将欧洲马鹿的Y染色体与梅花鹿的各条染色体进行了比对,发现同源区很少,且同源片段很短。因此,在基因组比对分析中,欧洲马鹿Y染色体的存在基本不会对常染色体以及X染色体的比对分析产生影响。其次,对基因共线性的分析发现,由于Y染色体上基因数相较于本研究发现的12 629个同源基因来说很少,因此,缺少Y染色体的基因分析对研究结果产生的影响基本上是可以忽略不计的。综上所述,本研究中因为不同性染色体带来的差异基本不会对研究结果产生影响。
本研究利用基因共线性结果分析得到直系同源基因的同义突变率Ks,计算出梅花鹿与欧洲马鹿的分化时间为0.318 MYA。这个结果与Kuwayama和Ozawa[32]基于细胞色素b(Cytb)进行系统发育分析得到的欧洲马鹿与梅花鹿之间的分化时间为0.80 MYA稍有差距。这可能是因为线粒体基因组代表的是母系遗传,在研究物种的起源分化时,物种的全基因组分析会比单独的母系遗传研究更加系统,更加全面。也可能是两个物种基因组测序或者组装质量的问题,但是基于两个物种染色体共线性和基因共线性的同源关系来看,二者的分化时间应该是极为接近的。
染色体倒位(chromosomal inversion)是常见的基因组结构变异,常常导致基因排列顺序的改变,能够抑制基因重组,在物种进化过程中扮演了重要的作用[33]。Yang等[7]通过对陆地棉At和Dt两个亚基因组非共线性区域的基因富集到几种代谢途径,包括维生素B6代谢和糖胺聚糖降解,这些合成代谢促进胚性愈伤组织发育。本研究对梅花鹿与欧洲马鹿染色体上倒位基因进行GO和KEGG功能富集到的主要的生物学过程及分子功能有:嗅觉中化学刺激的检测(GO:0050911)、嗅觉感觉(GO:0007608)、嗅觉感受器活动(GO:0004984)、信号传感器活动(GO:0004871)以及嗅觉传导(hsa04740)信号通路。通过功能富集结果推测,梅花鹿与欧洲马鹿在进化过程中嗅觉表型可能受到染色体倒位事件的影响,从而发生了不同的选择性进化。Fan等[34]对大熊猫、狗和猫的染色体倒位基因进行功能富集,同样发现富集到的基因功能主要与嗅觉感觉表型相关,而对于梅花鹿与欧洲马鹿嗅觉表型的差异暂时没有相关内容的研究报道,但是因为不同的生长环境和采食偏好性[35],梅花鹿与欧洲马鹿在嗅觉表型上应该会发生不同的选择性进化。就像驯鹿为了可以在寒冷的环境下生存,嗅觉进化的非常发达,以至于可以发现埋在厚厚积雪下的食物[36]。关于导致梅花鹿与欧洲马鹿嗅觉表型差异的主效基因的研究将会是我们后续的研究内容。
4 结 论
本研究基于比较基因组学的方法,以梅花鹿与欧洲马鹿两个物种染色体水平基因组为研究对象进行分析,初步获得了两个物种基因组的同源关系以及在进化过程中发生的染色体倒位现象,估算出梅花鹿与欧洲马鹿的分化时间为0.318 MYA,并借助功能富集分析将染色体倒位现象与嗅觉表型的差异联系起来,为鹿属动物染色体进化研究提供更多的理论基础。
——以多重共线性内容为例

