探索功能性脑网络差异的可视分析系统设计与实现
张振兴,吴亚东,廖 竞,王 娇
(1.西南科技大学 计算机科学与技术学院,四川 绵阳 621010; 2.四川轻化工大学 计算机科学与工程学院,四川 自贡 643000;3.西南科技大学 信息工程学院,四川 绵阳 621010)
0 引言
人脑的功能和结构极其复杂,理解大脑的运转机制,是人类当前面临的巨大挑战之一。近年来,研究人员将fMRI(功能性核磁共振成像)构建成功能性脑网络进行分析。功能性脑网络是大脑连接的一种简单表示,大脑可以按功能分为不同的脑区,这些脑区被定义为功能性脑网络中的节点;脑区之间存在功能上的关联,这种功能连接被定义为功能性脑网络中的边。对于阿尔默茨海默症等精神疾病而言,其病理基础不只涉及单一的脑区,而是大脑多个区域间的协同工作出现问题。因此将复杂网络理论应用到大脑研究中,可以从全脑不同脑区间的交互去分析患者大脑机制,从网络的角度来研究其大脑的运转[1,2]。
在神经科学领域,对功能性脑网络常用的研究思路是基于特定假设驱动的群组差异性分析。研究人员对患有疾病的被试者与健康状态下的被试者进行对照,揭示患者和健康人群之间的功能性连接差异[3]和脑网络特征参数差异[4],探索其在疾病状态下的变化规律,寻找客观的影像学指标以辅助临床诊断。此外,功能性脑网络具有模块化结构[5],这种模块化结构是根据脑区间的协同工作,将脑网络分成不同的具有自相似性的若干个模块,具有不同功能的模块之间通过有效的功能整合来维持正常的人脑功能。
因此,在神经科学领域中不同状态间功能性脑网络的模块化结构差异、功能性连接差异和网络特征参数差异是当前的热点问题,研究人员常用统计模型和深度学习方法对上述内容进行分析,然而这些方法只能寻找支持某种驱动假设的证据。并且现有的可视化工具也主要用于传达科学发现,功能较为单一。为解决以上问题,我们开发了用于fMRI的交互式可视分析系统——BrainDVis。BrainDVis将降维视图、网络特征参数分析视图、GIB布局视图和邻接矩阵视图相关联,研究人员在没有明确的解决方案时依赖多种交互实现自动分析,结合可视化视图寻找差异,由系统产生的视觉反馈来制定早期的理论方案或拒绝假设。
1 相关研究
由于功能性脑网络的高维度性,神经学家需要一个有效的可视分析工具用于探索其数据。对功能性脑网络进行可视化研究是为了观察脑区间的连通性和相互作用,进而用于比较不同状态下被试者功能性连接的差异,探究疾病带来的功能性连接变化。Xia等人开发了专用于研究功能性脑网络的可视分析工具BrainNet Viewer[6],其使用球棍模型展示功能性脑网络脑区间的功能性连接,保留了脑区在大脑空间的真实位置,提供基本的交互功能。但是这种可视化布局会导致混乱和遮挡,影响视觉效果。时磊等人使用NodeTrix可视化方法展示功能性脑网络[7],这是一种结合节点链接图和矩阵的混合可视化方法,该方法极大的降低了功能性脑网络的视觉复杂性。
但是要对功能性脑网络进行更深入的探索,还需要对其模块化结构进行详细的分析。复杂网络有一个共同的性质——模块化结构,模块化结构也称为社团结构。社团是网络中节点的集合,一个复杂网络是由若干社团构成的。功能性脑网络是一种典型的复杂网络,研究其模块化结构对分析各脑区功能之间的协作关系有非常重要的意义。Connectome Visualization Utility(CVU)是用于探索功能性脑网络模块化结构的工具[8]。但是,CVU仅限于在单个规模/级别上识别和可视化模块化结构,没有对模块间的协作进行分析。总体来说当前关于功能性脑网络模块化结构的可视分析系统研究进展甚微。
基于此,本文专注于解决目前没有可视化系统可以同时对功能性脑网络进行模块化差异分析和功能性连接差异分析的问题,开发了一个可视分析系统——BrainDVis。
2 可视化任务描述与系统架构
2.1 可视化任务
通过与神经学家的交流,总结出BrainDVis系统的设计思路,具体如下。
T1:不同状态的功能性脑网络是否存在较大的差异?因此对多个被试间的功能性脑网络差异要有整体直观的感知。
T2: 疾病是否会让功能性脑网络的网络特征参数发生改变?变化规律是怎样的?因此要对不同状态下功能性脑网络的网络特征参数进行分析与对比。
T3:疾病是否会导致功能性脑网络的模块化结构发生改变?因此需要一个直观的方式对功能性脑网络的模块化结构进行展示,并需要一种准确的方法对变化程度进行评估。
T4:某种疾病给功能性连接带来什么变化?需要一种直观的方式来展现功能性连接变化最大的是哪几个脑区。
T5:为了更好地结合神经学家的先验知识,系统应该具有多视图协同交互能力支持他们自由探索。
2.2 系统整体架构
本文的探索功能性脑网络差异分析框架如图1所示。系统采用B/S架构,使用Vue + D3 +Echarts作为系统框架,以Mysql作为后台数据库。共有3大模块,分别是可视分析视图模块、数据的处理与计算模块以及存储模块。
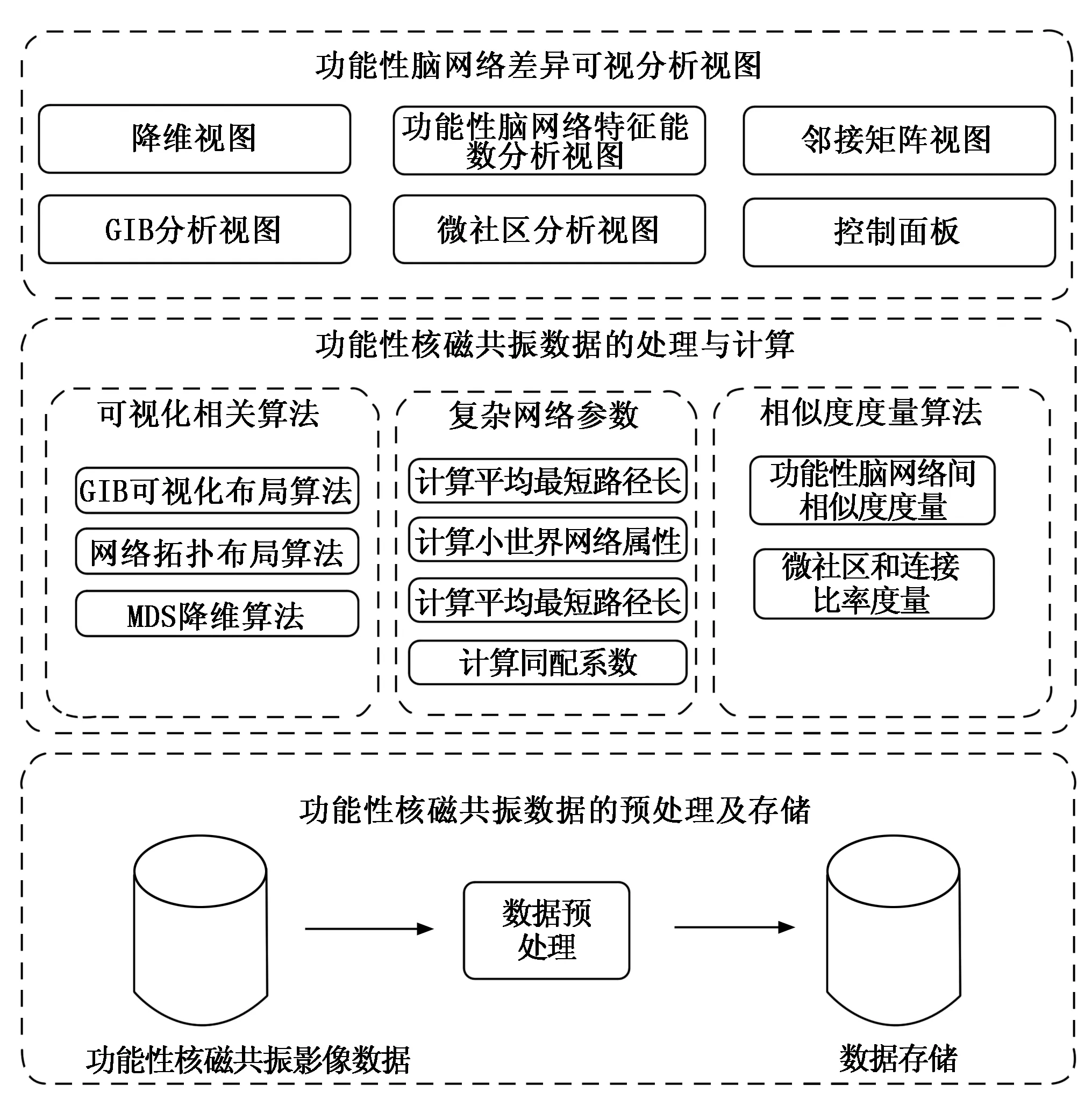
图1 探索功能性脑网络差异分析框架
数据的预处理及存储模块是将功能性核磁共振数据转化为计算机可以识别的数值型数据,再存储在数据库中。数据的处理与计算是将表示功能性脑网络的矩阵数据转换为结构化的数据,便于可视化算法、计算复杂网络参数和相似度度量算法的输入。可视化模块将数据映射为可视化视图,展示被试间的多方面差异,并提供点击、框选等交互操作。
2.3 数据预处理
将fMRI图像构建成功能性脑网络,分为以下3个步骤[9],如图2所示。

图2 数据处理流程图
网络构建详细步骤描述如下:
1)数据预处理:采用SPM(statistical parametric mapping)对影像数据进行时间校正、空间配准、标准化、平滑等预处理,通过滤波处理最后得到全脑时间序列数据。预处理后的fMRI图像都会产生数十万个体素点,对这些体素点按标准坐标归类到具体的某一脑区。
2)相关分析:以标准脑模板分割出来的脑区为单位,计算脑区内体素对应的时间序列的平均值,用来表示该脑区的时间序列和脑活动功能属性。脑区时间序列间的同步性表示脑区之间的功能连接关系,计算各脑区的相关性的方法是Pearson相关系数,公式为:
3)功能性脑网络的构建:相关系数用来判断脑功能网络的两个脑区节点间是否存在连接边。如果两个脑区间的相关值大于阈值时,在对应的节点间建立连接边;反之则不建立连接边。
4)平均功能性脑网络的计算:本文需要用到被试组的平均功能性脑网络。计算每个被试组内所有被试90个脑区的每个脑区的相关系数求平均值,继而得到每个被试组的均值相关矩阵,通过该矩阵可以构建平均功能性脑网络。
3 可视化系统实现
BrainDVis可视分析系统包含以下6个部分:(A)降维视图;(B)控制面板;(C)功能性脑网络特征参数分析视图;(D)邻接矩阵视图;(E)group-in-a-box分析视图;(F)微社区分析视图。
3.1 降维视图
由于功能性脑网络的高维复杂性,研究人员难以找到一种直观准确的方法比较大量被试间功能性脑网络的差异。他们常用的方法是将所有被试按照实验任务划分为两个被试组,对两组的平均功能性脑网络进行对比,寻找差异。但是被试组的平均脑网络会掩盖个体的特殊性,让我们忽略其中潜在的发现。
MDS降维算法[10]可以将高维度的功能性脑网络映射到低维空间中,使高维空间结构在低维空间得以保持。差异较大的功能性脑网络在低维空间下的映射点距离也较大,反之较小,即可以通过降维将被试之间的脑网络差异投射到低维空间。本文结合MDS降维算法,提出了一种功能性脑网络相似度度量方法。首先将所有的功能性脑网络矩阵A1,A2,…,An转换为向量X1,X2,…,Xn,然后计算向量两两之间的欧式距离,得出一个距离矩阵D,通过MDS算法计算出功能性脑网络在二维平面上的坐标。
MDS算法流程:
1)计算原始空间中数据点的距离矩阵。
2)计算内积矩阵B。
3)对矩阵B进行特征值分解,获得特征值矩阵Λ和特征向量矩阵V。


图3 降维视图
如图3所示,MDS算法计算出所有被试个体在二维空间上的坐标后,以点的形式映射在二维平面上,每个节点都代表一个被试,节点的颜色代表被试所属的组别,图中节点间的距离代表被试间功能性脑网络差异的大小。如果被试间具有组内差异小和组间差异大的特征,说明组内被试个体的功能性脑网络具有相似性,而组间被试个体的差异性较大。可以推断疾病会导致功能性脑网络发生改变。
3.2 功能性脑网络特征参数分析视图
研究人员利用降维视图从整体上对比被试间功能性脑网络的差异后,还需要挖掘产生差异的潜在因素。复杂网络理论提供的网络特征参数可以从不同角度对网络的拓扑属性进行分析。BrainDVis系统对功能性脑网络的特征参数进行了对比分析,使用到的特征参数包括:
1)小世界网络(δ):研究表明大脑具有“小世界”拓扑结构[4]。
2)平均最短路径(Lp):平均最短路径越短,在一定程度上可以表明整个网络之间信息传递的效率越高[11]。
3)平均聚类系数(Cp):整个网络的聚类系数定义为各个节点的聚类系数的均值,它可以描述网络中节点之间的连接紧密程度[11]。
4)同配系数(Ap):网络中连接度大的结点总是倾向于与连接度大的结点相连,那么称该网络为同配网络。
为了便于挖掘不同状态下被试个体功能性脑网络特征参数的异同,我们设计了良好的交互功能。如图4(a)所示,点击控制面板的brush按钮,降维视图中出现一个随鼠标操作而改变大小和位置的选择框。当选择框选中降维视图中一个或多个节点时,可以在数据表格中看到所选被试的特征参数和基本信息。
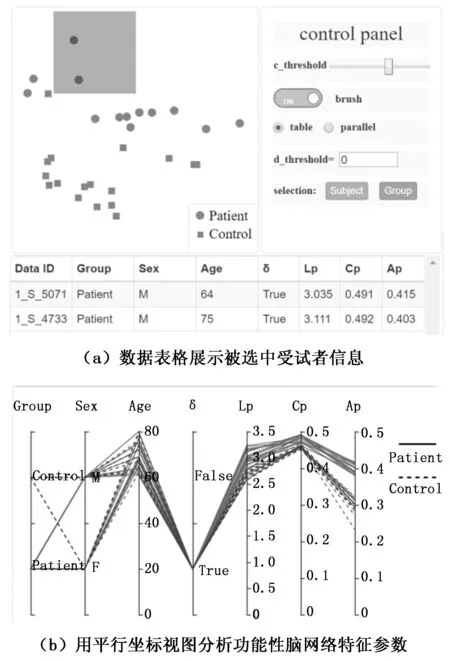
图4 功能性脑网络参数分析视图
在操作面板点击parallel后,数据表格转换为平行坐标视图。和上面的交互方法一致,在降维视图中选择感兴趣的被试,所选被试的数据将被加载到平行坐标图中。如图4(b)所示,每一个被试在平行坐标图中用一条折线所表示,折线颜色代表所属组别。用户根据分析需要,对维度轴中的属性值进行刷选,选择感兴趣的数据区域,对特征参数的差异做出进一步探究。在平行坐标上完成各维度轴的刷选后,系统会统计该数据区域中各组被试的数量。
3.3 功能性脑网络模块化分析
系统从模块划分和微社区两个层面上帮助研究人员对功能性脑网络模块化结构进行探索。
3.3.1 模块化结构分析视图
为解决节点和边缘数量过多时带来的视觉混乱问题,系统采用了Chaturvedi等人提出的GIB(group-in-a-box)可视化布局[12],GIB布局算法实现流程:
1)完成社区划分后,先使用treemap空间填充技术[13]为每个模块绘制一个单独的区域框,用于显示各个模块,矩形的面积由该模块内包含的节点数量来确定。
2)然后在每个矩形框内,使用力导向布局算法对模块内部节点进行布局;
3)添加图中的点和边。
如图5所示,利用Louvain算法[14]将功能性脑网络划分为多个模块,通过GIB布局将每个模块内的脑区节点放在同一个矩形框内,不同模块具有不同的颜色。这种布局方式保持了模块间的视觉距离,并且避免了由于脑区节点和功能连接数量过多而隐藏每个模块内部的网络结构,便于研究人员观察功能性脑网络每个模块内部的网络结构。

图5 基于GIB布局的模块划分视图
3.3.2 微社区分析视图
为了进一步探究疾病带给功能性脑网络的模块化结构的变化,本文提出了“微社区”和“连接比率”的概念,对不同状态下模块化结构的差异进行评估。
定义1 (微社区):对不同的脑网络运用同种模块划分算法,若某些脑区节点的组合同时出现在各个被试脑网络的某个模块中,那么我们把这些脑区节点的集合定义为微社区。如G1表示的节点组合同时出现在健康组模块二和抑郁症组模块二中,或G2表示的节点组合同时出现在健康组模块二和抑郁症组模块六中,我们定义G1和G2为微社区。
“连接比率”用于分析微社区之间的关系,连接比率的数学定义为:
Cij代表微社区i与微社区j之间所存在的实际边数。Mij为微社区i与微社区j之间的理论最大连边数。连接比率反映了两个微社区之间的紧密程度,3个微社区N1、N2、N3,如果存在I12>I13,即N1与N2的连接比率高于N1与N3的连接比率,因此我们认为N1与N2之间的关系比N1与N3更为紧密。
我们可以通过微社区和连接比率的概念间接分析各被试组模块化结构间的差异。载入多组被试组数据后,系统将计算出所有的微社区,并在列表中显示,如图6所示。当点击列表中的微社区后,被选中的微社区将被计算两两间的连接比率。节点代表所选微社区,边缘的厚度代表了微社区间连接比率的强弱。
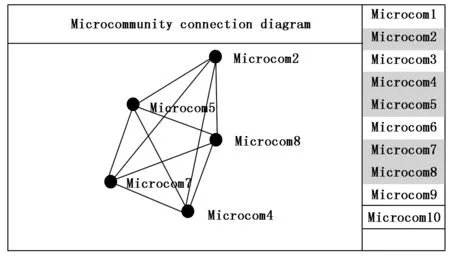
图6 微社区分析视图
3.4 邻接矩阵视图
邻接矩阵视图用于描述脑区之间如何相互关联,用于快速评估功能性脑网络的连接情况。如图7所示,邻接矩阵表示90个脑区之间的功能性连接情况。矩阵中的(i,j)位置表示脑区i与脑区j的功能性连接,第i行/第j列所对应元素如果具有颜色映射,则代表第i个脑区和第j个脑区之间具有功能性连接,且连接强度由颜色编码。

图7 邻接矩阵视图
3.5 功能性连接差异分析视图
大脑功能性连接用于衡量脑区之间的相关性。如果两个脑区之间的功能性连接系数越高,那么这两个脑区间的相关性也就越高。神经学家对特定原因造成的功能性连接变化非常感兴趣,比如随着年龄的增长或身体损伤,脑功能性连接是如何变化的。系统使用了GIB视图分析脑功能性连接的差异/变化,推测由疾病导致的功能性脑网络交互异常。
此时在GIB视图中关注的是功能性连接差异,当分析健康状态和疾病状态下功能连接差异时,GIB视图中的模块结构是疾病状态下的,节点间边的系数是疾病状态和健康状态下对应边系数的差值。如图8所示,如果边为红色,说明疾病状态较正常状态,功能性连接得到了增强。系统设置了阈值来寻找两者之间功能连接变化最显著的区域,即受疾病影响最大的脑区。对threshold进行调节,步长为0.01,此时视图中只显示功能性连接系数差值大于threshold的边。

图8 被试间功能性脑连接差异视图
4 系统应用
4.1 数据描述
本文采用的实验数据均来自LONI Image Data Archive (IDA)。在实验1中,我们使用了30例被试的静息态功能磁共振数据(resting-state fMRI),数据分为两个类别,其中健康被试(HC1)为 15例,阿尔茨海默病被试(AD)为15例。实验2使用了30例被试的静息态功能磁共振数据,其中健康被试 (HC2) 为 15例,抑郁症被试(MDD)为15例。所有数据都在3.0T Philips Achieva 扫描仪采集获得。两组被试在性别、年龄上没有显著差异。两组被试详细的基本信息见表1。

表1 被试者基本信息
4.2 阿尔莫茨海默症患者与健康者的差异
研究人员通过BrainDVis系统寻找AD症患者和健康者(HC1)间功能性脑网络的差异,用于发现AD症对大脑产生的影响。首先,使用降维视图从整体上对大量被试间的功能性脑网络差异进行分析。如图9(a)所示,降维视图中红色节点倾向于视图上方,而绿色节点在视图下方,形成了较为显著的聚类结构,具有组内被试差异小,组间被试差异大的特征。根据这种现象,研究人员验证了AD症导致患者大脑发生改变的结论。
如图9(b)所示,红色折线代表AD组,蓝色折线代表HC1组。可以发现无论是AD组还是HC1组,参数都为True。说明无论是否患病,脑网络都具有小世界性,这与已有的研究结果一致[7]。还可以发现AD患者的Lp、Cp、Ap三个参数均高于HC1组,说明虽然AD患者的小世界属性没有改变,但其功能性脑网络的内部特征发生改变。
AD患者的功能性脑网络平均聚类系数和平均路径长度均显著高于正常人,这意味着AD患者远距离脑区之间的整合能力降低,信息传递能力下降,这可能是AD患者大脑反应迟钝的重要原因之一。我们的结果与He等对AD患者功能性脑网络的研究结果一致[11]。尽管相配系数均为正数,我们的结果显示AD患者的相配系数均高于HC1组的相配系数。这表明AD患者功能性脑网络中节点度较大节点间的耦合更加紧密,即大脑在病变的过程中,关键节点间的耦合变得比正常人更加紧密。
最后研究人员利用功能性连接差异分析视图寻找受AD症影响最大的脑区。如图9(c)所示,针对AD症被试组和HC1被试组的均值功能性连接差值,将阈值调高,发现左颞上回脑区与其他几个脑区连接依然存在且为红色,说明左颞上回脑区的功能性连接强度比HC1组有显著增强。颞上回脑区主要参与听觉的处理、语言接收和自我监控[15]。有研究指出AD患者功能连接的增强是认知功能的补偿性重新分配或者补充[16],研究人员推测AD患者颞上回功能连接的增强暗示了语言理解和自我监控功能的受损和补偿。
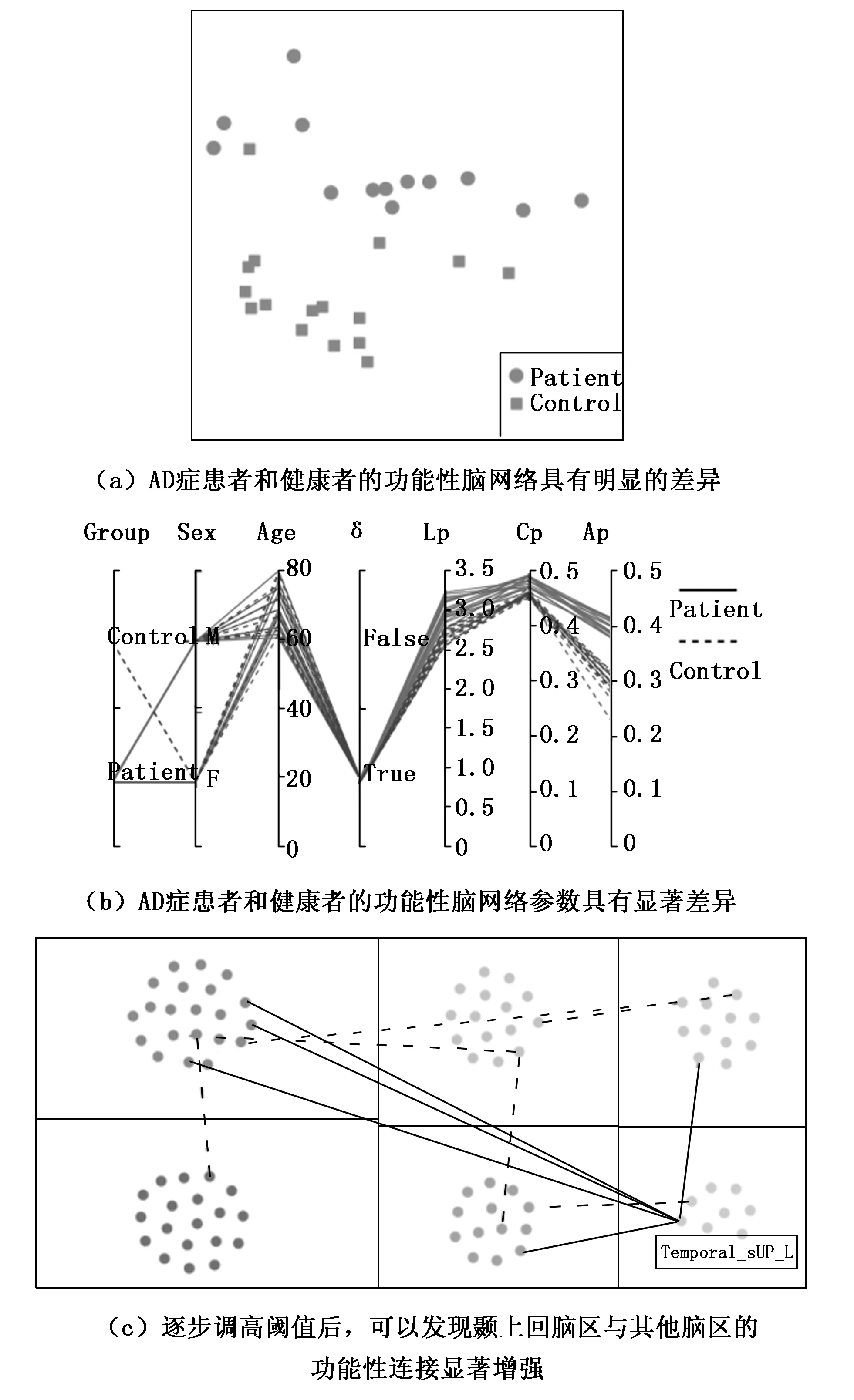
图9 AD症患者与健康者的差异性对比
4.3 抑郁症患者与正常人差异
研究人员通过分析抑郁症患者组(MDD)和健康组(HC2)组间的模块化差异,探究MDD症给功能性脑网络带来的变化。
点击操作面板中的Group键,系统计算两组的平均功能性脑网络后,将其载入到模块化分析视图中。如图10(a),(b)所示,抑郁症组和对照组的功能性脑网络都被分成了6个模块,由节点的颜色和数量可以发现,疾病使大脑模块划分发生了改变。
根据微社区概念,计算出两组平均脑网络包含10个微社区。基于抑郁症的患病机理和现有研究结论,研究人员选择了五个微社区进行分析,这些微社区包含的脑区涉及情感、意识和知觉等。如图10(c),(d),从对五个微社区的分析中,我们可以看到HC2组中微社区5和微社区7的连接比率显著高于MDD组相应的连接比率。微社区5主要包括的脑区有海马、杏仁核等,其涉及的功能主要是情感和记忆。微社区7的脑区主要牵涉到情感强化表达等。说明抑郁症患者这部分脑区间的交流减少,信息传导不畅,从而导致了患者在情感方面的障碍。

图10 MDD症患者组与HC2组模块分析展示
5 结束语
本文基于功能性核磁共振数据,结合多种算法与可视分析方法,设计并实现了探究不同状态间功能性脑网络差异的可视分析系统BrainDVis,解决了现有工具功能单一的问题。最后通过与神经学家合作的两个案例证明,BrainDVis可以满足研究人员的分析需求,这种综合性的分析工具提高了他们探索功能性脑网络的能力。随着人类对大脑研究的不断深入,这种基于交互式的可视分析工具会越来越重要。

