蓖麻LBD基因家族全基因组鉴定、进化和表达分析
段 强,何智彪,李国瑞,赵秀平,张 帅,韩雯毓,陈永胜
(1.内蒙古民族大学 蓖麻育种国家民委重点实验室,内蒙古通辽 028043;2.中国科学院长春光学精密机械与物理研究所应用光学国家重点实验室,吉林 长春 130000;3.内蒙古自治区高校蓖麻产业工程技术研究中心,内蒙古 通辽 028043;4.内蒙古自治区蓖麻育种重点实验室,内蒙古 通辽 028043;5.内蒙古自治区蓖麻产业协同创新中心,内蒙古 通辽 028043;6.蓖麻产业技术创新内蒙古自治区工程研究中心,内蒙古 通辽 028043;7.通辽市农牧科学研究所,内蒙古 通辽 028043)
转录因子(transcription factor,TF)是一类能够以序列特异性方式结合DNA 的蛋白质分子,通过调节转录水平指导基因表达,在各种生物学过程中发挥着重要的调控作用。LBD(lateral organ boundaries domain)是一类具有LOB(lateral organ boundaries)结构域的植物特有的转录因子基因家族[1],也称作AS2/LOB 基因,这类基因家族在植物的侧生组织原基中特异性表达。LBD 基因参与调控植物的生长发育与代谢过程并在其中起着重要作用,包括植物响应激素、营养元素、生物和非生物胁迫以及植物侧生器官发育、氮素营养的吸收代谢、次生代谢等[2]。
LBD 通常包含4 个高度保守的半胱氨酸基序CX2CX6CX3C(C 为保守的半胱氨酸残基、X 为不保守的氨基酸残基),这是LBD 家族最明显的特征[3]。同时,也发现一些LBD 含有甘氨酸组成的GAS 基序和类亮氨酸拉链基序LX6LX3LX6L。根据这些保守结构域,将LBD 转录因子家族分为两个亚家族Class 1 和Class 2[3]。当前已经明确了一些LBD 基因的功能,但仍有部分基因的功能尚待验证。在拟南芥中,AtLBD4 以及CHALFUN-JUNIOR 等[4]研究发现的AtLBD41 基因参与调控植物叶片发育。LBD 中的另一成员AtAS2 基因,在幼嫩花器官近轴面特异表达,从而调控植物的花器官发育[5-7]。SCHEIBLE 等[8]和RUBIN 等[9]研究发现,拟南芥中第2 类LBD 成员AtLBD37、AtLBD38、AtLBD39 以及ALBINSKY 等[10]在水稻中发现的OsLBD37 基因参与调控氮素代谢。ZENTELLA 等[11]研究发现,赤霉素抑制LBD 成员AtASL37 的表达;而IKEZAKI 等[12]研究发现,LBD的另一成员AtAS2 基因能够促进赤霉素合成。BERCKMANS 等[13]研究发现,拟南芥AtLBD33 和AtLBD18 基因能够通过激活E2Fa 的表达,促进侧根发育。杨树中与拟南芥AtLBD1 同源的LBD1 基因,能够与抑制物SRDX 结构域平移融合,降低直径增长,抑制韧皮部的发育,调控次生生长[14]。有研究发现,在拟南芥中,AtLBD16、AtLBD17、AtLBD18 和AtLBD29 是诱导愈伤组织初始体形成的关键调控因子,抑制LBD 基因表达会阻止愈伤组织的形成。
蓖麻(Ricinuscommunis)为大戟科(Euphorbiaceae)蓖麻属(Ricinus)一年生或多年生灌木。与大多数大戟科植物类似,蓖麻的适应能力特别强。2010年,蓖麻基因组草图的绘制使其成为大戟科第1 个完成基因组测序的物种[15];2021年,蓖麻基因组组装至染色体水平,为通过比较基因组学在染色体水平研究蓖麻耐胁迫的分子机制创造了条件[16]。本研究基于组装至染色体水平的蓖麻基因组对蓖麻LBD 基因家族进行全面鉴定,并在此基础上分析其基因结构、理化性质、进化关系及表达模式,旨在为解析蓖麻LBD基因家族功能奠定基础。
1 材料和方法
1.1 材料
绿蓖1 号、淄蓖5 号种子来自内蒙古通辽市农牧科学研究所,在实验室中种植于田园土∶蛭石∶营养土=1∶2∶1 的基质中。随机选择生长三叶期的蓖麻植株幼苗为试验材料,以喷施正常浓度除草剂(90 g/hm2)——25%砜嘧磺隆的2 种蓖麻群体为处理组;以未加除草剂处理,喷施等量清水的2 种蓖麻群体为对照组。每个处理随机取样4 株,采取每单株的叶片、茎段、根部分,将样品记为绿蓖1 号处理组(LB_T)、绿蓖1 号对照组(LB_C),淄蓖5 号处理组(ZB_T)、淄蓖5 号对照组(ZB_C),每组织部位设置4 次重复,分别命名为L1~L4、S1~S4、R1~R4。放入液氮冷冻,置-80 ℃保存。后续对处理组和对照组样品进行处理,包括总RNA 的提取、纯化,cDNA 文库构建,转录组测序及相关分析等。
1.2 数据来源
本研究中,蓖麻全基因组序列、蛋白质序列、编码序列与注释文件来自Oil Plant Database(http://oilplants.iflora.cn),拟南芥LBD 基因家族的基因与蛋白质序列获取于TAIR 数据库(Araport11,https://www.arabidopsis.org/),麻疯树获取于NCBI(RJC1_Hi-C,https://www.ncbi.nlm.nih.gov),玉米获取于NCBI(B73 RefGen_v4.0,https://www.ncbi.nlm.nih.gov),水稻获取于NCBI(Build 4.0,https://www.ncbi.nlm.nih.gov),番茄获取于NCBI(SL3.0,https://www.ncbi.nlm.nih.gov),烟草获取于NCBI(Ntab-TN90,https://www.ncbi.nlm.nih.gov),蓖麻的相关表达量数据来自Gene Expression Omnibus Database(https://www.ncbi.nlm.nih.gov)。
1.3 蓖麻LBD 基因家族成员鉴定及蛋白质理化性质分析
使用蓖麻蛋白质序列文件在本地建立数据库,以拟南芥全部的43 个LBD 基因家族成员序列作为query 序列进行本地BLASTP 比对(E-value≤1e-10)[17]。由Pfam 数据库(http://pfam.xfam.org/)获得LBD 基因家族典型结构域的隐马尔科夫模型(PF03195),以此为query 序列进行本地HMMER 比对[18]。将BLASTP 与HMMER 比对结果进行汇总,去除冗余序列。分别使用NCBI CDD 工具(https://www.ncbi.nlm.nih.gov/cdd/) 与EBI InterPro 工具(http://www.ebi.ac.uk/interpro/)对候选序列进行结构域检测[19-20],使用Clustal Omega 软件进行多重比对[21],使用Jalview 可视化比对结果[22],手动剔除结构域缺失与不完整的序列,对于同一基因存在多个转录本的情况,选择第1 个转录本作为代表序列,从而确定蓖麻LBD 基因家族的成员。通过ExPASy 的ProtParam 工具(https://web.expasy.org/protparam/)对已确定的蓖麻LBD 基因家族成员进行理化性质分析[23],同时使用Plant-mPLoc 进行蛋白质亚细胞定位[24]。
1.4 蓖麻LBD 基因结构、蛋白质保守基序分析
利用蓖麻基因组注释文件,通过编写的脚本提取LBD 基因家族成员的基因、内含子与外显子位点信息,并计算内含子与外显子长度。使用MEME 在线工具(http://meme-suite.org/tools/meme)对蓖麻LBD 蛋白进行保守基序分析[25],基序数目设置为10 个,通过编写的脚本提取保守基序的位点信息。蓖麻LBD 基因家族的结构、蛋白质保守基序均使用Evoview 在线工具进行可视化[26]。使用SWISS-MODEL在线工具对具有代表性的蓖麻LBD 蛋白进行同源三维建模[27]。
1.5 蓖麻LBD 基因家族染色体定位及复制分析
使用Circos 软件绘制基因在染色体上的分布图[28],从而确定在染色体上的位置。使用MCScanX 软件(http://chibba.pgml.uga.edu/mcscan2/) 对蓖 麻LBD基因家族在基因组中的复制模式进行分析[29],同时对蓖麻LBD 与拟南芥LBD 基因进行共线性分析,使用本地BLASTP 对蓖麻与拟南芥的蛋白质序列文件进行建库,以此蛋白质序列文件作为query 序列进行BLASTP 比对,设置E-value 为1e-10。判定相互匹配部分的长度大于较长序列长度的80%且相互匹配部分的相似性大于80%的紧密相连的基因中,只参与1 次复制事件,同时结合基因在染色体上的位置,判断其复制类型,蓖麻与拟南芥的种内、种间共线性关系使用Circos 软件进行可视化。使用KaKs_Calculator 2.0 软件对复制基因对进行进化选择压力分析[30],计算片段重复基因对的非同义替换率(Ka)和同义替换率(Ks)及其比例(Ka/Ks),剔除不符合阈值的基因对,使用ggplot2 对符合阈值的基因对的分析结果进行可视化[31]。
1.6 LBD 基因家族进化发育分析
对蓖麻LBD 蛋白质序列及拟南芥、麻疯树、玉米、水稻、番茄、烟草LBD 蛋白质序列进行ClustalW多重序列比对,对齐保守结构域两端序列,使用MEGA X 软件以邻接法(NJ)构建蓖麻种间系统发育树,使用PhyML 软件以最大似然法(ML)构建蓖麻LBD 基因家族种内系统发育树[32-33],最大似然法采用LG+G Model,邻接法采用JTF Model,设置Bootstrap 检验1 000 次,其余采用默认参数,使用Evolview 在线工具对系统发育树可视化[26]。
1.7 蓖麻LBD 基因家族表达模式分析
从NCBI GEO 数据库(https://www.ncbi.nlm.nih.gov/geo/)获取蓖麻不同组织的相关表达量数据,包括Ⅱ/Ⅲ期胚乳、Ⅴ/Ⅵ期胚乳、萌芽种子、叶片、雄花[34]。非生物胁迫数据来自内蒙古民族大学蓖麻育种国家民委重点实验室的前期研究(数据未公布),包括经过除草剂喷施与未喷施的蓖麻叶片、茎段和根。使用编写的脚本对统计得到的reads count 值进行FPKM标准化处理,在计算过程中,将基因所有外显子的长度之和定义为有效的基因长度。为了了解蓖麻LBD基因家族的表达模式,对所有样本中表达量为0 的基因进行剔除,并对剩余的蓖麻LBD 基因家族进行层次聚类。使用R-4.1.0 软件(https://www.r-project.org/) 的Pheatmap 包进行基因表达量热图绘制(https://cran.r-project.org/web/packages/pheatmap/)[35],为使结果更加准确,对表达量数据进行标准化处理。
2 结果与分析
2.1 蓖麻LBD基因家族成员鉴定及蛋白质理化性质分析
为确定蓖麻LBD 基因家族成员,分别进行了本地BLASTP 与HMMER 比对,由TAIR 获取了拟南芥LBD 基因家族成员的蛋白质序列,以此作为query 序列进行本地BLASTP 比对,结果显示,有33 条符合阈值的蓖麻LBD 基因家族候选序列(E-value≤1e-10);经确认,LBD 典型结构域的Pfam 登记号为PF03195,下载其隐马尔科夫模型,经本地HMMER比对也获得33 条符合阈值的候选序列(图1)。使用NCBI CDD 与EBI InterPro 工具对候选序列进行结构域检测,使用Clustal Omega 进行多重序列比对,手动剔除类锌指结构CX2CX6CX3C 缺失或不完整的序列,从而确保结果的准确性,对于同一基因存在多个转录本的情况,选择第1 个转录本作为代表序列;最后,共鉴定得到30 个蓖麻LBD 基因家族成员,按照基因在染色体上的顺序命名为RcLBD1~RcLBD30。对蓖麻LBD 基因家族成员进行了系统评估,包括基因长度、氨基酸数目、蛋白质分子量、等电点、亚细胞定位等。由表1 可知,30 个蓖麻LBD 基因家族的氨基酸数目与分子量差异较小,但有个别成员差异较大,其中,RcLBD29 只有73 个氨基酸数目,分子量最小,为8 513.10 D;RcLBD12 则有1 486 个氨基酸数目,分子量最高,为166 377.47 D。等电点范围波动较大,其中,RcLBD21 等电点最低,为4.53;RcLBD29 等电点最高,为9.72。除RcLBD29 外,疏水系数(grand average of hydropathy,GRAVY)均为负值,表明其他蓖麻LBD 蛋白均为亲水性蛋白。亚细胞定位结果显示,30 个蓖麻LBD 基因家族的蛋白质全部位于细胞核内,RcLBD12 既位于细胞核内也位于细胞膜中。

表1 蓖麻LBD 基因家族信息
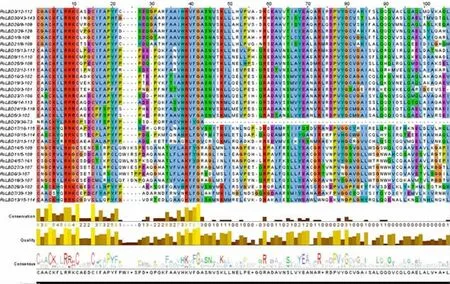
图1 蓖麻LBD 基因家族的序列特征
2.2 蓖麻LBD 基因结构、蛋白质保守基序分析
为探究蓖麻LBD 基因家族的基因结构,基于注释文件,使用编写的脚本进行了基因结构分析,30 个蓖麻LBD 基因的外显子、内含子分布情况见图2。由图2 可知,30 个蓖麻LBD 基因的外显子数目为2~7 个,内含子数目为1~7 个。其中,RcLBD12 与RcLBD28 含有7 个外显子和内含子,RcLBD10 含有5 个外显子和4 个内含子,RcLBD25 含有4 个外显 子和3 个内含子,RcLBD5、RcLBD8、RcLBD14、RcLBD20、RcLBD24 均含有3 个外显子和2 个内含子,而RcLBD17 仅含有1 个外显子并没有内含子,除此以外,其他所有成员均含有2 个外显子和1 个内含子。
基序,亦称模序、模体,是指DNA、蛋白质等生物大分子中的保守序列,是介于二级和三级结构之间的另一种结构层次。MEME 分析结果显示,30 个蓖麻LBD 基因家族成员中共找到10 个Motif(图2),仅前5 个Motif 的E-value 小于0.05,单个成员含有的Motif 范围在1~6 个。其中,所有成员均具有Motif2, 经比较发现,Motif2为类锌指结构CX2CX6CX3C,这也是LBD 基因家族的标志;绝大多数成员同时具有Motif1 与Motif3,经比较发现Motif1 为GAS 基序,Motif3 为类亮氨酸拉链结构LX6LX3LX6L。值得注意的是,RcLBD7 具有Motif2与Motif3 而缺失了Motif1,RcLBD29 仅具有Motif2并且第1 个半胱氨酸突变为天冬酰胺,更有趣的是,RcLBD4、RcLBD6、RcLBD11、RcLBD14、RcLBD18、RcLBD27 均缺失了Motif3,而在相同位点以Morif4(LLWSGNWHVCQAAVETVLRGGPIRPL)取代。可以明显看出,蓖麻LBD 基因家族拥有较为简单的蛋白质保守基序结构,与对应的基因结构基本一致。
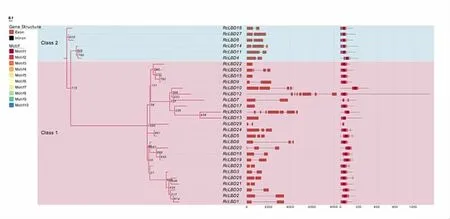
图2 蓖麻LBD 基因的种内系统发育树、基因结构和蛋白质保守基序
为进一步探究蓖麻LBD 蛋白的结构,使用SWISS-MODEL 对具有代表性的蓖麻LBD 蛋白进行同源建模(图3),其中,A 为RcLBD2,属于Class 1,具有完整的LOB 结构域;B 为RcLBD28,属于Class 2,缺失类亮氨酸拉链;C 为RcLBD29,属于Class 1,但缺失类亮氨酸拉链且GAS 基序不完整。如建模结果所示,无论属于哪个亚族,蓖麻LBD 蛋白的空间结构都呈现出近似对称的“Y”字结构,但RcLBD29是例外的,RcLBD29 只保留了上部的近似“V”的结构,这意味着RcLBD29 可能在进化过程中丢失了部分功能。

图3 蓖麻LBD 蛋白同源建模
2.3 蓖麻LBD 基因家族染色体定位及复制分析
基因在染色体上的分布与染色体在表达过程中的参与程度以及在植物生长发育中的重要性密切相关。为探究蓖麻LBD 基因家族的复制模式与进化机制,进行了染色体定位分析,以往蓖麻多在Scafford水平进行分析,难以有效地探明其关系,得益于测序技术的进步,二代测序结合三代测序已将蓖麻基因组组装至染色体水平(图4),30 个蓖麻LBD 基因分布在9 条染色体上,在RcChr6 上没有分布。此外,除RcLBD7 位于RcChr3 外,其余染色体上均有多个成员分布,RcChr8 上最多,有7 个成员分布,分别是RcLBD18~RcLBD24。
通过对蓖麻和拟南芥进行共线性分析,MCScanX 结果显示,在蓖麻中有7 对同源基因对,分别是RcLBD4 和RcLBD11、RcLBD6 和RcLBD27、RcLBD7 和RcLBD17、RcLBD9 和RcLBD15、RcLBD11和RcLBD14、RcLBD13 和RcLBD28、RcLBD18 和RcLBD27。对这些共线性区域分析时发现,在蓖麻的30 个RcLBD 基因中仅RcLBD19、RcLBD20 为串联重复基因,RcLBD1、RcLBD2 可能来自小规模的转座,或者是由某些其他基因的串联复制和插入产生的,RcLBD4、RcLBD6、RcLBD7、RcLBD9、RcLBD11、RcLBD12、RcLBD13、RcLBD14、RcLBD15、RcLBD17、RcLBD18、RcLBD27、RcLBD28 可能来源于全基因组复制或片段复制,其他的RcLBD 基因可能来自转座,如“复制型转座”“非复制型转座”或“保守转座”等。在拟南芥中发现6 对同源基因对,分别是AtLBD1 和AtLBD11、AtLBD10 和AtLBD36、AtLBD16和 AtLBD29、AtLBD37 和 AtLBD38、AtLBD37 和AtLBD39、AtLBD38 和AtLBD39。同时,蓖麻与拟南芥物种间存在25 对同源基因对,主要分布在拟南芥的AtChr1、AtChr3 上,表明了LBD 基因家族的高度保守性。可以发现,LBD 基因家族的部分成员在蓖麻和拟南芥染色体上的排列极为接近,聚集成簇,说明在进化历程中可能发生了大规模的片段复制事件。根据Circos 软件对共线性结果进行了可视化,其结果见图4。

图4 蓖麻LBD 基因染色体定位及共线性分析
蓖麻LBD 基因家族进化选择压力分析表明,所有基因对的Ka 值均小于Ks 值,所有Ka/Ks 值均小于0.4(图5),表明蓖麻LBD 基因家族在进化过程中经历了较强的纯化选择,以减少片段重复后的有害突变。
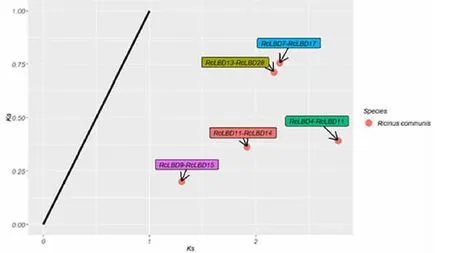
图5 蓖麻LBD 基因家族进化选择压力分析
2.4 LBD 基因家族进化发育分析
种内系统发育树(图2)结果显示,30 个蓖麻LBD 基因分为2 个亚族Class 1 和Class 2。亚族Class 1 与Class 2 的成员基序结构都较为简单,值得注意的是,亚族Class 1 中RcLBD7 与RcLBD29是特殊的,在Class 1 中除RcLBD7 缺失GAS 基序和RcLBD29 缺失GAS 基序与类亮氨酸拉链外,所有的Class 1 成员均具有包含类锌指结构、GAS 基序与类亮氨酸拉链在内的完整结构,这可能是在进化过程中发生了丢失。有趣的是,RcLBD13 与RcLBD28 在进化上明显晚于其他蓖麻LBD 基因家族成员,二者的类亮氨酸拉链结构出现了明显的氨基酸突变。
种间系统发育树(图6)包含30 个蓖麻LBD 蛋白、43 个拟南芥LBD 蛋白、40 个麻疯树LBD 蛋白、45 个玉米LBD 蛋白、34 个水稻LBD 蛋白、42 个番茄LBD 蛋白、92 个烟草LBD 蛋白。进化分析结果显示,7 个物种共计326 个LBD 蛋白被分为2 个亚族,命名为Class 1 和Class 2,在种间系统发育树中可以看出,30 个蓖麻LBD 蛋白分布于2 个亚族之中,与种内系统发育树结果一致。在所有亚族中均含有蓖麻与拟南芥的LBD 蛋白,在Class 2 中也包含6 个拟南芥LBD 基因家族成员,分别是AtLBD37、AtLBD38、AtLBD39、AtLBD40、AtLBD41 和AtLBD42,这也与拟南芥LBD 基因家族的分类一致,意味着两者可能在进化上比较相似。有趣的是,RcLBD29 在7 个物种中都是独特的,RcLBD29 仅具有1 个类锌指结构CX2CX6CX3C、类亮氨酸拉链缺失、GAS 基序发生氨基酸突变,但在进化中被归于Class 1,在1 000 次Bootstrap 检验的最大似然法树中自展值为1 000。总的来看,LBD 基因家族在7 个物种中高度保守。
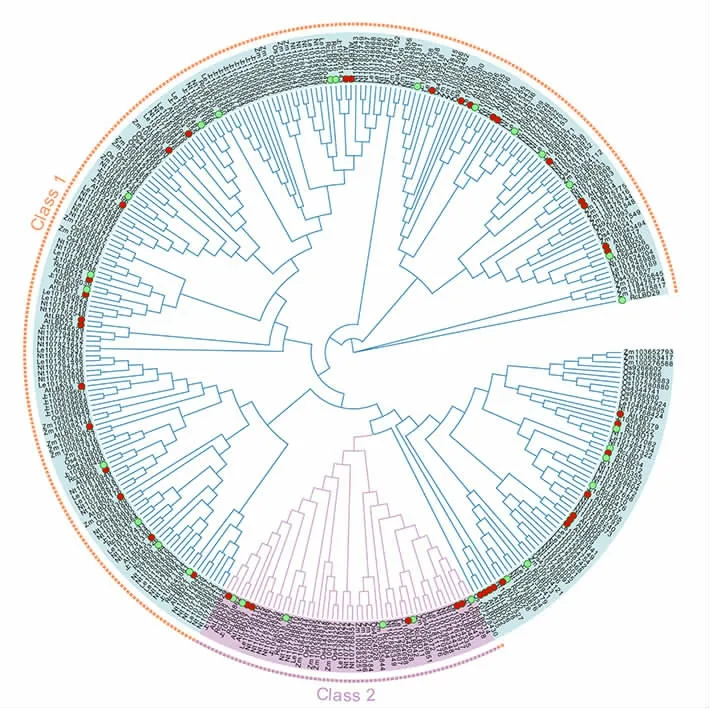
图6 蓖麻LBD 基因种间系统发育树
2.5 蓖麻LBD 基因家族表达模式分析
为研究蓖麻LBD 基因家族的表达模式,探究基因功能,从NCBI GEO 数据库中获取了蓖麻的表达量数据,包括Ⅱ/Ⅲ期胚乳、Ⅴ/Ⅵ期胚乳、萌芽种子、叶片、雄花,命名为A 组,其结果见图7。此外,为了探究蓖麻LBD 基因家族在非生物胁迫的功能,对内蒙古民族大学蓖麻育种国家民委重点实验室前期的测序数据进行了探究(数据未公开),包括经过除草剂喷施与未喷施的蓖麻叶片、茎段和根,命名为B 组,其结果见图8。
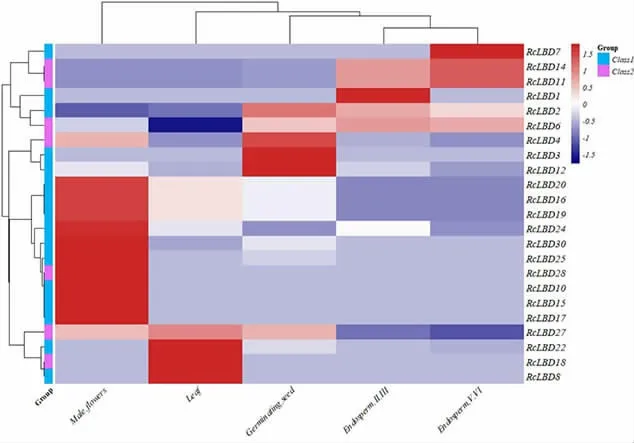
图7 蓖麻5 个组织中LBD 基因的表达量数据

图8 喷施除草剂后的蓖麻LBD 基因表达量数据
在A 组数据中,蓖麻LBD 基因在不同组织部位中具有不同的表达模式,表现出了显著的组织特异性。大多数的蓖麻LBD 基因家族仅在特定的组织部位中高表达。有研究显示,在拟南芥中ASL1基因参与调控了花的细胞分化从而调控花序的发育进程[4],在蓖麻雄花组织中RcLBD10、RcLBD15、RcLBD16、RcLBD17、RcLBD19、RcLBD20、RcLBD24、RcLBD25、RcLBD28、RcLBD30 高表达,意味着这些LBD 基因可能参与了蓖麻雄花的形态建成。在叶片组织中,RcLBD8、RcLBD18、RcLBD22、RcLBD27 高表达,有研究显示,在拟南芥中,AtAS2 通过抑制KNOX 基因表达来调控拟南芥近轴面区域的细胞增殖,从而介导两面对称的平展叶形成[36],AtLBD36 参与调控了叶的形态建成[37],说明蓖麻LBD 基因可能在蓖麻叶片发育中扮演了重要角色。此外,部分蓖麻LBD 基因在种子与胚的不同发育时期出现高表达,说明蓖麻LBD 基因在蓖麻发育前期也起到了重要作用,这与AtLBD30 参与拟南芥胚胎发生、ZmLBD19 调控玉米胚珠发育一致[36,38]。此外,值得注意的是,有7 个蓖麻LBD 基因在5 个组织中均为表达,分别是RcLBD5、RcLBD9、RcLBD13、RcLBD21、RcLBD23、RcLBD26 和RcLBD29。
在B 组数据中,展示了蓖麻受到除草剂砜嘧磺隆喷施后在根、茎、叶3 个组织部位中响应非生物胁迫的状况,同时,由于增加了茎段与根的样本数据,对A 组数据来说是一个有力的补充。可以明显看出,在蓖麻中绝大多数的LBD 基因在根中高表达。有研究表明,在拟南芥中,植物激素调节因子ARF 通过激活AtLBD16、AtLBD18、AtLBD29 从而调控了拟南芥的侧根发育过程[39-41],在水稻中,OsCrl11 也参与调节了水稻不定根的发生[42],这与蓖麻中LBD 基因一致。有研究发现,TaLBD34、TaLBD54 在受到冷胁迫的小麦中高表达[43],GmLBD12 在干旱、盐胁迫与植物激素诱导的大豆中高表达[44]。在蓖麻中,RcLBD6、RcLBD16、RcLBD27 在除草剂处理的蓖麻叶片中上调表达,其中,RcLBD6 上调近10 倍、RcLBD27 上调2 倍;RcLBD6、RcLBD12、RcLBD16、RcLBD23、RcLBD27 在除草剂处理的蓖麻根部中上调表达,其中,RcLBD6 上调3 倍、RcLBD27 上调2 倍;RcLBD6、RcLBD8、RcLBD14、RcLBD20、RcLBD27、RcLBD28 在除草剂处理的蓖麻茎段中上调表达,其中,RcLBD6 上调5 倍、RcLBD8 上调7 倍、RcLBD27上调2 倍。这表明蓖麻LBD 基因在抵御非生物胁迫的过程中发挥了重要作用,并且存在明显的组织特异性。值得注意的是,RcLBD6 与RcLBD27 在喷施过除草剂的蓖麻根、茎、叶3 个部位中均上调表达,有趣的是,RcLBD6 和RcLBD29 都与拟南芥AtLBD37、AtLBD38、AtLBD39 基因同源,这些都表明LBD 基因家族可能在植物逆境胁迫中起到了重要作用。此外,蓖麻中Class 1 与Class 2 的LBD 基因家族成员在表达模式上并没有明显的差别。
3 结论与讨论
转录因子普遍存在于高等植物之中,在植物生长发育、逆境胁迫等方面发挥着重要作用,侧生器官边界域基因LBD 是一类广泛存在于绿色植物中的特异性转录因子,参与调控植物生长发育与代谢过程并起着重要作用。
本研究首先在蓖麻中鉴定了33 条LBD 蛋白质序列,由于在蓖麻LBD 基因中存在多个转录本,对于同一基因存在多个转录本的情况,选择第1 个转录本作为代表序列,在剔除类锌指结构域缺失与不完整的序列之后,共获得30 条蛋白质序列,并以此作为蓖麻LBD 基因家族的成员。基因复制与进化选择压力分析显示,蓖麻与拟南芥LBD 基因家族在进化上高度保守,在进化历程中出现了大规模的片段复制事件并经历了较强的纯化选择来减少片段重复后带来的有害突变。蓖麻种内系统发育树显示,从基因结构与蛋白质保守基序来看,全部的RcLBD 基因被分为2 个亚族(Class1、Class2),2个亚族的RcLBD 基因的结构差异较小,但蛋白质保守基序差异明显。从蓖麻与拟南芥、麻疯树、玉米、水稻、番茄、烟草的种间系统发育树显示,7 个物种共计326 个LBD 蛋白被分为2 个亚族(Class1、Class2),可以看出,种间系统发育树与种内系统发育树有着极高的相似性,LBD 基因家族在进化上较为保守。值得注意的是,在进化过程中部分LBD 蛋白的GAS 基序与类亮氨酸拉链发生了氨基酸突变,类似RcLBD13与RcLBD28,可能暗示了一种进化趋势,此外,RcLBD29 在7 个物种中都是特殊的,更像是进化过渡阶段的产物。表达模式分析结果显示,蓖麻LBD基因家族可能在蓖麻的花、叶片、根、胚等组织发育过程中起到了重要的作用,RcLBD6 与RcLBD27在喷施过除草剂的蓖麻根、茎、叶中均上调表达且差异显著,被推测可能参与了蓖麻响应非生物胁迫的过程。
综上所述,与以往对蓖麻的基因家族研究不同,本研究是在染色体水平上进行的。本研究采用生物信息学手段,从全基因组筛选与鉴定了蓖麻LBD 基因家族成员,同时分析了基因结构、理化性质、蛋白质保守基序、进化发育、表达模式等,研究结果为解析蓖麻LBD 基因家族功能奠定了基础,并为遗传育种工作提供了参考。

