面向原子制造的框架核酸研究进展*
杨蓓 李茜 柳华杰† 樊春海‡
1) (同济大学化学科学与工程学院, 上海自主智能无人系统科学中心, 先进土木工程材料教育部重点实验室, 上海 200092)
2) (上海交通大学化学化工学院, 上海 200240)
1 引 言
以原子为出发点, 探索原子尺度精准性对于物质性能的重塑性, 挑战自最基本层次开始的材料与器件制造, 一直是全世界研究人员追求的目标.当使用原子作为材料的基本构筑单元时, 许多物理和化学特性都将显著有别于宏观状态, 原子制造的材料不能再通过经典力学、宏观统计分析等理论来解释, 而是以量子理论作为核心基础进行探讨, 因此原子制造将极大程度上有别于传统制造, 并产生许多新颖特性.例如, 在拓扑绝缘体表面掺入磁性原子形成长程铁磁序, 无需外加磁场, 就能形成稳定的基本没有耗散的量子反常霍尔效应[1,2]; 利用原子作为量子比特的载体, 有望发展全新的量子计算机, 有效屏蔽外界干扰, 高速并行处理大量数据[3];此外, 最近发展的单原子催化剂基于单个金属原子能级结构和电子结构的根本变化, 较一般的金属团簇或纳米颗粒催化剂有更为高效的催化效率和原子利用率[4,5].
与纳米制造类似, 原子制造既可以通过“自上而下”的原子操纵技术, 也可以通过“自下而上”的原子组装技术实现.原子操纵最早借助于扫描隧道显微镜(scanning tunnelling microscopy, STM)一个一个地精准控制原子的捕获、移动和放置来实现.例如, 1990 年IBM 的科学家在人类历史上首次通过STM 针尖操控35 个氙原子, 拼出了“IBM”三个字母[6], 随后又在铜表面均匀排布了48 个原子的圆圈构成“量子围栏”[7].另一种捕获并操纵单原子的方法, 则需要在超高真空环境下利用磁光阱将原子冷却到接近绝对零度并困住, 再采用一个非常小的光阱来捕获单个原子[8].以上两种技术需要极低温和超真空的特殊操作环境, 控制原子的技术成本很高, 这极大地制约了原子制造的进一步推广.近些年来, 自下而上的自组装技术在材料构筑,特别是纳米材料和器件的制备方面吸引了越来越多的注意力.自组装不仅从技术上解决了许多微观世界的构筑难题, 也从理念上给了科学家更多的启发, 尤其是在利用交叉领域知识迎接新挑战方面.早在1959 年, 著名物理学家Richard Feynman 谈及微观世界的操控技术, 就曾指出可通过自下而上的组装手段构建原子微型机器, 在原子尺度上进行各种机械操作, 为原子制造描绘了宏伟的蓝图[9].
核酸作为一类生物大分子, 传统上被认为是生物携带遗传信息的物质, 在生物遗传、变异和蛋白质的生物合成中具有极其重要的作用.然而, 基于对核酸结构的深入思考, 可以发现其本身就是具有原子级精准度的纳米自组装材料.核酸从化学结构上可认为是核苷酸的聚合物, 自然界中的核酸根据核苷酸中五碳糖的不同分为脱氧核糖核酸(deoxyribonucleic acid, DNA)和核糖核酸(ribonucleic acid, RNA)两类.以DNA 为例, 它是由四种脱氧核苷酸组成的链状聚合物, 每个核苷酸包含一个磷酸基团, 一个脱氧核糖和一个碱基, 其中碱基可以是腺嘌呤(A)、鸟嘌呤 (G)、胞嘧啶 (C)或胸腺嘧啶(T).需要指出的是, DNA 精确的分子识别特性决定了它结构组装上的精准性.遵循Watson-Crick配对原则, A 与T, G 与C 之间通过氢键相互配对, 再借助碱基对之间的π-π 堆积作用及磷酸骨架的亲水作用, 自组装形成具有双螺旋结构的DNA分子.这些双链DNA 分子具有精确纳米尺寸的螺旋直径(2 nm)和螺距(3.4 nm, 约10.5 碱基对),结构中的每个原子和化学键都具有确定的位置和数量.此外, 除了常见的DNA 双螺旋结构, 在细胞内存在的核糖开关[10]、核酶[11]、环状RNA[12], 以及在端粒基因组中发现的更为复杂的四链折叠结构G 四链体[13]和i-motif[14]等, 也都是精准自组装的产物, 在发挥重要生物学功能的同时, 也反映出核酸结构的灵活多样性与可调控性, 更丰富了可利用的核酸材料范围.
因此, 对核酸分子的研究启发了物理、化学和材料科学家, 20 世纪80 年代初Seeman 教授基于“利用DNA 框架进行蛋白质结构解析”这一愿景,首先提出DNA 用作可编程纳米构筑材料的概念,发展出“DNA 纳米技术”这一新领域[15,16].基于具有原子级精度的DNA 分子, 通过序列设计对DNA杂交配对过程进行程序化控制, 研究人员成功构建了多样的人工DNA 纳米结构, 如纳米瓦块阵列[17,18]、纳米结[19]、多面体[20,21]等.2006 年Rothemund[22]开发的DNA 折纸技术进一步推动了DNA 纳米技术的发展, 该方法利用几百条短单链(订书钉链)引导一条几千碱基的长单链(骨架链)按设计的形状折叠, 可以获得特定的百纳米级大小的二维甚至三维结构.利用DNA 折纸术强大的图案化能力,可以构筑可寻址的核酸框架结构对纳米颗粒、蛋白质、聚合物等基元进行精准组装实现功能化, 以及将其自身作为模块进行更高级组装[23].这种具有精确框架结构并具备特殊物理、化学和生物学功能的框架核酸(framework nucleic acids, FNAs), 提供了在纳米甚至原子尺度上进行力学、光学和电学等物理性质, 以及单分子水平化学与生化反应的精准调控手段.发展至今, DNA 自组装结构因其良好的组装效率和可靠性在纳米制造领域已经得到广泛的认可, 基于框架核酸的理念, 人们已经实现了多种分子或材料精确的空间组织, 以探究其在纳米机器[24]、信息处理[25]、生物传感[26]和疾病诊疗[27]等领域的广泛应用.
由此可见, 框架核酸理念的出现赋予了核酸分子新的深层次价值.精细设计和组装获得的一维到三维的框架结构作为空间可寻址的平台, 有望从应用层面对单个至多个原子进行精准定位、有序排布或定向移动, 逐步实现从原子到宏观的精确功能化, 对于探索原子制造接轨实际应用具有重要意义.本文将围绕框架核酸与原子制造两个前沿方向的交叉融合, 进行简单的全景描述与未来展望.首先将介绍体现原子精准性的框架核酸结构构建, 然后描述利用框架核酸进行功能化的一般策略, 最后着重介绍其在器件构筑方面的研究进展, 并探讨面向原子制造的未来发展方向, 利用框架核酸进行原子级精准构筑的优势和前景.
2 框架核酸的结构组装与功能化
2.1 框架核酸结构组装
人工设计的框架DNA 核酸结构最早是DNA瓦块(tile)结构[28].DNA 瓦块单元指由几条至几十条DNA 短链(几十个碱基)按照化学等比例退火合成获得的纳米级组装模块, 通常被设计为具有多个分支状, 并在双链分支末端添加单链区.单个瓦块结构中所有原子数量与位置都是精确可控的,通过合理设计单链黏性末端的序列, DNA 瓦块单元之间可以进行自组装(图1(a)), 并获得高级的一维或三维组装结构, 例如一维纳米线和纳米管[29]、二维周期性阵列[30,31]、三维四面体[20](图1(b))、立方体[32]、类富勒烯结构[21](图1(c))、甚至DNA 晶体[33]等.同样, 用RNA 瓦块单元参与自下而上的组装过程, 可以构建一些RNA 纳米结构[34], 如RNA棱柱体[35]、多面体[36]等.但是, 核酸瓦块的组装过程中需要严格控制组分的化学计量比, 组装效率不高, 反应过程耗时且容易出错, 另外组装结构的复杂性也受限制.
2006 年Rothemund[22]开发的DNA 折纸(origami)技术很好地弥补了瓦块组装的不足, 可以快速、精确地组装出矩形、正方形、三角形、五角星、笑脸等复杂的纳米图案(图1(d)).同年, 中国的研究团队[37]利用类似技术成功组装了首个不对称的二维平面结构—仿中国地图.此后, 出现了更多二维甚至三维的折纸结构, 例如纳米盒子[38]、蜂窝结构[39]、线框结构[40−42](图1(e))、纳米花瓶[43](图1(f))等.多个团队还开发了Tiamat[44], caDNAno[45]和DAEDALUS[46]等设计软件, 方便更多研究人员的参与.单个DNA 折纸结构的大小受到骨架链长度的限制, 因此研究人员进一步探索了将多个折纸单体组装为更大结构[47−50].例如, 在近期的一项工作中, Tikhomirov 等[50]以类似拼图的形式将64 个方形DNA 折纸模块分形组装, 构建出蒙娜丽莎、公鸡和细菌等微米级别的复杂图案(图1(g)).
除了静态的自组装框架结构, 核酸的杂交过程可编程特性更赋予了这些框架作为纳米机器的潜力.动态核酸结构的调控依赖于其构象变化, 驱动机制包括核酸杂交、底物识别和环境响应刺激等.最典型的核酸机器基于DNA 链置换反应驱动, 由于不同长度和序列DNA 单链之间互补配对的结合能力不同, 通过添加特定的DNA 单链作为燃料链, 驱动DNA 分子机器构象变化[51].由于一些特定核酸序列对离子、小分子和蛋白质的特殊识别作用, 还可以设计出具有底物识别作用的核酸机器[52],例如核酸适配体就是一类可识别特定底物发生构象转变的核酸材料.此外, 某些核酸序列还具有环境响应性, 例如富含C 碱基的一些序列可在酸性条件下形成四链i-motif 结构, 而碱性环境又回复到单链结构, 因此可设计出pH 驱动的核酸机器[53].
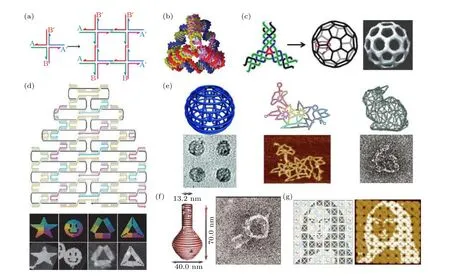
图1 典型框架核酸结构: DNA 瓦块和DNA 折纸结构 (a) DNA 瓦块组装成的二维晶格[28]; (b) DNA 四面体[20]; (c) 类富勒烯结构[21]; (d) DNA 折纸设计图及几种二维平面折纸结构[22]; (e) 球形[40]、花鸟图案[42]和兔子[41]线框DNA 折纸结构; (f) DNA 纳米花瓶结构[43]; (g) 16 个折纸模块组成的蒙娜丽莎图案[50]Fig.1.Typical FNAs: DNA tile and DNA origami: (a) DNA four-way junction[28]; (b) DNA tetrahedron[20]; (c) DNA buckyball selfassembled by three-point-star DNA tiles[21]; (d) 2D DNA origami structures[22]; (e) sphere[40], flower-and-bird pattern[42] and bunnyshape[41] wireframe DNA origami structures; (f) nanoflask DNA origami structure with complex curvatures[43]; (g) a Mona Lisa pattern self-assembled by 16 DNA origami tiles[50].
随着DNA 折纸等复杂框架结构的出现, 对核酸机器的研究进入了新的阶段.折纸结构具有更多的精准定位位点、更好的构象变化可操控性以及维度转换的能力, 因此适用于更复杂机器构建以及多功能体系的集成研究.Andersen 等[38]设计了一个可开合的DNA 纳米盒子, 为框架核酸的可控装载功能开发提供了新思路.Lund 等[54]将二维DNA折纸作为基底, 构建了可在特定路径上行走的DNA仿蜘蛛机器人.Turberfield 课题组[55]设计了一种可以在DNA 折纸表面构筑的线性轨道上进行连续匀速步进的DNA 分子马达.
2.2 框架核酸结构的精准功能化
利用框架核酸结构的原子精准性, 进行功能基元的精准定位, 能够充分体现框架核酸在功能调控方面的优势.框架核酸进行功能化定位的方式一般有两种: 一是通过核酸化学合成及修饰技术将荧光基团、化学及生化偶联基团、具有光电等性质的合成分子等各种化学基团共价修饰到核酸链上; 二是设计互补序列杂交, 实现无机纳米颗粒、蛋白质、功能核酸等大分子在框架核酸上的精准组装.两种方法共同实现了功能基元在框架核酸各个位置上高效率的空间定位, 且框架核酸的结构稳定性和核酸分子的特异性杂交使得定位的抗噪性得到了很好保障.Tinnefeld 课题组[56]在DNA 折纸的指定位点进行荧光基团标记, 可作为纳米标尺来校准超分辨显微镜.Li 等[57]开发了一种以DNA 四面体作为刚性节点的分形DNA 框架, 构建了低串扰、多色编码的超复合荧光放大器, 用于识别单分子及活细胞.Lu 等[58]在DNA 四面体边缘的特定位置修饰亚甲基蓝和二茂铁分子, 来探讨四面体框架空间介导的电荷输运机制.而将脂质体这类两亲分子在DNA 框架模板上的组装, 可构建出高度单分散的100 nm 以下的单膜脂质囊泡[59].
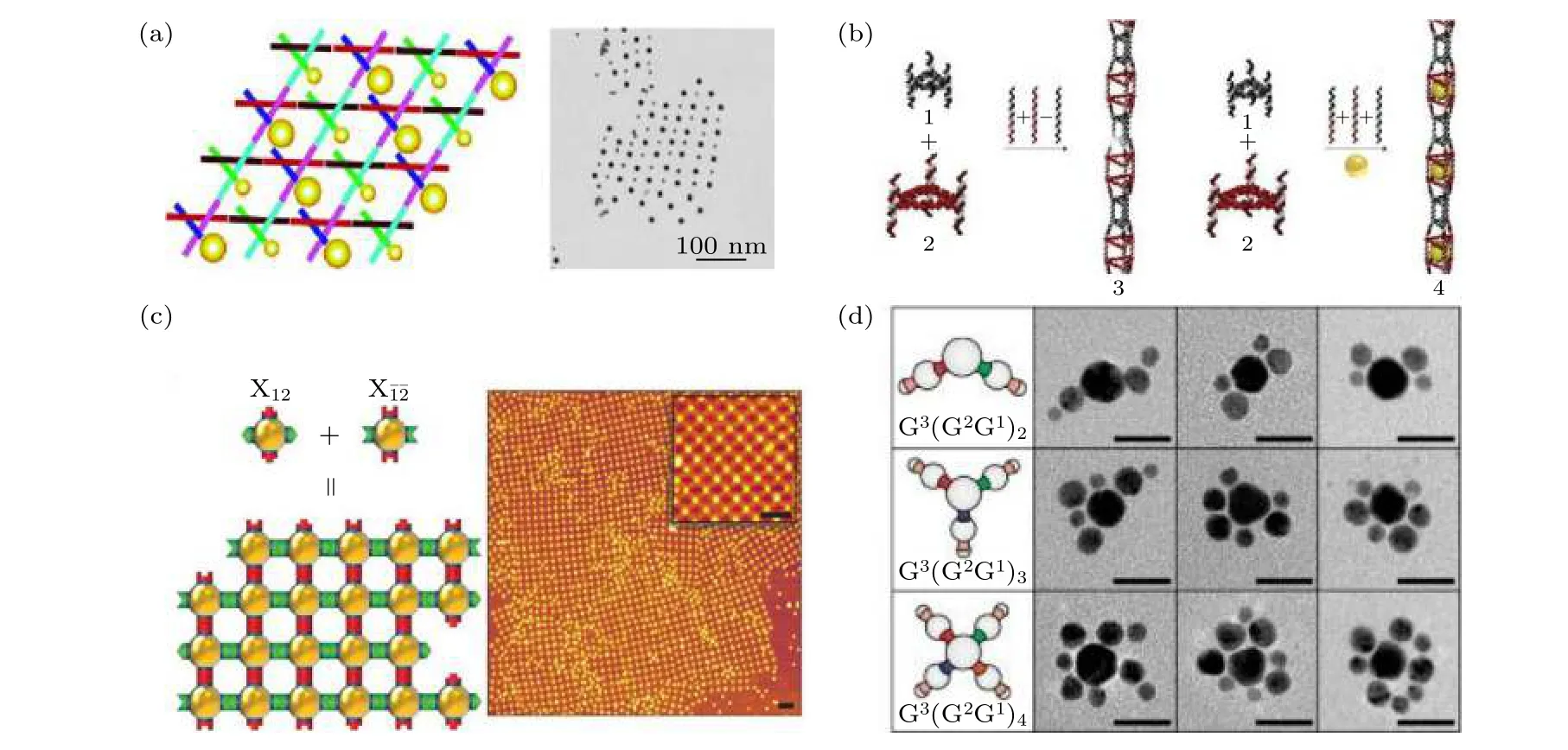
图2 框架核酸介导纳米颗粒组装 (a) DNA 瓦块介导AuNPs 组装成二维阵列[60]; (b) 三角形DNA 纳米管封装的AuNP 线[62];(c) DNA 折纸模块介导AuNPs 形成平面阵列[63]; (d) DNA 单链编码的AuNPs 组装成分支状类分子结构[65]Fig.2.FNAs-directed nanoparticles assembly: (a) 2D AuNP arrays self-assembled by DNA tiles [60]; (b) AuNP lines size-selective encapsulated within triangular DNA nanotubes[62]; (c) 2D AuNP arrays directed by DNA origami tiles[63]; (d) branched molecule-like structures self-assembled by single-stranded DNA encoded AuNPs[65].
金纳米粒子(gold nanoparticles, AuNPs)等更大粒径的颗粒也可实现在核酸框架结构上的精准排布, 形成各种寡聚体、一维至三维晶格等.最初, Seeman 课题组[60]利用DNA 瓦块构建了AuNP二维平面阵列(图2(a)), 之后Sharma 等[61]在将AuNPs 组装到二维DNA 瓦块阵列时发现AuNPs间的静电斥力会诱导DNA 纳米管的形成, 产生多种环状与螺旋状的AuNP 阵列.Lo 等[62]组装了一种空腔大小交替排列的三角形DNA 纳米管, AuNPs被尺寸选择性地封装到大空腔中(图2(b)).DNA折纸框架的出现为构建更复杂有序的AuNP 组装体指明了方向.Gang 课题组[63]将AuNPs 预先嵌入平面DNA 折纸框架模块中, 利用DNA 折纸模块之间选择性组装, 设计了周期排列的平面介观纳米粒子结构(图2(c)).他们还选用三种不同的多面体DNA 折纸框架, 将AuNPs、量子点和蛋白质作为客体分子封装在折纸框架中, 通过DNA折纸框架模块之间自组装间接构建纳米粒子超晶格结构[64].除此之外, Yao 等[65]还利用单链DNA将AuNPs 编码成具有正交价键的类原子纳米粒子, 通过DNA 链间的杂交反应将AuNPs 自组装成具有不同组成、大小、形状的低配位类分子结构(图2(d)).
基于核酸分子本身的性质, 还可以通过静电、配位和嵌入等多种方式与其他物质进行相互作用,并进而引导原子的原位聚集与成核生长.原位金属化反应是研究较为广泛的一类功能化方法.利用无差异性的DNA 链负电性与金属离子的库仑力,铜、银等金属可在框架核酸的表面进行生长[66,67].将预先组装的小颗粒作为晶种, 可以提升金属的生长程度, 甚至进行“浇筑”[68].利用某些基团与金属离子的选择性作用, 还能够实现金属的定位生长.例如, Pal 等[69]在利用Tollens 反应在DNA 折纸表面定点合成了水溶性的荧光银纳米团簇(图3(a)).最近, Jia 等[70]开发了一种无修饰的定位金属化反应技术, 他们利用DNA 单双链在金属离子存在下凝聚能力的不同, 设计了差异化的金属化反应, 并成功获得了仅有5 nm 线宽的金属图案生长(图3(b)).值得注意的是, 这种技术能够实现金属异质图案的“套印”, 即在DNA 框架模板的不同位置生长出不同种类的金属.无机非金属材料同样可在框架核酸上进行原位生长.Liu 等[71]利用框架核酸诱导硅烷团簇预水解的方法, 诱导二氧化硅在框架核酸模板的磷酸骨架周围形成沉积层, 制备了精确可控的DNA-二氧化硅复合材料(图3(c)).最近, Shang 等[72]利用表面伸出的双链, 又开发了一种在DNA 折纸模板上定点合成二氧化硅纳米结构的方法(图3(d)).
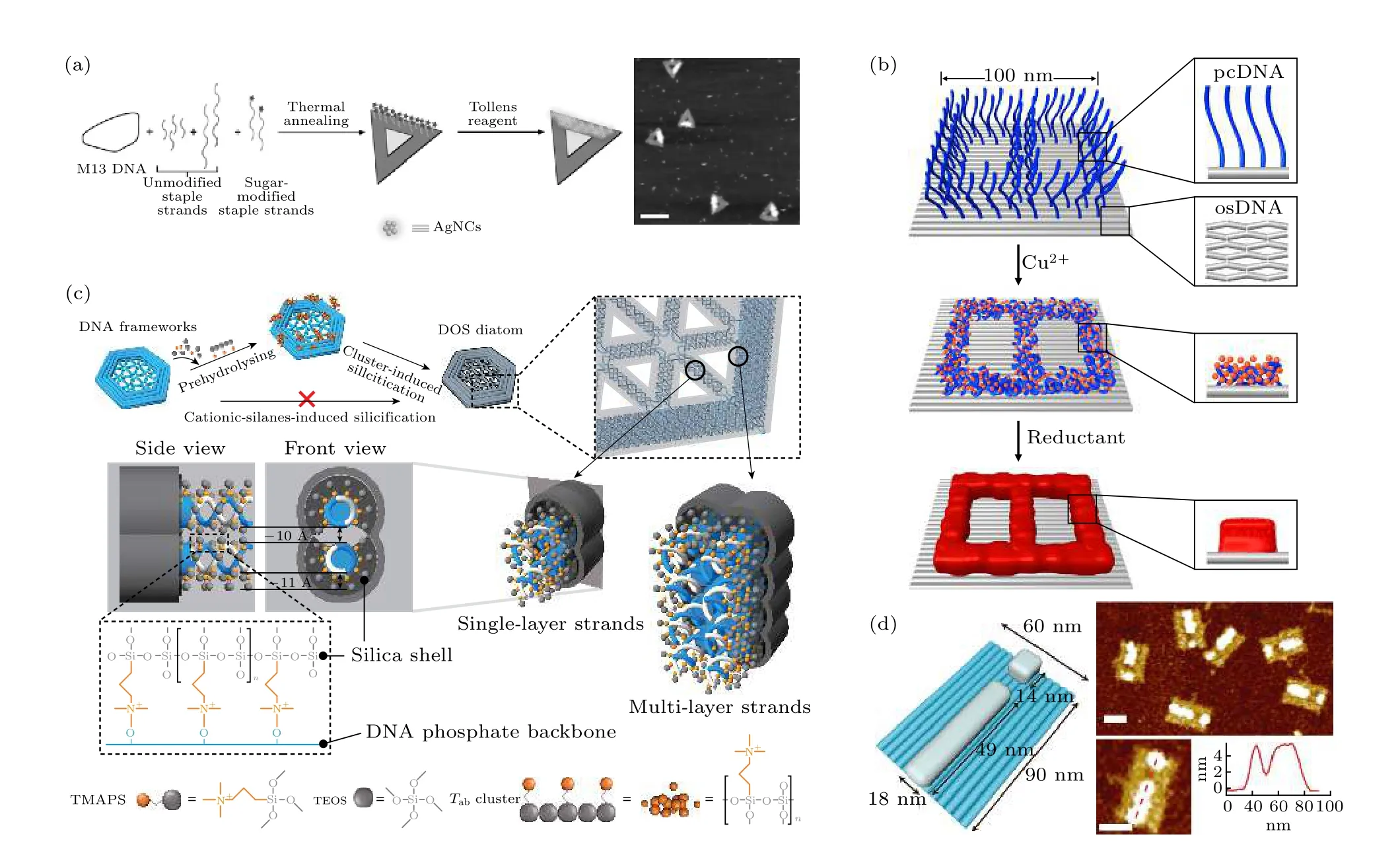
图3 框架核酸介导原位生长 (a) 三角形DNA 折纸上定点金属化形成银纳米簇[69]; (b) DNA 折纸上选择性金属化构建8 字形图案[70]; (c) DNA-二氧化硅复合材料的制备[71]; (d) DNA 折纸上定点合成“i”形二氧化硅纳米结构[72]Fig.3.FNAs-directed in-situ growth of nanomaterials: (a) Silver nanoclusters synthesized on DNA origami[69]; (b) selective DNA condensation and metallization on DNA origami for fabricating a digit 8 pattern[70]; (c) DNA origami silicification diatom-mimicking structures[71]; (d) site-specific synthesis of “i-pattern” silica nanostructure on DNA origami[72].
3 基于框架核酸的器件构筑
3.1 单分子反应器
框架核酸原子级精准性的特点, 为少至单个分子的准确定位提供了可能, 在此基础上, 使我们能够进一步在单分子尺度上对化学及生化反应进行研究[73].Gothelf 课题组[74]首先展示了二维折纸上的单分子化学反应, 他们在折纸表面选取了12 个不同位点, 每个位点分别修饰一个连接基团, 当体系中存在某种基团切断试剂时, 与之对应位置上的单个连接基团将被切断, 从而实现了对单个分子的定位剪切反应(图4(a)); 另一方面, 他们用同样的平台还设计了单分子连接反应, 可将单个分子定点连接在特定位置.此外, 他们还展示了控制大分子低聚物的形成, 首先将树状大分子连接到DNA 折纸表面形成环状图案, 再使相邻树状大分子单体间共价聚合, 制得了可控寡聚数量的大分子聚合图案[75].采用类似的思路, Weil 课题组和Wu 课题组等通过在DNA 折纸表面延伸G 四链体序列, 模拟过氧化物酶的活性催化多巴胺的聚合反应[76];或在G 四链体结构中嵌入光敏剂, 通过光诱导多巴胺聚合[77], 均可产生尺寸、形状精确可控的聚多巴胺图案(图4(b)).
自然界的生命体中存在着广泛的限域反应, 对于生命活动至关重要.一直以来人们希望构建分子级的化学反应器来精确调控反应进程及选择性, 并进一步提高反应产率, 框架核酸的精准定位及结构可编程性恰好满足了基本要求.Gothelf 课题组[81]曾基于DNA 的Holliday 结构构建了体积仅有10–24L的限域反应器.更多的工作关注于对酶级联反应在体外的限域调控, 例如, Willner 研究组[82]首先构建了葡萄糖氧化酶(glucose oxidase, GOx)与辣根过氧化物酶(horseradish peroxidase, HRP)周期性阵列结构, 并发现了反应效率的提升.Yan 课题组[83]发现这两种酶的级联反应活性依赖于酶间距,当酶间距很近时酶活性显著增强, 相距超过20 nm时酶活性则急剧下降.由于该体系调节酶通路的过程相对缓慢且效率低, Yan 课题组[78]在前面的基础上又通过调控DNA 折纸上的底物通道来快速有效地定向调节多种酶通路, 他们在折纸上以一定间距定位三种脱氢酶, 并加入了活性物质修饰的单链DNA 人工摆动臂调节不同酶通路的相对活性(图4(c)).Fu 等[79]进一步构建了DNA 折纸纳米管结构, 通过管的限域作用, 调控GOx和HRP 酶级联反应中间产物的定向输运, 使反应效率显著提升(图4(d)).此外, Xin 等[80]还设计了一个动态DNA机器, 通过调控GOx和HRP 之间的距离, 原位可逆地调节酶级联反应(图4(e)).

图4 框架核酸构建单分子反应器 (a) DNA 折纸上单分子化学键断裂反应[74]; (b) DNA 折纸上光诱导多巴胺聚合反应[77];(c) DNA 折纸上酶通路调控系统[78]; (d) DNA 纳米管中GOx 和HRP 的酶级联反应[79]; (e) DNA 机器可逆调控酶级联反应[80]Fig.4.FNAs used for single molecule reactors: (a) Single-molecule chemical cleavage reactions on DNA origami[74]; (b) lighttriggered polydopamine formation on DNA origami[77]; (c) enzyme pathway regulation system on a rectangular DNA origami platform[78]; (d) bienzyme cascade of GOx and HRP in a DNA origami nanotube[79]; (e) reversible regulation of enzyme cascade reaction by a DNA machine[80].
3.2 单分子传感器
框架核酸具有精准的空间可寻址性, 同样可以应用到对单个分子的捕获与识别, 构建单分子传感器.以DNA 折纸为例, 其具有类似芯片的几何特征, 每条订书钉链都可以作为分子识别的探针进行修饰和延伸, 原则上最多可产生200 多个分辨率约为6 nm 的探针位点, 因此可以作为单分子传感的理想平台.Ke 等[84]利用矩形DNA 折纸来模拟基因芯片, 以纳米级的精度在折纸上延伸单链探针,目标RNA 可与单链探针杂交形成坚固的“V”形DNA-RNA 双链, 很容易在单分子水平上被原子力显微镜(atomic force microscope, AFM)表征出来, 从而实现高细胞基因表达下的RNA 标签检测(图5(a)).基于这种核酸分子识别的设计原理还可制备单核苷酸多态性(single nucleotide polymorphisms, SNPs)分型检测传感器, Seeman 课题组[85]在DNA 折纸上设计了代表4 个碱基A, T, G, C的字母图案, 可用AFM 直接成像, 在目标核苷酸存在时通过立足点链置换反应使特定的字母图案被移除, 最终SNP 分型的结果经AFM 可直观清晰地得到(图5(b)).Zhang 等[86]利用DNA 折纸设计了一组形状ID, 能特异性靶向基因组序列, 并经AFM 成像进行单分子水平的SNP 直观检测.
除了DNA 折纸, DNA 四面体因其具有很高的结构刚性和组装效率, 且定向有序、间距可控、稳定性高, 非常适合应用于单分子传感.Pei 等[87]设计了顶点延伸探针DNA 的DNA 四面体结构,通过底部3 个顶点修饰的巯基固定在金电极上, 探针DNA 杂交结合目标DNA 后, 系统将监测的杂交信号转换为电化学信号输出, 结果表明该传感器对单碱基错配有显著的选择性 (图5(c)).若进一步设计能识别不同生物活性分子的DNA 四面体探针, 使其具备显著的并行性和高通量分析能力, 则有利于提高检测的灵敏度和特异性[90].

图5 框架核酸构建单分子传感器 (a) DNA 折纸上设计的V 形探针检测目标RNA[84]; (b) 字母图案的DNA 折纸用于SNP 检测[85]; (c) DNA 四面体探针检测目标DNA[87]; (d) AuNP 二聚体检测染料分子的SERS 信号[88]; (e) AuNP 四聚体对SERS 信号的单分子水平定点、定量检测[89]Fig.5.FNAs used for single molecule sensing: (a) Detection of the target RNA by hybridization with V-shaped probe stretched from a DNA origami[84]; (b) SNP detection with alphabetic patterned origami structures [85]; (c) recognition of the target DNA with a DNA tetrahedral structured probe [87]; (d) DNA origami-templated AuNP dimers for SERS[88]; (e) DNA origami-templated tetrameric Au nanoclusters for quantizing single-molecule SERS[89].
利用框架核酸组装AuNPs 构建的纳米等离激元结构同样适合检测单分子, 这主要是由于两个相邻粒子的局部表面等离激元耦合, 共振波长的光照射在粒子之间产生强电磁场, 使得位于纳米粒子间隙中等离激元“热点”处分子的荧光信号或拉曼散射强度增强[91].Lohmüller 课题组[88]构建了一个等离激元纳米天线, 将两个40 nm 的AuNPs 固定在刚性DNA 折纸块的两侧, 两个AuNPs 间隔距离即为折纸块的厚度(6 nm), 之后对嵌入在DNA折纸热点区域的分子进行了表面增强拉曼光谱(surface enhanced raman spectroscopy, SERS)测量, 发现分子的拉曼信号强烈增强(图5(d)).在随后的工作中, 他们通过光热诱导收缩将两个AuNP之间的间隙减小到1—2 nm, 从而进一步增强间隙处的场效应, 并定量描绘出热点处单个分子的SERS 信号强度的增强随AuNP 间距的函数变化[92].Fang 等[89]进一步开发出适用于更大粒径AuNPs定位组装的普适性方法, 他们将一组80 nm 的AuNPs锚定在DNA 折纸框架上的特定位点, 形成菱形四聚体纳米金团簇, 在金团簇的间隙热点处产生具有类Fano 共振特性的强电磁场, 他们将特定数量的染料贩子精确定位在此热点处, 首次实现了对SERS信号的单分子水平定点、定量检测(图5(e)).
3.3 分子装载与输运
将动态DNA 核酸框架作为基础, 还可以设计出单分子、单颗粒水平的输运机器, 构建纳米装配线.研究人员设法在DNA 折纸上设计精准可控的DNA 行走机器, 使其按指令沿特定的纳米装配线行走和搬运货物.依照链置换反应原理, Gu 等[93]最早设计了一个可进行单个AuNP 输运的DNA行走机器(图6(a)).而Qian 课题组[94]设计的DNA机器人不仅可以拾取货物, 还可执行复杂的货物分拣任务.在电场的驱动下, Kopperger 等[95]在DNA折纸平台上设计的DNA 纳米机械手臂可进行快速旋转, 并成功运送荧光基团和金纳米棒(图6(b)).磁场同样可以驱动DNA 分子机器, Lauback 等[96]将磁珠修饰到一个包含杠杆、转子和铰链的刚性DNA 折纸结构上, 通过施加旋转磁场, 纳米转子可以以2 Hz 的频率连续旋转.
利用DNA 折纸作为可控释放平台是研究的另一项热点.基于pH 变化来调控货物释放是一种常用的手段, Ijäs 等[99]设计并组装了一种可重构的DNA 折纸纳米胶囊, 利用Hoogsteen 键配对形成的DNA 三链结构作为闩锁, 通过急剧的pH 变化来控制胶囊结构的可逆开/关, 实现胶囊空腔内功能分子运载、封装等过程.Burns 等[100]构建的pH 响应的中空DNA 折纸立方盒, 可对功能蛋白封装、转移及在活细胞中释放.另一种调控手段是利用适配体的特异性识别作用, Li 等[97]将核蛋白的适配体序列作为紧固链, 使负载有凝血酶的DNA 管状折纸结构在核蛋白高表达的肿瘤血管内皮细胞表面富集、打开并暴露出凝血酶, 从而激活肿瘤部位的凝血功能导致肿瘤部位坏死并抑制其生长(图6(c)).除了响应外界刺激的可控释放, 框架核酸还可在DNA 双链上直接结合药物分子, 在体内实现有效释放.Wiraja 等[98]研究发现四面体DNA 折纸框架作为载体可以稳定、有效地渗透进真皮层并释放药物, 使药物积累和肿瘤抑制效果都得到提升(图6(d)).
3.4 纳米光学应用
利用框架核酸精确排布金属纳米颗粒、量子点和荧光团等功能部件, 对于创建具有定制光学特性和特殊功能的高级纳米光学系统至关重要, 比如荧光成像、光波导和手性光学系统等.Chen 等[101]通过调控DNA 四面体框架顶点处荧光染料分子之间的距离和排列方式创造性地构建了一个光流体激光器装置, 显著提高了激光效率(图7(a)).同样基于荧光分子的可控组装, 框架核酸在荧光成像方面存在应用潜力, 并由此发展了一种DNA-PAINT(DNA point accumulation for imaging in nanoscale topography)超分辨成像技术.荧光标记的短DNA 寡核苷酸(成像链)与DNA 纳米结构上延伸的靶互补链(对接链)动态结合和解离, 产生随机荧光闪烁, 之后进行单分子定位, 可实现DNA 纳米结构的超分辨成像[102](图7(b)).这种DNA-PAINT 技术具有纳米级的空间分辨率, 在此基础上发展了Exchange-PAINT 方法, 采用多条成像链对不同位点分别成像, 可表征多种靶标的空间结构.
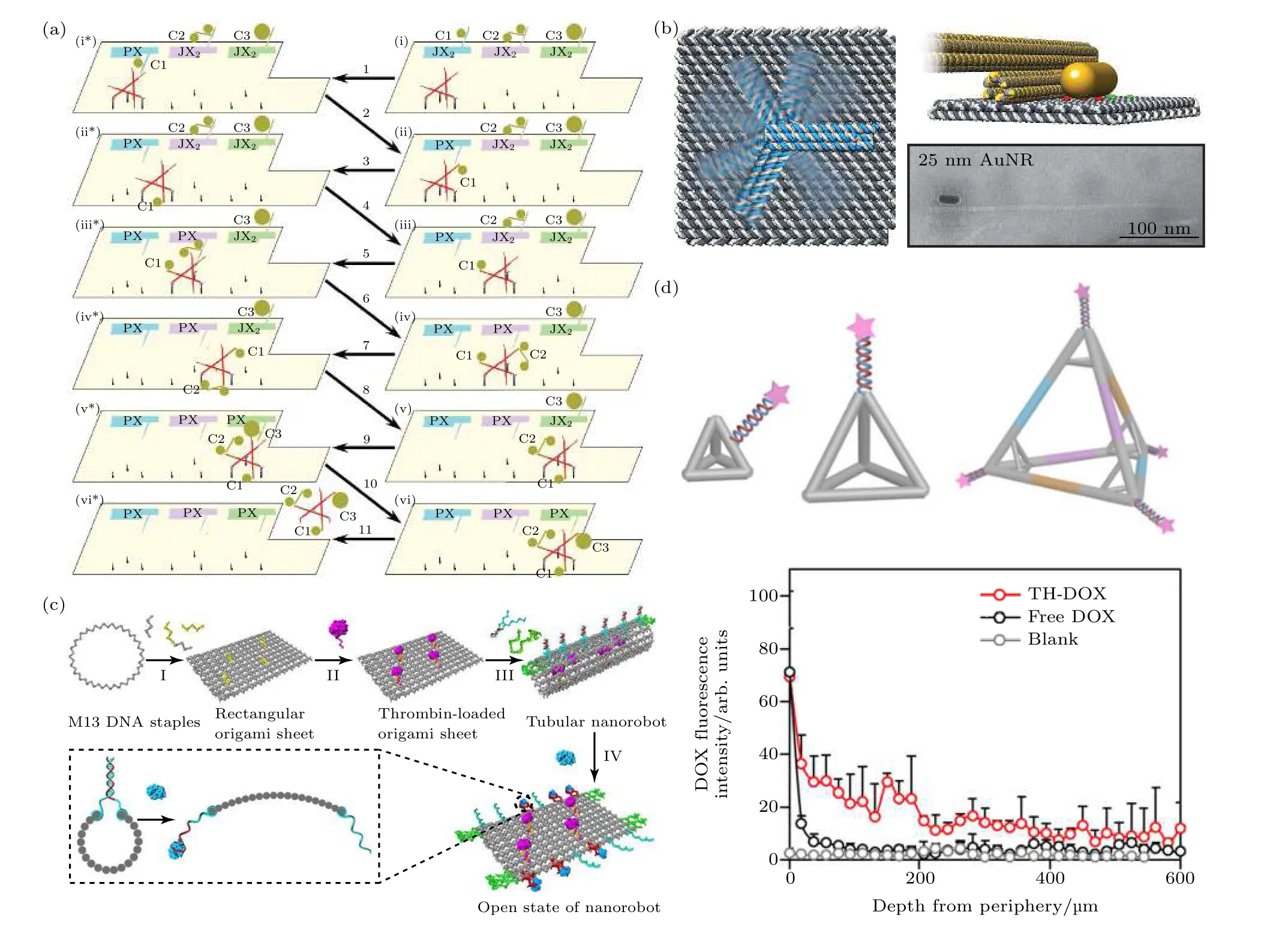
图6 框架核酸用于分子装载和输运 (a) DNA 折纸上的分子装配线[93]; (b) 电场驱动DNA 纳米机械臂旋转并使金纳米棒运动[95]; (c) 凝血酶功能化的DNA 纳米机器人[97]; (d) 3 种用于经皮给药的DNA 四面体结构[98]Fig.6.FNAs used for cargos loading and transporting: (a) Molecular assembly line on DNA origami[93]; (b) electrically actuated rotation of a nanorobotic arm, moving a gold nanorod[95]; (c) DNA origami nanocapsule actuated by changing pH[97]; (d) 3DNA tetrahedrons for transdermal drug delivery[98].
前文介绍了基于框架核酸构建纳米等离激元结构应用于单分子传感, 这些结构同样可应用于纳米光波导的构建.在金属纳米粒子链中当粒子间的间隙小于粒子半径时, 相邻纳米粒子的等离子激元产生强烈的共振耦合, 此时通过光与纳米粒子的相互作用就可限制、引导和操纵低于衍射极限的光,即光波导.利用框架核酸将AuNPs 定向自组装成线性阵列, 通过改变颗粒的空间排布和相邻颗粒间距可控制共振耦合频率[107], 若进一步改变中心AuNP 的位置, 就可形成可切换的等离子波导, 并在35—50 nm 传播范围内存在能量转移[103](图7(c)).但这种链波导每传播50 nm 就会产生10—20 dB的能量损耗, 为减少传播损耗, Gür 等[108]将AuNPs的间距减小到2 nm, 因而在62 nm 深亚波长的限制下将传播损耗降低到0.8 dB (每50 nm).这种基于框架核酸的组装等离子体波导的技术有望实现微米长的传播长度, 使其朝信息技术、传感电路和量子光学等领域迈出一大步.
利用框架核酸改变金属纳米离子的空间排布,可以实现手性光学性质的调控.其中, 球形AuNPs由于自身的各向同性便于无定向定位而被广泛应用.Kuzyk 等[104]利用24 个螺旋的DNA 折纸结构构建了左手和右手螺旋排列的AuNP 纳米线, 左旋和右旋都显示相应的手性信号(图7(d)).Ding课题组[109]设计的人工手性等离子体由矩形折纸上手性排布的最少数量的AuNPs 四聚体构成, 且具有显著的圆二色性.一些更具挑战性的手性等离子体结构可通过各向异性的金纳米棒进行组装.Lan 等[110]在二维DNA 折纸模板两侧设计“X”形排列DNA 捕获链用以定位金棒, 并进一步组装成金棒螺旋, 该结构可以产生增强的手性.为构筑更复杂的人工三维手性结构, Man 等[105]借助四面体DNA 折纸模板组装金纳米棒, 成功构建了11 种不同空间构型的手性等离子体超分子(图7(e)).在动态手性调控方面, Zhou 等[106]设计金纳米棒在DNA 折纸上进行循序渐进的定向行走, 即时产生动态的光学响应信号 (图7(f)).
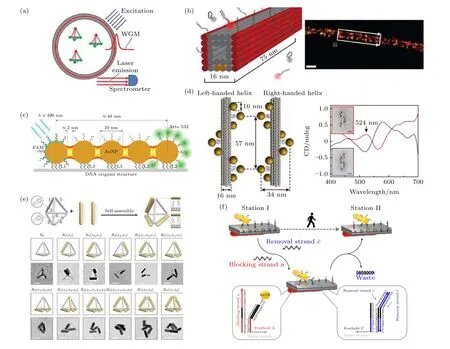
图7 框架核酸的纳米光学应用 (a) 基于DNA 四面体的光流体激光器实验装置[101]; (b) DNA-PAINT[102]; (c) DNA 折纸上线性排列的AuNPs 产生光波导[103]; (d) AuNPs 在DNA 折纸上的左旋和右旋排列[104]; (e) 四面体DNA 折纸组装的金纳米棒手性超分子[105]; (f) 金纳米棒在DNA 折纸上的动态行走[106]Fig.7.FNAs used for nanophotonics: (a) Optofluidic lasers based on a DNA tetrahedron[101]; (b) DNA-PAINT [102]; (c) waveguide on the line of AuNPs arranged by a DNA origami[103]; (d) AuNP helices on DNA origami[104]; (e) tetrahedral DNA origami-templated plasmonic metamolecules[105]; (f) Au nanorod walking on DNA origami[106].
3.5 纳电子器件
纳电子器件是另一个重要的研究方向, 框架核酸的原子级精准度在此方面具有重大的应用潜力[111].例如, 框架核酸组装结构可以作为高分辨率、低成本纳米制造的理想掩模板, 与纳米光刻技术结合,有望解决纳电子器件精细制造的难题.将DNA 模板置于基底上, 利用直接金属沉积[112]或阴影沉积[113]等方法, 可成功制得纳米级分辨率的规则图案或线性沟槽.为实现DNA 折纸掩膜到SiO2基底的直接图案转移, Diagne 等[114]利用DNA 折纸调节SiO2表面HF 气相刻蚀的反应速率, 合理控制反应条件, 成功将一个小于10 nm 的孔从DNA折纸转移到SiO2基底上(图8(a)).
框架核酸还可通过自下而上组装聚合物、金属、碳纳米管(carbon nanotube, CNT)等电子材料来构造纳电子器件.Gothelf 课题组[115]合成了一种具有潜在导电性的刷状DNA 共轭聚合物线,将其固定在DNA 折纸上, 构建了U 形、线性等多种图案, 该方法原则上可以制造纳米尺度任意形状的电子或光学导线(图8(b)).利用DNA 瓦块作为支架, Tapio 等[116]成功将三个AuNPs 组装成单电子晶体管, 在4.2 K 到室温范围内都能表现出库仑阻塞行为(图8(c)).CNT 在高性能、高能量效率晶体管领域具有很大的应用潜力, Sun 等[117]开发了一种对半导体CNT 空间受限整合的超分子组装方法, 以DNA 折纸砖块为模板, 通过对DNA 包覆的CNT 进行排列, 成功构建了间距为10.4 nm的平行CNT 阵列(图8(d)).利用这种组装策略,他们成功制备了固态多通道半导体CNT 场效应晶体管, 关键传输性能指标提高了10 倍以上[118].此外, 在构建CNT 器件时, CNT 错位会引起杂散导电路径, 产生错误的逻辑功能, 为了校准CNT,Zhang 等[119]利用球形核酸介导CNT 在DNA 折纸表面定位, 最终实现CNT 精确对准和平行排布.
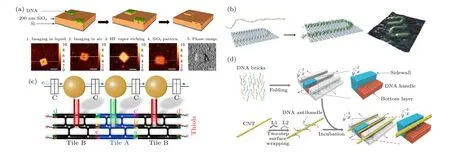
图8 框架核酸构建纳电子器件 (a) DNA 折纸到SiO2 基底的直接图案转移[114]; (b) 聚合物线在DNA 折纸上形成的“U”形图案[115]; (c) DNA 瓦块组装AuNP 构建单电子晶体管[116]; (d) DNA 折纸模板制备高度致密的CNT 平行阵列[117]Fig.8.FNAs used for nanoelectronics: (a) Pattern transferring from DNA origami into SiO2[114]; (b) polymer binding to the DNA origami with a “U” shaped pattern[113]; (c) DNA tile-templated single electron nanoelectronics[116]; (d) CNT alignment based on trench-like DNA templates[117].
3.6 信息处理器件
生命遗传信息的编辑与存储是核酸作为生物大分子的本质属性, 因此利用人工设计的框架核酸进行信息处理, 有望产生新的突破.早在1994 年,图灵奖获得者Adleman[120]开创性地用一维线性DNA 自组装解决含有7 个顶点的有向哈密顿路径问题, 证明DNA 分子具备强大的计算能力.多年来, DNA 的高度并行计算、高密度信息存储、运算速度快、能耗低等优势在DNA 计算[121]、信息存储、加密等领域得到了充分发掘[25].基于精准定位的特性, DNA 折纸可以作为逻辑电路的框架模板来控制DNA 分子或其他电路元件的排列.Liu 等[122]开发了一个DNA 分子计算器, 利用两条特定的DNA 序列分别代表一个数字输入进行乘法运算,输出库是预先定义的, 当两条输入链与溶液中的输出库混合时, 它们会绑定到一条正确的输出链上,形成稳定的三向连接, 再经转换器处理, 使计算结果在DNA 折纸上以数字形式显示出来(图9(a)).
将DNA 计算的概念融入DNA 纳米器件的设计中, 有助于在单分子尺度上开发智能DNA 分子机器.Wickham 等[126]设计的DNA 马达可在包含4 种可能路径的轨道网络中根据添加的外部指令或自身携带的信息来选择分路行走.在更为复杂的DNA 动态反应网络中, Chao 等[123]开发的DNA单分子巡航机器人系统成功在二维折纸上迷宫路径中找到了正确解.迷宫路径包含唯一入口、唯一出口、几个转角和交叉点, 体系中大量的单分子机器人通过杂交链式反应进行平行深度优先搜索共同寻找所有可能路径, 最后筛选出迷宫的正确路径, 并用原子力和超分辨率显微镜对这条路径进行单分子成像(图9(b)).
另一方面, 近年来掀起了对核酸信息存储安全问题的研究热潮.早期研究人员利用DNA 的序列信息发展DNA 隐写术, 将编码为DNA 链的信息隐藏在大量随机DNA 中[127].随着结构DNA 纳米技术的发展, 人们开始挖掘DNA 纳米结构在信息加密领域的应用潜力.Zhang 等[124]开发了一种DNA 折纸加密系统, 发送者将传递的信息加密成类似盲文的点阵图案, 通过骨架链杂交若干生物素修饰短链的组合将这些图案进一步加密.而接收者获得骨架链折叠的密钥及其他订书钉链, 将骨架链折叠成含正确的折纸图案, 并在原子力显微镜下识别, 最终明文信息被逐字解密成二进制数并解码(图9(c)).为了提高分子信息编码的安全级别,Song 课题组[128]利用可重构的DNA 折纸多米诺阵列编码信息, 阵列构象随密钥链的添加而改变,使加密的数据被转换成可见模式, 并进一步设计基于toehold 链置换反应的防伪方法, 以防止编码信息被解码和篡改[125](图9(d)).

图9 框架核酸构建信息处理器件 (a) DNA 折纸分子计算器[122]; (b) DNA 单分子巡航机器人解迷宫[123]; (c) DNA 折纸加密系统[124]; (d) DNA 折纸多米诺阵列编码信息[125]Fig.9.FNAs used for information processing: (a) DNA origami calculator[122]; (b) single-molecule DNA navigator for solving maze on the 2 D origami[123]; (c) DNA origami cryptography system[124]; (d) DNA origami domino array for coding information[125].
4 展 望
随着硅基集成电路技术后摩尔时代的来临,“自上而下”的光刻等制造手段已经无法满足电路高集成化及电子设备小型化的工艺要求, 人们迫切需要开发原子尺度的精准构筑技术, 以打破现有的技术限制, 推动未来科技和高端原件制造产业的发展.目前对于原子制造的研究主要集中在原子数可控团簇、低维材料原子级精准制造等凝聚态物理的研究方向, 而实现真正意义上的原子制造须以原子作为研究对象, 对其进行捕获、定向移动和精准定位.借助框架核酸这样一个结构有序、高度可编程、精准空间定位的可寻址平台, 有望实现原子级的精准构筑.例如, 通过化学修饰的手段将原子或螯合原子的配合物精准定位到框架核酸, 从而捕获数量可控的原子并进行有序排布; 利用动态DNA分子机器调控原子的定向移动过程, 有望对单个至多个原子进行可编程地操控.随着框架核酸的结构复杂性和空间尺寸进一步提升, 框架核酸作为一种高度集成化的组装平台, 有望推动原子自下而上构建更大规模、更为复杂的组装体, 并进一步制备功能器件或新材料.此外, 基于框架核酸对纳米颗粒、蛋白质、聚合物等功能基元精准组装的特性,赋予原子制造某些特殊性能, 或可推动其在生物医学、催化、纳米光学、量子科学和材料等交叉领域的发展, 例如基于框架核酸定位原子阵列制备高度集成的量子芯片, 发展更快运行速度和更高存储能力的量子计算机, 或制备以框架核酸为载体的高负载率、高活性的单原子催化剂.当然, 目前利用框架核酸进行原子制造的设想还停留在初步探索阶段, 一些机理和关键技术问题还急需解决, 比如原子制造过程中单原子、多原子的作用规律, 原子尺度的量子效应问题, 从原子到宏观的物理或化学特性调控问题等.此外, 适用于框架核酸原子制造的表征手段目前有限, 还需寻找新方法或通过荧光、酶催化反应间接表征框架核酸上的单个或多个原子.我们也期望框架核酸理念能促进原子水平操纵与表征技术的发展, 通过进一步的学科交叉, 降低与应用段接轨的技术门槛.

