INMT基因在肝癌中的表达及其机制研究
刘新会 罗越 林俊豪 庄雪芬 魏莉
肝癌是常见的恶性肿瘤之一,其发病率在全球癌症中居第5位,死亡率居第2位,每年可导致近700 000人死亡,严重威胁着人类的健康[1],而其中超过50%的病例发生在我国,最新的流行病学研究发现肝癌已经居我国60岁以下男性恶性肿瘤发病的首位[2-3],因此深入探究肝癌的发病机制,寻找其潜在的诊断和治疗靶点尤为重要。吲哚胺N-甲基转移酶(indolethylamine N-methyl-transferase,INMT)是甲基化的催化剂,主要与大脑活动相关[4]。近年来研究认为INMT与肿瘤的发生、发展相关,INMT在多种恶性肿瘤中均表达下调[5-7]。但INMT在肝癌中的表达和临床意义尚未见报道,其在肝癌中的作用及其机制仍不明确。癌症相关数据库(TCGA和GEO)可以通过对肝癌大样本的数据分析,为INMT基因在肝癌发生、发展中作用机制的深入研究提供客观依据。本研究采用TCGA肝癌数据库和肝癌基因芯片GSE102079,旨在应用生物信息学分析探讨INMT在肝癌发生、发展中的作用及可能的机制,为肝癌的防治提供生物信息学依据。
1 材料和方法
1.1 肝癌数据库来源 本研究从TCGA(https://www.cancer.gov/about-nci/organization/ccg/research/structuralgenomics/tcga,TCGA-LIHC)[8]下载肝癌RNA测序数据库及患者临床资料,包含371例肝癌患者的癌组织以及50例肝癌患者的癌旁组织。GSE102079肝癌基因芯片数据从 GEO(https://www.ncbi.nlm.nih.gov/geo/)下载[9],芯片共包含152例肝癌患者的癌组织和91例肝癌患者的癌旁组织。
1.2 INMT在肝癌数据库的表达分析 采用R语言分析TCGA数据库中INMT在癌组织和癌旁组织的表达情况,同时分析在GSE102079中INMT在肝癌组织和癌旁组织的表达差异,并采用Graphpad进行统计学分析和绘图。
1.3 INMT表达与肝癌患者的生存分析 采用Kaplan-Meier Plotter数据库分析[10],其中收录了肝癌患者总生存期(overall survival,OS)、无进展生存期(progress free survival,PFS)和目的基因的表达信息,在数据库中设置条件:Gene:INMT;Split patients by:Auto select best cutoff,分析数据库内INMT表达与肝癌患者OS和PFS的关系。
1.4 INMT相关基因的富集分析 采用R语言分析TCGA中INMT的相关基因,结果发现和INMT显著相关的基因303个。采用R语言分析GSE102079中INMT的相关基因,结果发现和INMT显著相关的基因421个。其中178个基因同时在两个数据库中为INMT相关基因(INMT-related genes,IRGS)。采用 DAVID(the Database for Annotation,Visualization and Integrated Discovery,http://david.abcc.ncifcrf.gov/)[11]在线分析数据库对IRGs进行KEGG通路[12]和GO通路[13]富集分析。
1.5 INMT相关基因的蛋白网络构建 STRING数据库是分析基因或蛋白相互作用的在线检索工具,将IRGS导入数据库,进一步构建INMT相关基因的蛋白网络,提供INMT相关蛋白的相互作用分析。
2 结果
2.1 INMT在肝癌组织和癌旁组织中的表达比较 TCGA数据库显示,INMT在肝癌组织中表达显著低于癌旁组织(P<0.01,图1a);进一步分析发现,和50例配对癌旁组织相比较,肝癌患者的癌组织中INMT仍然显著低表达(P<0.01,图1b)。为了在不同肝癌数据库中验证INMT的表达,笔者对肝癌基因芯片GSE102079进行数据分析,结果显示和癌旁组织相比较,INMT在肝癌组织中的表达显著降低(P<0.01,图1c),这提示INMT可能在肝癌中发挥抑癌基因作用。
2.2 INMT基因高表达与肝癌患者预后的相关性分析为明确INMT基因能否作为肝癌患者预后的分子标志物,笔者采用Kaplan-Meier Plotter数据库分析INMT基因表达与肝癌患者生存期的关系,结果发现和INMT基因低表达组(110例)相比较,INMT基因高表达组(254例)OS显著延长(P<0.01,图 2a);和 INMT基因低表达组(176例)相比较,INMT基因高表达组(194例)PFS也显著延长(P<0.01,图2b)。这提示INMT基因高表达可能作为肝癌患者预后较好的标志物。

图1 INMT在不同肝癌数据库中表达(a:TGCA肝癌数据库;b:TCGA配对肝癌数据库;c:GSE102079肝癌数据库;**P<0.01;INMT为吲哚胺N-甲基转移酶)

图2 INMT高表达和低表达肝癌患者的总生存期和无进展生存期比较(a:总生存期;b:无进展生存期;INMT为吲哚胺N-甲基转移酶)
2.3 INMT在肝癌中发挥抑癌基因作用的分子机制研究 为进一步研究INMT在肝癌中发挥抑癌作用的分子机制,笔者分析发现TCGA和GSE102079中有178个INMT相关基因(IRGs)重叠(图3a)。KEGG通路富集分析表明,IRGS主要涉及PI3K-AKT信号通路、细胞黏附分子、血管平滑肌收缩、肿瘤信号通路等(图3b);GO通路富集分析发现,IRGs主要涉及细胞外基质组成、细胞黏附、血管形成等生物过程,细胞学组成分析显示这些基因参与细胞外基质、细胞基底膜、细胞外泌体等的组成(图3c)。分子功能的变化主要涉及结合肝素、胶原、钙、整合素等,还参与细胞外基质结构等。
2.4 构建肝癌中INMT相关基因的蛋白相互作用网络采用STRING构建INMT相关基因的蛋白相互作用网络(图 4),发现 COL14A1、FBLN1/2、PECAM1、EMCN、FLT4等在INMT相关基因的蛋白相互作用网络中发挥关键作用(图5)。采用STRING构建的INMT相关基因的蛋白相互作用网络为进一步探讨INMT在肝癌中发挥作用的机制研究提供生物信息学依据。
3 讨论
肝癌早期诊断具有一定的难度,大多数肝癌患者就诊时已是中晚期,已经失去根治性治疗机会,病死率一直居高不下,且世界范围内超过50%的病例发生在我国,因此深入探究肝癌的发病机制,探索肝癌早期肿瘤标志物具有重要的临床意义[14]。INMT和肿瘤发生、发展相关,研究发现INMT在多种恶性肿瘤中均表达下调[5-7,15-16]。但INMT在肝癌中的表达和临床意义尚鲜见报道,其在肝癌中的作用及其机制仍不明确。本研究分析了肝癌TCGA和GEO数据库,发现和肝癌患者的癌旁组织相比较,癌组织中INMT表达显著下降,这和INMT在前列腺癌、肺癌、胃癌中表达下调一致,说明INMT可能在多种恶性肿瘤中发挥抑癌基因的作用,并进一步为INMT基因在肝癌中的深入研究提供依据。
目前INMT基因在肝癌中的表达及与预后的关系尚不明确,而且INMT在肿瘤中发挥抑癌作用的机制仍缺乏深入研究。本研究进一步分析发现INMT高表达肝癌患者的OS和PFS显著延长,这表明INMT基因高表达可能作为肝癌患者预后较好的标志物,可能在肝癌中发挥抑癌基因作用。进一步对两个肝癌数据库中的INMT显著相关基因进行分析,筛选出178个重叠基因(IRGs)。为了进一步了解INMT相关基因,笔者对重叠基因进行进一步的KEGG和GO通路富集分析,发现IRGS主要涉及PI3K-AKT信号通路、细胞黏附分子、血管平滑肌收缩、肿瘤信号通路等;生物过程变化主要有细胞外基质组成、细胞黏附、血管形成等生物过程,细胞学组成分析显示这些基因参与细胞外基质、细胞基底膜、细胞外泌体等的组成。分子功能的变化主要涉及结合肝素、胶原、钙、整合素等,还参与细胞外基质结构等。PI3K-AKT信号通路在肝癌发生、发展中发挥重要作用,其可能通过促进肝癌细胞增殖、侵袭和转移发挥促癌基因作用[17-19]。而细胞外基质以及黏附分子等也参与肝癌侵袭转移等过程[20-21]。这提示INMT可能通过参与PI3K-AKT信号通路或调控细胞外基质以及黏附分子表达在肝癌中发挥抑癌基因作用,这为进一步探讨INMT在肝癌中的作用机制提供了初步思路。

图3 在肝癌中与INMT表达相关的基因富集分析(a:TCGA和GSE102079数据库中INMT相关基因重叠的Venn图;b-c:INMT相关基因的KEGG和GO富集分析;INMT为吲哚胺N-甲基转移酶)
除此之外,笔者采用STRING构建了肝癌中INMT相关基因的蛋白相互作用网络,发现COL14A1、FBLN1/2、PECAM1、EMCN、FLT4等在INMT相关基因的蛋白相互作用网络中发挥关键作用。COL14A1是细胞外基质的重要组成成分,而且和肿瘤进展和转移相关。研究表明COL14A1可以显著抑制食管癌患者的增殖和侵袭从而发挥抑癌基因作用[22-23]。笔者研究亦发现在肝癌中INMT和COL14A1表达呈显著正相关。研究表明FBLN1/2、PECAM1、EMCN都是血管内皮的重要黏附分子,参与肿瘤的血管生存和侵袭转移[24-26]。本研究中,笔者发现在肝癌中INMT表达和FBLN1/2、PECAM1、EMCN呈正相关,说明INMT可能通过调控肝癌微环境中黏附分子的表达,从而参与肿瘤血管形成和发生、发展。采用STRING构建INMT相关基因的蛋白相互作用网络为进一步探讨INMT在肝癌中发挥作用的机制研究提供生物信息学依据。
综上所述,在本研究中,笔者发现INMT在肝癌组织中显著低表达,INMT高表达与肝癌患者预后较好相关,可能在肝癌中发挥抑癌基因作用,INMT可以作为肝癌早期诊断和预后的一个治疗靶点。此外,通过富集分析和STRING构建INMT蛋白互作网络初步探讨了INMT在肝癌发挥作用的潜在机制。为深入探讨INMT在肝癌中发挥抑癌基因的具体分子机制,还需要进行一系列的实验来验证本研究团队的预测结果。
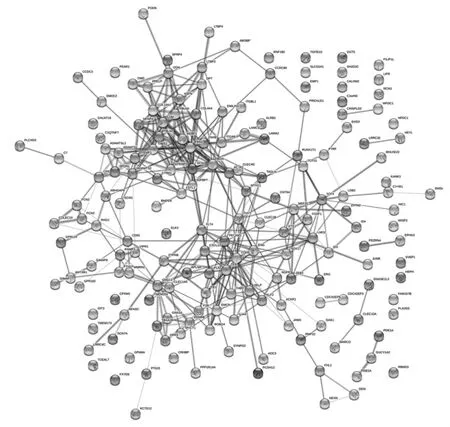
图4 INMT相关基因的蛋白相互作用网络(INMT为吲哚胺N-甲基转移酶)

图5 TCGA-LIHC 肝癌数据库中各基因与 INMT 相关性分析(a:COL14A1;b:FBLN1;c:FBLN2;d:PECAM1;e:EMCN;f:FLT4;INMT 为吲哚胺N-甲基转移酶)

