基于多任务神经网络模型的小肠淋巴瘤检测模型
谢 飞,王放舟,管子玉,段 群
(1.西北工业大学 计算机学院,陕西 西安 710129;2.西安电子科技大学 前沿交叉研究院,陕西 西安 710126;3.西北大学 信息科学与技术学院,陕西 西安 710127;4.咸阳师范学院 计算机学院,陕西 咸阳 712000)
原发性小肠恶性肿瘤是胃肠道恶性肿瘤的一种,其发生率仅占胃肠道恶性肿瘤的1%。传统的诊断方法主要依靠消化道钡餐透视,随着医疗影像的发展,CT和B超成为了其检测的主要方式[1-2]。过去,往往通过专家会诊的方式来对患者的CT图像进行分析,从而确定肿瘤的位置。但是由于医疗资源的不均衡,不同级别医院医疗从业人员水平的差异,导致一些患者无法得到有效的诊断而延误病情。近年来,基于深度学习的计算机视觉领域快速发展,越来越多医疗领域的人工智能研究开始出现。一方面医疗辅助诊断系统可以弥补一些基层医院因专业医疗人才匮乏、人才培养滞后而导致的漏诊或错诊,为患者治疗赢取时间,另一方面可以为医生的培养提供可靠的辅助工具。
目前,很少有人把目标检测的方法直接用于小肠淋巴瘤CT影像当中,但在其他疾病的诊断研究中,越来越多的检测方法被使用到。Yan等人提出了3DCE的网络结构,该结构利用了切片之间特征的空间关联性,将相邻切片按通道进行堆叠从而形成若干个三通道的数据模块,每个数据模块的中间通道含有标注的信息,这些数据模块送入网络将分别进行特征提取并进行特征融合,从而有利于模型检测效果的提升[3]。Li等人提出了MVP-Net网络模型,该模型结合了临床诊断的经验,将同一切片转化为不同窗口类型和窗口宽度的数据类型,并通过不同的分支将转化后的数据分别送入网络中进行特征提取和特征融合,有效提高了模型的检测精度[5]。Shao等人在FPN网络中加入了MSB模块,模块当中引入了膨胀卷积和注意力关注机制,提高了对病灶当中小样本的检测能力[4]。Ypsilantis等提出了ReCTnet网络模型,使用改进的resnet网络提取切片的特征,并使用LSTM来学习切片直接的关联特性,实验证明了利用这种关联特性可以显著提高肺结节检测的敏感度[6]。
以上研究虽然可以有效提高检测的精度,但是用于训练的数据均是有标签的含有病灶的数据,因此,在实际的应用过程当中会产生过高的假阳性率。本文为了减少假阳性率引入了正常人的数据作为训练样本,同时引入了分类模块剔除掉不包含小肠的切片。使用RetinaNet作为网络的主干结构,并在原始的FPN浅层的结构当中引入了分支结构用于提升对于微小病灶区域的检测精度。因为用于训练的数据较少,所以使用迁移学习的方式冻结浅层的参数特征,只对深层的参数进行微调。
1 相关工作
1.1 目标检测任务
目标检测是计算机视觉的主要任务之一,其主要包括两个方面:一是确定目标的位置,并用矩形框表示其范围;二是确定矩形框中目标的类别。主流的检测类型主要有两种,一种为一阶段方法,另一种为两阶段方法。一阶段方法如Yolo[8-10]系列和SSD[11],其主要思路是利用卷积神经网络直接对图片进行分类和回归,整个过程只需要一步。两阶段方法则是将检测过程分为两个阶段,第一个阶段是利用RPN或者selective search生成相应的候选框;第二个阶段则是利用这些候选框进行分类和回归,有代表性的模型则是RCNN[12]系列。两种方法在检测性能和检测速度方面各有优劣。近年来,随着RetinaNet[13]、FCOS[14]等方法的提出,一阶段检测模型不仅速度更快,而且在检测精度方面毫不逊色于两阶段的检测模型,因此得到了更加广泛的应用。
1.2 病理图像数据及数据预处理
数据来源于上海瑞金医院34名含有标注的小肠淋巴瘤患者CT切片,53名不含有标注的患者切片,以及16名正常人的切片。每名患者的切片数据是由相应的设备对病患的腹部进行逐层的断面扫描,得到的一系列沿z轴堆叠的三维图像。每名患者切片平均数量为40张左右,多的数量可以达到80张。每名患者切片当中有一部分是有淋巴瘤的切片,这一部分切片经过了医生的标注,我们需要把这一部分切片抽取出来,作为后续有监督学习训练的数据。切片数据为DICOM格式,医生标注肿瘤数据之后,产生的标注文件的格式也为DICOM格式。每个DICOM文件除了包含医学影像信息之外,还含有相应的DICOM数据头部信息,每个数据头部信息中包含了标识的相关信息。其中,病人淋巴瘤切片信息和医生标注信息中都含有唯一的标示信息,这些标示信息可以建立起病人淋巴瘤数据和医生标注数据一对一的映射关系,通过这一关系可以将经过医生标注后的淋巴瘤数据从患者的原始切片数据当中分离出去。图1展示了患者的原始切片和医生标注后的切片。使用Pydicom库分离淋巴瘤数据,最终得到医生标注好的淋巴瘤切片数据共954张。对于没有进行标注的患者数据,只抽取出其小肠部分的切片,用于后续的迭代训练。为了便于后续迁移学习的训练,将所有的DICOM格式文件转化为JPG格式,转化后的图片大小为512×512。

A 患者原始切片 B 医生标注切片
因为带有标注的数据相对较少,我们采用了随机裁剪、旋转、扭曲形变、添加高斯噪声这4种数据增强方式。最后选取80%的数据作为训练集,其余的数据作为测试集。为了更好评估实验得到的效果,采用多次实验取平均值的方式来评估实验的结果,每次实验会相应更新训练数据和测试数据。
1.3 模型预训练和迁移学习
深度学习是数据驱动的,好的结果往往需要大量的数据进行训练。小肠淋巴瘤作为一种小众疾病,一方面其数据量较少,并且涉及到病人隐私,数据收集困难;另一方面,由于小肠淋巴瘤的大小和形状不规则,在标注过程中会耗费医生大量的时间和精力。迁移学习是指将模型从其他样本学习到的浅层特征迁移到现有的任务中,迁移学习不仅可以加快模型的收敛,还可以提高模型检测的效果。ImageNet[19]是一个计算机视觉系统识别项目,是目前世界上图像识别最大的数据库,包含了1 000个类别的1 000多万张图片。目前,大多数检测模型的主干网络都是在该数据集下先进行预训练,然后再迁移到下游任务当中。但是,对于医疗影像数据来说,由于图像特征之间的差异,使用ImageNet做迁移学习并不能获得良好的效果。DeepLesion数据库[7]由美国国立卫生研究院临床中心(NIHCC)的团队开发,是通过从他们自己的图片存档和通信系统中挖掘历史医学数据而开发的。该数据库中的图像包括多种病变类型,如肾脏病变,骨病变,肺结节和淋巴结肿大。一共有30 000多个病人的数据,并且对于每个病人的病灶区域都有详细的标注。本文使用DeepLesion数据集作为预训练,将得到的参数迁移到下游任务中,冻结浅层的参数并对深层的参数进行微调。
2 模型结构
本文使用RetinaNet作为检测网络的主要结构,RetinaNet[13]是由何凯明提出的单阶段检测模型。模型主要由特征金字塔(FPN)和Focal loss两部分组成。特征金字塔(feature pyramid network,FPN)网络通过自底向上的卷积层来提取特征,对于不同的特征层,底层的特征层包含更多的位置信息,而顶层的特征层则包含更加丰富的语义信息。将顶层包含丰富语义信息特征层通过上采样与底层信息进行融合,可以使得模型对于不同尺度目标的检测更具有鲁棒性。特征的融合是逐层迭代进行的,这种方式可以使得底层特征层包含每个特征层的信息。
本文网络结构如图2所示。其中:C2、C3、C4、C5是从模型主干网络中取出的不同深度的特征图;P5、P4、P3、P2是自顶向下通过逐层特征融合得到的特征图;P6、P7由C5通过卷积和池化操作得到。本文还在特征金字塔的底部又增加了一个分支结构P1用于加强对小目标的检测。这个分支结构没有进行特征融合,由C2通过一个1*1的卷积层和一个3*3的卷积层得到。最终将P1~P7分别送入ClassificationModel和RegressionModel进行分类和回归预测,得到的结果会和全局分类模块得到的结果一并送入到筛查模块当中,输出最终的检测结果。

图2 网络结构图
2.1 全局分类子网络
目标检测模型往往选用含有标注信息的图片进行训练,而在医学辅助诊断实际应用过程当中,送入检测模型的切片往往是复杂的,包含人体各个横切面的组织和器官,而肝脏、心脏、肺部、骨盆等器官不在检测范围之内。因此,这些数据没有参与相应的训练,在检测过程中会产生大量的假阳性结果,影响实际的用户体验。本文构建了一个用于全局分类的子网络模型,模型在切片级别对样本进行分类,只保留潜在患病区域切片的结果,滤除掉其他包含非发病区域组织和器官的切片。全局分类子网络结构如图3所示。

图3 全局分类子网络结构图
网络将C5层作为模型的输入,C5层包含了网络中最丰富的语义信息,不需要和底层的特征进行融合。经过一个2*2的最大池化层和3*3的卷积层将通道数降低为256,之后分别进行最大池化和平均池化,将得到的结果在通道上进行拼接,再采用两层全连接层并进行dropout防止出现过拟合,最后,通过Softmax得到每一类别的检测分数。
2.2 网络主干结构和SEmodel
模型使用ResNeXt101[17]作为网络的主干结构。ResNeXt在ResNet的基础上借鉴了Inception的思想,将模型分成了多个分支结构再进行融合。不同于Inception的是,模型的每个分支结构都是完全相同的,其基本结构如图4所示。

图4 ResNeXt基本网络结构图
SEmodel[18]可以通过学习为不同的通道赋予相应的权重,对重要的特征赋予更高的权重,同时抑制不重要的特征。其主要分为两个操作,Squeeze和Excitation。Squeeze利用全局平均池化的方式,将单个通道的空间特征用其平均值来表示,如式(1)所示,
(1)
其中:Fsq表示uc到zc之间的映射关系;uc表示在c通道上的特征图;zc表示对该通道特征表征的数值。将一个大小为H×W×C的特征图经过Squeeze操作变成了1×1×C大小。之后会经过Excitation操作,通过一个瓶颈结构融合不同通道的特征,最终通过Sigmoid得到每个通道的注意力分数(attention factor)。其主要结构由图5所示。

图5 SEmodel主要结构图
2.3 网络损失函数
与通用的目标检测任务相比,医疗影像数据的病灶检测区域往往较小,特别是对于小肠淋巴瘤数据来说,其病灶个数为1到2个,且比较集中,因此,在实际检测过程当中就会产生大量的负样本。Focal Loss可以减少因正负样本失衡而造成的检测精度下降的问题。其公式如式(2)所示,
(2)
其中:y′表示模型预测的概率得分;α和γ表示相应的平衡因子,α用来平衡正负样本的重要性,γ降低易于分类样本的损失,使模型可以更加关注于较难分类的样本。
模型的总体损失函数如式(3)所示,
Loss=Lf1+Lf2+βLf3。
(3)
损失由3部分构成:Lf1代表模型在局部切片的分类损失;Lf2代表模型的回归损失;Lf3代表模型对整张切片的分类损失;β代表平衡因子,用于平衡不同损失之间的权重。
3 实验与分析
3.1 未标注病人数据的利用
本实验共有34位带有标注的病人数据。其中26例病人用于训练,8例病人用于测试。此外还有58位医生确认患病,但没有进行标注的患者数据。本文使用已有标注数据训练好的模型,对未标注的患者数据进行检测,检测得到的结果中选取置信度高的做为模型的训练数据。为了保证得到结果的准确性,本文选择3个不同网络结构的模型用于检测,通过比较各个模型的结果,以手动筛查的方式得到最终的结果,筛查主要依据同一病人相邻切片的关联性。模型经过了两次迭代,每次迭代包括了上次训练通过筛查得到的数据,最终得到了标注数据603张,得到的数据将全部用于训练。本文使用改进的Retinanet模型分别测试了只使用标注数据和使用标注数据和未标注数据训练在测试集上的结果,使用平均精准度(average precision, AP)衡量训练结果,如表1所示。可以看到使用未标注数据在测试集的结果AP值提高了3%。

表1 两种不同数据集训练的结果
3.2 正常人数据的利用
模型用于训练的数据均为患者数据,正常人数据中一些基础疾病在CT影像上的表征(如小肠囊肿、肠梗阻、肠道溃烂)与淋巴瘤的特征具有高度的相似性,缺乏正常人数据的训练会使得检测系统在实际应用过程中产生过高的假阳性。检测模型需要相应的标注来指导模型的训练,正常人数据当中缺乏相应的标注。一种方法可以通过本文提出的全局分类子网络来区分含肿瘤数据和未含肿瘤数据,但是因为肿瘤是细粒度信息,使用深层网络提取到的特征进行分类可能会丢失这些信息。而且含有病灶的切片包含小肠部分,不含病灶区域除了有包含小肠部分的切片,还有包含其他器官的切片,所以会使模型更倾向于将包含小肠的部分认定为有病灶。本文将正常人数据分为了3种类型,如图6所示。

A 类别0 B 类别1 C 类别2
类别0主要包含肝脏等器官,类别1包含小肠区域,类别2包含骨盆区域。3种类别在结构上具有较大的差异性,便于分支网络进行分类。同时因为类别0和类别2不会出现病灶区域,且其中包含的器官相对较大,因此在训练过程中不产生相应的锚框用于分类和回归。对于类别1的数据,为了减少anchor的数量,同时使模型的训练更具有针对性,将对anchor的产生范围进行限定。正常人的数据中主要关注的是那些易于分类错误的数据,使用检测模型找到这些容易分错数据的boundingbox,将这些boundingbox按一定比例放大并作为标签送入网络训练。在模型进行损失函数的计算过程中IOU大于0.5的,我们认为其为负类,将其值设定为0,而IOU小于0.5的,将其设定为1,也就是这些anchor不参与后续的训练。
3.3 实验设置
本文的实验数据包含3部分:带有标注的小肠淋巴瘤数据;没有标注的小肠淋巴瘤数据;正常人的数据。其中带有标注的小肠淋巴瘤数据为904张,通过上文方式得到的伪标注的小肠淋巴瘤数据603张,正常人的数据1 350张。为了保证实验结果的准确性,伪标注数据将全部用于训练,其余数据80%用于训练和验证,20%的数据用于测试。
本文使用Pytorch作为实验的框架,采用改进版的RetinaNet模型。模型选用Adam(adaptive moment estimation)优化器,初始学习率设置为0.000 01。使用Pytorch的工具类ReduceLROnPlateau对训练过程的学习率进行调整。其中,patience设置为4,即模型连续四次迭代损失不下降就降低学习率。每次更新学习率时,将新的学习率变为上一次迭代学习率的80%。损失函数中,将 设置为0.25,设置为2,β设置为0.12。为了增加对小目标的检测精度,将anchor尺度设置为[0.25,0.5,1.2]。
3.4 实验评价指标
因为测试数据包含患者数据和正常人数据,因此实验的评价指标共分为两部分,召回率(recall)和假阳性率(false positive rate)。召回率表示样本中正例(指肿瘤)被正确预测的比例。主要是用于评价模型对于肿瘤的敏感程度。肿瘤的置信度设置为0.6,即置信度大于0.6的都会被认定为阳性。IOU则设置为0.5,召回率用式(4)表示为
(4)
假阳性率则表示样本中被错误认为为正例的个数占整个样本的比例,主要反映正常人被误诊的概率,用式(5)来表示,
(5)
其中:TP表示被正确的识别为正样本;FN表示被错误的识别为负样本;FP表示被错误的识别为正样本;N表示样本的总数。
3.5 实验结果与分析
Re代表了初始的RetinaNet网络模型,由ImageNet来作为预训练。其中:G代表使用间质瘤作为预训练数据;D代表使用DeepLesion作为预训练数据;All代表同时使用两者作为预训练数据。接着比较了两种不同的对正常人数据训练方式得到的结果。M1表示将正常人也按照检测标注的方式建立标签,只是将相应的boundingbox标签所有数字设置为0,标签的类别设置为负类;M2方式与3.2节提到的方法相同。S代表网络中加入了SE模块;C代表加入了分支模块;P1代表加入了P1层。实验结果如表2所示。

表2 实验结果
从结果上来看,使用DeepLesion数据集和间质瘤数据集比使用ImageNet数据集做预训练在召回率上有17.6%左右的提升。DeepLesion数据集没有间质瘤效果好可能原因是间质瘤的部分特征与小肠淋巴瘤有高度的相似性。模型中加入了SE模块后,模型召回率提高了约6.4%,说明SE模块提高了模型特征的提取能力。Re+All+SE有最好的召回率的结果,但其在正常人测试数据集上有很高的假阳性。Re+All+S+M1中尽管模型的假阳性率得到了显著的下降,但是模型的召回率也下降了约19.2%,可能原因是过高的负类样本对实际的检测效果产生了影响。Re+All+S+C+M2降低了假阳性的同时,还能保证模型检测的精度。最终的改进检测模型进一步提高了检测模型的检测精度并降低了模型的假阳性率。
图7为RetinaNet与本文提出的方法在小肠淋巴瘤患者检测的对比图。蓝色矩形框为医生实际的标注,根据检测分数的高低,分别用红色、黄色和绿色矩形框表示模型得到的结果。图7中A列为RetinaNet模型得到的结果,B列为本文提出改进模型得到的结果。通过对比可以看出改进模型得到的结果相比于原模型,其置信度分数会更高,同时其检测得到的结果与医生标注的结果重合度会更高。在小目标检测方面,改进后的模型对小目标检测效果更加显著。
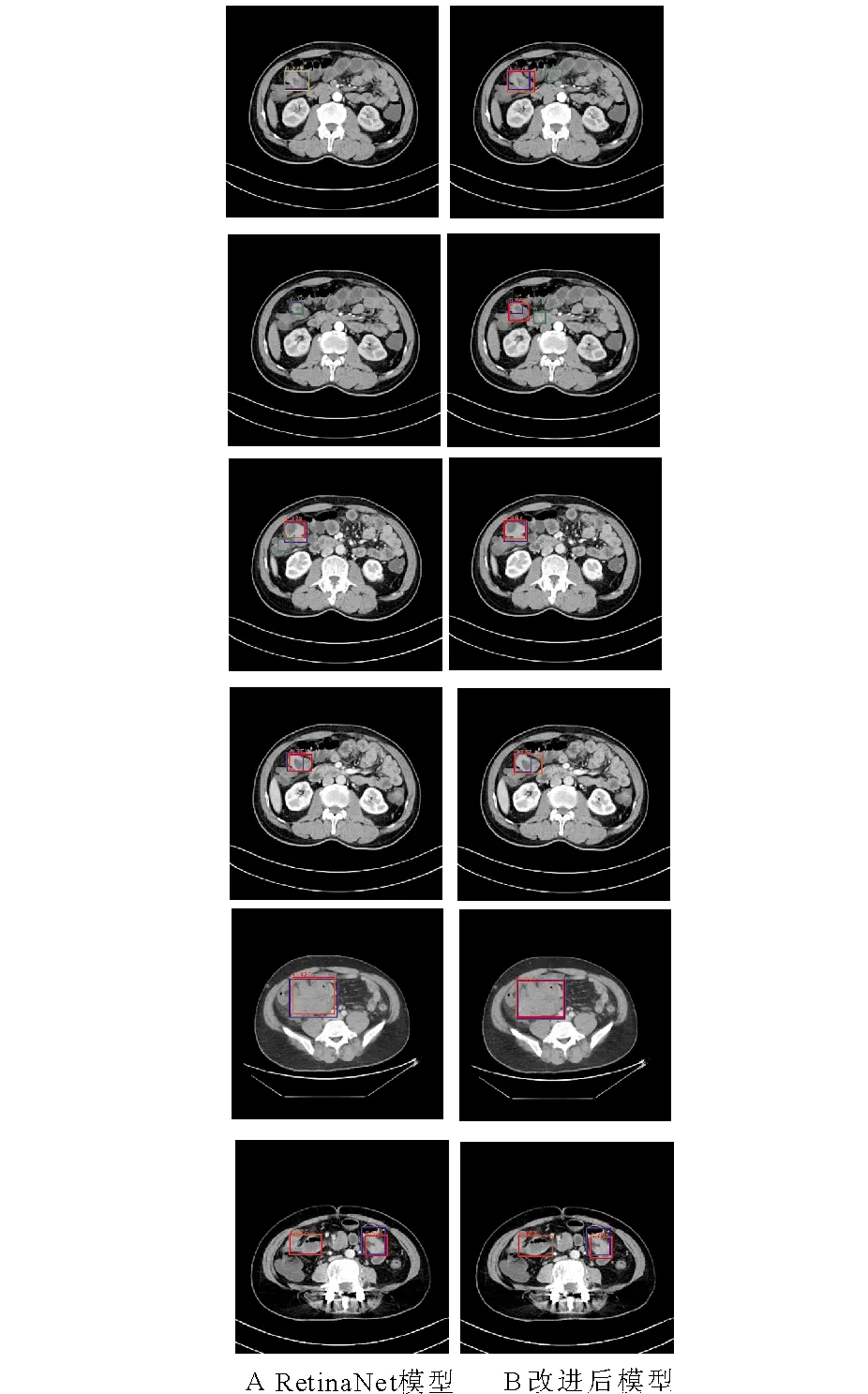
图7 RetinaNet与改进模型试验结果对比图
4 结论
本文提出了基于RetinaNet的多任务检测模型,该检测模型在原模型的基础上加入了分类分支模块,该模块可以用于剔除数据中非小肠的区域,同时便于引入无 boundingbox的正常人小肠区域的数据,用于降低模型的假阳性率。结合本文提出的对于无标注数据的使用策略,通过这些方式,在保证模型检测灵敏度的同时,降低了模型检测结果中产生的假阳性率。为解决肿瘤形态差异大,部分肿瘤较小的问题,本文在原有模型的基础上加入了SE模块,并在网络底层增加P1层用于提高对肿瘤的检测精度,实验表明本文的策略可以达到良好的效果。

