苏尼特羊不同生长时期肌肉组织lncRNA的差异表达分析
王瑞雪 刘佳森 李蕴华 成立新 岳林芳 马万山 赵艳红
(1.内蒙古自治区农牧业科学院 畜牧研究所,呼和浩特 010031;2.内蒙古农业大学 动物科学学院,呼和浩特 010018)
lncRNA作为2012年度新兴起的生命科学研究之一[1],在生物众多生命活动中起着至关重要的调控作用,是继mRNA和miRNA之后最热门的分子生物学研究领域之一。lncRNA 是一类长度均>200 nt且不具备或很少具有蛋白质编码能力的 RNA[2],并缺乏明显的开放阅读框。最初有研究认为 lncRNA 是基因组转录过程中形成的“噪音”,在生物体内不发挥任何生物学功能[3]。但近期的研究表明,lncRNA可参与细胞核和细胞质中的 X 染色体失活、染色体结构改变、基因印记、转录激活/沉默和核内运输等关键生命活动[4]。Cesana等[5]通过研究人和小鼠的骨骼肌生长发育时发现,特异性表达的长链非编码 RNA linc-MD1可以结合miR-133和miR-135来调控肌肉特异性基因MAML1和MEF2C的表达。Zhu等[6]研究发现,lnc-mg 通过结合miR-125b调控骨骼肌发生的关键调节因子-IGF2的蛋白质水平,进而调节肌肉的生长发育。近年来,在畜禽领域逐渐开展了lncRNA的相关研究。Ren等[7]在猪的胎儿滋养层鉴定出一种新的lncRNA,发现其在猪胎儿骨骼肌中表达上调,并显著影响猪胎儿骨骼肌的发育。Li等[8]通过RNA-seq技术和生物信息学分析方法,在鸡基因组中鉴定出281种与鸡骨骼肌发育相关的新lncRNA。
绵羊肌肉发育相关的lncRNA研究正处于起步阶段,刘瑞凿等[9]利用转录组测序技术对乌珠穆沁羊和特克赛尔羊腓肠肌进行lncRNA测序,发现lncRNA TCONS_00102859只在特克赛尔羊腓肠肌中表达,并且随着发育其表达量逐渐增高。进一步研究显示,TCONS_00102859的过表达会引起肌肉分化相关基因MyoD和MyoG表达上调,MSTN表达下调,证明TCONS_00102859可能在成肌细胞的增殖分化中发挥作用。吴明明[10]通过对胚胎期绵羊90 d和120 d背最长肌样本进行高通量测序,共鉴定出1 114个lncRNA,其中有466个差异表达lncRNA,并进一步证明,差异表达lnc-SEMT在肌肉发育中起正调控作用。Li等[11]对新疆地方品种-哈萨克羊进行了系统的分析,在哈萨克羊出生前后骨骼肌的比较中鉴定了1 566个lncRNA,其中差异表达的lncRNA为404个。苏尼特羊,也叫戈壁羊,主要产于内蒙古自治区锡林郭勒盟苏尼特左旗和右旗,是经过长期的自然选择和人工选育而形成的地方良种。苏尼特羊肉肌肉饱满,瘦肉率高,蛋白质含量高、脂肪低,膻味轻,深受国内外用户的好评。苏尼特羊肌肉发育相关lncRNA的研究尚未有相关报道,因此,本研究以苏尼特羊肌肉为研究对象,对120 d胎儿和5月龄羔羊背最长肌进行RNA-seq测序分析,以期为解析苏尼特羊肌肉发育的分子机制提供理论基础,同时为分子辅助育种提供参考。
1 材料与方法
1.1 样品采集及RNA提取
试验样本所用群体来自内蒙古苏尼特右旗某苏尼特羊原种场。对妊娠120 d苏尼特羊和5月龄苏尼特羊羔羊静脉注射40 mg/kg BW的戊巴比妥钠麻醉,屠宰,采集120 d苏尼特羊胎儿背最长肌样本3个、5月龄羔羊背最长肌样本2个,样品采集后立即置于液氮中,然后储存于实验室-80 ℃冰箱中。利用Trizol法分别提取胎儿120 d和5月龄羔羊背最长肌的总RNA。利用1%的琼脂糖凝胶电泳分析RNA降解程度以及是否有污染,Nanodrop 2 000检测RNA的纯度及浓度。
1.2 文库构建、测序及转录本的拼装
按照测序要求进行lncRNA文库构建,由于采集样本的日期不同,首先对3个120 d 胎儿背最长肌RNA样本进行等量混池测序(MFH_MLcn),然后对 5月龄羔羊背最长肌进行并行测序(S5BZ_MS)。测序平台为Illumina HiseqTM2500。利用fastx_toolkit(V0.0.13)软件对测序得到的Raw reads进行质控过滤。使用Tophat 2(V2.0.9)将过滤后的测序序列比对到绵羊参考基因组,应用cufflinks软件对比对后的bam文件进行转录本的拼接。
1.3 差异表达lncRNA的筛选和靶基因预测
首先通过5步筛选法进行基本的筛选:转录本exon个数筛选、转录本长度筛选、转录本已知注释筛选、转录本表达量筛选和编码潜能筛选,然后利用CPC、CNCI和pfam蛋白结构域及PhyloCSF分析方法进行编码潜能的分析,最后取交集鉴定新的候选lncRNA。从候选lncRNA中筛选出差异表达的lncRNA,差异表达分析使用Cuffdiff软件,差异表达分析标准为∣log2(Fold change)∣≥1且q<0.05。使用Cuffdiff(http:∥cufflinks.cbcb.umd.edu/manual.html#cuffdiff)软件对筛选所得到的lncRNA进行定量分析,从而得到各样品中lncRNA的FPKM信息,对差异表达lncRNA的cis作用靶基因设定为lncRNA的上下游100 kb邻近基因。
1.4 GO富集和KEGG通路分析
利用GOseq软件[12]对预测的靶基因进行GO分析。使用KOBAS软件[13]进行KEGG 通路分析。
1.5 lncRNA的荧光定量PCR验证
随机选取8个lncRNA,进行实时荧光定量PCR反应。内参基因选择β-actin,引物均由生工生物工程(上海)股份有限公司合成,引物序列信息如表1所示。荧光定量PCR反应总体系20 μL:上、下游引物(10 μmol/L)各0.8 μL,cDNA 2 μL,ddH2O 6 μL,TB Green Premix Taq Ⅱ 10 μL,Rox reference dye Ⅱ 0.4 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 10 s, 60 ℃ 30 s,共40个循环,3个重复。用2-ΔΔCt法计算羔羊lncRNA的相对表达量,并与转录组测序结果进行比对验证。

表1 lncRNA引物序列信息Table 1 lncRNA primer sequence information
2 结果与分析
2.1 原始数据分析与质控
对测序得到的原始数据(Raw reads)进行过滤分析,如表2所示,2组样本的总Clean reads 的大小分别为17.94 G和 25.27 G。Q20和Q30的比列均在95%以上,表明测序质量可靠。
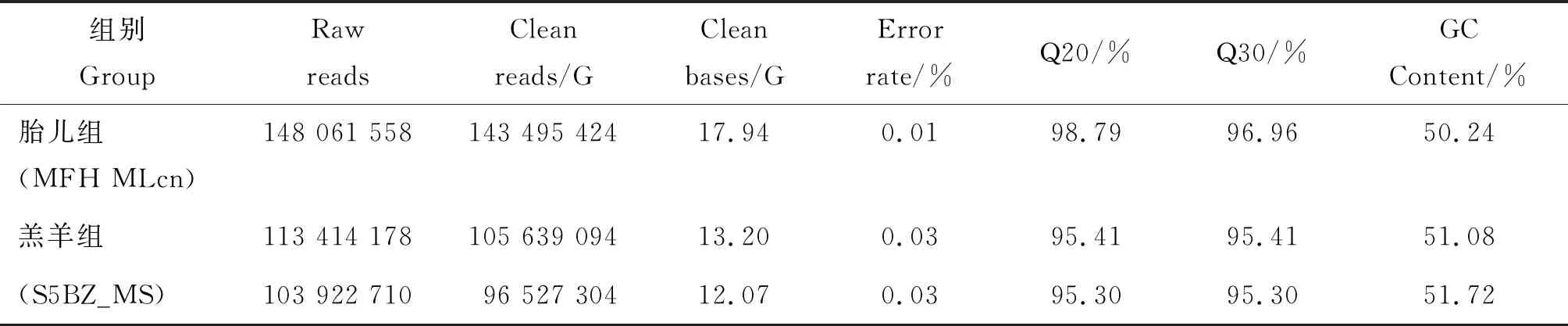
表2 数据产出质量情况Table 2 Statement of data output quality
2.2 序列比对与拼接
质控后的Clean reads比对到绵羊参考基因组结果见表3。结果显示,120 d胎儿组(MFH_MLcn) 样本中Clean reads的86.42%能定位到基因组上的测序序列,4.67%在参考序列上有多个比对位置,reads在参考序列上唯一比对率为81.75%;羔羊组(S5BZ_MS)样本中的Clean reads比对到基因组上的比例均为80%以上,比对到参考序列上多个比对位置的比例为5.3%~5.64%, reads在参考序列上唯一比对率为75.06%~77.17%。
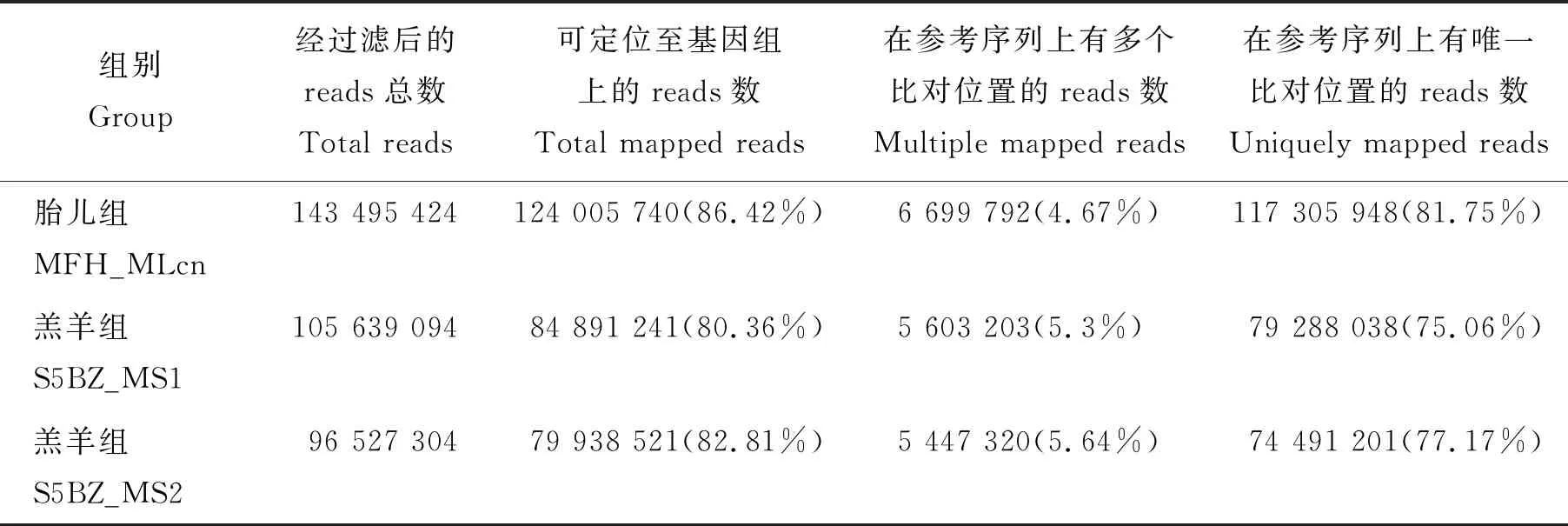
表3 Reads 与参考基因组比对Table 3 Summary of the alignment of reads with the reference genome
2.3 lncRNA转录本的鉴定
利用cufflinks对转录片段进行拼接,初步筛选出10 3307个转录本。然后对这些转录本进行分步筛选,最终鉴定到1 112个候选lncRNA转录本,其中包括586个已知lncRNA和526个新lncRNA,详细结果见图1(a)。进一步对候选lncRNA进行分类,如图1(b)所示,基因间型lncRNA占90.3%,反义型lncRNA 占9.7%。未发现有内含子型的lncRNA。
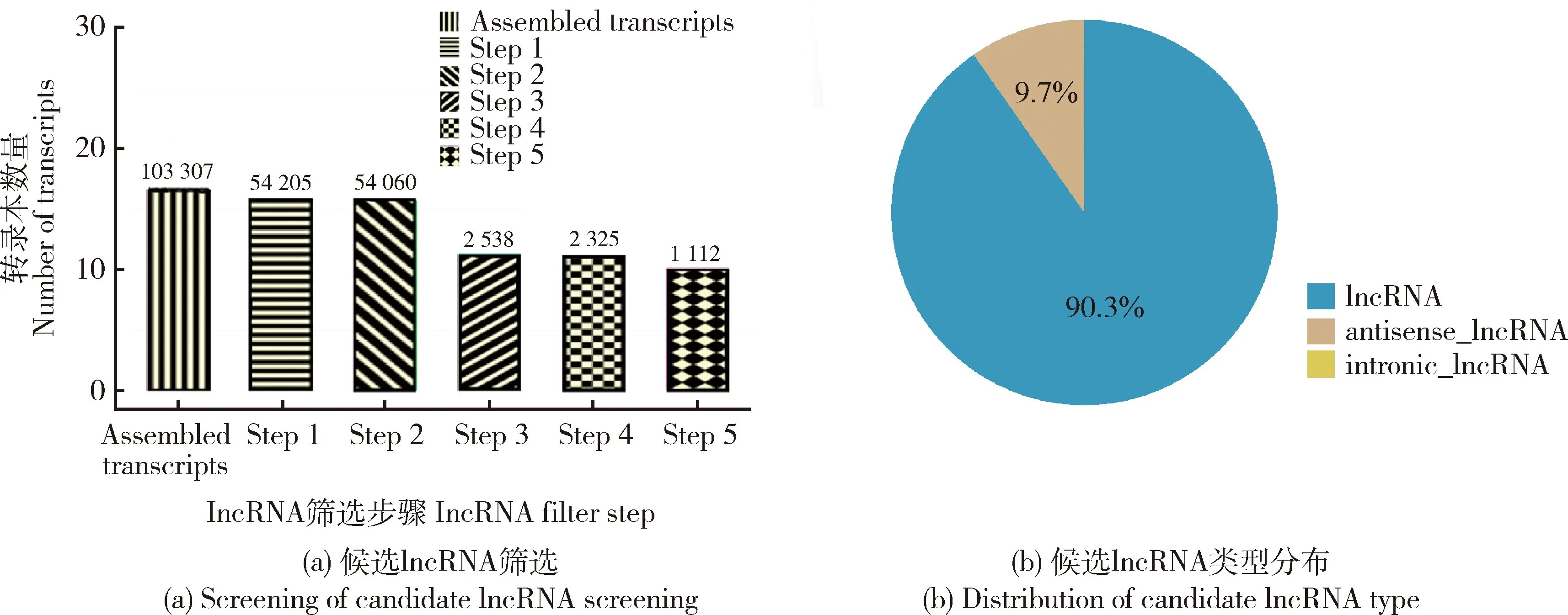
图1 候选lncRNA 筛选与类型分布Fig.1 Screening and type distribution of candidate lncRNA
2.4 lncRNA结构与特征分析
为研究苏尼特羊肌肉中 lncRNA 的基本特征,对比分析了 lncRNA 与 mRNA的转录本长度、外显子数、ORF长度及转录本表达水平。结果显示,与mRNA相比,lncRNA长度区间在200~3 000 bp的lncRNA数为890个,占总数lncRNA的80.0%。每个lncRNA平均长度为2 517 bp,平均2.5个外显子数(图2(a))。包含2个和3个外显子的lncRNA数占所有lncRNA的87.3%(971个)(图2(b))。对lncRNA的开放读码框(ORF)进行分析发现,多数lncRNA的长度在500 bp以内,平均数为123 bp。如图2(c)所示,mRNA的ORF长度明显大于lncRNA。这一结果也提示lncRNA在转录及转录后等表观调控方面起着重要的作用。对测序获得的 lncRNA与 mRNA 的表达量进行比较,发现mRNA的整体表达量明显高于lncRNA(图2(d))。
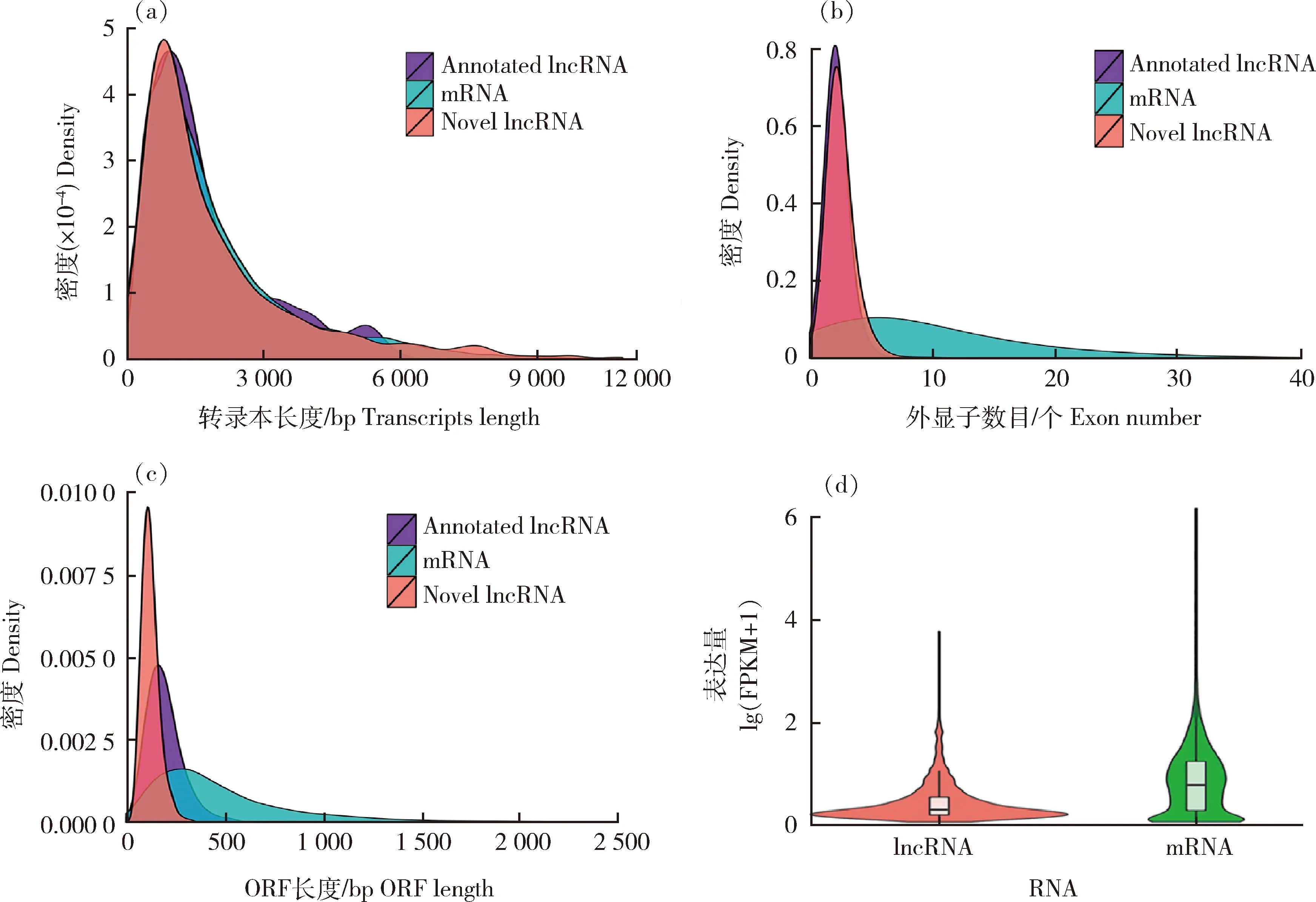
(a) lncRNA与mRNA的转录本长度;(b) lncRNA与mRNA外显子数目;(c) lncRNA与mRNA ORF长度;(d) lncRNA与mRNA转录本表达量;纵坐标中的密度表示对应横坐标轴数值的因子数/检测因子的总数;FPKM表示每百万测序碱基中每千个转录子测序碱基中所包含的测序片段数。(a) lncRNA and mRNA transcripts length; (b) lncRNA and mRNA exon number; (c) lncRNA and mRNA ORF length; (d) lncRNA and mRNA transcript expression level; Density in Y-axis represent the number of factors corresponding to the value of the abscissa axis/the total number of detection factors; FPKM represent number of sequenced fragments contained in every thousand transcripts of sequenced bases per million of sequenced bases.图2 lncRNA与mRNA的结构特征比较分析Fig.2 Comparative analysis of structural characteristics between lncRNA and mRNA
2.5 lncRNA的差异表达分析
在|log2(Fold change)|>1和q<0.05条件下,2组样本之间的差异表达结果见图3。结果显示,在胎儿120 d组和 5月龄羔羊组中获得399个差异表达的 lncRNA,其中212个表达上调,187个表达下调。
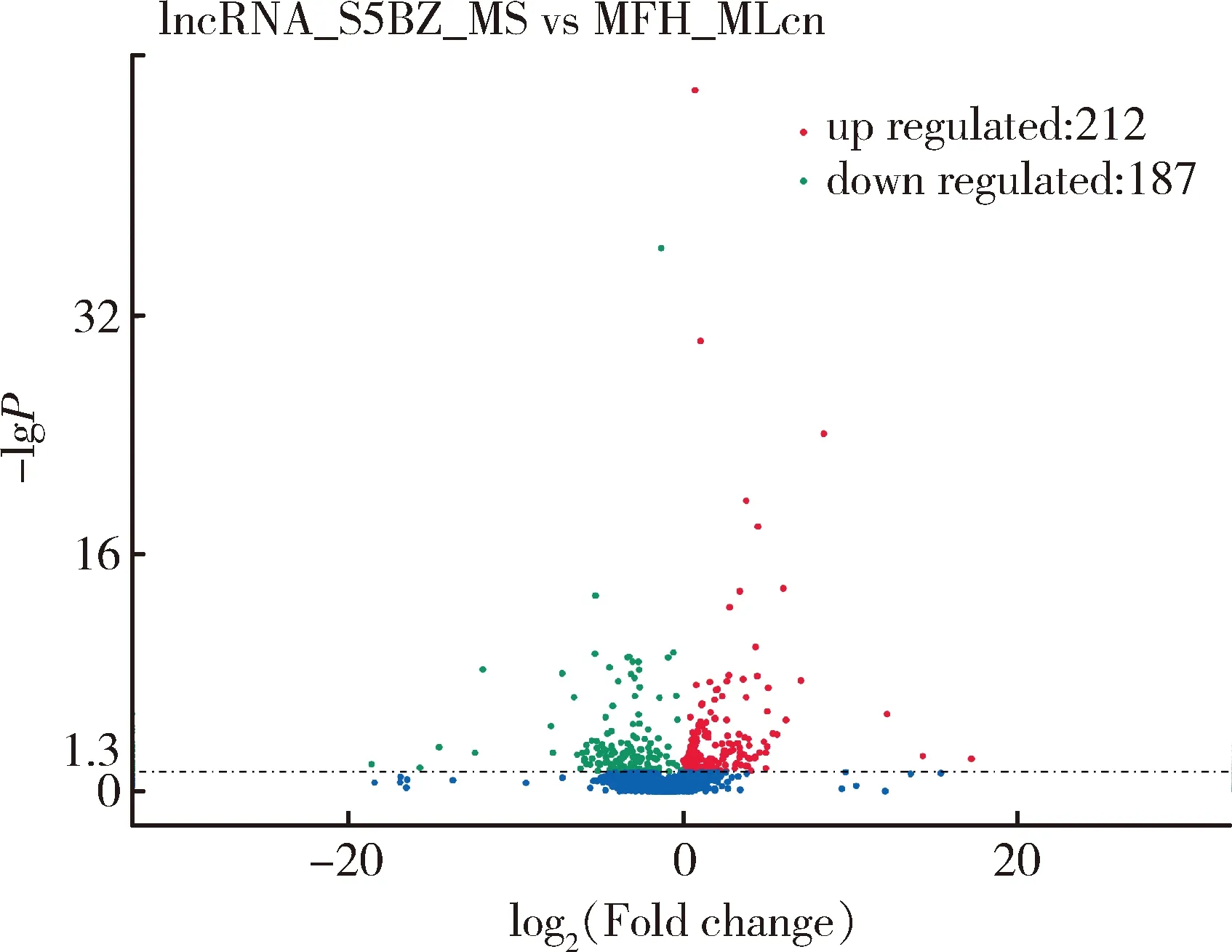
图3 差异表达 lncRNA 火山图Fig.3 Volcano map of differentially expressed IncRNA
对差异表达的lncRNA进行层次聚类分析来判断差异转录本在不同实验条件下的表达模式,由图4(a)可知,羔羊组样本间聚类在一起,与胎儿组差异明显。此外,胎儿组上调基因对应下调基因,而羔羊组与胎儿组基因表达模式有不同,羔羊组lncRNA的表达模式下调基因多于上调基因。之后,两两之间的差异表达组之间进行了韦恩图(Venn)分析,结果如图4(b)所示,共有11个lncRNA在所有差异表达组中被检测到。差异表达lncRNA在不同样本中的表达量见图5,值得注意的是ENSOART00000028118、ENSOART00000028271、LNC_000424和LNC_000978转录本在2个组中都有较高表达。此外,LNC_000422在胎儿120 d中有表达,在5月龄羔羊中无表达;LNC_000942在5月龄羔羊中有表达,在胎儿120 d中无表达。ENSOART00000028384和LNC_000421在胎儿120 d中的表达量更高;LNC_000311和LNC_000719在5月龄羔羊中的表达量更高。
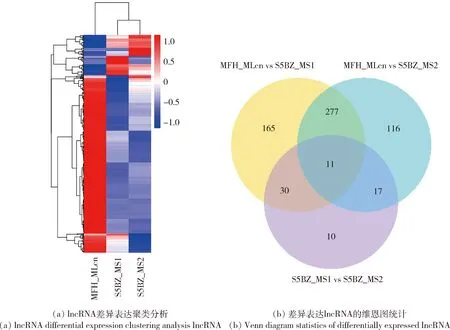
图4 差异表达lncRNA分析Fig.4 Differential expression analysis of lncRNA
2.6 lncRNA 的荧光定量PCR验证
为进一步验证测序结果,并检测其在绵羊肌肉中的表达,随机选择8个差异表达的lncRNA,其中LNC_000074, LNC_000196, LNC_000588在转录组数据中表现为上调,LNC_000233, LNC_000276,LNC_000408, LNC_000586和 LNC_000649在转录组数据中表现为下调,并通过qRT-PCR检测其在胎儿及羔羊肌肉中的表达。由图6可知,经 qRT-PCR 验证,LNC_000074,LNC_000196和LNC_000588均上调表达,与RNA-seq测序结果上调一致;LNC_000223,LNC_000276,LNC_000408,LNC_000586和LNC_000649均下调表达,与RNA-seq测序结果下调一致。证明实时荧光定量结果与转录组测序结果表达趋势一致。
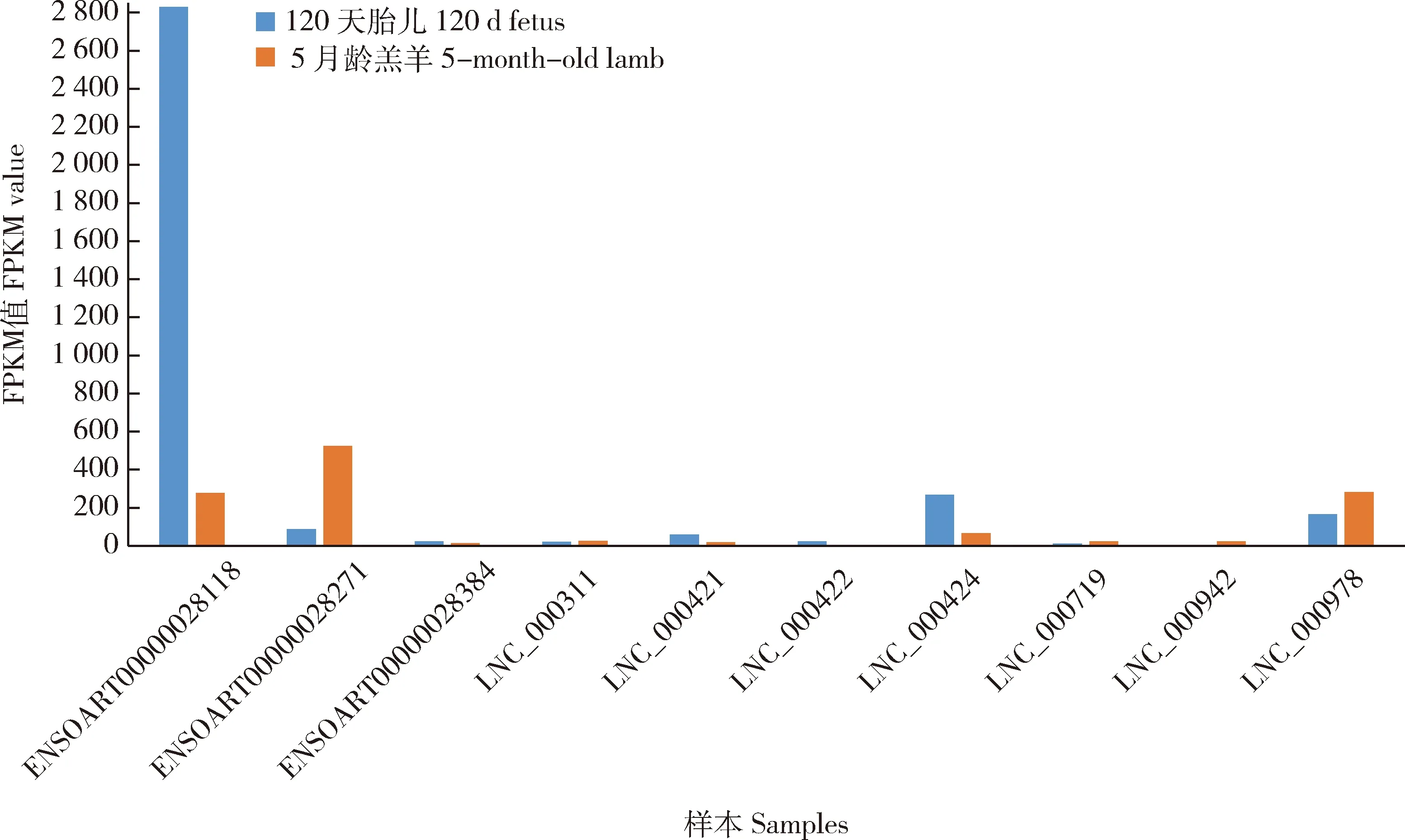
图5 差异表达lncRNA在不同样本中的表达量Fig.5 Expression of differentially expressed lncRNA in different samples
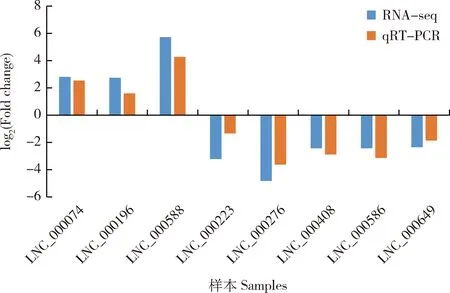
图6 差异表达lnRNA qRT-PCR与RNA-seq比较分析Fig.6 Comparison analysis of differentially expressed lncRNA by qRT-PCR and RNA-seq
2.7 差异表达lncRNA靶向mRNA预测
利用lncRNA和mRNA的定位来预测lncRNA在顺式调控关系中的潜在目标。将共同位置的阈值设置为lncRNA的上下游100KB,结果表明,626个差异表达lncRNA中503个lncRNA存在邻近基因。对应于1 273个靶基因。其中多个lncRNA具有共同的靶基因,如LNC_000421和LNC_000422的靶基因为RTL1等。也有lncRNA具有多个可变剪切靶向多个编码基因,如编码基因TNNT1,PPP1R12C,EPS8L1是LNC_000311的潜在目标等,并且这些lncRNA呈现不同方向的调控作用。
2.8 靶基因的功能注释分析(GO和KEGG)
对这些邻近基因进行功能注释分析,GO结果分别从生物学过程(Biological process,BP)、细胞组分(Cellular component,CC)和分子功能(Molecular function,MF)3个条目进行分类。结果如图7显示,MFH_MLcn vs S5BZ_MS差异表达lncRNA的靶基因显著富集到了60个GO term条目中,其中,涉及代谢过程的调控,生物合成BP,蛋白结合GC以及免疫反应MF等相关。另外,在其他GO term条目中也发现了包括肌肉结构发育、骨骼肌细胞分化,骨骼肌收缩、横纹肌细丝,肌肉组织发育等与骨骼肌发育紧密相关的GO term,提示这些lncRNA在肌肉发育的不同阶段起着重要的调控作用。
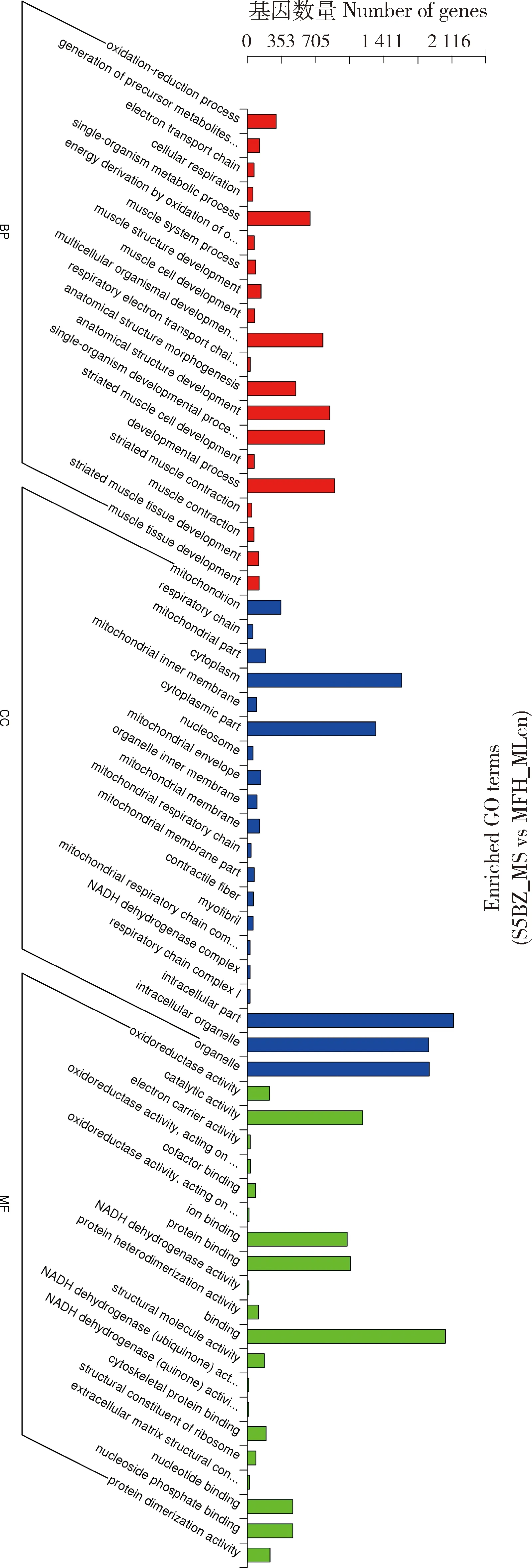
图7 差异lncRNA 的GO富集分析Fig.7 GO enrichment analysis of differential lncRNA
对这些靶基因基因进行KEGG通路分析,结果如图8所示,这些靶基因主要涉及Wnt信号传导途径、Notch信号通路、Gap连接和甲状腺激素合成等,都可能与骨骼肌的生长,发育和功能有关。这些结果表明lncRNA可能在骨骼肌的发育和生长中起重要作用,可根据以上功能注释信息对这些lncRNA开展肌肉发育相关的研究提供了重要的信息。
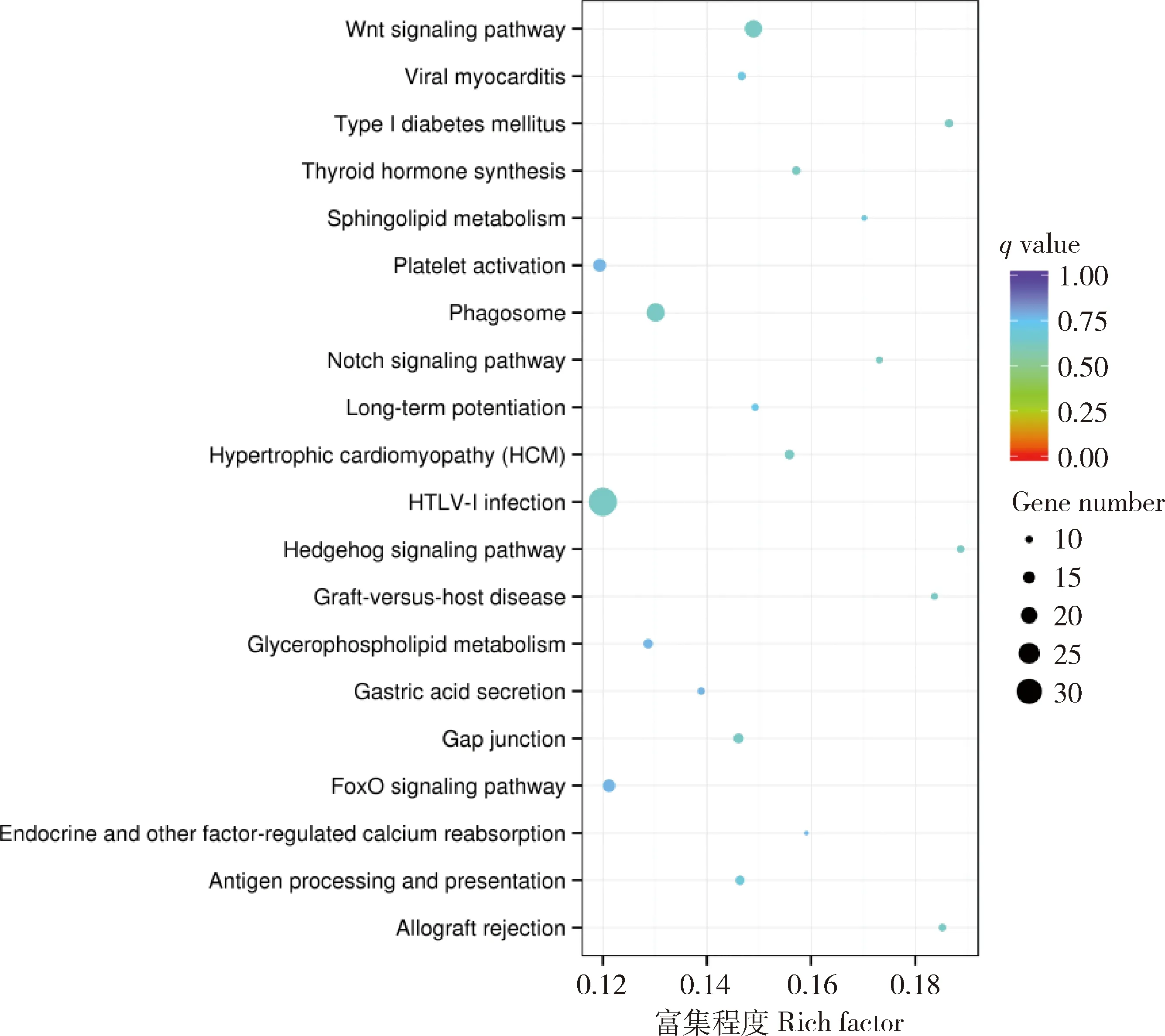
图8 差异表达lncRNA靶基因KEGG分析Fig.8 KEGG analysis of differentially expressed lncRNA target gene
3 讨 论
近年来,随着绵羊基因组研究的不断深入,对其基因组进行了大量的注释和功能分析,但对内蒙古地方品种—苏尼特羊长链非编码RNA的研究鲜有报道。因此,本研究以苏尼特羊肌肉为研究对象,对120 d胎儿和5月龄羔羊背最长肌进行RNA-seq测序分析,以期为解析苏尼特羊肌肉发育的分子机制提供理论基础,同时为分子辅助育种提供参考。
在本研究中,通过对苏尼特羊胎儿120 d和5月龄苏尼特羊背最长肌组织中的lncRNA进行转录组测序分析,进一步预测lncRNA的靶基因以揭示他们的功能。经过生物信息学分析,对比分析了 lncRNA 与 mRNA 的长度、外显子数、外显子大小及表达水平,发现与mRNA相比,lncRNA长度区间在200~3 000 bp的lncRNA数占总数lncRNA的80.0%,说明lncRNA保守性较差。包含2个和3个外显子的lncRNA数占所有lncRNA的87.3%,平均为2.5个外显子数,外显子数量比较少,与Cabili等[14]的研究结果一致。mRNA的ORF长度明显大于lncRNA,说明蛋白质编码基因具有更长的ORF。对测序获得的 lncRNA与 mRNA 的表达量进行比较,发现mRNA的整体表达量明显高于lncRNA,可能与lncRNA没有编码蛋白质的能力有关。Li等[11]通过对绵羊的lncRNA和mRNA进行特征分析,结果发现,与mRNA相比,lncRNA的外显子数量集中分布在2个或3个,且ORF长度较短,表达水平较低。本研究与Li等的研究结果一致,这些相似之处说明本研究中分析的lncRNA可靠性较高。在本研究中总共鉴定了 1 112 个候选lncRNA,包括586个已知lncRNA和526个新lncRNA,得到靶向mRNA编码基因为1 273个,这些基因也为今后的研究提供了数据基础。
进一步对对胎儿120 d组(MFH_MLcn)和 5月龄羔羊组(S5BZ_MS)进行差异表达分析,共获得399个差异表达的 lncRNA,其中212个表达上调,187个表达下调,说明不同发育时期苏尼特羊肌肉的生长发育具有不同的lncRNA调控机制。Zhan等[15]通过构建山羊胎儿(45、60和105 d妊娠)和出生后(出生后3 d)背最长肌的RNA文库,在不同发育阶段骨骼肌库之间的配对比较中,共发现577个差异表达转录本。苏尼特羊lncRNA和山羊lncRNA之间的差异可能归结于品种和阶段特异性。通过对lncRNA在不同样本中的FPKM值进行比较,结果说明ENSOART00000028118、ENSOART00000028271、LNC_000424和LNC_000978均在胎儿120 d和5月龄羔羊中有表达,ENSOART00000028271、LNC_000978、LNC_000311和LNC_000719在5月龄羔羊中的表达量较高,ENSOART00000028118和LNC_000424则与之相反。其中,ENSOART00000028118和LNC_000424在5月龄羔羊组中的表达量显著上调,该结果进一步证明ENSOART00000028118和LNC_000424在不同发育时期的苏尼特羊肌肉组织中具有不同的调控作用,调控机制可能为:在胎儿120 d肌肉组织发育中起负调控作用,在5月龄羔羊肌肉组织发育中起正调控作用。ENSOART00000028271、LNC_000311和LNC_000719在5月龄羔羊肌肉组织中的表达量显著下调,可能说明其在胎儿120 d肌肉组织发育中起正调控作用,在5月龄羔羊肌肉组织发育中起负调控作用。说明lncRNA的表达调控在不同时期有不同的表达方向,但仍需要进一步的实验验证。
文献研究显示,在本研究鉴定到显著差异的lncRNA中, ENSOART00000028118、 ENSOART00000028384、ENSOART00000027214、ENSOART00000028894、LNC_000196、LNC_00023、LNC_000269、LNC_000307、LNC_000311、LNC_000421、LNC_000422、LNC_00459 和 LNC_000879分别靶向对应与绵羊肌肉生长发育相关的基因PPP1R12C、EPS8L1、RTL1、SIRT1、TRIM7、KLHL40、TNNI1、MEF2C、FOSB、JPH2和MYH1。研究表明,RTL1基因被证实参与促进小鼠肌肉的增长[16],并且可显著调控绵羊的肌肉肥大[17]。研究发现SIRT1基因对肌肉和脂肪的发育均具有调控作用[18]。TRIM7为糖原蛋白相互作用蛋白(GNIP),并被证明在骨骼肌中高表达[19]。Chao等[20]的研究发现,KLHL40基因被预测为LOC105603392的一个顺式调控基因,并且被证明为肌肉的特异转录基因位点[21]。而作为慢骨骼肌钙蛋白基因,TNNT1、TNNI1和TNNC1在绵羊肌肉中具有一致的表达模式[22],同时有研究证明他们是维持缓慢肌纤维的重要基因[23]。Potthoff等[24]研究发现,骨骼肌中 MEF2C 的缺失会导致肌节组织出现异常。此外,FOSB作为FOS家族的一员,在细胞增殖、分化和骨骼发育中起着重要的作用。FOS家族编码亮氨酸拉链蛋白,与JUN家族的蛋白质二聚体结合,形成转录因子复合物AP-1,参与细胞的增殖、分化和转化[25]。研究也表明JPH2基因被认为是心肌细胞内T-小管成熟的关键调节物,其突变可导致扩张型心肌病[26],由此可以证明JPH2具有调节骨骼肌中生物学功能的作用。此外发现 120 d 羔羊胎儿与5月龄羔羊中LNC_000421和LNC_000422与其靶基因RTL1之间呈正调控,说明LNC_000421和LNC_000422通过对靶基因RTL1的调控来促进肌肉生长。ENSOART00000028118与其靶基因IGF2之间呈正调控,LNC_000311与其对应的靶基因TNNI3之间也呈现正调控趋势,说明ENSOART00000028118和LNC_000311参与了苏尼特羊胚胎期至性成熟期发育阶段骨骼肌的发育过程。这些结果进一步揭示了lncRNA及其靶基因的协同关系,提示接下来可以侧重研究这些lncRNA在苏尼特羊肌肉中的功能及作用机制。
在对差异表达的lncRNA进行GO和KEGG富集分析发现,在一些lncRNA在肌肉调节中起重要作用的通路中显著富集。主要涉及Wnt信号通路[27]、Notch信号通路、Gap连接[28]和甲状腺激素合成[29]等相关通路,均与肌肉发育相关,证明这些差异表达的lncRNA可以通过以上信号通路对肌肉的生长发育进行调控。Li等[11]通过对哈萨克羊进行KEGG分析发现主要涉及Wnt信号传导途径、钙信号传导途径、Gap连接、河马信号传导和甲状腺激素合成等信号通路,其可能与骨骼肌的生长,发育和功能有关。本研究结果与Li的部分相似,进一步提高了本研究预测关键基因及其相关通路的准确性。
4 结 论
本研究利用高通量测序技术对苏尼特羊不同发育阶段肌肉进行了初步的鉴定和分析,发现在苏尼特羊肌肉中存在大量的lncRNA,其表达水平在不同的发育阶段存在着一定的差异。通过GO和KEGG富集分析,这些差异表达lncRNA涉及代谢过程的调控,生物合成,基因表达,蛋白结合以及mTOR信号通路、FoxO信号通路、胰岛素信号、肌动蛋白细胞骨架的调节、Wnt信号通路、间隙连接等信号通路。并筛选出13个与不同发育阶段骨骼肌相关的候选lncRNA和靶基因,为从lncRNA的角度更好地理解苏尼特羊肌肉生长发育的遗传调控机制提供了理论基础,也为绵羊分子辅助育种提供参考。

