化肥减施对花生根际土壤细菌群落结构和多样性的影响
黄志鹏,吴海宁,唐秀梅,贺梁琼,韩柱强,钟瑞春,熊发前,刘 菁,唐荣华,蒋 菁
(广西农业科学院经济作物研究所,广西 南宁530007)
中国是世界最主要的化肥生产与施用大国[1],化肥的过量施用已造成作物品质下降、肥料利用率低[2-3]及土壤细菌多样性和活性下降[4]等诸多不利影响。 目前,农业生产集约化水平不断提高,土壤养分平衡盈余,化肥减施是今后的必然趋势[5-6]。花生是我国重要的油料和经济作物,其单产近年来徘徊不前,为了维持、提高产量,过量施肥普遍存在,不仅提高了生产成本,而且引发了诸多环境问题[5,7]。 前人研究表明,适量减施化肥对作物产量、品质无明显负向影响[8-9],开展化肥减施相关研究对花生合理施肥具有重要的实践意义。
化肥可迅速补充和及时满足作物对养分的需求,对作物产量的贡献起关键性作用[3],并通过对土壤营养和环境条件的改变,直接或间接影响土壤微生物区系[6,10-11]。 细菌约占土壤微生物数量的70%~90%,在土壤养分循环及维持农田微生态系统稳定等方面发挥重要作用[12],其多样性及群落组成在一定程度上决定土壤肥力的高低[13]。 根际是土壤微生物最活跃的区域,土壤细菌比真菌和放线菌更偏好在根际区域生长并且对肥料的敏感性更高[14],其细菌群落结构和多样性易受施肥条件的影响[10,15-16]。 前人关于肥料对土壤细菌影响的报道较多,主要集中在化肥单独及其与各种有机肥料配合施用对土壤细菌数量、群落结构及多样性的影响[16-18],关于化肥减施对花生根际细菌群落结构影响的研究报道较少。 因此,本文以单作花生为研究对象,采用Illumina Miseq测序平台,对化肥梯度减施条件下的根际土壤进行16S r RNA 高通量测序分析,研究化肥减施对根际土壤细菌群落结构和多样性的影响,为花生减肥增效提供理论参考依据。
1 材料与方法
1.1 试验地概况
试验在广西农业科学院武鸣里建科研基地进行,土壤理化性质:有机质17.7g/kg、全氮1.03g/kg、全磷0.63g/kg、全钾2.5g/kg、水解性氮85mg/kg、有效磷12.6mg/kg、速效钾138 mg/kg、交换性钙7.7 cmol/kg、p H 值6.8。
1.2 试验方案
选用桂花1026为供试材料,设5个化肥减施梯度处理:施用三元复合化肥(N∶P2O5∶K2O=15∶15∶15)450 kg/hm2(F1:习惯施肥,化肥用量设为100%);施用三元复合肥382.5 kg/hm2(F2:化肥减施15%);施用三元复合肥315kg/hm2(F3:化肥减施30%);施用三元复合肥225 kg/hm2(F4:化肥减施50%);施用三元复合肥0 kg/hm2(F5:化肥减施100%,即不施)。 采用宽窄行起垄种植,垄距90cm,其中:宽行距55cm、窄行距35cm,穴距16cm,每穴1粒,随机区组设计,3次重复,田间管理与一般花生高产田相同。
1.3 根际土壤样品的采集
在结荚期采集各处理植株的根际土壤:随机选取正常生长的花生植株,拔起后抖落根部表土,用无菌毛刷将附着在根系表面1~4 mm 的土壤刷入无菌离心管中,每处理小区取5份根际土壤充分混合后作为该小区的根际土样品,保存于-80℃冰箱,用于根际土壤细菌高通量测序分析。
1.4 根际土壤细菌多样性分析
1.4.1 土壤总DNA 提取及PCR 扩增程序
土壤总DNA使用FastDNA SPIN Kit for Soil试剂盒,每个根际土样品称取0.5 g鲜土,依照试剂盒说明提取土壤总DNA。 DNA 浓度和纯度利用NanoDrop2000进行检测,利用1%琼脂糖凝胶电泳检测DNA 提取质量。
使用土壤细菌16S r RNA(V3-V4)基因的通用PCR 引物:上游引物5’-GTACTCCTACGGGAGGCAGCA -3’; 下 游 引 物5’-GTGGACTACHVGGGTWTCTAA-3’,进行扩增,PCR 反应体系:模板DNA 20 ng,正反向引物(5μmol/L)各0.8μL,BSA(2 ng/μL)2.4μL,2×Taq Plus Master Mix 10μL,dd H2O 6μL,总 体 积20μL。PCR 扩增程序:95℃5 min;94℃30s,55℃30s,72℃45s,32个循环;72℃8min;4℃保存。 扩增产物于1%的琼脂糖凝胶中电泳(120 V,30 min)并使用凝胶成像系统检测后经胶纯化试剂盒进行纯化回收。 MiSeq文库构建、上机测序和数据分析由北京百迈客生物科技有限公司完成。
1.4.2 数据处理
对原始数据进行过滤、拼接、去除嵌合体,得到最终有效数据(Effective Tags)。 使用QIIME(version 1.8.0)软件中的UCLUST 对Tags在97%的相似度水平下进行聚类、获得OTU,并基于Silva(细菌)和UNITE(真菌)分类学数据库对OTU 进行分类学注释。 将OTU 的代表序列与微生物参考数据库进行比对可得到每个OTU 对应的物种分类信息,进而在各水平(phylum,class,order,family,genus,species)统计各样品群落组成,利用QIIME软件生成不同分类水平上的物种丰度表,再利用R 语言工具绘制成样品各分类学水平下的群落结构图。 使用Mothur(version 1.30)软件,对样品Alpha多样性指数进行评估,用QIIME软件进行Beta多样性(Beta diversity)分析。 部分数据采用Excel 2017和DPS(v7.05)进行处理分析,多重比较采用LSD 法[13]。
2 结果与分析
2.1 根际土壤细菌高通量测序结果
运用Illumina MiSeq技术对15个花生根际土壤样品的细菌进行高通量测序分析,各处理原始序列数、优化序列数、有效序列数、覆盖率指数统计分析详见表1,覆盖率指数均大于0.99,说明制备的MiSeq文库覆盖了土壤样品中99%以上的细菌类群,库容足够大,能客观反映土壤样品中细菌的真实信息。 稀释性曲线(Rarefaction Curve)用于验证测序数据量是否足以反映样品中的物种多样性,随着测序数据量的增大,样品的稀释曲线(图1)趋于平坦,说明测序结果基本覆盖了样品中绝大多数细菌的生物学信息,可用于后续分析。
2.2 根际土壤细菌群落丰度与多样性指数
各处理根际土壤的OTUs和Alpha多样性指数(表2)分析表明:习惯施肥(F1)和减施处理(F2、F3、F4)土壤的OTUs、菌群物种丰度指数(Ace、Chao1)明显大于不施肥处理(F5),F4(减施50%)处理条件下的菌群物种丰度均为最高并显著大于不施肥处理;各处理土壤的Simpson指数随化肥用量的减小呈降低趋势,Shannon指数却随化肥用量的减小而增大,不施肥土壤的菌群物种多样性指数(Simpson、Shannon)与习惯施肥处理间显著差异,但不施肥处理土壤的多样性指数与各减施处理间无显著差异。 以上结果说明,习惯施肥提高了花生根际土壤菌群物种丰度但降低其多样性,化肥减施在增加土壤菌群物种丰度的同时多样性无变化。 化肥减施50%处理条件下,花生根际土壤菌群物种丰度最高,并且其群落多样性与不施肥土壤类似。

表1 不同处理测序结果Table 1 Sequencing results of the treatments

表2 不同处理细菌群落丰度与多样性指数Table 2 Bacterial community richness and diversity indices relative to treatments

图1 不同土壤样品OTUs的稀释曲线Fig.1 Rarefaction curves of the OTUs number at 97%similarity for different soil samples
2.3 根际土壤细菌群落组成
细菌16S r DNA 高通量测序序列经物种注释后共获得49个门、101个纲、200个目、360个科和552个属类细菌。 图2所示,在门水平上,F1、F2、F3、F4和F5处理相对丰度排名前10的细菌分别占细菌总数的97.97%、97.63%、97.52%、97.15%和97.86%,其中优势菌群为变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、放线菌门(Actinobacteria)和芽单胞菌门(Gemmatimonadetes),各处理的平均相对丰度分别为38.76%、17.41%、12.53%、10.93%和6.97%。在属水平上,F1、F2、F3、F4和F5相对丰度排名前10的细菌分别占细菌总数的23.98%、25.25%、25.34%、24.23%和25.03%,其中优势菌属为鞘氨醇单胞菌属(Sphingomonas)、玫瑰弯菌属(Roseiflexus)、Haliangium、硝化螺旋菌属(Nitrospira)、Candidatus_Solibacter、H16、芽单胞菌属(Gemmatimonas)、RB41,各处理平均相对丰度分别为5.70%、3.40%、3.02%、2.11%、2.10%、2.06%、1.97%、1.91%。
各处理的菌群相对丰度显著性差异分析表明:F2与F3在门和属水平上的细菌群落构成差异不显著,F5 与F4 间的差异亦不显著(表3)。F1与F5处理间菌群相对丰度差异显著的门属分别为83个,F1与F4间菌群相对丰度差异显著的门属分别为65个,其余处理间菌群相对丰度差异显著的门属均少于3个。 结果说明,习惯施肥能显著改变根际土壤细菌在门和属水平上群落组成和结构,少量(15%)和中量(30%)减施化肥明显影响根际土壤细菌的群落组成和结构,大量减施化肥(50%)则对根际土壤细菌的群落组成和结构影响较小。

图2 不同土壤样品根际细菌在门水平(A)和属水平(B)上的优势物种丰度图Fig.2 Dominant species abundance map at the phylum(A)and genus (B)levels relative to soil samples
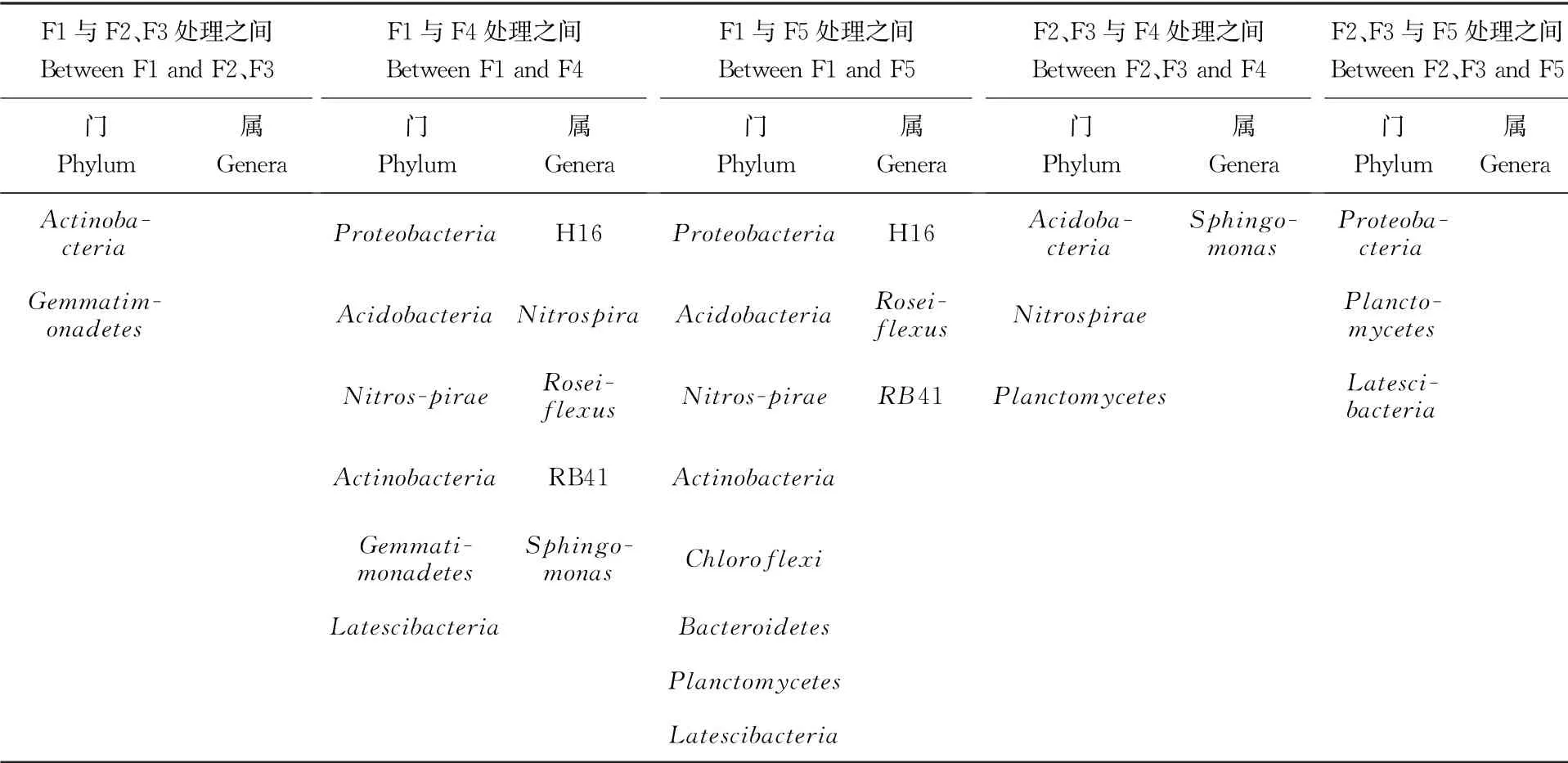
表3 不同处理间相对丰度达到差异显著的细菌门属列表Table 3 List of soil bacterial phylumgenera with significant difference between treatments
2.4 根际土壤细菌的Venn图分析
习惯、减施、不施三种化肥施用方式总OTU数分别为2120、2136 和2095 个,共 同 包含的OTU 有2081个(图3)。 在OTU 数量种类方面,减施肥料处理不仅包含习惯施肥和不施肥,并拥有2个代表特有细菌属的独特OTU。 结果说明,化肥减施条件下的根际土壤能兼容不同类型细菌的生存需求,增加根际土壤细菌的组成种类。
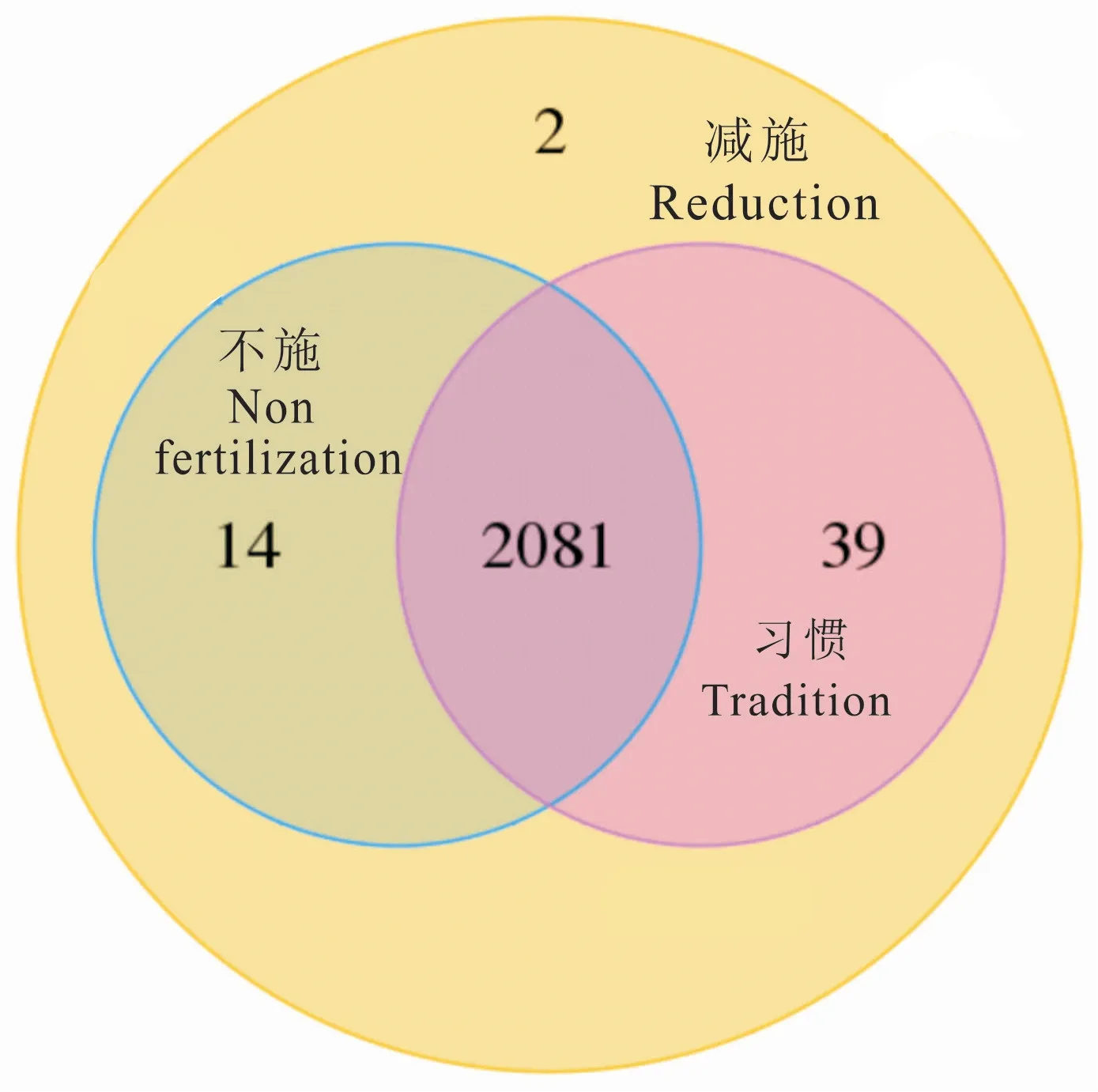
图3 根际土壤细菌OTUs数量Venn图Fig.3 Venn diagram of the No.of bacterial OTUs in rhizosphere soil

图4 基于非度量多维标定法(NMDS)(A)和非加权组平均法(UPGMA)(B)的群落结构分析Fig.4 Community structure analysis based on NMDS (A) and UPGMA (B)
2.5 根际土壤细菌群落的相似性分析
采用非度量多维标定法(NMDS)和非加权组平均法(UPGMA)对15个样本的花生根际土壤细菌进行分析,研究不同处理间细菌群落结构和物种组成的相似性。 NMDS排序结果(图4A)显示,细菌群落结构按化肥施用方式分为3个组群,每种化肥施用方式的土壤细菌群落分别聚为1个组群,表明习惯、减施、不施处理间的细菌群落结构有较明显的差异。 UPGMA 分析表明(图4B),除个别样品外,细菌群落物种组成分为两大类,即习惯处理与减施、不施处理,说明习惯施肥使菌群物种组成发生明显变化;F2、F3处理被聚为1个分支,F4、F5处理被聚为另外一个1个分支,说明F4与F5有较高的菌群相似性。 结果说明,施肥能明显改变细菌群落结构和物种组成,减施处理的菌群结构和物种组成介于习惯施肥与不施肥处理之间,随减施量的提高,土壤菌群物种组成逐步趋同于不施处理。
3 讨 论
土壤微生物组成和功能直接受养分的影响[19],土壤中细菌是陆地生态系统中生物多样性的主要代表,与碳固存、营养元素循环等生态系统功能密切相关[20]。 施肥是维持土壤肥力的重要措施,无机肥能直接改变土壤速效养分含量[18]。 化肥施用对不同生态系统土壤细菌群落组成和多样性有不同的影响[6,9,12-13,16]。 本研究中,与不施肥处理相比,习惯施肥能明显改变花生根际土壤的菌群物种丰度和多样性,这与孙瑞波等[21]在砂姜黑土和荀卫兵[16]在旱地红壤上的研究结果相似,但亦有研究得出不同结论[22],其原因可能与化肥种类、土壤类型和用量等因素有关[23]。 化肥减施作为一种短期的无机肥施用管理措施,能使根际土壤细菌的群落结构和多样性产生可被观察到的变化,这与李杨[17]、Guo等[24]的研究结果类似。 对比习惯施肥,化肥减施增加了菌群物种丰度,随着减施量的增大,根际土壤细菌多样性也随之提高,这与吴海宁等[25]对单作花生的研究类似。 化肥减施50%条件下,花生根际土壤菌群物种丰度最高并显著大于不施肥处理,其菌群多样性与不施肥处理差距很小但显著大于习惯施肥处理,说明适宜的化肥减施水平有利于菌群丰度与多样性的提高[22]。
本研究中,与不施肥处理相比,化肥习惯施用能显著改变根际土壤细菌在门和属水平上的群落组成和结构,两者间相对丰度排名前10且差异显著的细菌门属分别为83个。 土壤p H 被认为是影响土壤微生物群落演替变化的最主要因子[16],N-P-K 复合肥的大量使用会导致土壤酸化改变[12],可能是土壤细菌群落结构发生显著变化的原因,这也与孙瑞波[21]、岑剑[23]等研究结果类似。 本试验中,少量(15%)、中量(30%)和大量(50%)减施化肥处理间的群落构成差异不大,即肥料用量对土壤细菌群落构成影响不大,与廖莎[26]的研究结果类似。 大量减施化肥条件下的根际土壤细菌群落构成与不施肥处理间无显著差异,这可能与大量减施化肥使土壤养分含量处于相对均衡状态有关,尚需深入研究。
本研究中,减施条件下的根际土壤能兼容不同类型细菌的生存需求,在组成种类方面,减施肥料处理不仅包含习惯施肥和不施肥处理,并拥有2个代表特有细菌属的独特OTU。 化肥减施在降低施肥对根际土壤菌群影响的同时仍可满足不同种类细菌生存所需的养分,在增加菌群丰度,保持其多样性方面的效果明显。
4 结 论
习惯施肥显著改变了根际土壤细菌的群落组成和结构,提高了花生根际土壤的菌群物种丰度但多样性降低;化肥减施在进一步增加菌群丰度的同时维持其原有多样性,化肥减施50%条件下,花生根际土壤菌群丰度最高,其群落构成和多样性与不施化肥土壤类似。

