睡莲叶片胎生发育转录组分析
苏群 田敏 李春牛 李先民 卢家仕 黄展文 李杰梅 卜朝阳 王虹妍



摘 要:睡莲叶片胎生现象是繁育途径的重要补充,对种群的传播、扩散和生态环境的适应性有重要作用。通过转录组测序技术筛选和分析睡莲叶片胎生现象相关的代谢路径和调控基因,为深入认识睡莲叶片胎生发育的分子机制提供参考。以叶片具有胎生现象的‘小花睡蓮’(X)和叶片无胎生现象的‘蓝星睡莲’(L)为材料,利用RNA-Seq技术对叶片4个发育阶段的叶脐部分进行生物信息学分析。分析对照(L)和样品(X)叶片不同发育阶段测序结果:筛选出的差异表达基因(DEGs)中,34 909个基因(48.65%)表达上调,36 850个基因(51.35%)表达下调。DEGs分析显示,随着叶片的发育,X和L上调基因和下调基因数均呈增加趋势。对L1-vs-X1、L4-vs-X4阶段的GO和KEGG功能富集分析表明,DEGs主要富集在质膜和膜相关成分、胞外区域、细胞壁等相关的细胞组分中,涉及到代谢过程、生物合成和应急响应等;Pathway代谢通路表明,DEGs主要参与到植物激素信号转导、苯丙烷类生物合成、氨基酸类代谢、类黄酮生物合成、甘油磷脂类代谢以及细胞周期相关等过程。对DEGs进一步分析,克隆出了4个可能参与睡莲叶片胎生发育的转录因子。
关键词:睡莲;叶片胎生;转录组分析;差异表达基因;代谢通路
中图分类号:Q949.746.1 文献标识码:A
Abstract: Leaf vivipary in water lily (Nymphaea) is an important supplement to breeding, which plays an important role in the propagation and adaptability of species. Transcriptome sequencing technology was used to screen and explore genome-wide analysis of regulatory genes and metabolic pathways involved in leaf vivipary in water lily. This study would lay a foundation for further understanding the molecular mechanism of leaf viviparous development. Viviparous N. micrantha (X) and non-viviparous N. colorata (L) were selected as the experimental materials. By using RNA-Seq technology, four stages of leaf development of the leaf stalk and stem and stem join were tested and analyzed using a series of bioinformatics analysis. The result showed that 34 909 (48.65%) DEGs were up-regulated and 36 850 (51.35%) DEGs were down-regulated. DEGs analysis showed that up-regulated genes and down-regulated genes both increased with the leaves development of N. micrantha amd N. colorata. GO and KEGG enrichment analysis of L1-vs-X1 and L4-vs-X4 stage showed that DEGs were mainly enriched in plasmalemma, cytomembrane and extracellular domain cytoderm associated with metabolic process, biosynthesis and stimulus response. Pathway metabolism pathways indicated that DEGs were mainly involved in plant hormone signal transduction, phenylpropanoid biosynthesis, amino acids metabolism, flavonoid biosynthesis, glycerolipid metabolism and cell cycle and other processes. Four transcription factors potentially involved in leaf viviparous development of water lily were cloned based on DEGs analysis.
Keywords: water lily; leaf vivipary; transcriptome sequencing; DEGs; metabolic pathway
DOI: 10.3969/j.issn.1000-2561.2021.12.009
睡莲为睡莲科(Nymphaeaceae)睡莲属(Nymphaea L.)多年生草本植物,花色丰富,花期长,适应性与抗逆性强,栽培容易且分布广泛[1]。睡莲属为睡莲科中最大的属,约50余种(含变种),可分为subg. Nymphaea、subg. Anecphya、subg. Brachyceras、subg. Hydrocallis和subg. Lotos 5个亚属[2-3]。睡莲因其极高的观赏价值和在植物进化、分类中的重要地位而越来越受到爱好者、育种家和植物学家的追捧[4-7]。部分热带睡莲的叶片除进行光合作用外,还具有叶片胎生(vivipary)的特性,在其叶脐部位(叶片与叶柄连接处)长出新的植株[1]。胎生是植物另一种繁殖途径,可使其种群在较短时间内快速传播和扩散,更易适应多变而复杂的自然环境,此外胎生对生物多样性保护,生态平衡的发展具有重要意义[8]。与睡莲叶片胎生现象相关的内部代谢路径和调控基因的研究则鲜见报道。
本研究利用Illumina测序平台,以叶片具有胎生现象的‘小花睡莲 ’(Nymphaea micrantha, X)和叶片无胎生现象的‘蓝星睡莲’(Nymphaea col-orata, L)为材料,对叶片4个发育阶段的叶脐部分进行转录组测序,并重点比对分析了L1-vs-X1和L4-vs-X4阶段的差异表达基因和代谢通路富集,以期筛选睡莲叶片不同发育模式下的相关代谢路径和参与叶片胎生发育的调控基因,为睡莲叶片胎生发育的分子遗传机制研究提供参考。
1 材料与方法
1.1 材料
‘小花睡莲’(X)和‘蓝星睡莲’(L)均种植于广西壮族自治区农业科学院花卉研究所睡莲资源圃内,采用缸栽形式,水缸尺寸为口径85 cm,深度50 cm,水深常年维持在25 cm左右。取‘X’(样品)叶片和‘L’(对照)叶片4个不同发育阶段的叶脐部位为材料(图1),将相同基因型的材料6株混合取样,并重复取样1次。取样后迅速放入液氮中保存。
1.2 方法
1.2.1 RNA提取、文库构建和转录组测序 委托攸归(上海)生物科技有限公司完成RNA提取、文库构建和转录组测序工作。采用CTAB法提取睡莲总RNA并采用Agilent 2100进行质检。RNA样品检测合格后,富集并打断成短片段,用六碱基随机引物(random hexamers)合成一链cDNA,然后加入缓冲液、dNTPs、DNA polymerase I和RNase H合成二链cDNA,随后利用AMPure XP beads纯化双链cDNA。纯化的双链cDNA先进行末端修复、加A尾并连接测序接头,再用AMPure XP beads进行片段大小选择。最后进行PCR扩增,并用AMPure XP beads纯化PCR产物,得到最终的文库。采用Illumina NovaSeq 6000平台进行双末端测序。
使用NGS QC Toolkit[9]软件对测序得到的原始数据进行质控以得到高质量的clean reads。使用hisat2[10]将clean reads与‘蓝星睡莲’的参考基因组进行比对(ftp://download.big.ac.cn/gwh/Plants/ Nymphaea_colorata_Nym_GWHAAYW00000000/ GWHAAYW00000000.genome.fasta.gz),比对结
果以二进制binary文件即bam文件进行储存。之后使用Cufflinks[11]对基因定量获取FPKM值。采用htseq-count[12]软件获取落到各个样本中基因的reads数目。
1.2.2 差异表达基因(DEGs)检测、功能注释和富集分析 使用STEM (Short Time-series Ex-pression Miner)软件对不同发育阶段的睡莲叶片中基因的表达模式进行分析。并采用R中的pheatmap函数进行热图聚类分析。使用DESeq (2012) Rpackage的estimateSizeFactors函数对数据进行标准化,并使用nbinomTest函数计算差异比较的P value和fold change值。以P<0.05且fold change值2倍以上(上调或下调)作为筛选差异基因的标准。利用Blast2GO[12]软件和KAAS软件(https://www.genome.jp/tools/kaas/)分别进行GO和KEGG[13]注释和富集分析。重点选择比对了L1-vs-X1、L4-vs-X4阶段基因的差异表达情况,统计同种材料不同时期和不同材料同一时期转录组的DEGs,并进行GO和KEGG富集分析,以判定差异基因主要影响的代谢途径和信号通路。
2 结果与分析
2.1 总RNA质量检测和组装结果分析
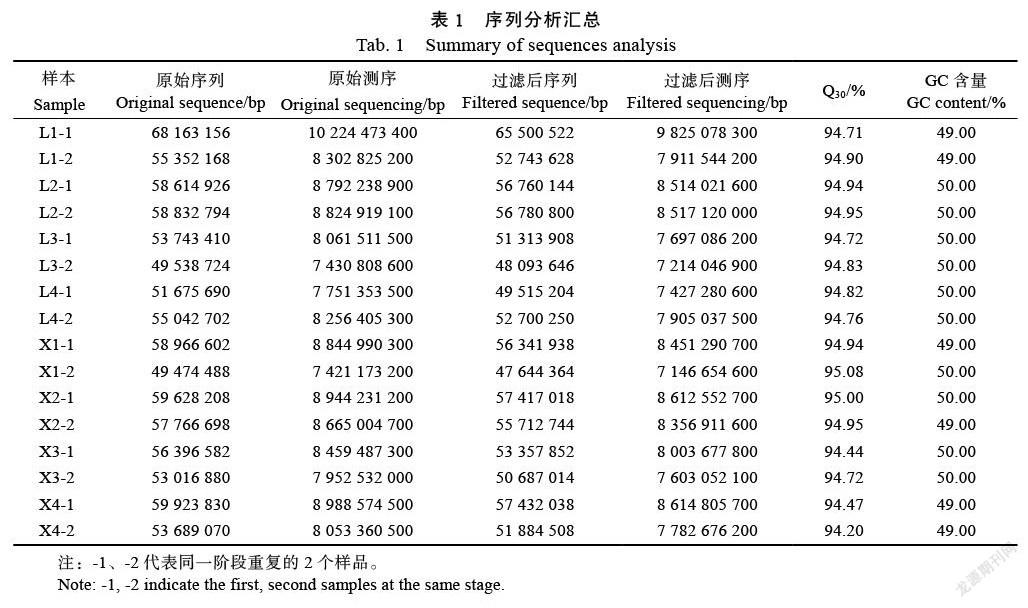
‘小花睡莲’叶片和‘蓝星睡莲’叶片4个不同发育阶段总计16份样本,经检测,样品总RNA质量达到建库要求,其中浓度为70~1230 ng/µL,A260/280为2.1~2.2,28S/18S为1.1~1.9,RIN≥7。
转录组测序共获得134.97 Gb原始数据(表1),各样本Q30值达到94%以上,GC≥49%,说明测序结果准确度较好,可用于后续分析。
2.2 转录组基因表达数据分析
为了分析‘小花睡莲’和‘蓝星睡蓮’睡莲叶片转录组中基因表达差异情况,做了全转录组表达基因热图。从图2可看出,‘小花睡莲’和‘蓝星睡莲’中不同发育阶段的叶片中基因表达差异较为明显,L1-vs-X1、L4-vs-X4阶段有着较为显著的差异表达基因。同时进一步分析‘小花睡莲’和‘蓝星睡莲’叶片中不同表达基因的时空表达模式,利用STEM软件对‘小花睡莲’和‘蓝星睡莲’8个组织进行了基因表达趋势(Profile)分析。结果显示共检测到60个表达趋势,其中‘小花睡莲’和‘蓝星睡莲’各30个(图3)。在‘小花睡莲’4个发育阶段叶片中,表达趋势8、12、4、18、0、14、25、13、21是显著富集(P<0.05),分别包含2337、1302、1254、1157、1008、953、913、662、643个表达基因;在‘蓝星睡莲’4个发育阶段叶片中,表达趋势4、8、25、0、21、12是显著富集(P<0.05),分别包含了3773、1919、1491、1153、838、675、530个表达基因。这些基因的大量表达,提示它们在此发育阶段发挥重要作用。
2.3 差异表达基因的数据分析
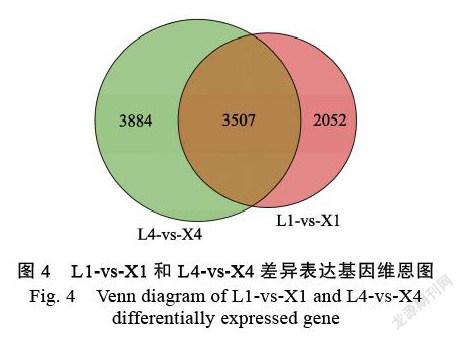
如表2所示,在检测到的DEGs中,上调差异基因34 909个(48.65%),下调差异基因36 850个(51.35%)。随着叶片的发育,‘小花睡莲’和‘蓝星睡莲’上调基因和下调基因数均呈增加趋势,各阶段比对分析显示,L1-vs-X1阶段的DEGs为5559个,其中2033个基因表现为上调,3526个基因表现下调,下调基因数明显多于上调基因数;L4-vs-X4阶段的DEGs最多,高达7391个,其中2979个基因上调,4412个基因下调,同样也是下调基因多于上调基因。L1-vs-X1和L4-vs-X4的DEGs韦恩图分析表明,3507个DEGs在2个比对阶段皆有表达,3884个DEGs在L4-vs-X4中差异表达,2052个DEGs在L1-vs-X1中表达(图4)。根据试验设计及数据结果初步推测3884个基因中可能存在睡莲叶片胎生发育相关的基因。
2.4 差异表达基因的GO功能分析
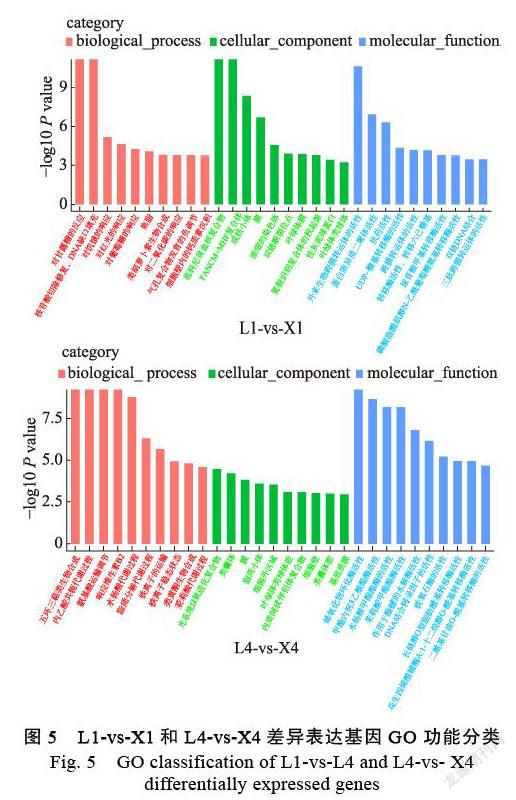
为了进一步了解睡莲叶片胎生发育分子机制,重点分析了L1-vs-X1阶段的5559个DEGs以及L4-vs-X4阶段的7391个DEGs的GO富集分析top30(筛选3种分类中对应基因数目大于2的GO条目,按照每个条目对应的-log10 P value由大到小排序的各前10条)。从图5可知,DEGs在L1-vs-X1及L4-vs-X4阶段均主要富集在细胞组分中,具体到质膜和膜相关成分、核小体、类囊体、胞外区域、细胞壁等的细胞组分中,涉及到代谢过程、生物合成等;在生物过程中,较多的DEGs归类为次生代谢、光合呼吸反应、防卫反应、铁离子运输、应急响应及DNA复制和修复等;在分子功能分类中,参与结合、转运和催化活性的DEGs最多。
2.5 差异表达基因的KEGG功能分析
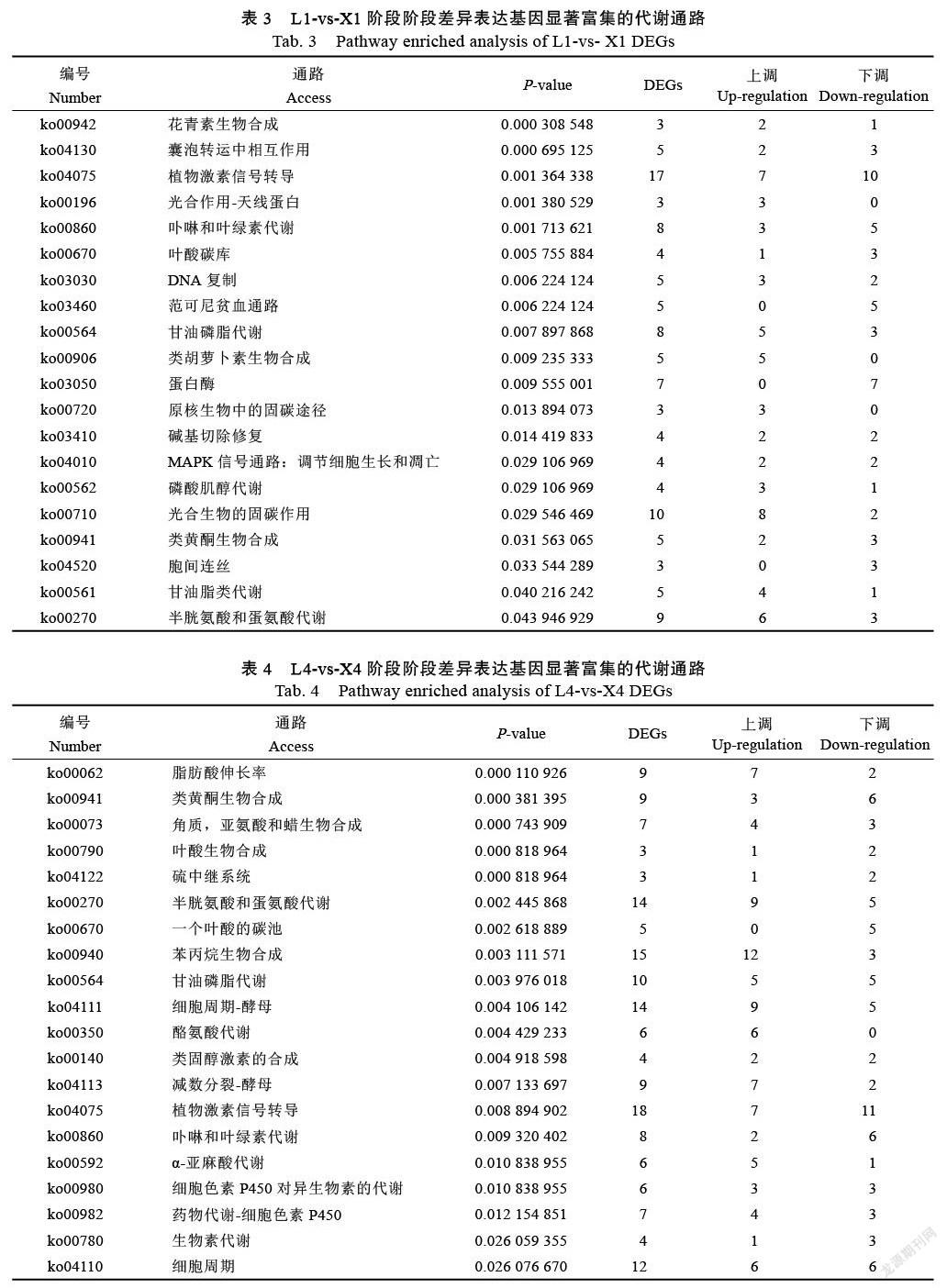
KEGG是有关Pathway的主要公共数据库,可以利用KEGG数据库对差异Unigene进行Pathway富集分析(结合KEGG注释结果)。L1-vs-X1阶段以及L4-vs-X4阶段的KEGG富集分析top20(以P≤0.01为筛选标准,同时过滤掉差异基因数目小于3的条目)如表3~表4所示。Pathway代谢通路表明DEGs主要参与到植物激素信号转导、苯丙烷类生物合成、氨基酸类代谢、细胞周期、光合作用、类黄酮生物合成以及磷脂类代谢等相关过程。
L1-vs-X1阶段植物激素信号转导通路富集DEGs最多达到17个,7个基因表达上调,10个基因下调表达;其次,光合作用固碳路径中有8个基因上调,2个基因下调;氨基酸代谢路径富集9个DEGs,6个基因上调,3个基因表达下调;磷脂类代谢及叶绿色代谢类路径各有8个DEGs,此外囊泡转运互作路径、DNA复制、类胡萝卜素生物合成等路径DEGs均达到了5个。L4-vs-X4阶段植物激素信号转导通路富集DEGs最多达18个,其中7个基因表达上调,11个基因表达下调;苯丙烷生物合成富集DEGs 15个,12个基因表达上调,3个基因表达下调;重点指出的是参与细胞周期-减数分裂相关的DEGs多达35个,其中22个基因上调,13个基因下调。此外,甘油磷脂类代谢路径DEGs有10个,上调和下调表达基因各有5个;富集参与到脂肪酸代谢、类黄酮代谢、类固醇激素代谢及叶绿素代谢通路的DEGs也较多。综上可知,L1-vs-X1阶段及L4-vs-X4阶段的植物激素信号转导通路富集DEGs都是最多,且下调基因数多于上调基因数。
2.6 激素信号转导途径中显著差异表达基因的筛选
为了探寻参与睡莲叶片胎生的内部调控机制的基因,通过重点分析‘小花睡莲’和‘蓝星睡莲’转录组数据,筛选‘小花睡莲’显著上调转录因子,并通过转录组reads组装获得基因全长。
以此为基础,设计相关基因扩增引物,通过PCR克隆方法,得到4个转录因子基因,分别为ERF1B(NCBI登录号:MT542693)、ERF105(NCBI登录号:MT974544)、RAP2-3(NCBI登录号:MT799791)和WRKY22(NCBI登录号:MT 799792)。
3 讨论
叶片通常被认为是植物进行光合作用、呼吸作用和蒸腾作用的主要场所,部分叶片也具有观赏价值和营养价值[14]。一些植物叶片还能够从叶缘或者叶片中央部位长出新的完整植株,如伽蓝菜属的大叶落地生根(Kalanchoe daigremontiana)[15]和睡莲属(Nymphaea spp.)的一些种(品种)[1, 8]等。叶片、花朵等植物营养器官的胎生是植物另一种繁殖途径,可使其种群在较短时间内快速传播和扩散,更易适应复杂而多变的自然环境。此外胎生对生物多样性保护及生态平衡的发展具有重要意义,这种胎生能力受内部基因的调控,具有可遗传性,植物通过这种方式可将进化的优良性状一代代遗传固定下来[8]。部分热带睡莲也多以叶胎生的形式繁殖,但其形态发育和分子水平的内部调控的机理尚不清楚,有待于进一步研究。
本研究中L1-vs-X1和L4-vs-X4阶段的植物激素信号转导通路富集的DEGs都最多,说明在调控睡莲叶片胎生苗发育过程中,植物激素信号一直发挥着重要作用,这与Kane等[16]探讨植物激素对睡莲胎生苗的研究的结果一致。在L1-vs-X1阶段上调基因7个,下调基因10个;L4-vs-X4阶段上调基因7个,下调基因11个,2个比对阶段均有较多的上下调基因,维持着动态平衡。此外通过胎生和非胎生睡莲种间转录组的比较试验,发现生长素(AUX/IAA)、生长素应答因子(ARF)、植物生长素酰胺合成酶(GH3)和生长素上调小RNA(SAUR)的显著差异调节。Aux/IAA是早期反应基因,能够精确快速地觸发基因重编程[17]。ARF转录因子在生长素感知时被激活,并启动下游信号通路,包括SAUR基因。SAURs调节许多生长素介导的反应,特别是通过细胞伸长的组织生长[18]。这些基因可能通过参与细胞分裂、扩大和分化而直接参与睡莲胎生外部生长的发育[17]。生长素与细胞因子的结合也有助于细胞分化,细胞分裂素(CRE)的高表达可能是胎生细胞分化的原因之一,因为其对茎顶端分生组织的活性是已知的[19]。此外,有研究表明其他激素如乙烯等和IAA的互作来调控胎生苗根系的发育[20]。郭书磊等[21]利用RNA-Seq技术研究玉米叶片形态建成也得出多种植物激素相互作用通过参与调控玉米叶片宽窄的形态建成研究相同。综上所述,植物激素间的相互作用和动态平衡对睡莲叶片胎生发育有着重要的影响。
随着近年乡村振兴和休闲旅游的兴起,睡莲因其迷人的外表而得到快速的推广应用,但性状优良的睡莲品种往往不具有胎生能力,种苗繁育困难,导致价格居高不下,严重制约了其进一步的推广应用。虽然目前在胎生睡莲的形态解剖、分子水平上取得了一些进展,但引起胎生现象的内部调控机制并没有得到系统解答,仍需进一步在分子水平上探究形成机理和挖掘重要功能基因。转录因子在植物的生长发育及其对外界环境的反应中起着重要调控作用,其序列和结构的多样性决定了其功能的多样性。通过睡莲种间胎生和非胎生转录组的比较研究,挖掘出4个显著差异表达的转录因子,分别为ERF1B、ERF105、RAP2-3和WRKY22。通过前人的研究得知AP2/ERF家族有多个成员能够调节体细胞胚胎发生[22],其中被研究得最多的是BABY BOOM (BBM)基因[23-27]和WOUND INDUCEDDEDIFFERENTI ATION 1 (WIND1)或RAP2.4基因[28]。也有研究表明WRKY家族转录因子通过参与其他激素介导的信号转导来间接调节植物体细胞的发育[29-30]。AP2/ERF基因家族和WRKY等基因家族成员多而复杂,成员之间的功能差异很大,需具体到特定物种中进行功能验证,如在苹果中ERF1B基因能够通过调控LOX途径进行香气的合成[31]。而在棉花中则可能参与抗黄萎病的调控[32]。
本研究通过转录组角度分析出植物激素间的相互作用和动态平衡对睡莲叶片胎生发育有着重要的影响,并结合RT-PCR技术克隆了ERF1B、ERF105、RAP2-3和WRKY22等4个可能参与叶片胎生过程调控的转录因子基因,以期为睡莲叶片胎生的内部代谢和基因调控提供一定理论基础,今后还需对克隆出的4个转录因子作进一步的研究探讨。
参考文献
[1] 黄国振, 邓惠勤, 李祖修. 睡莲[M]. 北京: 中国林业出版社, 2009: 1-22.
[2] Borsch T, Löhne C, Wiersema J H, et al. Towards a com-plete species tree of Nymphaea: shedding further light on subg[J]. Telopea, 2011, 13(1-2): 193-217.
[3] 李淑娟, 尉 倩, 陈 尘, 等. 中国睡莲属植物育种研究进展[J]. 植物遗传资源学报, 2019, 20(4): 829-835.
[4] Kirie S, Iwasaki H, Noshita K, et al. A theoretical morpho-logical model for quantitative description of the three- di-mensional floral morphology in water lily (Nymphaea)[J]. PLoS One, 2020, 15(10): e0239781.
[5] Povilus R A, Dacosta J M, Grassa C, et al. Water lily (Nym-phaea thermarum) genome reveals variable genomic signa-tures of ancient vascular cambium losses[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(15): 8649-8656.
[6] Zhang L, Chen F, Zhang X, et al. The water lily genome and the early evolution of flowering plants[J]. Nature, 2020, 577(7788): 79-84.
[7] 苏 群, 杨亚涵, 田 敏, 等. 睡莲种质资源遗传多样性分析及DNA指纹图谱构建[J]. 热带作物学报, 2020, 41(2): 258-266.
[8] 艾星梅, 陈龙清, 李宇航, 等. 植物营养体胎生研究进展[J]. 热带亚热带植物学报, 2020, 28(2): 209-216.
[9] Patel R K, Jain M. NGS QC Toolkit: a toolkit for quality control of next generation sequencing data[J]. PLoS One, 2012, 7(2): e30619.
[10] Langmead B, Salzberg S L. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 2012, 9(4): 357-359.
[11] Trapnell C, Roberts A, Goff L, et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nature Protocols, 2012, 7(3): 562- 578.
[12] Conesa A, Götz S, García-Gómez J M, et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 2005, 21(18): 3674-3676.
[13] Kanehisa M, Araki M, Goto S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Re-search, 2008, 36(database issue): D480-D484.
[14] 严 松, 严长杰, 顾铭洪. 植物叶发育的分子机理[J]. 遗传, 2008, 30(9): 1127-1135.
[15] 鐘天秀. 大叶落地生根胎生苗差减cDNA文库的构建及相关基因的克隆[D]. 北京: 北京林业大学, 2015.
[16] Kane M, Kauth P. Use of plant growth regulators to enhance viviparous waterlily propagation[J]. Water Garden Journal, 2006, 21(4): 7-10.
[17] Luo J, Zhou J J, Zhang J Z. Aux/IAA gene family in plants: molecular structure, regulation, and function[J]. International Journal of Molecular Sciences, 2018, 19(1): 259.
[18] Charles C. SAUR15 connects auxin perception to lateral and adventitious root formation[J], Plant Physiology, 2020, 184(2): 558-559.
[19] Inoue T, Higuchi M, Hashimoto Y, et al. Identification of CRE1 as a cytokinin receptor from Arabidopsis[J]. Nature, 2001, 409(6823): 1060-1063.
[20] Kulka R G. Hormonal control of root development on epi-phyllous plantlets of Bryophyllum (Kalanchoe) marnieria-num: role of auxin and ethylene[J]. Journal of Experimental Botany, 2008, 59(9): 2361-2370.
[21] 郭书磊, 鲁晓民, 齐建双, 等. 利用RNA-Seq发掘玉米叶片形态建成相关的调控基因[J]. 中国农业科学, 2020, 53(1): 1-17.
[22] Mantiri F R, Kurdyukov S, Chen S K, et al. The transcription factor MtSERF1 may function as a Nexus between stress and development in somatic embryogenesis in Medicago truncatula[J]. Plant Signal Behav, 2008, 3(7): 498-500.
[23] Boutilier K, Offringa R, Sharma V K, et al. Ectopic expres-sion of BABY BOOM triggers a conversion from vegetative to embryonic growth[J]. Plant Cell, 2002, 14(8): 1737-1749.
[24] Horstman A, Li M, Heidmann I, et al. The BABY BOOM transcription factor activates the LEC1-ABI3-FUS3-LEC2 network to induce somatic embryogenesis[J]. Plant Physiology, 2017, 175(2): 848-857.
[25] Liu D, Chen X, Liu J, et al. The rice ERF transcription factor OsERF922 negatively regulates resistance to Magnaporthe oryzae and salt tolerance[J]. Journal of Experimental Botany, 2012, 63(10): 3899-3911.
[26] Lowe K, Wu E, Wang N, et al. Morphogenic regulators Baby boom and Wuschel improve monocot transformation[J]. Plant Cell, 2016, 28(9): 1998-2015.
[27] Zhang Z, Zhao H, Li W, et al. Genome-wide association study of callus induction variation to explore the callus formation mechanism of rice[J]. Journal of Integrative Plant Biology, 2019, 61(11): 1134-1150.
[28] Ikeuchi M, Sugimoto K, Iwase A. Plant callus: mechanisms of induction and repression[J]. Plant Cell, 2013, 25(9): 3159- 3173.
[29] Shang Y, Yan L, Liu Z Q, et al. The Mg-chelatase H subunit of Arabidopsis antagonizes a group of WRKY transcription repressors to relieve ABA-responsive genes of inhibition[J]. Plant Cell, 2010, 22(6): 1909-1935.
[30] Zou X, Seemann J R, Neuman D, et al. A WRKY gene from creosote bush encodes an activator of the abscisic acid sig-naling pathway[J]. The Journal of Biological Chemistry, 2004, 279(53): 55770-55779.
[31] 岳璇璇. 蘋果乙烯响应因子ERF1B调控LOX途径香气合成的机理研究[D]. 泰安: 山东农业大学, 2020.
[32] 刘 坤. 海岛棉ERF族B3和B1亚组转录因子基因的克隆和特征研究[D]. 北京: 中国农业科学院, 2011.
责任编辑:黄东杰

