围垦对崇明东滩湿地全程氨氧化微生物的影响①
宋怡轩,金 锐,张鑫磊,张 洁,贾仲君,张耀鸿*
围垦对崇明东滩湿地全程氨氧化微生物的影响①
宋怡轩1,2,金 锐1,张鑫磊1,张 洁1,贾仲君2,张耀鸿1*
(1南京信息工程大学气象灾害预报预警与评估协同创新中心/江苏省农业气象重点实验室,南京 210044;2土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所),南京 210008)
全程氨氧化细菌(complete ammonia oxidizers,Comammox)的发现根本改变了学术界对硝化过程的认识,但其地理分异规律及对氮转化过程的贡献仍不清楚。本研究选择长江口崇明东滩不同围垦年限(0、27、51、86 a)稻田表层耕作土壤,采用好氧培养试验测定土壤硝化潜力;通过标靶功能基因实时荧光定量硝化微生物的数量变异特征,包括全程氨氧化细菌(Comammox)、氨氧化细菌(ammonia-oxidizing bacteria,AOB)和氨氧化古菌(ammonia-oxidizing archaea,AOA)。结果表明,与围垦0 a的自然滩涂湿地相比,围垦27、51、86 a的水稻土净硝化速率从N 2.24 mg/(kg·d)分别增加至N 19.3、11.6和11.4 mg/(kg·d),增幅高达5.1 倍~ 8.7倍。AOA的丰度与围垦年限显著正相关。自然滩涂湿地中AOA和AOB的数量分别为0.34×107copies/g和1.14×107copies/g,围垦86 a后增幅最高可达27.9倍。自然滩涂湿地中Comammox Clade A和Clade B的基因拷贝数高于围垦稻田土壤,且Comammox Clade A随着围垦年限增加其丰度显著增加。统计分析发现,AOB与土壤硝化速率显著正相关,可能在围垦水稻土氨氧化过程中发挥了重要作用;而Comammox Clade A和Clade B与土壤总有机碳(TOC)、铵含量(NH4+)呈显著负相关关系,可能更适应于营养贫瘠的滩涂自然湿地土壤。
围垦;全程氨氧化细菌;氨氧化
氨氧化作用作为土壤氮素转化的关键过程之一, 是硝化过程的限速步骤。一直以来,人们普遍认为硝化作用由两类不同的化能自养型细菌:氨氧化细菌(ammonia-oxidizing bacteria, AOB)和亚硝酸盐氧化细菌(nitrite-oxidizing bacteria, NOB)完成的两步氮循环过程[1-2]。2004年,化能自养型氨氧化古菌(ammonia- oxidizing archaea, AOA)被发现,随后的许多研究表明,这类微生物在土壤和水生系统的硝化过程中发挥着重要作用,丰富了驱动好氧氨氧化过程的微生物类型。从热力学和动力学角度来看,相比于经典分步硝化,独立完成氨氧化和亚硝酸盐氧化的全程氨氧化过程产生的能量更多,在产率较高而生长速率较低的条件下具有更大优势[3-4]。2015年,Daims等[3]和van Kessel等[5]发现了硝化螺菌属(spp.)中有微生物能全程完成硝化作用,即一步将氨氮转化为硝态氮,这类微生物统称为全程氨氧化细菌(complete ammonia oxidizers, Comammox)。该细菌的发现使人们对氮循环过程有了一个全新的认识。通过分析宏基因组学数据发现Comammox细菌与硝化螺旋菌属spp.中其他微生物生境相似,广泛分布于土壤、淡水湿地、地热温泉、污水处理厂和饮用水处理系统中[3, 5-6]。然而,Comammox在滨海湿地生态系统中的分布特征和生态学重要性却鲜有报道。
崇明东滩位于长江口崇明岛的最东端,是长江口规模最大、发育最完善的河口型潮汐滩涂湿地。入海径流携带的泥沙在河口附近沉积,塑造了广阔的潮滩湿地。随着经济的发展,人类对土地的需求越来越迫切,滩涂围垦改农田成为解决土地问题的有效途径。一方面,围垦区的土壤来自于滩涂淤积的泥沙,土壤发育时间相对较短,其土壤本底较为均一;另一方面,围垦后的湿地不再受潮汐影响,与近海间的物质能量交换基本消失,特别是水稻种植、翻耕、施肥等农业管理措施,使围垦区生境发生显著变化,形成了独特的湿地类型:围垦区稻田湿地,特别是大量的农业活性氮进入稻田生态系统后,使其成为氮循环的热区。因此,开展滨海河口湿地土壤氨氧化过程及其相关功能微生物的研究,有助于深入认识围垦造田和农业氮输入对氮关键转化过程的作用机理,具有重要的理论意义与科学价值。
基于此,本文以典型滨海河口湿地——崇明东滩为研究区,采用空间代替时间的研究方法,研究围垦造田条件下滨海湿地土壤的好氧氨氧化过程,探讨其空间变异特征及主要影响因素;通过微生物分子生物学研究方法,分析湿地土壤中全程氨氧化微生物(Clade A 和 Clade B)和经典氨氧化微生物(AOA 和 AOB)的丰度变化,评估这些微生物功能基因的生态位分化及其对硝化作用的相对贡献,以期为长江口湿地环境保护与生态治理提供重要的参考依据。
1 材料和方法
1.1 土壤样品采集
采样地点为上海市崇明岛(121°09′ ~ 121°54′E,31°27′ ~ 31°51′N),属于典型的亚热带季风气候,终年温热,降水充沛,年均温15.3 ℃,年降水量1 003.7 mm[7]。本研究选取东滩湿地保护区滩涂土壤为围垦 0 a土壤作为对照,其他3个水稻土的围垦年限分别为27、51、86 a。围垦植稻不同年限的实验样点根据Cui等[8]的参考文献选取。为了使采样点能更准确反映不同的土壤发育年限,更具代表性,在每个样区内以S形设置6个采样点,各采样点间距约为10 ~ 15 m,用土钻取表层0 ~ 20 cm鲜土,并将该6个采样点的土壤均匀混合为1个混合样本,于保温箱加冰袋保存运回实验室冷冻保存备用。
1.2 土壤理化性质测定
土壤理化性质的分析测定主要参考《土壤农业化学分析方法》[9]。土壤全氮含量采用半微量凯氏定量法测定。土壤总有机碳采用浓硫酸–重铬酸钾消煮,硫酸亚铁滴定法测定。土壤NH4+-N和NO– 3-N用2 mol/L KCl溶液浸提后,采用AA3流动分析仪测定;亚硝态氮采用重氮反应–比色法测定。土壤pH采用水土比为2.5︰1提取水溶液后,用数字酸度计(PHSJ- 6L,上海仪电科学仪器股份有限公司)测定;土壤电导率用电导法测定,采用水土为5︰1提取水溶液后用电导率仪(DDSJ-319L,上海仪电科学仪器股份有限公司)测定。
硝化速率的测定采用好气培养法[10],取沉积物土样10 g(干基),加15 ml去离子水,将土样分散均匀,形成悬浊液。称取总重量并记录。25 ℃避光预培养7 d以便激活土壤微生物。预培养结束后,将每个土样分为两组:第一组的3个锥形瓶,直接测定其中的NO– 3-N含量(0)。第二组的6个锥形瓶中,用移液枪加入(NH4)2SO4溶液使N最终浓度达到200 mg/L,再根据重量法补加去离子水,使之去离子水总量为20 ml。然后在恒温培养箱中25 ℃避光培养7 d后,取出锥形瓶,测定其中的NO– 3-N 含量(= 7)。
硝化速率的计算公式为:

式中:为硝化速率(N, mg/(kg·d));(NO– 3)和(NO– 3)0分别为7 d和0 d 时NO– 3-N含量。
1.3 土壤DNA提取
利用Fast DNA Spin kit for soil提取试剂盒 (MPBiomedicals, USA) 提取土壤样品中的总DNA,取0.5 g土壤样品,按试剂盒提供的试剂和步骤提取DNA,FastPrep®FP120 细胞破碎仪以速度 6 m/s,时间 40 s进行细胞破碎。取部分DNA提取液用分光光度计(NanoDrop -1000 UV-Vis)测定DNA浓度和纯度( OD260 /OD280和OD260 /OD230)。土壤DNA保存于–80 ℃冰箱待用。
1.4 实时荧光定量PCR
在C1000TMReal-Time System扩增仪上进行实时荧光定量PCR扩增。测定氨氧化古菌(AOA)、氨氧化细菌(AOB)和全程氨氧化细菌(Comammox Clade A与总Comammox)的基因拷贝数。AOB、AOA、总Comammox和Comammox Clade A的基因定量 PCR 所用扩增引物和扩增条件见表1[6,11-14]。反应体系均为20 μl,包括DNA样品1 μl、Taq DNA聚合酶10 μl、前后引物各0.5 μl、无菌水8 μl。

表 1 AOA, AOB, Comammox Clade A和总Comammox 的amoA基因引物序列及其扩增条件
提取AOA、AOB、Comammox Clade A和总Comammox的基因的重组质粒,并通过测序验证,再用分光光度计( NanoDrop ND-1000 UV-Vis)测定质粒浓度,并用无菌水将质粒稀释6 ~ 8个梯度,用于制作定量PCR的标准曲线,根据标准曲线计算出目的基因的拷贝数。Comammox Clade B基因的拷贝数为总Comammox 与Comammox Clade A的差值。
1.5 数据统计分析
用IBM Statistics SPSS 16.0软件进行统计分析,通过单因素方差分析(one-way ANOVA)和Pearson分析土壤理化性质、氨氧化微生物丰度的差异性以及相关性检验,显著性水平= 0.05。
2 结果与分析
2.1 不同围垦年限湿地土壤的理化性质变化特征
崇明岛湿地不同围垦年限土壤理化性质如表2所示。长期植稻显著提升了水稻土无机氮含量,降低了硫酸盐含量。围垦稻田土壤NO– 3含量为11.79 ~ 18.67 mg/kg,是未围垦滩涂湿地土壤的1.8倍 ~ 2.9倍;围垦稻田土壤NH4+含量为14.48 ~ 23.37 mg/kg,比未围垦滩涂湿地土壤高出1.1倍 ~ 2.7倍;且围垦51 a稻田土壤的NH4+和NO– 3含量均显著高于其他3个采样点。土壤NO– 2含量在不同采样点之间没有显著差异。土壤SO2– 4 浓度与EC值的变化趋势一致,也随着围垦年限增加而明显减小,且未围垦滩涂湿地土壤显著高于围垦稻田。围垦稻田的土壤总有机碳(TOC)介于15.29 ~ 18.34 g/kg,显著高于未围垦的自然滩涂湿地,且随围垦年限增加有显著增加的趋势。土壤全氮(TN)含量介于0.84 ~ 1.12 g/kg,4个采样点之间没有显著差异。未围垦滩涂湿地土壤的pH为7.86,显著高于围垦稻田。土壤电导率随着围垦年限的增加显著减小,且未围垦滩涂湿地土壤的EC值是围垦稻田的3.2倍 ~ 6.9倍。
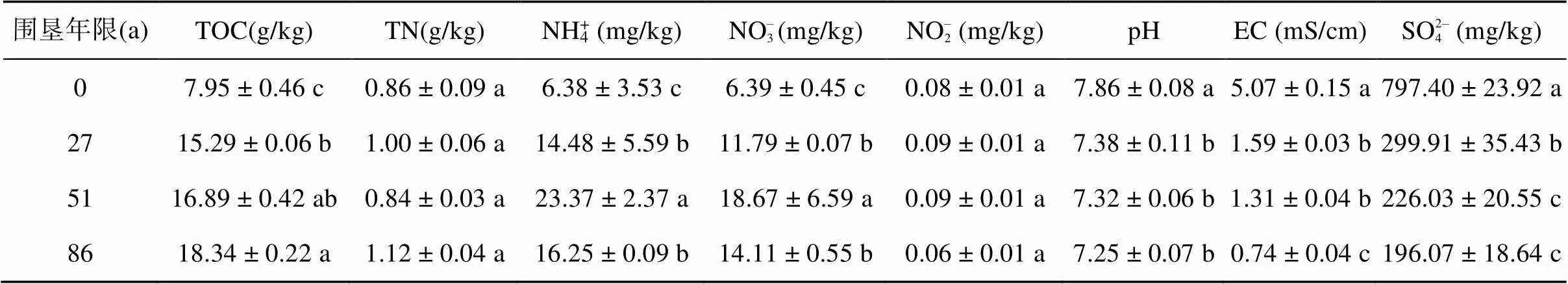
表2 不同围垦年限土壤的理化性质
注:同一列数据小写字母不同表示不同围垦年限处理间差异达<0.05显著水平,下表同。
2.2 不同围垦年限湿地土壤的硝化速率
如图1所示,围垦0 a的自然滩涂湿地硝化速率最低,仅为N 2.22 mg/(kg·d),围垦27、51、86 a后水稻土硝化速率分别为N 19.3、11.6和11.4 mg/(kg·d),增幅高达8.7倍、5.2倍和5.1倍。其中,围垦27 a稻田土壤的硝化速率显著高于围垦51 a和86 a土壤,分别高出66% 和67%。

图1 不同围垦年限土壤的硝化速率
2.3 氨氧化细菌和古菌的丰度特征
由图2可知,围垦0、27、51、86 a的土壤AOA的基因拷贝数(以每克干土计)分别为0.34×107、1.10×107、4.07×107、9.95×107copies/g,随着围垦年限增加而显著增大(< 0.05)。围垦27、51、86 a的稻田土壤AOB的基因拷贝数(以每克干土计)为8.43×107、6.96×107、6.69×107copies/g,分别是围垦0 a滩涂湿地土壤的7.4倍、6.1倍和5.9倍。相关性分析发现,4个采样点的AOB基因拷贝数与土壤硝化速率呈显著正相关(= 0.96*)。
2.4 全程氨氧化细菌的丰度特征
近年来国外研究者发现了具备单独将NH4+氧化为NO– 3的可纯培养细菌,均属于亚硝酸盐氧化细菌中的硝化螺菌属,被称为全程氨氧化细菌。由图3可知,围垦0、27、51、86 a土壤的Comammox Clade A 拷贝数分别为1.04×107、0.14×107、0.64×107、1.00×107copies/g。其中,围垦稻田土壤的Clade A 拷贝数随着围垦年限增加而显著增大,但均低于围垦0 a的滩涂湿地土壤。Comammox Clade B在4个采样点中的丰度特征与Clade A变化特征有所不同,其拷贝数分别为5.73×106、1.78×106、0.48× 106、2.04×106copies/g;其中,围垦稻田土壤的Clade B 拷贝数均显著低于围垦0 a的滩涂湿地土壤。

图2 不同围垦年限湿地土壤AOA和AOB amoA功能基因拷贝数
2.5 不同氨氧化微生物之间的丰度比值
由表3可见,崇明岛围垦年限0、27、51、86 a的土壤中,不同氨氧化微生物之间的丰度比值AOA/AOB介于0.13 ~ 1.49,表明这两类微生物的功能基因拷贝数大致在一个数量级水平;Clade A/Clade B介于0.77 ~ 13.35,表明Clade A的功能基因与Clade B的变化范围较大;AOA、AOB与Clade A 的功能基因拷贝数的比值介于0.33 ~ 61.43。AOA、AOB与Clade B 的功能基因拷贝数的比值介于0.60 ~ 146.18。

图3 不同围垦年限Comammox amoA功能基因拷贝数
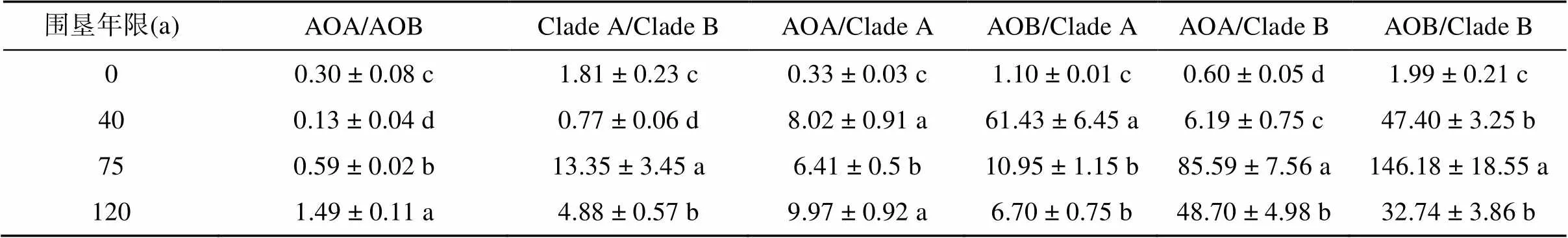
表 3 不同氨氧化微生物的丰度比值
2.6 氨氧化微生物丰度与土壤理化性质的相关性
对围垦0、27、51、86 a的湿地土壤理化性质与氨氧化微生物丰度进行了相关性分析。由表4可见,AOB基因拷贝数与NH4+、NO– 3呈正相关,且与NO– 3含量达显著水平;而Clade A和Clade B均与NH4+、NO– 3呈负相关,且与NH4+含量都达到显著水平。推测全程氨氧化微生物(Clade A、Clade B)更适应于营养贫瘠条件,而AOB可能在活性氮源充足条件下主导硝化作用。TOC、NO– 2与AOA、Clade A、Clade B之间呈负相关关系,而与AOB呈正相关,且TOC与AOB、Clade A、Clade B的相关性达到显著水平。4类氨氧化微生物的基因拷贝数均与pH呈正相关,且与AOA的相关性显著。SO2– 4、EC与AOB丰度呈显著负相关,与Clade B表现为显著正相关。
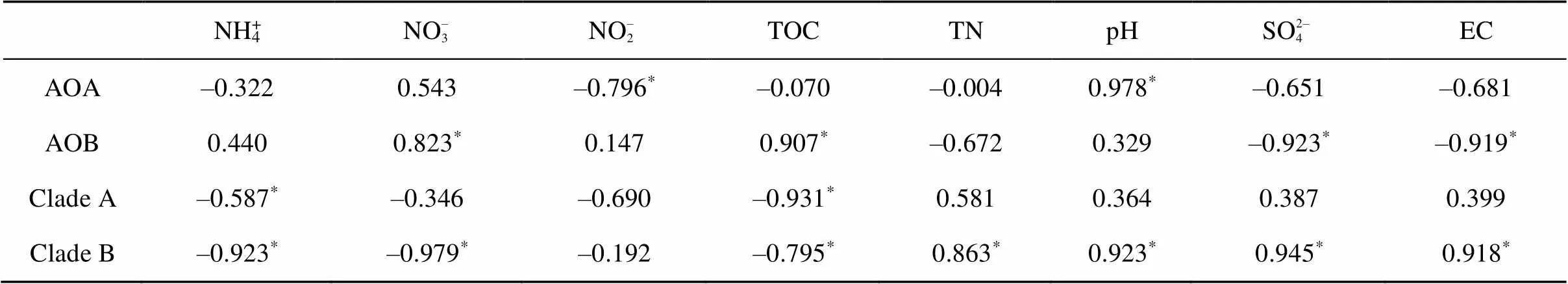
表 4 氨氧化微生物丰度与土壤基本理化性质的相关性
注:* 表示相关性达< 0.05显著水平。
3 讨论
本试验以长江口崇明岛东滩湿地为对象来研究典型氨氧化微生物(AOB、AOA)与全程氨氧化细菌(Comammox)的分布及丰度特征,对了解长江口湿地生态系统中硝化作用微生物分布及数量特征具有重要意义。本研究中,不同围垦年限(0、27、51、86 a)的湿地土壤中均检测到全程氨氧化细菌,证实了全程氨氧化细菌存在于长江口崇明岛围垦稻田和自然滩涂湿地中。国外研究者基于宏基因组数据库比对功能基因,推测全程氨氧化细菌广泛存在于稻田土壤[3,5]、湿地[3]、淡水生态系统、工程环境[6]等。Wang等[2]发现不同地区自来水处理厂的样本中Comammox丰度远高于AOA和AOB的的丰度,达到2倍 ~ 5倍。不同围垦年限的AOA、AOB与全程氨氧化细菌(Clade A或Clade B)的功能基因拷贝数的比值远远大于1(表4),表明在此生境中经典氨氧化微生物比全程氨氧化细菌在数量上更具优势。本研究的采样区域为海陆交错带,其全程氨氧化细菌两个分支Clade A与Clade B 的功能基因的检测结果,对于揭示长江入海河口区域中是否存在全程氨氧化细菌可提供重要的参考数据。在本研究中,Clade A基因拷贝数与AOA、AOB的数量级相同,均为107copies/g,且Clade A与Clade B比值范围为0.8 ~ 13.4,说明在长江口围垦稻田湿地与自然滩涂湿地中全程氨氧化细菌主要以Comammox Clade A居多。万琪慧等[15]在紫色水稻土中也检测到Comammox Clade A和Clade B,且Clade A/Clade B比值范围为16 ~ 34,表明两个分支以Clade A为主,本试验结果与此一致。新近,国外研究者在意大利农田土壤中检测到了两分支的Comammox丰度值均高于104genecopies/ng(以DNA计),其中Comammox Clade A的基因拷贝数比Comammox Clade B高出2 ~ 3个数量级[6],暗示了这些生境中Comammox Clade A在氮硝化过程中可能发挥着重要的作用。
通常认为,氮素含量丰富,且中性至碱性的土壤有利于AOB生长繁殖,施加氮肥的稻田土壤往往以AOB起主导作用进行硝化过程。钟文辉等人[16]研究发现长期施用氮肥提高氨氧化细菌数量可能是施氮土壤硝化率和硝化势较高的原因之一。本试验中,相关性分析发现AOB与土壤TOC、NO– 3呈显著正相关;随着围垦年限增加,硝化速率和AOB的基因拷贝数的变化趋势一致,两者呈显著正相关,表明AOB对氨氧化过程起主导作用。黄蓉等[17]在提高酸性红壤的pH后,发现AOB提高2个数量级,而硝化速率也显著增加,本试验结果与此一致。Comammox的基因拷贝数均明显低于AOB,且与硝化速率呈显著负相关,暗示了Comammox对硝化速率的相对贡献较小。两类全程氨氧化细菌即Comammox Clade A与Comammox Clade B有相似规律,未开垦滩涂湿地的基因拷贝数普遍高于围垦稻田土壤,暗示这类微生物在滩涂自然湿地的氮转化过程中可能起着重要的作用。
大量的研究结果发现,AOA比AOB对氨具有更高的亲和力,因此当氨浓度很低时,AOA对氨的竞争远胜于AOB。Martens等[18]发现在海洋中的AOA可以通过调整细胞机制在低于10 nmol/L的底物浓度环境中进行生长。最近的研究发现Comammox对氨的亲和力大于AOA。本研究中,Comammox分支A和B(Comammox Clade A与Comammox Clade B)的A基因拷贝数与TOC、NH4+浓度均呈显著负相关(表4),推测在没有围垦的自然滩涂湿地中,TOC、活性氮含量少的贫营养环境可能更有利于Comammox的生长繁殖。
综上所述,长江口湿地中检测到ComammoxA基因,且与AOA和AOB共同生存,可能存在对底物和碳源的竞争。如要精确计算三者对表观硝化速率的相对贡献,需要进一步考察其RNA反转录、蛋白质等代谢水平[19],进一步从宏基因组、转录组、蛋白质组、代谢组等方面探究其生理生化特性,从而可以准确评估其实际对硝化作用的贡献率。
4 结论
1)本试验在4种围垦年限的稻田土壤中检测到全程氨氧化细菌(Comammox)A基因,表明Comammox广泛分布在该围垦地区,且以分支Clade A为主。
2)未开垦滩涂湿地中Comammox Clade A 和Clade BA基因普遍高于围垦稻田土壤。
3)半程氨氧化细菌(AOB)与硝化速率随围垦年限的变化一致,可能对硝化速率贡献最大。
[1] Nunes-Alves C. Do it yourself nitrification[J]. Nature Reviews Microbiology, 2016, 14(2): 61.
[2] Wang Y L, Ma L P, Mao Y P, et al. Comammox in drinking water systems[J]. Water Research, 2017, 116: 332–341.
[3] Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504–509.
[4] Costa E, Pérez J, Kreft J U. Why is metabolic labour divided in nitrification?[J]. Trends in Microbiology, 2006, 14(5): 213–219.
[5] van Kessel M A H J, Speth D R, Albertsen M, et al. Complete nitrification by a single microorganism[J]. Nature, 2015, 528(7583): 555–559.
[6] Pjevac P, Schauberger C, Poghosyan L, et al. AmoA- targeted polymerase chain reaction primers for the specific detection and quantification of comammox Nitrospira in the environment[J]. bioRxiv, 2017, DOI:10.1101/096891.
[7] Compilation Committee of Chongming Annals. Chongming county annals[M]. Shanghai: Shanghai People’s Publishing House, 1989: 42.
[8] Cui J, Liu C, Li Z L, et al. Long-term changes in topsoil chemical properties under centuries of cultivation after reclamation of coastal wetlands in the Yangtze Estuary, China[J]. Soil and Tillage Research, 2012, 123: 50–60.
[9] 鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000.
[10] Chu H Y, Fujii T, Morimoto S, et al. Community structure of ammonia-oxidizing bacteria under long-term application of mineral fertilizer and organic manure in a sandy loam soil[J]. Applied and Environmental Microbiology, 2007, 73(2): 485–491.
[11] Beman J M, Francis C A. Diversity of ammonia-oxidizing Archaea and bacteria in the sediments of a hypernutrified subtropical estuary: Bahía del Tóbari, Mexico[J]. Applied and Environmental Microbiology, 2006, 72(12): 7767– 7777.
[12] Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia- oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704–4712.
[13] Yu C D, Hou L J, Zheng Y L, et al. Evidence for complete nitrification in enrichment culture of tidal sediments and diversity analysis of clade a comammox Nitrospira in natural environments[J]. Applied Microbiology and Biotechnology, 2018, 102(21): 9363–9377.
[14] Wang M Y, Huang G H, Zhao Z R, et al. Newly designed primer pair revealed dominant and diverse comammox amoA gene in full-scale wastewater treatment plants[J]. Bioresource Technology, 2018, 270: 580–587.
[15] 万琪慧, 王书玲, 赵伟烨, 等. 重庆紫色水稻土中“全程”和“半程”氨氧化微生物的垂直分异[J]. 微生物学报, 2019, 59(2): 291-302.
[16] 钟文辉, 蔡祖聪, 尹力初, 等. 种植水稻和长期施用无机肥对红壤氨氧化细菌多样性和硝化作用的影响[J]. 土壤学报, 2008, 45(1): 105–111.
[17] 黄蓉, 张金波, 钟文辉, 等. 土地利用方式对万木林土壤氨氧化微生物丰度的影响[J]. 土壤, 2012, 44(4): 581–587.
[18] Martens-Habbena W, Berube P M, Urakawa H, et al. Ammonia oxidation kinetics determine niche separation of nitrifying Archaea and Bacteria[J]. Nature, 2009, 461(7266): 976–979.
[19] Liu S, Shen L D, Lou L P, et al. Spatial distribution and factors shaping the niche segregation of ammonia- oxidizing microorganisms in the Qiantang River, China[J]. Applied and Environmental Microbiology, 2013, 79(13): 4065–4071.
Effect of Reclamation on Abundance of Comammox Microorganism in Chongming Wetland
SONG Yixuan1,2, JIN Rui1, ZHANG Xinlei1, ZHANG Jie1, JIA Zhongjun2, ZHANG Yaohong1*
(1 Nanjing University of Information Science and Technology, Collaborative Innovation Center for Forecast and Evaluation of Meteorological Disasters/Jiangsu Provincial Key Laboratory of Agricultural Meteorology, Nanjing 210044, China; 2 State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China)
The discovery of comammox broadens our understanding of the nitrification process, but little is known about its natural geographical distribution and its contribution to nitrogen conversion process. In this study, the surface soils of the paddy fields with different reclamation years (0, 27, 51 and 86 a) were selected in the Chongming wetland of the Yangtze River estuary, soil nitrification rates and its influencing factors were determined by aerobic incubation, and the target functional genewas analyzed by real-time fluorescent quantitative PCR to analyze the quantitative variation characteristics of Comammox, ammoxidox bacteria (AOB) and ammonia archaea (AOA). The results showed that the net nitrification rates of paddy soils with 40, 75 and 120 a of reclamation were N 19.26, 11.63 and 11.43 mg/(kg·d), respectively, which were significantly 5.1-8.7 times higher than that of natural tidal wetland (<0.05). Thegene copy number of soil ammonia archaea AOA was between 0.34×107and 9.95×107copies/g, which increased significantly with the increase of reclamation year. Thegene copy number of soil ammonia bacteria AOB ranged from 1.14×107to 8.43×107copies/g, which was significantly positively correlated with soil nitrification rate (= 0.96*,<0.05), indicating that AOB might play a dominated role in nitrification process. The Comammox Clade A and Clade B copy number of the soils in the paddy fields (27, 51 and 86 a) were lower than that of tidal flat wetland soil (no reclamation), and Clade A increased significantly with the increase of the reclamation year. Comammox Clade A/Clade B was significantly negatively correlated with soil total organic carbon (TOC) and ammonium content (NH4+), indicating that the ammonia-oxidizing microbes (Clade A, Clade B) are more suitable for nutritional infertility. In summary, Comammox bacteria is widely present in the coastal wetland soils of the Yangtze River estuary, which possibly may play an important role in nitrogen transformation process of tidal flat natural wetlands.
Reclamation; Comammox; Ammonia oxidation
S154
A
10.13758/j.cnki.tr.2020.06.014
宋怡轩, 金锐, 张鑫磊, 等. 围垦对崇明东滩湿地全程氨氧化微生物的影响. 土壤, 2020, 52(6): 1196–1202.
国家自然科学基金项目(41671247, 41103039),江苏省自然科学基金项目( BK20171455) 和江苏省大学生创新创业训练计划项目(201810300094X, 201710300018)资助。
(yhzhang@nuist.edu.cn)
宋怡轩(1995—),女,四川南充人,硕士研究生,主要从事农业应对气候变化研究。E-mail: 2287784164@qq.com

