FOXM1688-748结构域相互作用蛋白的鉴定
谭拥军 杨仕平 余雳 黄小芹 陈燕 谭桂湘

摘 要:为了鉴定转录因子FOXM1转录激活结构域(C-端第688位到748位氨基酸序列)的互作蛋白,以FOXM1的cDNA为模板,采用聚合酶链式反应(Polymerase Chain Reaction,PCR)方法扩增获得其转录激活结构域序列,克隆进入原核表达载体pGEX-4T2;利用大肠杆菌原核表达获得融合谷胱甘肽S-转移酶(Glutathione S-Transferase,GST)標签的重组蛋白GST-FOXM1688-748;通过GST-pulldown结合质谱方法鉴定FOXM1688-748的互作蛋白,成功构建了原核表达质粒pGEX-4T2-FOXM1688-748,获得了原核表达的重组蛋白GST-FOXM1688-748. 将质谱鉴定获得的互作蛋白进行分析和归类,预示一些互作蛋白可以参与激活FOXM1的转录活性,如RPN2、MISP、MCM7蛋白,一些互作蛋白可以参与调控FOXM1蛋白的稳定性,如USP9Y、CUL4A、HSPB1、BAG2蛋白. FOXM1蛋白的转录激活结构域与许多不同功能的蛋白发生相互作用,暗示该结构域具有重要的分子生物学作用,期望为以FOXM1为靶点的临床药物研发提供实验依据.
关键词:转录因子FOXM1;FOXM1688-748结构域;原核表达;相互作用
中图分类号:Q786 文献标志码:A
Identification of FOXM1688-748 Domain-interacting Proteins
TAN Yongjun†,YANG Shiping,YU Li,HUANG Xiaoqin,CHEN Yan,TAN Guixiang
(College of Biology,Hunan University,Changsha 410082,China)
Abstract:To identify the interacting proteins of transactivation domain of forkhead box protein M1 which is located on the 688th to the 748th amino acid sequence of C-terminal end,the cDNA of FOXM1 was used as a template, the transcription activation domain sequence was amplified by polymerase chain reaction and cloned into the prokaryotic expression vector pGEX-4T2. The recombinant protein GST-FOXM1688-748 fused with glutathione S- transferase tag was obtained by Escherichia coli prokaryotic expression. The interacting proteins were identified by GST-pulldown assay and mass spectrometry. The pGEX-4T2-FOXM1688-748 prokaryotic expression plasmid was constructed successfully and the FOXM1688-748 recombination protein was obtained. According to the mass spectrometry results, the interacting proteins were analyzed and classified to show that some of them activated the transcriptional activity of FOXM1, such as RPN2, MISP, and MCM7 proteins, and some of them regulated the stability of FOXM1 protein, such as USP9Y, CUL4A, HSPB1, and BAG2 proteins. The transcription activation domain of FOXM1 protein interacts with many proteins participating in different pathways, indicating that this domain has an important molecular biological role, which can expect to provide experimental basis for clinical drug development targeting FOXM1.
Key words:FOXM1;FOXM1688-748 domain;prokaryotic expression;interaction
FOXM1(Forkhead box M1)是转录因子FOX家族的成员,该家族成员都有一个保守的翼状螺旋DNA结合结构域[1]. FOXM1几乎在所有肿瘤细胞中高表达,是基因表达和细胞循环机制以及其他重要生理过程的关键调节器[2],参与肿瘤细胞的增殖、迁移、侵袭、转移、上皮-间质转化、肿瘤血管生成、防止细胞过早衰老等过程[3-7],它与癌症的发生和发展存在着至关重要的关系,已被确立用于癌症诊断、治疗和预后[8]. FOXM1蛋白主要包含3个结构域,分别是保守的DNA结合结构域(DBD)、N-端抑制结构域(NRD)、C-端转录激活结构域(TAD)[9]. FOXM1激活靶基因转录的机制中,通过直接结合靶基因启动子后招募其他共激活因子,激活基础转录机器,从而启动转录[10]. 通常情况下,FOXM1的N-端抑制结构域与C-端转录激活结构域相互作用,抑制FOXM1的转录活性[11-12]. FOXM1需要发挥转录激活作用时,主要依赖细胞周期蛋白依赖性激酶(CDKs)和PLK1激酶,对FOXM1的TAD进行磷酸化,使TAD构象发生变化,诱导NRD释放TAD. 随后磷酸化的TAD可募集共激活剂CBP,实现激活基因转录[13].
目前,已发现FOXM1与许多蛋白发生相互作用. 一些互作蛋白作为FOXM1的正调节因子,通过激活FOXM1的功能,增强其在癌细胞中的活性,如NPM、MELK、Pin1等蛋白[14]. 另一些互作蛋白可作为FOXM1的负调节因子,通过抑制FOXM1的功能来减弱其在癌细胞中的活性. 如在蛋白毒性应激下,HSP70与FOXM1的相互作用抑制了FOXM1的DNA结合能力[15]. 此外,FOXM1与其他蛋白相互作用,发挥介导这些蛋白激活或增强其致癌通路的功能. 如在胶质瘤细胞中,FOXM1直接结合β-catenin,增强了β-catenin的核定位和转录活性,从而激活WNT信号通路[16].
近几十年来,对FOXM1的转录激活研究较多集中在其转录激活结构域的氨基酸修饰上,而对直接调控转录激活结构域的研究较少. 为了拓展FOXM1转录激活结构域对其转录活性影响的认识,本文通过体外纯化该结构域的方法,来研究其他可能通过TAD结构域调控FOXM1转录活性的机制,这对揭示FOXM1蛋白的分子功能具有重要的生物学意义.
1 材料与方法
1.1 材 料
质粒pGEX-4T2、FOXM1 CDS序列由本实验室保存;乳腺癌细胞MDA-MB-231购自美国模式培养物保藏所(American Type Culture Collection,ATCC);胎牛血清(FBS)购自GIBCO公司;感受态细胞DH5α、BL21 Rosetta(DE3)、DNA聚合酶、PCR引物、1 kb DNA maker购自北京擎科生物技术有限公司;T4 DNA连接酶购自Ferments公司;限制性内切酶XhoI、BamH1购自Thermo Fisher公司;Glutathione Sepharose 4B珠子、GST抗体、Rabbit二抗购自碧云天生物技术公司;Mouse二抗购自Bio-Rad公司;FOXM1抗体购自CST(cell signaling technology)公司;BAG2抗体购自上海生工生物工程股份有限公司.
1.2 方 法
1.2.1 pGEX-4T2-FOXM1688-748载体的构建
在NCBI中检索FOXM1的CDS区序列,截取其第688位到748位氨基酸对应的核苷酸序列并结合载体pGEX-4T2的MCS位点确定FOXM1688-748的引物. 其上游引物为 GCGGATCCATGTCCCCG
GAGCCACAGGTT,下游引物为GCCTCGAGCTA
CTGTAGCTCAGGAAT,划线部分为限制性内切酶BamH1、XhoI的酶切位点. 以FOXM1的CDS序列为模板,扩增出FOXM1688-748片段. 扩增条件为98 ℃,2 min;98 ℃,10 s;60 ℃,15 s;72 ℃,15 s;35个循环. 将PCR产物克隆入pGEX-4T2空载体中,经BamH1、XhoI双酶切鉴定后送擎科生物有限公司测序.
1.2.2 重组蛋白GST-FOXM1688-748的表达和纯化
将pGEX-4T2-FOXM1688-748、pGEX-4T2质粒转化入感受态细胞BL21中,挑选阳性单克隆,加入含有氨苄的LB培养基中,37 ℃、220 r/min,扩大培养至OD值为0.6~0.8的产物,加入终浓度为0.8 mmol/L IPTG,28.5 ℃诱导过夜,4 000 r/min,离心20 min,收集菌体,加入终质量浓度为200~400 mg/mL溶菌酶冰上裂解30 min. 超声破碎,离心收集上清,加入GST珠子,4 ℃孵育2 h. 离心弃上清,用PBS将珠子洗5遍,GST Elution Buffer洗脱纯化的蛋白,收集蛋白并用考马斯亮蓝染色检测其纯化效果.
1.2.3 细胞培养
MDA-MB-231细胞用体积分数为10%的FBS和1%双抗(100 mg/L青霉素和链霉素)的DMEM完全培养基培养,置于37 ℃、体积分数为5%的CO2、相对湿度为90%的细胞培养箱中培养,当MDA-MB-231细胞密度达到90%后用于Pulldown实验.
1.2.4 GST-pulldown
收集MDA-MB-231细胞,加入500 μL的IP裂解液,以体积比1 ∶ 100的比例加入蛋白酶抑制剂,冰上裂解1 h,12 000 r/min,离心5 min,收集上清. 加入30 μL GST珠子,4 ℃预孵育1 h. 1 000 r/min离心2 min,收集上清,一分为二,分别加入50 μg原核纯化的GST、GST-FOXM1688-748蛋白、30 μL GST珠子,4 ℃孵育2 h. 1 000 r/min,离心2 min收集GST珠子,用预冷的PBS漂洗4次,加入30 μL 1xLoading Buffer,98 ℃变性15 min后进行SDS-PAGE和考马斯亮蓝检测.
1.2.5 质谱分析
将Pulldown拖下来的蛋白通过聚丙烯酰胺凝胶电泳(polyacrylamide gel electrophoresis,PAGE)分離后,切下来送上海胡珀生物有限公司打质谱. 将实验组与对照组蛋白去重后,选择质谱分数大于2的蛋白用UniProtKB在线网站(https://www.uniprot.org/)分析分子功能,将结果进行整理和归类,得到不同功能的蛋白. 为了验证质谱结果的可靠性,挑选潜在的互作蛋白BAG2进行互作验证,方法同1.2.3节中的方法.
2 结果与分析
2.1 pGEX-4T2-FOXM1688-748载体的构建
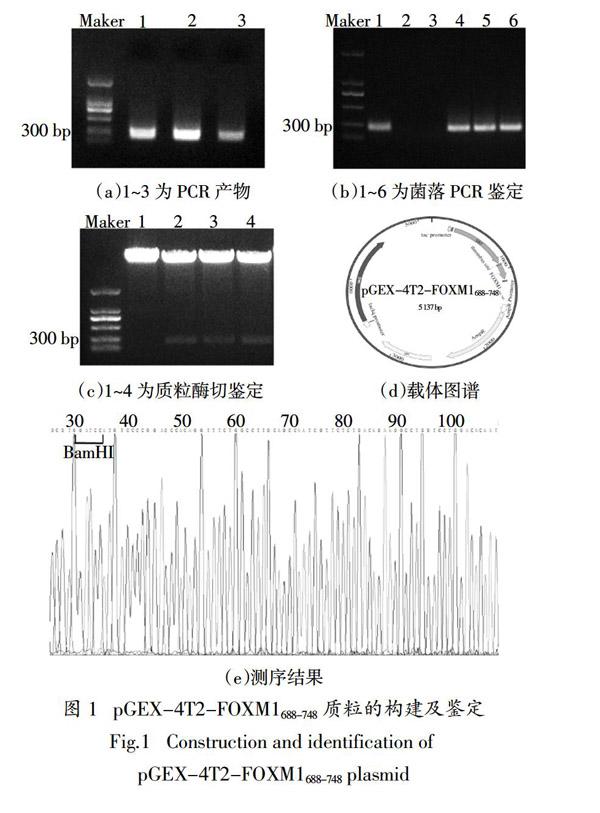
以FOXM1 CDS序列为模板,扩增出FOXM1688-748目的条带,其大小为200 bp左右(图1(a)). 将该片段克隆入经酶切后的pGEX-4T2载体中,进行菌落PCR和双酶切鉴定,琼脂糖凝胶电泳显示其在100~300 bp有明显的条带(图1(b)(c)),pGEX-4T2-FOXM1688-748载体示意图如图1(d)所示. 将构建好的载体进行序列比对,结果显示与本文设计结果相同(图1(e)). 由此可知,本文成功构建了pGEX-4T2-FOXM1688-748原核表达载体.
2.2 重组蛋白GST-FOXM1688-748的表达与纯化
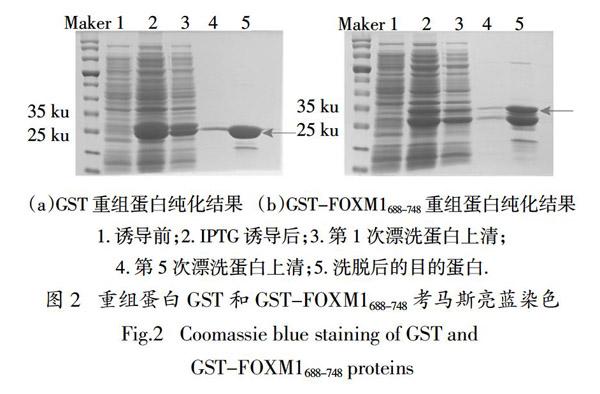
将克隆成功的pGEX-4T2-FOXM1688-748质粒转化入大肠杆菌感受态细胞BL21 Rosetta,挑选阳性单克隆扩大培养,用GST珠子纯化,考马斯亮蓝染色展示纯化结果如图2所示. 由图2可知,GST和GST-FOXM1688-748蛋白的纯化效果较好,并且可以得到较高浓度的GST、GST-FOXM1688-748蛋白,为后续的Pulldown实验提供实验材料.
2.3 FOXM1688-748与FOXM1全长蛋白相互作用
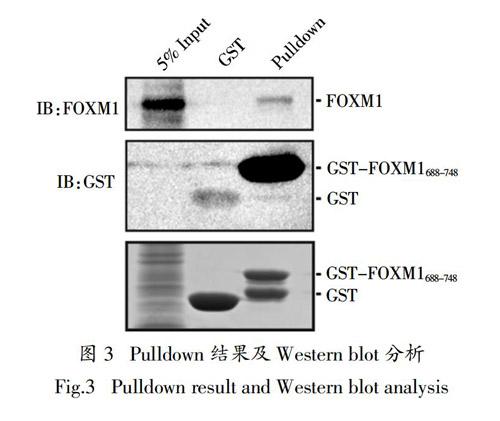
为了验证FOXM1688-748与FOXM1全长蛋白的作用关系,通过GST-pulldown的方法来验证它们的相互作用. 用预清除的MDA-MB-231细胞裂解液分别与GST、GST-FOXM1688-748重组蛋白孵育,用FOXM1、GST抗体进行免疫印记检测. 重组蛋白GST-FOXM1688-748可以拖下其全长蛋白FOXM1,而GST蛋白在同一位置未检测到FOXM1条带,表明FOXM1688-748可以与其全长相互作用(图3),证实了纯化的重组蛋白FOXM1688-748具有良好的生物活性.
2.4 FOXM1688-748互作蛋白的鉴定
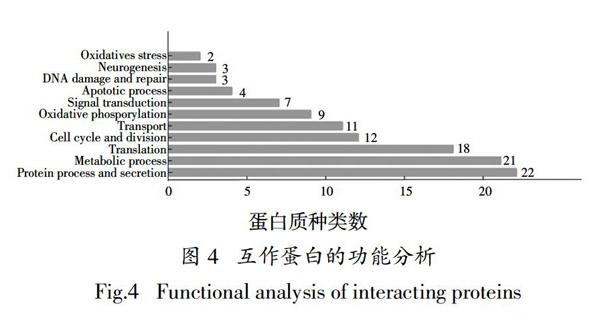
为了研究FOXM1688-748可能的生物学作用,将GST-pulldown拖下来的蛋白送公司进行质谱鉴定,取质谱分数大于2的蛋白进行分析,得到112个蛋白,其中有2个蛋白已被证明与FOXM1的转录激活结构域互作,分别是CDK1和FOXM1. 对质谱中的蛋白进行分类和整理,发现这些蛋白分子功能较多集中于蛋白质的加工和分泌、细胞周期和分裂、翻译过程(图4). 在蛋白质的加工和分泌方面,捕获到的蛋白有HSP90AB1、HSPB1、BAG2、USP9Y、DERL1、UBB、FAM129A等,其中HSP90AB1、HSPB1、BAG2与蛋白质的折叠有关,USP9Y、DERL1、UBB与蛋白质的泛素化有关,FAM129A参与翻译后蛋白质的磷酸化. 在细胞周期和分裂方面,捕获到的蛋白有CDK1、FOXM1、MCM7、RPN2、MISP、NCAPG等,这些蛋白在细胞分裂过程中发挥了重要的作用. 在翻译方面,捕获到的蛋白有GCN1、RACK1、LARS、DDX39A、HNRNPA0等,GCN1、RACK1、LARS参与蛋白质的合成过程,而DDX39A、HNRNPA0分别参与mRNA的前期剪接和细胞因子mRNA的转录后调控.
2.5 FOXM1688-748与BAG2的互作验证
为了验证质谱结果的可靠性,从质谱中挑选潜在的互作蛋白BAG2进行互作验证. 在Pulldown实验结果中,原核表达的FOXM1688-748 可与MDA-MB-231细胞中内源的BAG2相互作用(图5). 上述结果证实了质谱结果的可靠性,并为研究FOXM1 C端互作蛋白的功能及分子机制提供实验依据.
3 讨 论
Pulldown是一种广泛用于体外检测两种或多种蛋白质之间的物理相互作用的方法,以确认预测的蛋白质-蛋白质之间的相互作用或捕获新的互作蛋白[17]. 在这项研究中,本文成功纯化了FOXM1转录激活结构域FOXM1688-748,并利用Pulldown实验证实它能够与FOXM1全长蛋白互作,这是由于FOXM1的NRD可以与其TAD相互作用[11]. 由于内源FOXM1的TAD需要在细胞周期蛋白依赖性激酶的磷酸化后才能被NRD释放,为了直接获得与FOXM1转录激活结构域相互作用的蛋白,利用GST-pulldown方法,在不受NRD影响的情况下,通过质谱鉴定得到一系列与其相互作用的蛋白. FOXM1在肿瘤的发生发展中具有至关重要的作用,这离不开各种互作蛋白对FOXM1的翻译后调控. 在质谱结果中,我们捕获到一些已知的FOXM1互作蛋白,如CDK1. 在细胞分裂的S期或M期,CyclinB1/CDK1复合物结合FOXM1的转录激活结构域,诱导CDK1磷酸化FOXM1,磷酸化后的FOXM1招募共激活因子CBP,从而激活FOXM1的转录活性[10]. 根据质谱结果中蛋白质的功能,预示RPN2、MISP、MCM7、FAM129A蛋白可能参与调节FOXM1的转录活性.
除了对FOXM1翻译后的修饰实现的调控外,一些互作蛋白还可以参与维持FOXM1蛋白的稳定性,它们通过对FOXM1的去泛素化作用来增强FOXM1的稳定性,如USP21. 在基底样乳腺癌细胞中,USP21与FOXM1互作,使FOXM1去泛素化免受蛋白酶体的降解[18]. 质谱结果中也存在一些泛素化分子,如USP9Y、CUL4A,预示这些蛋白参与FOXM1蛋白的泛素化或去泛素化,从而调节其蛋白质的稳定性. 此外,一些与蛋白质折叠有关的分子伴侣也出现在质谱结果中,如HSPB1、USP9Y、BAG2. Pulldown实验也证实BAG2与FOXM1的互作,预示这些蛋白可能通过对FOXM1蛋白的折疊来维持其稳定性.
FOXM1作为经典的转录因子,其主要的功能是结合到下游靶基因的启动子上,招募一些转录复合体,实现对靶基因的转录激活[19]. 质谱中也出现了一些与转录相关的蛋白,如RuvBL1. RuvBL1是NuA4组蛋白乙酰转移酶复合物的成分,主要通过对核小体组蛋白H4和H2A的乙酰化参与靶基因的转录激活,这种修饰既可以改变核小体与DNA的相互作用,又可以促进修饰的组蛋白与其他可正向调节转录的蛋白质的相互作用[20]. 预示RuvBL1可能被招募到转录复合体中,参与FOXM1对下游靶基因的激活.
綜上所述,利用FOXM1688-748重组蛋白为原料,通过Pulldown结合质谱的方法获得了FOXM1转录激活结构域的潜在互作蛋白,并对这些潜在互作蛋白的分子功能进行分析归类. 这为后续研究FOXM1转录激活结构域的互作蛋白提供可靠的实验依据,对探究FOXM1分子的生物学功能具有重要的意义.
参考文献
[1] LI Y M,WU F,TAN Q,et al. The multifaceted roles of FOXM1 in pulmonary disease[J]. Cell Communication and Signaling,2019,17(35):1—16.
[2] NANDI D,CHEEMA P S,JAISWAL N,et al. FOXM1:repurposing an oncogene as a biomarker[J]. Seminars in Cancer Biology,2018,52:74—84.
[3] ZHONG S B,ZHOU A Y,QI F H,et al. Downregulating forkhead box M1 inhibits proliferation by inhibiting autophagy in the SW480 cell line[J]. Biomedical Reports,2017,7(1):47—50.
[4] ZHANG J,NIU Y,HUANG C. Role of FOXM1 in the progression and epithelial to mesenchymal transition of gastrointestinal cancer[J]. Recent Patents on Anti-Cancer Drug Discovery,2017,12(3):247—259.
[5] JIANG L,WANG P,CHEN L,et al. Down-regulation of FOXM1 by thiostrepton or small interfering RNA inhibits proliferation,transformation ability and angiogenesis,and induces apoptosis of nasopharyngeal carcinoma cells[J]. International Journal of Clinical and Experimental Pathology,2014,7(9):5450—5460.
[6] CHOI Y W,NAM G E,KIM Y H,et al. Abrogation of B-RafV600E induced senescence by FOXM1 expression[J]. Biochemical and Biophysical Research Communications,2019,516(3):866—871.
[7] ZHANG Z Y,TU K J,LIU F Y,et al. FOXM1 promotes the migration of ovarian cancer cell through KRT5 and KRT7[J]. Gene,2020,757:144947.
[8] ALVAREZ-FERNÁNDEZ M,MEDEMA R H. Novel functions of FOXM1:from molecular mechanisms to cancer therapy[J]. Frontiers in Oncology,2013,3(30):1—5.
[9] LIAO G B,LI X Z,ZENG S,et al. Regulation of the master regulator FOXM1 in cancer[J]. Cell Communication and Signaling,2018,16(57):1—15.
[10] MAJOR M L,LEPE R,COSTA R H. Forkhead box M1B transcriptional activity requires binding of CDK-cyclin complexes for phosphorylation-dependent recruitment of p300/CBP coactivators[J]. Molecular and Cellular Biology,2004,24(7):2649—2661.
[11] PARK H J,WANG Z,COSTA R H,et al. An N-terminal inhibitory domain modulates activity of FOXM1 during cell cycle[J]. Oncogene,2008,27(12):1696—1704.
[12] ANDERS L,KE N,HYDBRING P,et al. A systematic screen for CDK4/6 substrates links FOXM1 phosphorylation to senescence suppression in cancer cells[J]. Cancer Cell,2011,20(5):620—634.
[13] MARCEAU A H,BRISON C M,NERLI S,et al. An order-to-disorder structural switch activates the FOXM1 transcription factor[J]. Elife Sciences,2019,8:e46131.
[14] GARTEL A L. FOXM1 in cancer:interactions and vulnerabilities[J]. Cancer Research,2017,77(12):3135—3139.
[15] HALASI M,VÁRALJAI R,BENEVOLENSKAYA E,et al. A novel function of molecular chaperone HSP70:suppression of oncogenic FOXM1 after proteotoxic stress[J]. Journal of Biological Chemistry,2016,291(1):142—148.
[16] ZHANG N,WEI P,GONG A,et al. FOXM1 promotes β-catenin nuclear localization and controls WNT target-gene expression and glioma tumorigenesis[J]. Cancer Cell,2011,20(4):427—442.
[17] LOUCHE A,SALCEDO S P,BIGOT S. Protein-protein interactions:pull-down assays[J]. Methods in Molecular Biology,2017,1615:247—255.
[18] ARCECI A,BONACCI T,WANG X X,et al. FOXM1 deubiquitination by USP21 regulates cell cycle progression and paclitaxel sensitivity in basal-like breast cancer[J]. Cell Reports,2019,26(11):3076—3086.
[19] WIERSTRA I. Chapter three-the transcription factor FOXM1 (Forkhead box M1):proliferation-specific expression, transcription factor function, target genes, mouse models, and normal biological roles[M]. San Diego:Elesvier Academic Press Inc,2013:164-167.
[20] DOYON Y,SELLECK W,LANE W S,et al. Structural and functional conservation of the NuA4 histone acetyltransferase complex from yeast to humans[J]. Molecular and Cellular Biology,2004,24(5):1884—1896.
收稿日期:2020-12-10
基金項目:国家自然科学基金资助项目(81472718),National Natural Science Foundation of China(81472718)
作者简介:谭拥军(1967—),男,湖南长沙人,湖南大学教授,博士生导师
通信联系人,E-mail:yjtan@hnu.edu.cn

