中国春小麦肌醇磷脂依赖的磷脂酶C基因的全基因组鉴定及表达分析
司旭阳,贾哓玮,张洪艳,贾羊羊,田士军,张科,潘延云
中国春小麦肌醇磷脂依赖的磷脂酶C基因的全基因组鉴定及表达分析
司旭阳1,贾哓玮1,张洪艳1,贾羊羊1,田士军1,张科2,潘延云1
(1河北农业大学生命科学学院/河北省植物生理与分子病理学重点实验室,河北保定 071000;2河北农业大学农学院/省部共建华北作物改良与调控国家重点实验室/河北省作物生长调控实验室,河北保定 071001)
【】探知小麦基因组序列中肌醇磷脂依赖的磷脂酶C(PLC)的编码基因,解析小麦中PLC基因的结构与进化特征,揭示TaPLC基因在小麦各组织中的表达模式及其响应盐胁迫和干旱胁迫过程中的表达规律,以便深入分析小麦TaPLC基因调节小麦应答盐或干旱胁迫中的生理作用。基于Ensembl Plants全基因组数据库,以水稻和拟南芥PLC基因为参考序列,检索小麦TaPLC基因家族(http://plants.ensembl.org/index.html),并利用在线蛋白结构分析工具(Pfam、CDD和SMART)对TaPLC基因进行结构分析和鉴定;运用ExPASy(http://cn.expasy.org/tools)分析TaPLC基因的分子和生化特征;使用WoLF PSORT(https://www.genscript.com/wolf-psort.html)预测TaPLC基因的亚细胞定位;运用MEGA 7.0软件进行系统进化树分析;并利用实时荧光定量PCR分析TaPLC基因在不同组织以及幼苗期在盐或干旱胁迫下的表达特征。在小麦基因组中鉴定了11个TaPLC基因序列,其编码蛋白与其他植物PLC结构相似,均具有X和Y保守催化结构域以及C2结构域;系统进化分析表明11个TaPLC基因可分为4组,即、、、,均为植物PLC基因家族的直系同源基因,在进化方面具有保守性,但也存在一定的分歧;通过与小麦祖先种比对分析,发现B亚基因组中的在异源六倍体形成前就已经缺失,进一步表明小麦亚基因组间进化的不对称性;亚细胞定位预测TaPLC基因定位于叶绿体、线粒体或细胞质中;TaPLC基因家族各成员的启动子普遍存在植物激素响应和应激响应相关的顺式调控元件;实时定量结果表明,不同的TaPLC基因在不同的组织中有不同的表达水平,其主要在根、叶、穗和籽粒中表达;且多数基因参与小麦对盐和干旱胁迫的响应,无论是在耐旱和耐盐品种中,还是在对照品种中,TaPLC基因均受到胁迫的诱导表达,并在12 h内迅速达到峰值。在小麦基因组中共鉴定11个TaPLC基因,编码典型的植物PLC蛋白,在进化上具有保守性,但亚基因组间也存在进化的不对称性。不同的TaPLC基因有各自的组织和细胞表达特征,在小麦生长发育过程中起调节作用;TaPLC基因在应答盐或干旱胁迫中起重要作用。
小麦;肌醇磷脂依赖的磷脂酶C;盐胁迫;干旱胁迫
0 引言
【研究意义】小麦()是世界上最重要的作物之一。随着世界人口的不断增长,粮食安全已成为一个主要问题。干旱、土壤盐分以及极端温度等渗透胁迫是影响植物生长最主要的不利环境[1-2]。植物肌醇磷脂信号系统在应答渗透胁迫,调节植物生长中发挥着重要的作用[3-4]。肌醇磷脂依赖性磷脂酶C(phosphatidylinositol-dependent phospholipase,PI-PLC,简称为PLC)是肌醇磷脂信号系统中的关键酶之一[3-4],研究小麦基因组中编码PLC的基因及其功能对于澄清小麦在胁迫中的应答反应和改良小麦在不利环境中的生长都有重要的理论和应用价值[1,3]。【前人研究进展】在哺乳动物中已鉴定出5种PLC亚型,即β、γ、δ、ε和ζ,被激活的PLC可以水解质膜上的4,5-磷酸磷脂酰肌醇(phosphatidylinositol 4,5-bisphosphate,PIP2)产生二酰甘油(diacylglycerol,DAG)和三磷酸肌醇(inositol 1,4,5-trisphosphate,IP3)双信使分子,分别激活蛋白激酶C(protein kinase C,PKC)和释放胞内钙库Ca2+,进而调控一系列靶蛋白和靶酶的活性,参与细胞生长、增殖、代谢、分泌、收缩等发育过程和生理过程的调节[2]。植物PLC的研究始于20世纪80年代,目前已经在多个物种——拟南芥[5]、大豆[6]、烟草[7-8]、水稻[9]、马铃薯[10]、玉米[11]、百合[12]和绿豆[13-14]中克隆获得PLC基因序列。每个植物物种几乎都由多个PLC基因组成基因家族,而各个成员可能参与不同的生理过程。以拟南芥为例,其基因组编码9个AtPLC家族成员(—),其中可能编码质膜相关蛋白[15],并负调控幼苗对盐的耐受性[16]。是组成型表达基因,其不仅参与对幼苗生长发育和内质网应激反应的调控[17],而且还通过生长素信号调节雌雄配子体的发育[18]。参与初级和次级根的生长调控,其过表达提高了植物的抗旱性[19]。也参与了根系的发育[20],和的过表达都可增加植物对干旱的耐受性,并在ABA调控的种子萌发和气孔运动中发挥作用[20-21]。【本研究切入点】与动物PLC不同,目前发现的植物PLC结构均与动物中最简单的PLC ζ亚型相似。植物PLC的作用模式与动物也不相同,植物细胞中缺乏受IP3调控的受体,IP3被进一步磷酸化形成六磷酸肌醇(inositol hexaphosphate,IP6),参与细胞内钙信号形成,但具体的作用机制尚不清楚;植物中目前也没有鉴定出PKC的同源类似物,DAG可用于磷脂的再生或继续被磷酸化形成磷脂酸(phosphatidic acid,PA),而PA被认为是植物特有的第二信使,调节植物的多种生理过程,但是目前也只鉴定了少数的PA结合蛋白,且蛋白与PA互作的结构域尚未确定[3,22-24]。因此,需要更广泛地分析植物PLC基因的功能,以诠释其作用机制。小麦TaPLC的研究起始较早,1987年,就在小麦根细胞的质膜中检测到PLC活性[25];1992年检测了小麦PLC酶活性的生化特征[26-27]。但目前仅有2个TaPLC基因被克隆,即和(GenBank:HM754654.1和HM754653.1),初步结果显示这两个基因在调节小麦幼苗生长中发挥着作用且表达受到盐或干旱的诱导[28]。基于小麦为异源六倍体的特征以及植物中PLC基因多是以多基因家族形式存在,小麦基因组中应该还有其他的TaPLC基因序列。【拟解决的关键问题】本研究通过全面检索小麦基因组,获得全部TaPLC基因序列,明确其结构、表达模式和进化特征,并检测TaPLC基因在各种组织中以及在干旱或盐胁迫下的表达模式,为全面探究TaPLC基因家族成员的生物学功能奠定基础。
1 材料与方法
1.1 植物材料
所用小麦品种有中国春,洛旱7#(LD)为抗旱小麦品种,科农199(KD)为对照小麦品种,小偃60#(XS)为耐盐小麦品种,石麦15#(SS)为对照小麦品种。
选取小麦两叶一心期的根、叶以及成熟后的茎、穗等材料,液氮速冻后,-80℃保存备用。
小麦在霍氏培养液水培至两叶一心期时,分别移至20% PEG 6000和200 mmol·L-1NaCl的培养液中,于0、0.5、1、2、6、12、24和48 h取叶片,液氮速冻后,-80℃保存备用。
1.2 TaPLC基因的鉴定
首先利用已知的水稻和拟南芥的PLC蛋白序列在全基因组数据库(Ensembl Plants)中查询小麦中的PLC同源基因的序列信息(http://plants.ensembl.org/ index.html)。将检索结果为PLC-like的蛋白序列分别利用Pfam(http://xfam.org/)、CDD(https://www.ncbi. nlm.nih.gov/Structure/cdd/wrpsb.cgi)和SMART(http:// smart.embl-heidelberg.de/)验证蛋白序列是否含有PLC结构保守域(X、Y和C2结构域),含有这3个保守域的序列被认定为小麦TaPLC基因序列。
1.3 基因的染色体定位及序列分析
利用Ensembl Plants网站查询TaPLC基因在染色体上的定位;利用在线工具ExPASy(http://cn.expasy. org/tools)分析TaPLC蛋白的分子和生化特征(如分子量和等电点等);利用MEME获得TaPLC基因基序,并利用Evolview绘制基因结构和基序示意图;在SMART和Pfam中得到TaPLC蛋白结构域,并在IBS软件中绘图;利用PLACE(http://www.dna.affrc.go.jp/ PLACE/)和Plant CARE(http://bioinformatics.psb.ugent. be/webtools/plantcare/html/)分析TaPLC基因启动子区的顺式作用元件。
1.4 多序列比对及进化分析
利用DNAMAN软件对TaPLC蛋白序列进行多序列比对。为了研究不同物种PLC之间的进化关系,从NCBI下载拟南芥、水稻、大豆等物种PLC氨基酸序列,用Clustal W进行多序列比对,然后用MEGA 7.0采用邻接法构建系统发育树,Bootstrap值设置为1 000。利用Evolview在线软件对进化树进行优化处理。
1.5 基因的表达分析
利用在线软件WoLF PSORT(https://www.genscript. com/wolf-psort.html)预测TaPLC蛋白的亚细胞定位;利用The Triticeae Multi-omics Center在线小麦表达数据库(http://202.194.139.32/expression/index.html? tdsourcetag=s_pcqq_aiomsg)以及URGI网站中Wheat Expression Browser(http://www.wheat-expression.com),选择Chinese Spring cv-1 Development,输入gene ID(电子附表1),获得TaPLC基因在根、茎、叶、穗和籽粒的表达数据(TPM值),利用R-3.6.3绘制热图。
利用qRT-PCR的方法检测和验证部分TaPLC基因的表达。首先利用检索到的TaPLC基因序列信息,以cDNA为模板扩增基因全长,并测序,进而根据序列信息设计区别于其他旁系同源基因的特异性定量引物(电子附表2)。
将所取材料研磨成粉末,提取总RNA,并反转录为cDNA,用于实时荧光定量PCR检测。以为内参基因,qRT-PCR的反应体系为模板1/3 μL、2×AceQ qPCR SYBR Green Master Mix 10 μL、上下游引物各0.5 μL(10 μmol·L-1),ddH2O补至20 μL,每个样品进行3次技术重复。反应程序为95℃5 min;95℃ 10 s,60℃ 30 s,40个循环,绘制融解曲线,95℃ 15 s,60℃ 60 s,95℃ 15 s。利用2-ΔΔCT法分析数据,试验设计3个生物学重复。
2 结果
2.1 小麦TaPLC基因家族成员的鉴定
为了全面鉴定小麦TaPLC基因家族成员,利用拟南芥AtPLC基因和水稻OsPLC基因的编码蛋白序列信息对小麦基因组数据库进行局部BLAST比对,并进行关键字搜索和蛋白质域搜索。发现小麦基因组序列中有11个序列同源基因(电子附表1),分别分布在第1、2、4和5染色体(表1)。小麦基因组为AABBDD六倍体,这11个TaPLC基因中有4组直系同源基因,每组直系同源基因分别有A、B和D 3个部分同源基因,分别将其命名为、、、、、、、、、和a。未能在第1B染色体上检出同源序列,推测是由于亚基因组间的不对称进化所导致。
为了进一步确定第1B染色体中TaPLC基因丢失的时间,利用和序列分别检索了AA()二倍体、DD()二倍体和AABB()四倍体小麦祖先种基因组。结果显示,在祖先种的AA和DD基因组中均检索到高度相似同源基因(TRIUR3_33781-T和AET1Gv20175500.1),但在AABB基因组中只在A亚组发现同源基因(TRIDC1AG009650.1),而没有检索到B亚组上的同源基因,揭示B亚基因组中的TaPLC基因在异源六倍体形成之前就已经缺失(电子附图1),暗示了小麦亚基因组间进化的不对称性。
根据Pfam、SMART和CDD的结果,获得的TaPLC基因编码序列都具有X、Y和C2结构域,并用IBS绘制TaPLC蛋白结构域示意图(图1)。通过分析TaPLC基因的分子和生化特性,发现这些基因的ORF长度范围为1 770—1 902 bp,其编码589—633 aa多肽,预测分子量范围为5.54—71.11 kD。理论等电点为5.37—6.07(表1)。
2.2 小麦TaPLC基因的系统发育分析
将检索到的TaPLC蛋白序列与目前已知的植物PLC蛋白序列进行比对和分析,包括9个拟南芥AtPLC基因、4个水稻OsPLC基因和6个番茄SlPLC基因等共15种植物的56个PLC蛋白序列,7种苔藓PLC、2种酵母PLC、2种微藻和3种动物ζ亚型的PLC等共计67个蛋白序列,结果显示,小麦TaPLC基因家族与已知PLC序列有很高的相似性,其中TaPLC2组(即TaPLC2A、TaPLC2B和TaPLC2D)、TaPLC3组和TaPLC4组同源关系较近,它们与水稻、玉米、百合和高粱等单子叶植物的PLC的相似性较高,但TaPLC1组的2个蛋白(TaPLC1A和TaPLC1D)则与双子叶植物(如拟南芥、大豆和番茄等)具有较高的相似度(图2)。结合在B基因组中丢失的现象,说明小麦中的TaPLC基因在进化进程中既具有保守性,也出现了分歧。

数字表示每个结构域在蛋白质序列中的位置
2.3 小麦TaPLC基因的结构特征
为了进一步了解TaPLC基因的发育关系,分析比较TaPLC基因的DNA序列和氨基酸序列的结构,及其外显子-内含子结构。发现同一组的基因(如//)彼此之间不仅核酸序列和氨基酸序列相似性都达到98%以上(电子附表1),而且外显子-内含子结构也具有高度同一性(图3),表明小麦TaPLC基因每组的部分同源基因是经历了基因复制而形成的。组的3个基因均有8个外显子和7个内含子,其他组的基因均有9个外显子和8个内含子,与目前发现的其他植物的PLC基因有着相似的剪接方式。
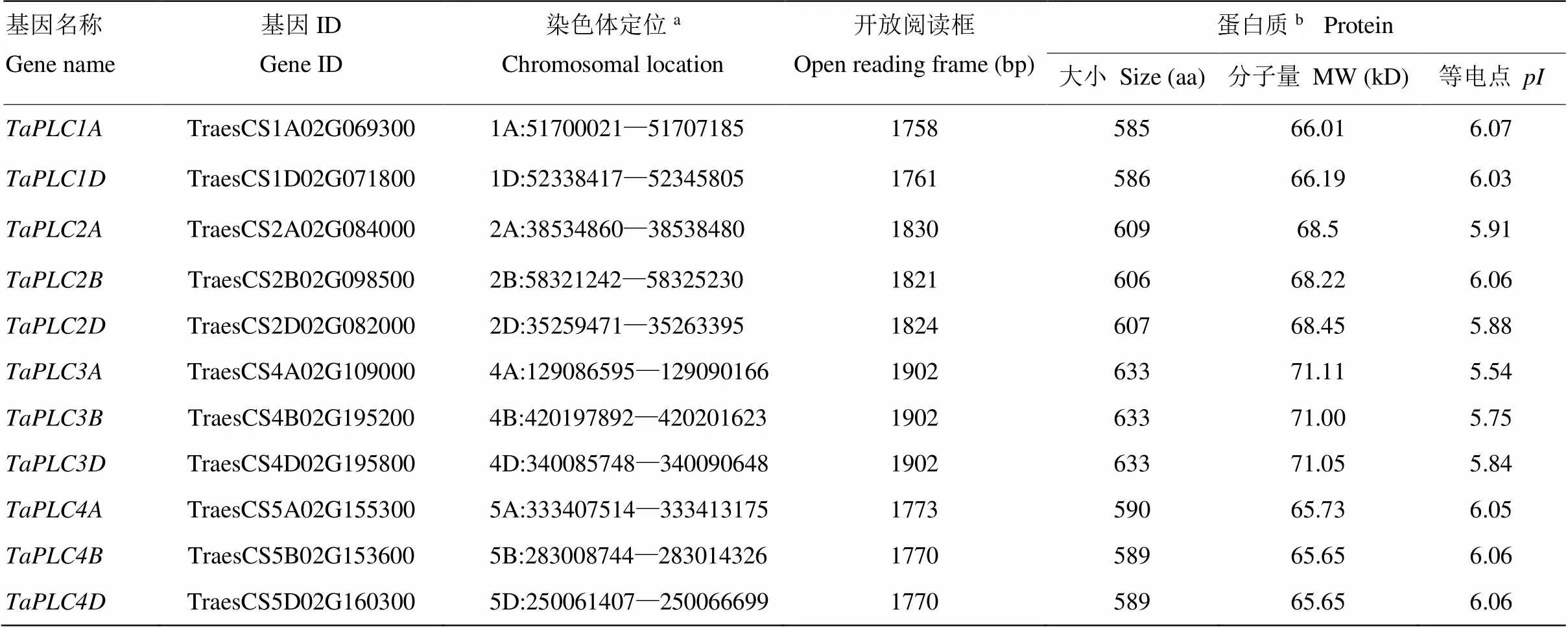
表1 小麦TaPLC基因家族各成员的分子和生化特征
a:染色体位置;b:每个TaPLC基因的核酸和氨基酸序列列于电子附表1
a: Chromosomal location;b: The Nucleic acid and deduced amino acid sequences of each TaPLC gene are listed in S1 Table

☆:双子叶植物;★:单子叶植物;○:松柏类植物;●:石松类植物;:苔藓植物;■:微藻;:酵母;▲:哺乳动物。Ta:小麦;At:拟南芥;Gm:大豆;Os:水稻;Pp:小立碗藓;Ps:云杉;Nt:烟草;Sl:番茄;Ld:百合;Sb:高粱;Pi:矮牵牛;St:马铃薯;Bn:油菜;Sm:卷柏;Mp:细小微胞藻;Ot:金牛鸵球藻;Hs:智人;Sp:裂殖酵母;Sc:酿酒酵母;Zm:玉米

A:TaPLC基因系统发育树;B:TaPLC基因外显子-内含子结构示意图;C:TaPLC蛋白基序示意图,基序的序列和logo图见电子附表3和电子附图2
利用MEME,在TaPLC基因家族中找到了14个不同的motif(图3-C)。亲缘关系较近的成员具有相似的motif组合。TaPLC基因共同具有10个motif,特有motif 13,特有motif 12,特有motif 13。14个motif的图示和序列在电子附表3和电子附图2中列出。
2.4 TaPLC基因启动子的顺式作用元件
为了探究能够影响TaPLC基因表达因素及TaPLC基因可能参与的调控途径,利用在线软件(PlantCARE和PLACE)分析了翻译起始位点(ATG)上游的1.5 kb区域的调控序列。结果显示,TaPLC基因都含有多种顺式作用元件,除了2个核心顺式元件T和C(分别是TATA box和CAAT box),还发现了6个植物激素响应元件,包括ABA响应元件、乙烯响应元件、2个GA响应元件及2个生长素响应元件;同时发现8个应激响应元件,即干旱响应元件、低温响应元件,热激响应元件和光响应元件等(图4和电子附表4),暗示TaPLC基因可能参与了植物对激素和逆境胁迫的响应过程。此外,一些部分同源基因之间,如、和之间,有着类似的顺式调控元件;而不同组的基因之间却有较大的差异,如组的元件数量,较组的多(图4),暗示小麦中不同的TaPLC基因,有些因受共同的因素调控而呈现相近表达模式的基因,也有的调控和表达方式彼此差异较大,从而能够在多种的生理过程中发挥作用。
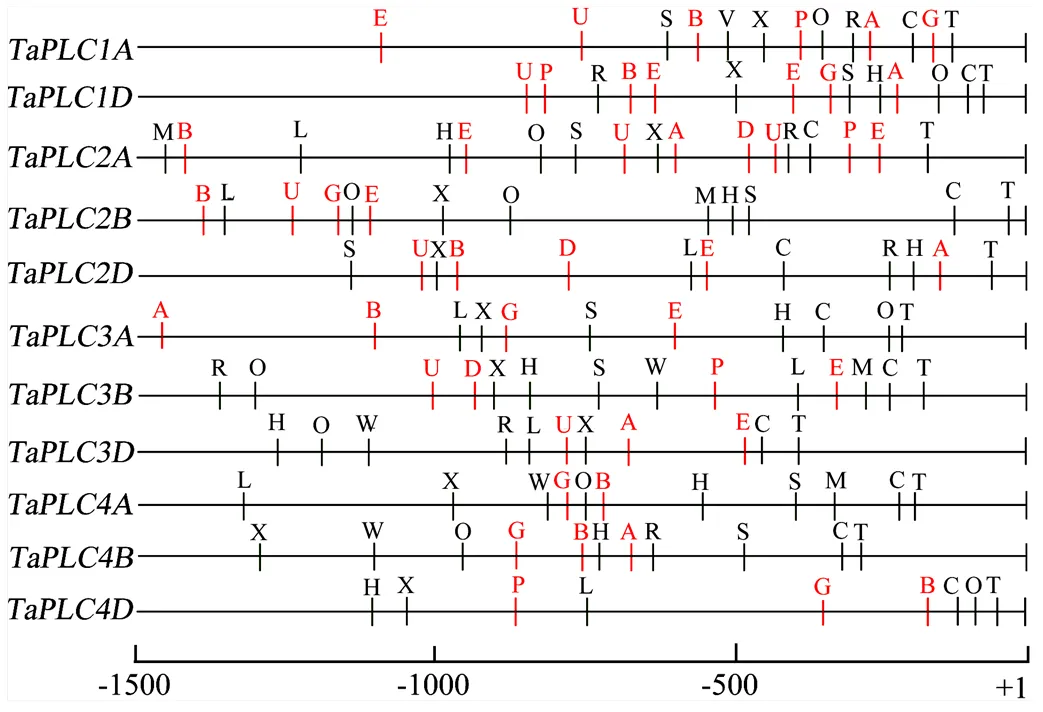
A:ABA响应元件;E:乙烯反应元件;G:赤霉素反应元件;P:赤霉素反应元件;B:参与茉莉酸反应的元件;D和U:生长素反应元件;W:伤害反应元件;L:低温响应元件;S:干旱反应相关的MYB结合位点;M:光反应相关的MYB结合位点;H:热激反应元件;R:光反应的顺式调控元件;O:光反应元件的一部分;X:应答病菌反应的WRKY蛋白结合位点。激素反应顺式元件为红色字母;胁迫反应顺式元件为黑色字母
2.5 小麦TaPLC基因在细胞、组织中的表达模式
利用WoLF POSRT分析结果显示,不同的TaPLC基因具有不同的亚细胞分布特征,其中组和组分别主要在线粒体中分布,组主要在叶绿体中分布,而组主要在细胞质中分布(表2)。
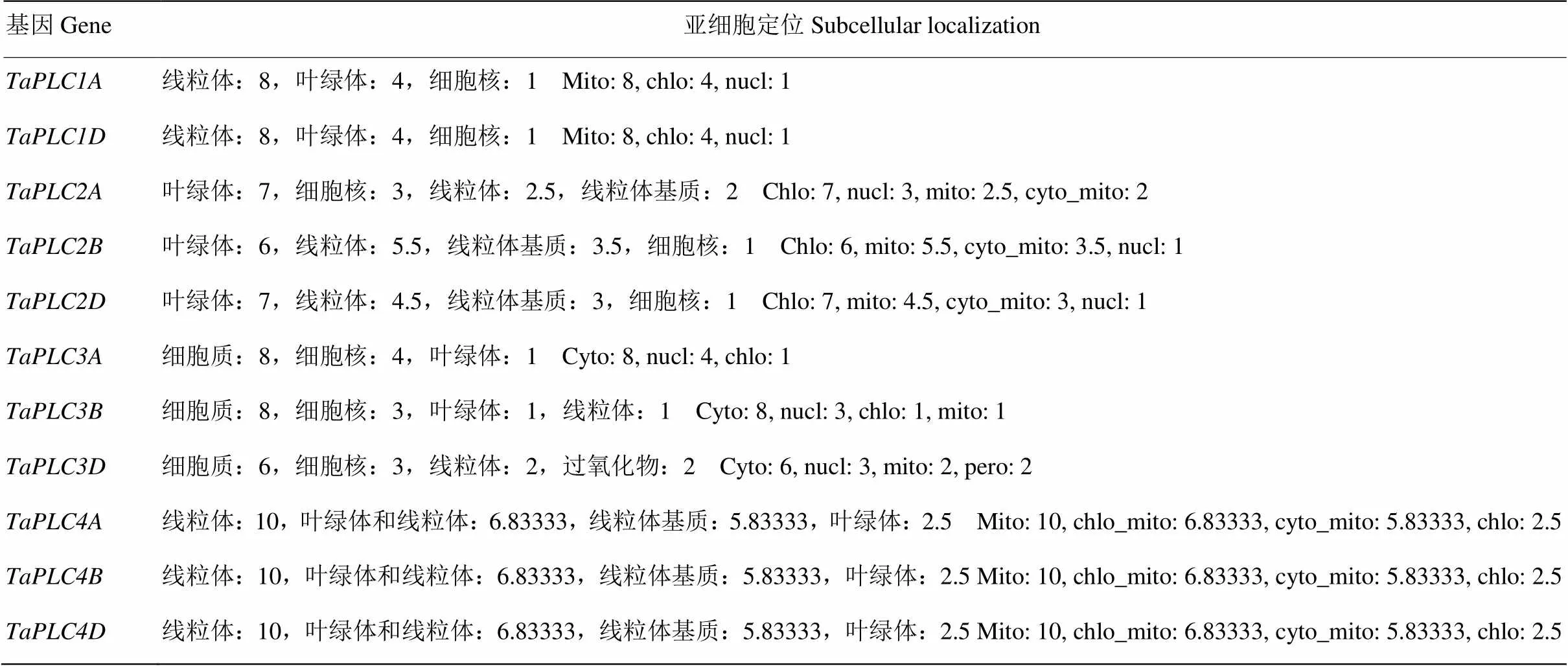
表2 TaPLC基因表达蛋白的亚细胞分布特征
为了更好地揭示TaPLC基因在异源六倍体小麦中的潜在功能,在2种分析网站上检测了该家族在小麦各个组织中的表达丰度。分析结果彼此相似,数据显示,()、(//)和(//)均在根中高表达,而、和则分别在穗、籽粒和叶中有较高丰度(图5-A和电子附图3)。
为了进一步验证转录组的结果,根据获得的、、和cDNA序列设计引物(电子附表2),用实时荧光定量PCR方法分析这些基因在不同组织的表达情况。结果显示,4个基因在5种组织(根、茎、穗、成熟叶和幼叶)中均有表达,但表达水平彼此不同。在所检测的组织中均为表达最高的,表达最低;、和均在根中表达最高,则是在穗中表达最高。qRT-PCR结果与转录组数据具有一定的相似性,如在穗中表达最高,其他基因均在根中表达最高(图5)。以上结果表明TaPLC基因在不同组织的表达模式具有相似性,但也存在差异,暗示该基因家族在小麦的生长发育过程中发挥着作用,同时也暗示不同成员之间可能发生了一定程度的功能分化。
2.6 非生物胁迫对TaPLC基因表达的影响
基于膜磷脂代谢的肌醇磷脂信号系统,在植物应答渗透胁迫的反应中发挥着重要的作用。检测了TaPLC基因在盐或干旱胁迫下的表达变化。结果显示,所检测TaPLC基因的转录均显著受到盐或干旱的诱导(图6)。干旱刺激0.5 h后,4个TaPLC基因的表达量均提高,上调最显著的是和;其中和在干旱6 h时表达量升至最高,而和,则在干旱12 h时达到高峰(图6-A),暗示TaPLC基因在参与植物对干旱胁迫响应的过程中受到了不同的调控策略。同样,该家族成员也受到盐胁迫的诱导,但对盐胁迫的敏感性也存在着一定的差异,如、和均在胁迫处理6 h时表达量最高,而则在受刺激后2 h即达到高峰(图6-B)。
为了解TaPLC基因受盐或干旱诱导表达是否具有普适性,选择抗旱小麦品种洛旱7#(LD)[29](以科农199(KD)为对照)和耐盐品种小偃60#(XS)[30](以石麦15#(SS)为对照)检测了TaPLC基因在胁迫下的表达特征。结果显示,无论是耐旱/盐的品种,还是相对不耐旱/盐的对照品种,TaPLC基因的表达均显著受到干旱/盐胁迫的诱导;另外,干旱胁迫下,抗旱的品种LD中,表达较对照KD更高;耐盐的品种中,等在盐的胁迫下也比对照有更高的表达量(图7)。

A:RNA-seq数据分析;B:qRT-PCR检测结果。小写字母表示各基因在不同组织的显著性水平(P = 0.05)
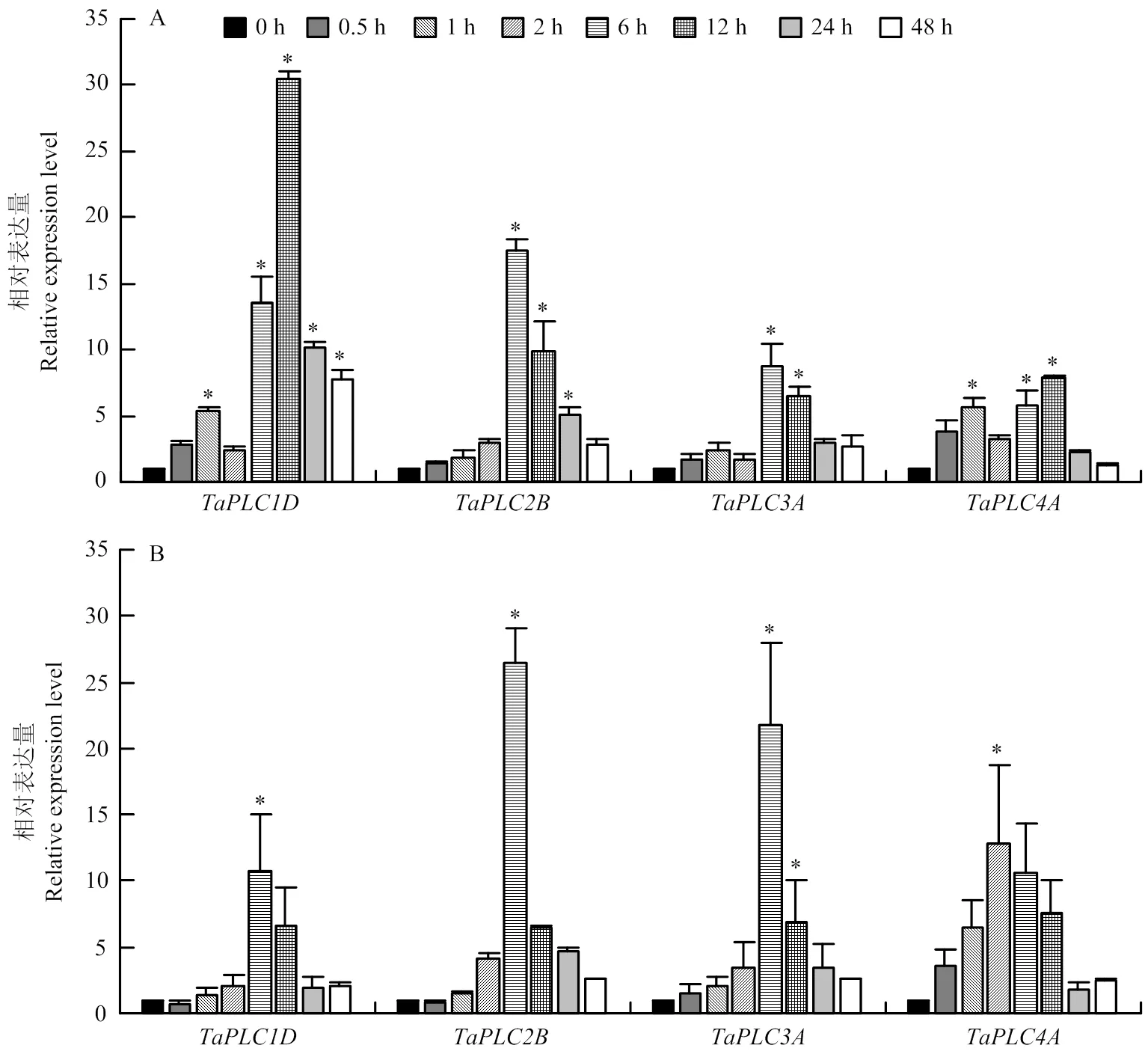
A:TaPLC基因干旱条件下表达分析;B:TaPLC基因盐条件下表达分析。星号表示与对照组比较差异显著(P= 0.05)
综上所述,TaPLC基因受到干旱和盐胁迫的诱导,且各基因对不同胁迫处理的敏感性存在差异,揭示家族成员在响应非生物胁迫过程存在着一定的功能分化。

A:耐旱品种中TaPLC基因在干旱条件下表达分析;B:耐盐品种中TaPLC基因在盐条件下表达分析。KD:科农199;LD:洛旱7#; SS:石麦15#;XS:小偃60#
3 讨论
3.1 小麦中的TaPLC基因家族
通过对小麦基因数据库的检索,在中国春小麦基因组中发现11个PI-PLC编码基因。生物信息学分析结果表明,该家族成员的基因结构、编码蛋白序列的结构以及生化特征等参数与目前已知的植物PLC基因相似[31],显示了TaPLC基因序列在进化上的保守性。
目前发现植物中PLC基因都是以多成员的家族形式存在,如拟南芥中9个成员、水稻中4个、番茄中6个等[31-33]。由于小麦异源六倍体的遗传特征,11个TaPLC基因根据其序列的同源性和结构特征,可以分成4组直系同源基因,即—,每组内包括3个分别位于A、B和D亚基因组中的部分同源基因,其序列相似性达到98%—99%(电子附表1)。另外,在B染色体上却没有拷贝,进一步检索AA二倍体,DD二倍体和AABB四倍体小麦祖先种基因组,发现B基因组中的在异源六倍形成之前就已经缺失(电子附图1)。组内的3个基因在4A、4B和4D染色体上的位置不尽相同。这些结果说明在小麦的进化过程中染色体可能丢失部分基因或发生了基因易位(表1)。通过系统进化分析发现,不同物种的PLC基因均存在于不同的分支中,即使在植物中,单子叶和双子叶的PLC也明显地被区分在不同的模块中,显示出PLC在不同物种中存在着进化上的分歧。然而,小麦和,与单子叶植物水稻和玉米序列的相似度却比双子叶植物的PLC基因序列相似性更高些,表明小麦的和,在进化上与其他的旁系同源基因有较大的差异(图2)。另一方面,根据外显子-内含子结构进行的聚类分析,组的2个基因,均有较长的第一个内含子,与其他3组TaPLC基因的相似度最低,反而与拟南芥和,以及水稻的结构相似[31,33]。外显子-内含子结构是基因演变的重要参数。因此这一组的基因同源关系较其他组较远,这与图3显示的进化树的结果也是一致的。
3.2 TaPLC基因的表达模式
植物PLC的研究最早始于小麦,早在1987、1992年人们分别从小麦中检测到了PLC的生物活性[25-27],河北省植物生理与分子病理学重点实验室前期根据文献报道的2个小麦和(GenBank:HM754654.1和HM754653.1)[34]序列进行了药物学和表达分析检测,发现高盐或干旱至少诱导了的转录和翻译水平的表达,并且施加PLC抑制剂U73122或edelfosine会损害幼苗生长并增强幼苗对干旱和高盐分胁迫的敏感性,表明参与了小麦对盐或干旱胁迫的应答反应[28]。
干旱和高盐导致的渗透胁迫是影响植物的生长发育,造成世界范围内的作物减产的重要因素。固着生长的植物进化出复杂的信号传导系统,以感受环境变化,调控基因表达,从而在细胞和分子水平上对环境胁迫做出响应[1,35]。磷酸肌醇信号传导途径在植物对渗透胁迫的响应中具有重要作用[4]。作为该途径的关键酶之一,植物PLC不仅在渗透胁迫下被诱导表达,而且它们的过表达植物在胁迫环境下还表现多种抵抗或耐受胁迫的表型。拟南芥有9个AtPLC家族成员,第一个被克隆的就是受干旱或盐强烈诱导的亚型[36];qRT-PCR检测结果显示,盐胁迫下、和3种亚型的转录水平提高最多,达5倍以上[37-38];遗传学试验也证实,负调节拟南芥的耐盐性,和的超表达均提高了拟南芥的耐旱性能[18,21-23]。玉米主要在根中表达,干旱胁迫可以诱导其表达上升[36],超表达转基因玉米,比野生型玉米表现出更好的耐旱性能[11]。NaCl处理后,水稻叶片中的PLC的底物分子PIP2的含量会在30 min内迅速增加至4倍[40];水稻的4种OsPLC基因中,在盐胁迫下表达上调,的表达则降低[22];介导Ca2+信号产生,建立了整个植株的耐盐性[8];两个敲除突变体和株系的幼苗在盐胁迫下的生长和成活率都降低,过表达可以提高高盐和缺水条件下的成活率[41]。另外,其他物种如烟草NtPLC基因、马铃薯StPLC基因和绿豆VrPLC基因等被克隆时,也检测到其表达受到盐、干旱等刺激诱导[8,10,13-14]。甘蓝型油菜的过量表达,增强了植物的抗旱性并促进早花和成熟[42]。以上表明,表达受到胁迫影响的PLC基因,在植物体内超表达或缺失后,都会改变植物抵抗或耐受盐或干旱刺激的生理性状。
本研究检索小麦基因组序列,获得了11个TaPLC基因序列后,检测了它们在高盐或干旱胁迫下的表达变化,发现所检测的4个TaPLC基因在转录水平都显著受到胁迫刺激的诱导,其中在干旱胁迫下表达上升最高,而受盐胁迫诱导最显著(图6)。通过序列比对,核实了即前述报道的(GenBank:HM754654.1),即(GenBank:HM754653.1)。另外有趣的是,这些TaPLC基因在耐旱/盐的小麦品种洛旱7#(LD)/小偃60#(XS)[29-30]以及不耐的对照品种中,表达均显著受到干旱/盐胁迫的诱导(图7)。综上,植物PLC以表达量变化的方式普遍参与的盐或干旱的应答响应。植物面对高盐或干旱甚至冷、热等渗透胁迫的物理性因子刺激时,一是可以在细胞的各个位置通过各种大分子和结构独立地感知,此外植物在应答这些胁迫信号时具有一些共同的特征,其中最主要的就是Ca2+作为主要应答的通用第二信使[1,4]。而植物PLC,作为调节膜脂分子代谢的酶类,不仅依附于细胞质膜存在,还在线粒体、叶绿体等细胞器中分布(表2),并且是钙信号形成的上游调控者[3-4]。结合TaPLC基因在不同品系的小麦中受盐或干旱诱导后的表达模式的相似性,推测PLC基因作为膜肌醇磷脂信号转导途径的关键因子,其部分功能可能是参与植物对胁迫信号的感知。另外,在TaPLC基因的调控序列区,除了共有的TATA box和CAAT box外,还有ABA、乙烯、赤霉素、生长素等激素反应的顺式作用元件,光反应相关元件以及低温、热激和干旱等渗透反应的响应元件(图4)。非生物胁迫触发的信号途径与激素对植物的调节过程有很多的cross talk[1,43]。以上诸多信息对于深入研究TaPLC基因家族的每个成员参与小麦应答盐或干旱生理过程的作用机制提供了十分有用的信息,为利用这些基因对小麦进行遗传改良奠定好的基础。
4 结论
TaPLC基因家族中有11个植物PLC直系同源序列,分为4组,每组内的3个(或2个)部分同源物为小麦六倍体演化过程中染色体复制形成的拷贝。不同的TaPLC基因有各自的组织和亚细胞水平的分布特征,并且每组的TaPLC基因成员至少有1个基因的转录受到盐或干旱的胁迫诱导。TaPLC基因的上游调控区存在激素调控的顺式元件及应激响应元件。
[1] Gong Z, Xiong L, Shi H, Yang S, Herrera-Estrella L R, Xu G, Chao D Y, Li J, Wang P Y, Qin F, LI J, DING Y, SHI Y, WANG Y, YANG Y, GAO Y, ZHU J K. Plant abiotic stress response and nutrient use efficiency., 2020, 63(5): 635-674.
[2] 江成, 周厚君, 赵岩秋, 何辉, 楚立威, 宋学勤, 卢孟柱. 干旱和高盐胁迫下钙离子依赖核酸酶基因CDD对银腺杨84K生长发育的影响. 林业科学, 2019, 55(2): 33-40.
JiAng c, zhou h j, zhao y q, he h, chu l w, song x q, lu m z. Effects of CDD gene on the growth and development of Populus alba× P. glandulosa ‘84K’ in response to drought and salt stresses., 2019, 55(2): 33-40. (in Chinese)
[3] Hou Q, Ufer G, Bartels D. Lipid signalling in plant responses to abiotic stress., 2016, 39(5): 1029-1048.
[4] Munnik T, Vermeer J E. Osmotic stress-induced phosphoinositideand inositol phosphate signalling in plants., 2010, 33(4): 655-669.
[5] TasmaIM, Brendel V, Whitham S A, Bhattacharyya M K. Expression and evolution of the phosphoinositide-specific phospholipase C gene family in., 46(7): 627-637.
[6] Shi J, Gonzales R A, Bhattacharyya M K. Characterization of a plasma membrane-associated phosphoinositide-specific phospholipase C from soybean., 1995, 8(3): 381-390.
[7] Pical C, Kopka J, Muller R B, Hetherington A M, Gray J E.Isolation of 2 cDNA clones for phosphoinositide-specific phospholipase C from epidermal peels (accession no. X95877) and guard cells (accession no. Y11931) of(PGR 97-086)., 1997, 114: 747-749.
[8] Tripathy M K, Tyagi W, Goswami M, Kaul T, Singla-Pareek S L, Deswal R, Reddy M K, Sopory S K. Characterization and functional validation of tobacco PLC delta for abiotic stress tolerance., 2012, 30(2): 488-497.
[9] Li L, Wang F, Yan P, Jing W, Zhang C, Kudla J, Zhang W. A phosphoinositide-specific phospholipase C pathway elicits stress-induced Ca2+signals and confers salt tolerance to rice., 2017, 214(3): 1172-1187.
[10] Kopka J, Pical C, Gray J E, Muller-Rober B. Molecular and enzymatic characterization of three phosphoinositide-specific phospholipase C isoforms from potato., 1998, 116: 239-250.
[11] Wang C R, Yang A F, Yue G D, Gao Q, Yin H Y, Zhang J R. Enhanced expression of phospholipase C 1 (ZmPLC1) improves drought tolerance in transgenic maize., 2008, 227(5): 1127-1140.
[12] Pan Y Y, Wang X, Ma L G, Sun D Y. Characterization of phosphatidylinositol-specific phospholipase C (PI-PLC) frompollen., 2005, 46(10): 1657-1665.
[13] Kim Y J, Kim J E, Lee J H, Lee M H, Jung H W, Bahk Y Y, Hwang B K, Hwang I, Kim W T. The Vr-PLC3 gene encodes a putative plasma membrane-localized phosphoinositide-specific phospholipase C whose expression is induced by abiotic stress in mung bean (L.)., 2004, 556(1): 127-136.
[14] Gnanaraj M, Udhayakumar N, Rajiv GR, Manoharan K. Isolation and gene expression analysis of Phospholipase C in response to abiotic stresses from(L.) Wilczek., 2015, 53(6): 335-341.
[15] Cao Z, Zhang J, Li Y, Xu X, Liu G, Bhattacharrya M K, Yang H, Ren D. Preparation of polyclonal antibody specific for AtPLC4, anphosphatidylinositol-specific phospholipase C in rabbits., 2007, 52(2): 306-312.
[16] Xia K, Wang B, Zhang J, Li Y, Yang H, Ren D.phosphoinositide-specific phospholipase C 4 negatively regulates seedling salt tolerance., 2017, 40(8): 1317-1331.
[17] Kanehara K, Yu C Y, Cho Y, Cheong W F, Torta F, Shui G, Wenk M R, Nakamura Y.AtPLC2 is a primary phosphoinositide-specific phospholipase c in phosphoinositide metabolism and the endoplasmic reticulum stress response., 2015, 11(9): e1005511.
[18] Li L, He Y, Wang Y, Zhao S, Chen X, Ye T, Wu Y, Wu Y.PLC2 is involved in auxin-modulated reproductive development., 2015, 84(3): 504-215.
[19] Zhang Q, van Wijk R, Zarza X, Shahbaz M, van Hooren M, Guardia A, Scuffi D, García-Mata C, Van denEnde W, Hoffmann-Benning S, Haring M A, Laxalt A M, Munnik T. Knock-down ofPLC5 reduces primary root growth and secondary root formation while overexpression improves drought tolerance and causes stunted root hair growth., 2018, 59(10): 2004-2019.
[20] Zhang Q, van Wijk R, Shahbaz M, Roels W, Schooten B V, Vermeer J E M, Zarza X, Guardia A, Scuffi D, García-Mata C, Laha D, Williams P, Willems L A J, Ligterink W, Hoffmann-Benning S, Gillaspy G, Schaaf G, Haring M A, Laxalt A M, Munnik T.phospholipase C3 is involved in lateral root initiation and ABA responses in seed germination and stomatal closure., 2018, 59(3): 469-486.
[21] van Wijk R, Zhang Q, Zarza X, Lamers M, Marquez F R, Guardia A, Scuffi D, García-Mata C, Ligterink W, Haring M A, Laxalt A M, Munnik T. Role forPLC7 in stomatal movement, seed mucilage attachment, and leaf serration., 2018, 9: 1721.
[22] Singh A, Kanwar P, Pandey A, TyagiAK, Sopory S K, Kapoor S, Pandey G K. Comprehensive genomic analysis and expression profiling of phospholipase C gene family during abiotic stresses and development in rice., 2013, 8(4): e62494.
[23] Singh A, Bhatnagar N, Pandey A, Pandey G K. Plant phospholipase C family: Regulation and functional role in lipid signaling., 2015, 58(2): 139-146.
[24] Pokotylo I, Kolesnikov Y, Kravets V, Zachowski A, Ruelland E. Plant phosphoinositide-dependent phospholipases C: variations around a canonical theme., 2014, 96: 144-157.
[25] Melin P M, Sommarin M, Sandelius A S, Jergil B. Identification of Ca2+-stimulated polyphosphoinositide phospholipase C in isolated plant plasma membranes., 1987, 223(1): 87-91.
[26] Melin P M, Pical C, Jergil B, Sommarin M. Polyphosphoinositide phospholipase C in wheat root plasma membranes, partial purification and characterization., 1992, 1123(2): 163-169.
[27] Pical C, Sandelius A S, Melin P M, Sommarin M. Polyphosphoinositide phospholipase C in plasma membranes of wheat (L.): Orientation of active site and activation by Ca and Mg., 1992, 100(3): 1296-1303.
[28] Zhang K, Jin C, Wu L, Hou M, Dou S, Pan Y. Expression analysis of a stress-related phosphoinositide-specific phospholipase C gene in wheat (TL.)., 2014, 9(8): e105061.
[29] 吴少辉, 张学品, 杨洪强, 冯伟森. GS旱地小麦新品种-洛旱7号. 中国农业科技导报, 2009, 11(S2): 118-120.
Wu s h, zhang x p, yang h q, feng w s. GS dryland wheat new variety-luohan 7., 2009, 11(S2): 118-120. (in Chinese)
[30] 张明明, 董宝娣, 乔匀周, 赵欢, 刘孟雨, 陈骎骎, 杨红, 郑鑫. 播期、播量对旱作小麦‘小偃60’生长发育、产量及水分利用的影响. 中国生态农业学报, 2016, 24(8):1095-1102.
Zhang m m, dong b d, qiao y z, zhao h, liu m y, chen q q, yang h, zhen x. Effects of sowing date and seeding density on growth, yield and water use efficiency of ‘Xiaoyan 60’ wheat under rainfed condition., 2016, 24(8): 1095-1102. (in Chinese)
[31] Tasma I M, Brendel V, Whitham S A, Bhattacharyya M K. Expression and evolution of the phosphoinositide-specific phospholipase C gene family in., 2008, 46(7): 627-637.
[32] Singh A, Kanwar P, Pandey A, Tyagi A K, Sopory S K, Kapoor S, Pandey G K. Comprehensive genomic analysis and expression profiling of phospholipase C gene family during abiotic stresses and development in rice., 2013, 8(4): e62494.
[33] Vossen J H, Abd-El-Haliem A, Fradin E F,van den Berg G C M, Ekengren S K, Meijer H J G, Seifi A, Bai Y, ten Have A, Munnik T, Thomma B P H J,Joosten M H A J. Identification of tomato phosphatidylinositol-specific phospholipase- C (PI-PLC) family members and the role of PLC4 and PLC6 in HR and disease resistance., 2010, 62(2): 224-239.
[34] Khalil H B, Wang Z, Wright J A, Ralevski A, Donayo A O, Gulick P J. Heterotrimeric Gα subunit from wheat (), GA3, interacts with the calcium-binding protein, Clo3, and the phosphoinositide-specific phospholipase C, PI-PLC1., 2011, 77(1/2): 145-158.
[35] Morran S, Eini O, Pyvovarenko T, Parent B, Singh R, Ismagul A, Eliby S, Shirley N, Langridge P, Lopato S. Improvement of stress tolerance of wheat and barley by modulation of expression of DREB/CBF factors., 2011, 9(2): 230-249.
[36] Hirayama T, Ohto C, Mizoguchi T, Shinozaki K. A gene encoding a phosphatidylinositol-specific phospholipase C is induced by dehydration and salt stress in., 1995, 92(9): 3903-3907.
[37] Lin W H, YE R, Ma H, Xu Z H, Xue H W. DNA chip-based expression profile analysis indicates involvement of the phosphatidylinositol signaling pathway in multiple plant responses to hormone and abiotic treatments., 2004, 14 (1): 34-45.
[38] Tasma I M, Brendel V, Whitham S A, Bhattacharyya M K. Expression and evolution of the phosphoinsitide-specific phospholipase C gene family in., 2008, 46 (7): 627-637.
[39] Zhai S M, Sui Z H, Yang A F, Zhang J R. Characterization of a novel phosphoinositide-speciWc phospholipase C fromand its expression in., 2005, 27 (11): 799-804.
[40] Darwish E, Testerink C, Khalil M, El-Shihy O, Munnik T. Phospholipid signaling responses in salt-stressed rice leaves., 2009, 50(5): 986-997.
[41] 邓先俊. 水稻磷脂酰肌醇特异性磷脂酶C4(OsPLC4)调节水稻渗透胁迫响应及生长发育[D]. 武汉: 华中农业大学, 2018.
DENG X J. Phosphatidylinositol-specific phosphatidylase C 4 (OsPLC4) regulates osmotic stress response and growth and development in rice [D]. Wuhan: Huazhong Agricultural University, 2018. (in Chinese)
[42] Georges F, DAS S, Ray H, Bock C, Nokhrina K, Kolla A, Keller W. Over-expression ofphosphatidylinositol- phospholipase C2 in canola induces significant changes in gene expression and phytohormone distribution patterns, enhances drought tolerance and promotes early flowering and maturation., 2009, 32(12): 1664-1681.
[43] 杜康兮, 沈文辉, 董爱武. 表观遗传调控植物响应非生物胁迫的研究进展. 植物学报, 2018, 53(5): 581-593.
Du k x, shen w h, dong a w. Advances in epigenetic regulation of abiotic stress response in plants., 2018, 53(5): 581-593. (in Chinese)
Genomic Profiling and Expression Analysis of Phosphatidylinositol-specific PLC Gene Families among Chinese spring Wheat
SI XuYang1, JIA XiaoWei1, ZHANG HongYan1, JIA YangYang1, TIAN ShiJun1, ZHANG Ke2, PAN YanYun1
(1College of Life Sciences,Hebei Agricultural University/Key Laboratory of Hebei Province for Plant Physiology and Molecular Pathology, Baoding 071000, Hebei;2College of Agronomy, Hebei Agricultural University/State Key Laboratory of North China Crop Improvement and Regulation/Key Laboratory of Crop Growth Regulation of Hebei Province, Baoding 071001, Hebei)
【】To characterize the gene family encoding phosphatidylinositol-specific phospholipase-C (PI-PLC), the gene number, structure, phylogenetic relationship and expression pattern of TaPLC genes were analyzed. In addition, we explored their relative transcription levels in various tissue and the expression pattern under drought and salt stress to further analysis the physiological role ofTaPLC genes in response to abiotic stress.【】Based on the genome database (Ensembl Plants, http://plants.ensembl.org/index.html), TaPLC genes were identified from, and the gene structures were analyzed by some bioinformatics tools, such as Pfam, CDD and SMART. The protein sequence characteristics of the members in TaPLC family were analyzed with the online server ExPASy (http://cn.expasy.org/tools). The subcellular localization of TaPLC genes were predicted using the WoLF PSORT (https://www.genscript.com/wolf-psort.html). Phylogenic tree analysis was performed by software MEGA 7.0. The real-time PCR was used to detect the gene expression levels in different tissues under abiotic stress. 【】A total of 11 TaPLC genesmembers within the heterologous hexaploid wheat genome were identified and analyzed. All TaPLC genes have conserved X-Y catalytic domains and C2 domains, just like other plant PLC genes. Subcellular localization showed that these proteins were located in the chloroplast or mitochondria. Phylogenetic analysis revealed that all of the TaPLC genes were unevenly classified into four numbered,,,, and, which were all orthologous genes of plant PLC genes in plants, and highly conserved during evolution. However, the deletion ofB subgenome indicates asymmetry evolution in different subgenomes. Further analysis of cis-regulatory elements elucidated that these members of TaPLCfamily shared similar elements, such as the phytohormone and the stress response cis-elements. Each PLC genehas a unique tissue expression pattern, and they are expressed mainly in roots, leaves, spikes, and gains. In addition, PLC genesmediate the response of wheat to salt and drought stresses, which are induced rapidly by salt or drought stress stimulation. 【】The TaPLCfamily incontains 11 members which have conserved X-Y catalytic domains and C2 domains. Although the TaPLC genes were highly conserved, they were asymmetrically evolved in different subgenomes. They had specific expression pattern in different tissues, and were induced by salt or drought stress, which suggested TaPLCgenes play a role in response to salt or drought in wheat.
; PI-PLC; salt stress; drought stress

10.3864/j.issn.0578-1752.2020.24.001
2020-03-23;
2020-06-02
河北省自然科学基金(C2017204095)、河北省高等学校科学技术研究项目(ZD2017039)
司旭阳,E-mail:sixuyanglove@163.com;贾哓玮,E-mail:jia999666@126.com;司旭阳和贾哓玮为同等贡献作者。通信作者潘延云,E-mail:pyycell@163.com。通信作者张科,E-mail:zhangke0126@163.com
(责任编辑 李莉)

