卵巢癌标志物溶血磷脂酸检测的研究进展
李年生 陈立 肖祚庥 杨禹祺 艾可龙
摘 要 卵巢癌的死亡率高,是严重威胁女性健康的重大疾病。当前,由于缺乏有效、便捷的早期诊断技术,大部分卵巢癌患者被发现时已经处于中晚期而错失最佳治疗时机。因此,对早期卵巢癌的快速、灵敏、低成本和准确检测具有非常重要的意义。在早期卵巢癌患者血液中存在溶血磷脂酸(Lysobisphosphatidic acids, LPA)浓度升高现象,而在其它疾病中却较少出现此情况,因此LPA可作为一种高特异性、有良好临床应用前景的卵巢癌标志物。近年来, 该领域的相关研究取得了较大的进展, 开发了很多LPA的检测技术和方法。本文对LPA的检测方法进行分类、归纳和总结,着重介绍了近年来该领域的重要进展,最后对LPA标记物检测在临床应用方面所面临的挑战进行了分析, 对其发展和前景进行了展望。
关键词 卵巢癌; 溶血磷脂酸; 癌症标志物; 质谱检测; 比色检测; 荧光检测; 评述
1 引 言
卵巢癌是严重威胁女性健康的疾病[1,2]。由于卵巢癌早期缺少症状,筛查方法有限,因此其早期诊断比较困难。75%的卵巢癌患者就诊时已为晚期,而晚期病例療效不佳(晚期卵巢癌患者5年生存率仅25%)[3~6]。因此,虽然卵巢癌的发病率居妇科恶性肿瘤的第三位,但其死亡率高居妇科癌症首位。值得注意的是,早期确诊的卵巢癌患者5年生存率为90%。因此,实现卵巢癌早期诊断,可以大幅度降低卵巢癌的死亡率。目前,临床上卵巢癌诊断的方法主要包括影像学检查(超声成像、计算机断层扫描、正电子发射断层扫描及核磁共振成像等)、组织病理切片等。影像学检查手段能够明确盆腔肿块是否存在,甚至能发现卵巢肿瘤,区分固体肿块和液体囊肿,判断肿块的尺寸,以及观察肿块内部的情况等。尽管如此,影像学检查无法判定卵巢肿块是良性或恶性,且其成本高、价格昂贵、普适性低。组织病理切片检查需要通过手术切除选取标本,制成病理切片,用显微镜检查病变。病理切片是卵巢癌确诊的主要手段,但其缺点是需要手术、创伤性大,且无法发现转移病灶。这些影像学检查、组织病理切片方法通常仅适用于中晚期卵巢癌病人的检查。
通过体液检测卵巢癌相关标志物是更为便捷、经济的方法[1,7,8]。卵巢癌标志物主要有人附睾蛋白4、癌抗原-125(Cancer antigen 125, CA-125)和间皮素等,其中CA-125在临床上已被用于卵巢癌的筛查[9~11]。CA-125是一种糖类蛋白质,中晚期卵巢癌患者的CA-125水平会升高,但是检测CA-125对早期卵巢癌的筛检并不适用,有些其它疾病或健康女性在特定生理周期也会引起血清中的CA-125水平升高。此外,在卵巢癌早期仅有25%左右的患者CA-125的水平升高[12]。因此,通过CA-125筛查早期卵巢癌具有高的假阳性和漏诊率。溶血磷脂酸(Lysobisphosphatidic acids, LPA)在卵巢癌细胞的增殖、侵袭等方面发挥了重要作用。在卵巢癌早期,新血管增生对肿瘤的增殖至关重要。Song等[14]研究发现,LPA能诱导卵巢癌细胞高度表达血管内皮生长因子(Vascular endothelial growth factor, VEGF),而VEGF能在肿瘤部位诱导血管新生。LPA能促使卵巢癌细胞增值转录因子(主要为叉头框蛋白M1, forkhead box M1)表达,而直接诱导卵巢癌增殖[15]。另外,LPA能促进基质金属蛋白酶9(Matrix metalloproteinase 9, MMP9)的表达,MMP9能促进卵巢癌转移和侵袭[16]。这说明LPA在卵巢癌的发生、发展中发挥了重要作用[16~18]。据报道,卵巢癌早期患者血清中的LPA升高率达90%,而中晚期患者可达100%[19]。LPA是一种脂类物质,易从血液、腹水、唾液等中提取,因而便于进行分析和检测。上述结果表明, LPA是具有临床应用前景的卵巢癌早期标记物。
基于LPA检测的重要意义,大量检测手段、生物探针被开发用于检测LPA[12,20~33]。本研究组也构建了聚丁二炔比色探针检测血清中的LPA[21]。当前, LPA的检测主要通过液相色谱-质谱联用、气相色谱与原子吸收光谱或质谱联用等方法实现[13,23,34~50]。这些方法具有灵敏度高、普适性好的优势,但缺点是耗时、操作复杂和成本高。LPA也可以通过荧光法、比色法和电化学发光等便捷、经济的方法进行检测,然而它们的特异性和灵敏度需要进一步提高。近年来,由于纳米技术、光谱技术的发展和应用,LPA检测方法在灵敏度、特异性等方面取得了很大的进展。本文对LPA检测主要方法进行分类阐述, 对相关检测机理和影响因素进行了深入的探讨,重点介绍了该领域最新进展,最后展望了LPA检测在临床应用中面临的挑战和机遇。
2 质谱法检测LPA
质谱技术具有灵敏度高、准确性高、响应时间快、需要的样本量少、通量高等优点,被广泛用于生物样品的分析和检测[51],已经成为生物样品中脂质分析的常规技术。
质谱检测LPA的准确性主要依靠分离技术。采用质谱检测体液(主要包括血液、尿液、唾液等)样本中的LPA通常按照如图1所示的流程[13],分8个步骤进行。首先是样本收集,然后经过脂质抽提和色谱分离后,再进入质谱仪进行检测和分析[52]。体液样本中最普遍采用的是血液样本。血液中LPA的丰度比溶血磷脂酰胆碱低两个数量级,同时还包含与LPA具有类似结构的溶血磷脂酰丝氨酸、磷脂酰胆碱等,这些脂质分子都会对质谱检测LPA造成干扰。近年来,在LPA的抽提和分离方面的研究也取得了很大的进展。对于抽提方法,已从传统的甲醇/氯仿脂质液-液提取法(Bligh & dyer方法)发展到固-液提取法。对于分离方法,也从早期的毛细管电泳法、薄层色谱法等发展到高效液相色谱法等。目前,高效液相色谱法已经成为分离LPA的常规方法。质谱技术中,常规的电子碰撞电离、电喷雾电离和基质辅助激光解吸电离技术都已被用于LPA的检测。 本文将从抽提、分离方法和电离方式三个方面对质谱法检测LPA的最新进展进行归纳和总结。
2.1 LPA的抽提方法
通过质谱法进行LPA检测时,获得高灵敏度和高准确性的最重要的一步是LPA抽提。不同于常见脂质分子,LPA是一种两亲性分子,具有较大的极性。除了常规脂质提取法(Bligh & Dyer方法)外,基于异丙醇、甲基叔丁基醚和丁醇等溶剂的方法也被用于提取LPA[53~56]。Triebl等[38]对比研究了Bligh & Dyer 方法、Bligh & Dyer盐酸法、甲基叔丁基醚提取法和丁醇提取法(图2) ,发现甲基叔丁基醚提取法效果较差,在提取过程中超过69%的LPA丢失;采用Bligh & Dyer 方法提取效果同样较差,主要因为LPA的强极性导致其在甲基叔丁基醚和氯仿中的溶解度不高;因HCl能导致LPA质子化而提高其在非极性溶剂中的溶解度,采用HCl修改的Bligh & Dyer 方法能大幅提高LPA的提取效率(提取率在91%~99%之间)。最近,Scherer等[42]发现,通过Bligh & Dyer HCl方法抽提LPA时,约2%的溶血磷脂酰胆碱在酸性条件下水解生成LPA。考虑到溶血磷脂酰胆碱在血清中的浓度比LPA高两个数量级,采用该方法会导致LPA水平人为升高。因LPA能在正丁醇中很好地溶解,通过正丁醇方法也能获得较好的LPA提取效果。2019年,Aristizabal-Henao等[34]采用正丁醇抽提法不仅获得了很好的LPA萃取效率, 且能大幅减少血样体积,仅用50 μL血清样本,即分离出LPA的多种异构体。
除了液-液抽提法,固相萃取法在近年来也被用于萃取LPA。Wang等[57]使用分子印迹聚合物作为固相萃取的固定相,通过固-液抽提实现对LPA的高效提取。分子印迹是在固体或凝胶中產生印记的过程,其大小、形状和电荷分布与模板分子相对应[58]。通常将模板分子与功能单体通过共价或非共价键结合,然后再高浓度交联聚合。去除模板后形成具有空位和目标物结合位点的分子印迹聚合物,可选择性地结合目标分子。通过这种方法,可快速和简单地提取高纯度的LPA,提取率超过94.6%,比液-液抽提法具有回收率更高、便捷和易于自动化操作的优点。
2.2 LPA的分离
高效液相色谱法广泛应用于生物、化学和环境等样品分析,其核心是高效液相色谱柱[52, 59]。高效液相色谱柱一般分为正相柱和反相柱。正相色谱柱一般选择极性的多孔球形硅胶填充。2016年, Cífková等[36]采用正相色谱柱分离LPA。通过对不同化学性质的色谱柱、流动相的pH值、流动相添加剂的类型和浓度进行深入研究,发现当采用氢化二氧化硅柱、酸化处理(pH 4)时,高效液相色谱能有效分离脂类物质,可获得血清样品中所有脂质类(包括LPA)良好的色谱峰形状。
相对于正相色谱柱,反相色谱柱具有更好的分离能力和稳定性。在反相色谱法中,极性化合物先被洗脱,而非极性化合物因与吸附剂表面的亲和性不同而被依次分离。目前,反相色谱柱已经被广泛用于分离磷脂分子[60~62]。LPA在反相柱中通常以长拖尾状宽峰的形式被洗脱,导致LPA的分离、检测和定量分析的效果较差。Ogiso等[45]通过使用含有低浓度H3PO4(5 μmol/L)和高比例水(40%)的起始流动相,可大幅减少LPA洗脱峰的拖尾,从而高效分离LPA。流动相中低浓度的H3PO4能促使LPA质子化,从而减少极性而减少洗脱峰的拖尾。然而,H3PO4的引入会对随后进行的质谱分析造成干扰。LPA上的羟基基团与三甲基硅烷化重氮甲烷(Trimethylsilyl chloride, TMS)发生专一性强、效率高的反应[35,40,63,64],易被甲基化而大幅减少分子极性,使其在反相色谱柱中易被分离。最近,王雪颖等[23]通过该反应对LPA进行甲基化修饰(图 3A),可大幅改善LPA的色谱行为。如图3B和3C所示,LPA的色谱峰具有很长的拖尾,而甲基化后的LPA色谱峰得到了明显改善,色谱峰的拖尾减少,色谱峰的强度也大幅提高。
2.3 LPA的质谱电离方法
在质谱分析中,样品电离一般在离子化器里进行。最常见的离子化器是电子离子化器。电子离子化是一种“硬”的电离方法,采用电子离子化器能产生大量碎裂片段而能提供详细的质谱信息,可为分子结构表征提供重要信息。电子离子化器主要通过由钨或铼加热灯丝产生的电子束与样品气体分子碰撞时,解离一个电子而产生电离。在正常大气压的空气中,有机物在加热条件下会迅速燃烧,因此电子离子化器通常与气相色谱联用(在真空条件下),但不能与液相色谱或高效液相色谱(一般在正常大气压下)联用。
通过高效液相色谱分离LPA时,一般采用较“软”的离子化器。电喷雾电离和基质辅助激光解吸电离是两种“软”电离方法[13]。这两种电离法通常不会或很少产生分子碎片。基质辅助激光解吸电离是利用基质吸收激光的能量使样品中的分子以最小的碎裂度产生离子[65,66]。例如,Tanaka等[31]采用氮分子激光器发射337 nm的激光作为能量来源以辅助电离样品。然而该方法重现性不佳,为了提高重现性,每个质谱样本需要多次激光激发(平均需采用256次)。电喷雾电离是将含有分析物的液体通过电喷雾形成精细的气溶胶,高温去溶剂后将分析物离子化。电喷雾电离的最大优势是能便捷地与液相色谱分离技术联用。相对于基质辅助激光解吸电离,当前LPA的质谱分析采用电喷雾电离更为普遍[19,67~73]。例如, Cífková等[36]通过高效液相色谱-电喷雾质谱联用实现了对复杂样品猪脑和肾中的LPA的分离和定量检测。
3 光学方法检测LPA
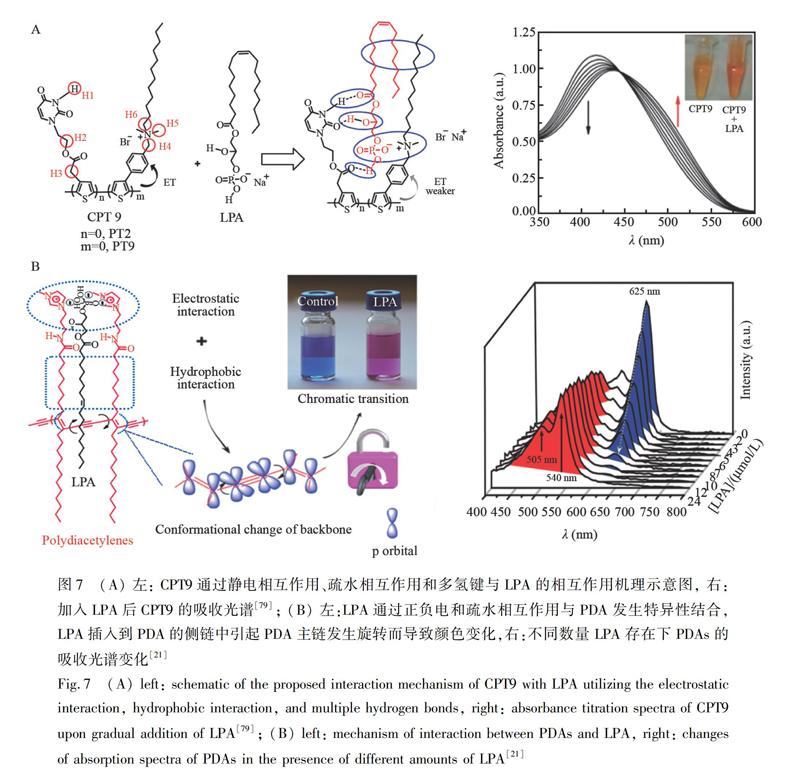
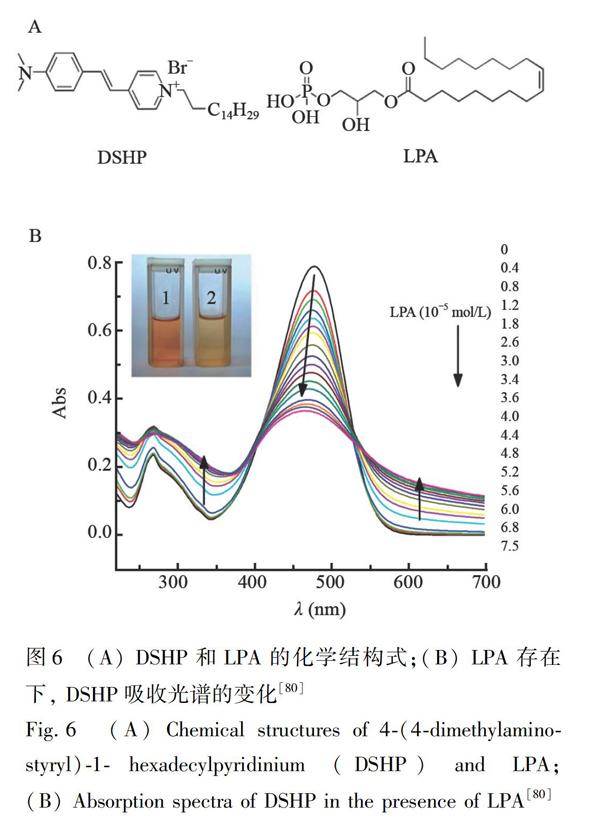
对于实际临床检验来说,发展一种相对简单有效检测LPA的方法非常必要,因此操作简便、低成本的光学方法因此受到广泛的关注。基于光学检测LPA的方法包括有电化学发光法、表面增强拉曼光谱法、荧光法和比色法等[12,19,21,22,26,30,33,56,74~80],其中, 以荧光法和比色法为主。光学方法最大的瓶颈是选择性和灵敏度。由于体液的成分复杂,光学探针应具有较好的抗干扰能力, 以确保对LPA的选择性响应。另外,卵巢癌患者血清的LPA浓度介于1.3~50 μmol/L之间,临界值为1.3 μmol/L[19]。因此,有临床应用价值的光学探针需要具有很高的灵敏度,其检出限应低于1.3 μmol/L。近年来,LPA光学探针在灵敏度、特异性等方面取得了很大的进展,本节对荧光法和比色法这两种光学法进行归纳和总结。
References
1 Shabaninejad Z, Vafadar A, Movahedpour A, Ghasemi Y, Namdar A, Fathizadeh H, Pourhanifeh M H, Savardashtaki A, Mirzaei H. J. Ovarian Res., 2019, 12(1): 84
2 Bray F, Ferlay J, Soerjomataram I, Siegel R L, Torre L A, Jemal A. Ca-Cancer J. Clin., 2018, 68(6): 394-424
3 Jang H I, Lim S H, Lee Y Y, Kim T J, Choi C H, Lee J W, Kim B G, Bae D S. Obstet. Gynecol. Sci., 2017, 60(2): 170-177
4 Ruhaak L R, Xu G, Li Q, Goonatilleke E, Lebrilla C B. Chem. Rev., 2018, 118(17): 7886-7930
5 Bast R C, Hennessy B, Mills G B. Nat. Rev. Cancer, 2009, 9(6): 415-428
6 Azvolinsky A. Nat. Med., 2013, 19(2): 117
7 Galenkamp N S, Soskine M, Hermans J, Wloka C, Maglia G. Nat. Commun., 2018, 9: 4085
8 Xu Y. Cancers, 2019, 11(10): 1523
9 Szymanska B, Lukaszewski Z, Hermanowicz-Szamatowicz K, Gorodkiewicz E. Talanta, 2020, 206: 120187
10 Saadati A, Hassanpour S, Bahavarnia F, Hasanzadeh M. Anal. Methods, 2020, 12(12): 1639-1649
11 Kim S I, Kang N, Leem S, Yang J, Jo H, Lee M, Kim H S, Dhanasekaran D N, Kim Y K, Park T, Song Y S. Cancers, 2020, 12(5): 1309
12 Minis E, Holcomb K, Sisti G, Nasioudis D, Kanninen T T, Athanasiou A, Frey M K, Chapman-Davis E, Caputo T A, Witkin S S. Eur. J. Obstet. Gynecol. Reprod. Biol: X, 2019, 2: 100012
13 Jelonek K, Ros M, Pietrowska M, Widlak P. Clin. Lipidol., 2013, 8(1): 137-150
14 SongY, Wu J, Oyesanya R A, Lee Z, Mukherjee A, Fang X. Clin. Cancer Res., 2009, 15(2): 492-501
15 Wierstra I. Adv. Cancer Res., 2013, 119: 191-419
16 Jesionowska A, Cecerska E, Dolegowska B. Anal. Biochem., 2014, 453: 38-43
17 Kipps E, Tan D S P, Kaye S B. Nat. Rev. Cancer, 2013, 13(4): 273-282
18 Mohammadi Z, Roodbari N H, Parivar K, Salehnia M. Cell J., 2020, 22(3): 358-366
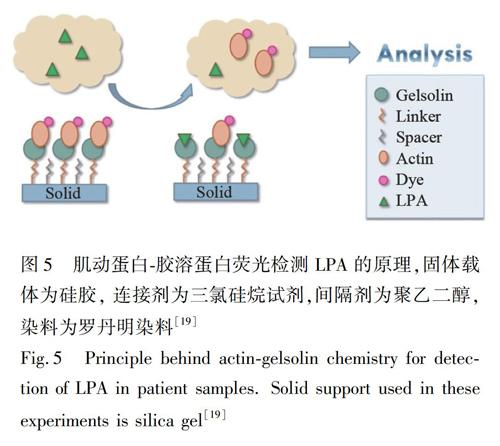
19 De La Franier B, Thompson M. Biosensors-Basel, 2020, 10(2): 13
20 Wu S, Min H, Shi W, Cheng P. Adv. Mater., 2020, 32(3): 1805871
21 Wang Y, Pei H, Jia Y, Liu J, Li Z, Ai K, Lu Z, Lu L. J. Am. Chem. Soc., 2017, 139(33): 11616-11621
22 Chen X, Gui W, Liu H, Ma Q. Analyst, 2017, 142(21): 4142-4149
23 WANG Xue-Ying, ZONG Zhao-Yun, JIAO Yu-Pei, XU Li-Na, WANG Yu-Song, LI Lin, LIU Xiao-Hui. Chinese J. Anal. Chem., 2019, 47(2): 249-255
王雪穎, 宗兆运, 焦玉佩, 许丽娜, 王昱淞, 栗 琳, 刘晓蕙. 分析化学, 2019, 47(2): 249-255
24 Huang Q, Lei H, Dong M, Tang H, Wang Y. Analyst, 2019, 144(13): 3980-3987
25 De La Franier B, Thompson M. Biosens. Bioelectron., 2019, 135: 71-81
26 Zheng Z, Geng W C, Gao J, Wang Y Y, Sun H W, Guo D S. Chem. Sci., 2018, 9(8): 2087-2091
27 Kumar N, Singh A K. J. Clin. Diagnostic Res., 2018, 12(4): QE1-QE7
28 Zhang Y, Liu Y, Li L, Wei J, Xiong S, Zhao Z. Talanta, 2016, 150: 88-96
29 Yin R, Yang T, Su H, Ying L, Liu L, Sun C. Cancer Biomark., 2016, 17(3): 259-269
30 Yao D, Lin Z, Wu J. ACS Appl. Mater. Interfaces, 2016, 8(9): 5847-5856
31 Yamashita R, Tabata Y, Iga E, Nakao M, Sano S, Kogure K, Tokumura A, Tanaka T. Lipids, 2016, 51(2): 263-270
32 Piccoli R, De Rosa N, Davis S. Gynecological Cancers: Genetic and Epigenetic Targets and Drug Development, Gynecological Cancers in press, 2016: 79-95
33 Seballos L, Zhang J Z, Sutphen R. Anal. Bioanal. Chem., 2005, 383(5): 763-767
34 Aristizabal-Henao J J, Fernandes M F, Duncan R E, Stark K D. Lipids, 2019, 54(8): 479-486
35 Lee J C, Byeon S K, Moon M H. Anal. Chem., 2017, 89(9): 4969-4977
36 Cífková E, Hájek R, Lísa M, Hollapek M. J. Chromatogr. A, 2016, 1439: 65-73
37 Wang J, Sibrian-Vazquez M, Escobedo J O, Wang L, Chu Y H, Moore R G, Strongin R M. Analyst, 2015, 140(22): 7572-7577
38 Triebl A, Trotzmüller M, Eberl A, Hanel P, Hartler J, Kfeler H C. J. Chromatogr. A, 2014, 1347: 104-110
39 Onorato J M, Shipkova P, Minnich A, Aubry A F, Easter J, Tymiak A. J. Lipid Res., 2014, 55(8): 1784-1796
40 Clark J, Anderson K E, Juvin V, Smith T S, Karpe F, Wakelam M J O, Stephens L R, Hawkins P T. Nat. Methods, 2011, 8(3): 267-272
41 Zhao Z, Xu Y. J. Chromatogr. B, 2009, 877(29): 3739-3742
42 Scherer M, Schmitz G, Liebisch G. Clin. Chem., 2009, 55(6): 1218-1222
43 Guan W, Zhou M, Hampton C Y, Benigno B B, Walker D, Gray A, McDonald J F, Fernandez F M. BMC Bioinform., 2009, 10: 259
44 Shan L, Jaffe K, Li S, Davis L. J. Chromatogr. B, 2008, 864(1-2): 22-28
45 Ogiso H, Suzuki T, Taguchi R. Anal. Biochem., 2008, 375(1): 124-131
46 Meleh M, Pozlep B, Mlakar A, Meden-Vrtovec H, Zupancˇicˇ-Kralj L. J. Chromatogr. B, 2007, 858(1): 287-291
47 Tanaka T, Tsutsui H, Hirano K, Koike T, Tokumura A, Satouchi K. J. Lipid Res., 2004, 45(11): 2145-2150
48 Sutphen R, Xu Y, Wilbanks G D, Fiorica J, Grendys E C, LaPolla J P, Arango H, Hoffman M S, Martino M, Wakeley K, Griffin D, Blanco R W, Cantor A B, Xiao Y J, Krischer J P. Cancer Epidemiol. Biomarkers Prev., 2004, 13(7): 1185-1191
49 Holland W L, Stauter E C, Stith B J. J. Lipid Res., 2003, 44(4): 854-858
50 Kim H, Yoon H R, Pyo D. Bull. Korean Chem. Soc., 2002, 23(8): 1139-1143
51 Mergner J, Frejno M, List M, Papacek M, Chen X, Chaudhary A, Samaras P, Richter S, Shikata H, Messerer M, Lang D, Altmann S, Cyprys P, Zolg D P, Mathieson T, Bantscheff M, Hazarika R R, Schmidt T, Dawid C, Dunkel A, Hofmann T, Sprunck S, Falter-Braun P, Johannes F, Mayer K F X, Juergens G, Wilhelm M, Baumbach J, Grill E, Schneitz K, Schwechheimer C, Kuster B. Nature, 2020, 579: 409-414
52 Zuvela P, Skoczylas M, Liu J J, Baczek T, Kaliszan R, Wong M W, Buszewski B. Chem. Rev., 2019, 119(6): 3674-3729
53 Kumari M, Schoiswohl G, Chitraju C, Paar M, Cornaciu I, Rangrez A Y, Wongsiriroj N, Nagy H M, Ivanova P T, Scott S A, Knittelfelder O, Rechberger G N, Birner-Gruenberger R, Eder S, Brown H A, Haemmerle G, Oberer M, Lass A, Kershaw E E, Zimmermann R, Zechner R. Cell Metab., 2012, 15(5): 691-702
54 Cao W, Cheng S, Yang J, Feng J, Zhang W, Li Z, Chen Q, Xia Y, Ouyang Z, Ma X. Nat. Commun., 2020, 11(1): 375
55 Holcˇapek M, Liebisch G, Ekroos K. Anal. Chem., 2018, 90(7): 4249-4257
56 Matyash V, Liebisch G, Kurzchalia T V, Shevchenko A, Schwudke D. J. Lipid Res., 2008, 49(5): 1137-1146
57 Wang J, Sibrian-Vazquez M, Escobedo J O, Lowry M, Wang L, Chu Y H, Moore R G, Strongin R M. Analyst, 2013, 138(22): 6852-6859
58 Yoshikawa M, Tharpa K, Dima S O. Chem. Rev., 2016, 116(19): 11500-11528
59 ZHAO Ying, WANG Nan, GAO Hua-Long, GUO Zi-Xuan, LU An-Xiang, GUO Xiao-Jun, LU Jing-Hua, LUAN Yun-Xia. Chinese J. Anal. Chem., 2020, 48(5): 662-669
趙 颖, 王 楠, 高华龙, 郭子轩, 陆安祥, 郭晓军, 卢静华, 栾云霞. 分析化学, 2020, 48(5): 662-669
60 Siriwardane D A, Wang C, Jiang W, Mudalige T. Int. J. Pharm., 2020, 578: 119077
61 Antonelli M, Benedetti B, Cavaliere C, Cerrato A, Montone C M, Piovesana S, Lagana A, Capriotti A L. Food Chem., 2020, 310: 125860
62 Zhang Q, Nong Y, Liu Z, Gong L. Anal. Chem., 2019, 91(22): 14458-14466
63 Bozelli J C, Epand R M. Proteomics, 2019, 19(18): e1900138
64 Lee H C, Yokomizo T. Biochem. Biophys. Res. Commun., 2018, 504(3): 576-581
65 McDonald W S, Jones E E, Wojciak J M, Drake R R, Sabbadini R A, Harris N G. Am. J. Clin. Pathol., 2018, 188(8): 1779-1793
66 Kaya I, Zetterberg H, Blennow K, Hanrieder J. ACS Chem. Neurosci., 2018, 9(7): 1802-1817
67 Eum J Y, Lee J C, Yi S S, Kim I Y, Seong J K, Moon M H. J. Chromatogr. A, 2020, 1618: 460849
68 Xu Y. Cancers, 2018, 10(7): 227
69 Shrestha P, Hussain D, Mulder R J, Taylor M C, Singh S P, Petrie J R, Zhou X R. Front. Plant Sci., 2018, 9: 1234
70 Inoue M, Okamoto Y, Atsumi Y, Shiojiri M, Hidaka M, Tanaka T, Tsutsumi T, Shirasaka N, Tokumura A. Biofactors, 2018, 44(6): 548-557
71 Hou T, Taylor M C, Shrestha P, Singh S, Zhang Z J, Zhou X R. J. Chromatogr. A, 2018, 1572: 100-105
72 Fukasawa K, Nakajima S, Gotoh M, Tanaka S, Murofushi H, Murakami-Murofushi K. J. Chromatogr. A, 2018, 1567: 177-184
73 Aldi S, Matic L P, Hamm G, van Keulen D, Tempel D, Holmstrom K, Szwajda A, Nielsen B S, Emilsson V, Ait-Belkacem R, Lengquist M, Paulsson-Berne G, Eriksson P, Lindeman J H N, Gool A J, Stauber J, Hedin U, Hurt-Camejo E. Mol. Ther. Methods Clin. Dev., 2018, 10: 17-28
74 Zhang S Y, Shi W, Cheng P, Zaworotko M J. J. Am. Chem. Soc., 2015, 137(38): 12203-12206
75 Wang L, Sibrian-Vazquez M, Escobedo J O, Wang J, Moore R G, Strongin R M. Chem. Commun., 2015, 51(9):1697-1700
76 Jiang T, Lu N, Yang J, Hang Y, Wang J, Zhao P, Hua J. RSC Adv., 2015, 5(124): 102863-102867
77 Correnti C, Clifton M C, Abergel R J, Allred B, Hoette T M, Ruiz M, Cancedda R, Raymond K N, Descalzi F, Strong R K. Structure, 2011, 19(12): 1796-1806
78 HAO Yu-Jia, ZHU Guang-Ming. Chinese J. Anal. Chem., 2020, 48(2): 164-173
郝瑜佳, 朱光明. 分析化學, 2020, 48(2): 164-173
79 Lan M, Liu W, Wang Y, Ge J, Wu J, Zhang H, Chen J, Zhang W, Wang P. ACS Appl. Mater. Interfaces, 2013, 5(6): 2283-2288
80 Zhao W, Liu W, Zhang W, Zeng L, Fan Z, Wu J, Wang P. Analyst, 2012, 137(8): 1853-1859
81 Hurjui I, Neamtu A, Dorohoi D O. J. Mol. Struct., 2013, 1044: 134-139
82 Zhytniakivska O, Trusova V, Gorbenko G, Kirilova E, Kalnina I, Kirilov G, Molotkovsky J, Tulkki J, Kinnunen P. J. Fluoresc., 2014, 24(3): 899-907
83 Noguchi K, Herr D, Mutoh T, Chun J. Curr. Opin. Pharmacol., 2009, 9(1): 15-23
84 Nag S, Ma Q, Wang H, Chumnarnsilpa S, Lee W L, Larsson M, Kannan B, Hernandez-Valladares M, Burtnick L D, Robinson R C. Proc. Natl. Acad. Sci. USA, 2009, 106(33): 13713-13718
85 Gremm D, Wegner A. Eur. J. Biochem., 1999, 262(2): 330-334
86 Xu Y, Shen Z, Wiper D W, Wu M, Morton R E, Elson P, Kennedy A W, Belinson J, Markman M, Casey G. J. Am. Med. Assoc., 1998, 280(8): 719-723
Progress in Detection of Biomarker of Ovarian
Cancer: Lysophosphatidic Acid
LI Nian-Sheng, CHEN Li, XIAO Zuo-Xiu, YANG Yu-Qi, AI Ke-Long*
(Xiangya School of Pharmaceutical Sciences, Hunan Provincial Key Laboratory of Cardiovascular Research,
Central South University, Changsha 410078, China)
Abstract Ovarian cancer is a serious threat to women's health, and its mortality rate is the highest in gynecological cancer. At present, there is a lack of effective and convenient early diagnosis technology for ovarian cancer. Most of the patients with ovarian cancer are found in the middle and late stage and miss the best time for treatment. Therefore, the rapid, sensitive, economic, and accurate detection of early ovarian cancer is of great significance. In recent years, lysophosphatidic acid (LPA) has been found to increase in serum in the early stage of ovarian cancer, but less in other diseases. Therefore, LPA is a promising and highly specific marker of ovarian cancer. To detect LPA, many detection techniques and methods have been developed. In recent years, great progress has been made in this field. This paper summarizes the detection methods of LPA, and focuses on the important progress in this field in recent years. Finally, the challenges and prospects of LPA detection in real clinical application are also prospected.
Keywords Ovarian cancer: Lysophosphatidic acid; Biomarker; Mass spectrometry; Colorimetry; Fluorescence detection; Review
(Received 20 June 2020; accepted 14 August 2020)
This work was supported by the National Natural Science Foundation of China (No.21974134) and the Innovation-Driven Project of Central South University , China (No. 202045005).
2020-06-20收稿; 2020-08-14接受
本文系國家自然科学基金项目(No.21974134)和中南大学创新驱动项目(No.202045005)资助
* E-mail: aikelong@csu.edu.cn

