CYP450在植物非生物胁迫及解毒代谢中的作用

摘要 CYP450在动植物、真菌和细菌等生物中扮演丰富的功能角色,主要表现为参与各种次生代谢物质的合成及环境中有毒化合物的降解,对生物的生长发育和环境适应方面具有重要意义。对细胞色素CYP450在植物非生物胁迫及解毒代谢方面的生物学功能和应用进行了简要综述,为进一步研究该基因家族及各成员之间的功能提供理论参考。
关键词 植物CYP450;非生物胁迫;解毒代谢;生物学功能
中图分类号 Q945文献标识码 A
文章编号 0517-6611(2020)22-0010-04
doi:10.3969/j.issn.0517-6611.2020.22.004
Functions of CYP450 in Plant Abiotic Stress and Detoxification Metabolism
JIA Yan-hua (School of Biological Engineering,Chongqing University,Chongqing 400044)
Abstract CYP450 plays a wide range of function roles in animals,plants,fungi and bacteria,and is mainly involved in the synthesis of various secondary metabolites and degradation of toxic compounds in the environment,which is of great significance for the growth and development of organisms and environmental adaptation.This article briefly reviewed the biological functions and applications of cytochrome CYP450 in plant abiotic stress and detoxification metabolism,and provided theoretical references for the further research on the function of this gene family and its members.
Key words Plant CYP450;Abiotic stress;Detoxification metabolism;Biological function
作者简介 贾燕华(1994—),女,四川安岳人,硕士研究生,研究方向:植物分子生物学。
收稿日期 2020-05-09
细胞色素P450(Cytochrome P450,CYP450)是一个成员丰富的超基因家族激酶系统,广泛存在于各种动植物、真菌及细菌中[1]。CYP450由于其还原态可与CO相结合,并在450 nm处有最强吸收光谱而得名[2]。CYP450被誉为“万能生物催化剂”,一旦被激活,可参与一系列催化反应,在信号传导、生物防御、非生物胁迫以及代谢产物的合成和降解中扮演重要角色。动物中最早发现的CYP450酶系始于1958年,存在于大鼠的肝微粒体中。而植物中首次发现的CYP450酶系则始于1969年,存在于农作物棉花之中。近年来,研究报道的植物CYP450的数量呈现暴发增长趋势,被功能鉴定的植物CYP450也越來越多[3]。植物CYP450的功能主要体现在2个方面:一方面是参与次生代谢物质的合成,包括萜类、黄酮类、生物碱、内源激素及信号分子等;另一方面是参与有毒化合物的降解,如除草剂、杀虫剂、环境毒素和有机染料等。随着现代生物技术的发展,数以万计的植物CYP450基因被识别,部分植物的CYP450基因功能也得到了验证,这就加快了植物CYP450基因的功能研究进展。笔者针对目前CYP450的研究现状,主要对近年来植物CYP450的分离及其在非生物胁迫及解毒代谢中的功能研究进行简要综述。
1 植物CYP450基因的分类
通常情况下,根据CYP450基因编码蛋白序列的相似性可对其进行分类,若相似性大于40%则为同一家族;若为40%~55%,则属于同一家族下的2个亚家族;若大于55%,则属于同一亚家族;若为等位基因,其相似性应不小于97%[4]。1990年,植物中第一个CYP450基因CYP71A1被克隆,这是一个与鳄梨成熟相关的基因[5]。截至目前,植物中
被公布的CYP450家族已达到127个[6]。根据系统进化关系,植物CYP450家族可划分为11个簇(clans),分别是7个单基因家族簇(CYP51_clan、CYP74_clan、CYP97_clan、CYP710_clan、CYP711_clan、CYP727_clan、CYP746_clan)和4个多基因家族簇(CYP71_clan、CYP72_clan、CYP85_clan、CYP86_clan)[7]。CYP450的家族成员数量通过祖先基因的重复不断扩大,基因重复后产生的分化也使得各家族成员间的功能变得纷繁复杂,这对于植物的适应性进化具有非常重要的意义。
2 植物CYP450基因的分离
随着各种植物基因组序列的相继公布,被分离和鉴定的CYP450也越来越多。近年来,研究人员已从拟南芥、葡萄、木瓜、苔藓、水稻和三叶杨等植物中鉴定出1 415个CYP450基因[8]。在所有参与植物代谢的酶基因家族中,CYP450是成员数目最大的一个家族,约占植物总蛋白编码基因的1%[7]。研究表明,大部分已知CYP450家族的存在比单子叶和双子叶植物的分化时间还早[9]。根据http://drnelson.ut mem.edu/Cytochrome P450.html上更新至2009年8月的CYP450基因数目来看,植物中被公布的CYP450数目最多,约占已公布物种CYP450总数的40%(图1)。截至目前,NCBI数据库上上传的CYP450基因数目正不断更新(http://www.ncbi.nlm.nih.gov/Genbank/),但其中仍只有少部分被功能鉴定[10]。
3 与植物非生物胁迫有关的CYP450基因
非生物胁迫(如干旱、盐碱、寒冷和热胁迫等)是植物生长过程中不可避免的自然环境因素。通常情况下,干旱和盐胁迫是植物最主要的2种非生物胁迫因素,它们严重影响植物的生长和发育[11]。由于环境条件的不断恶化和高污染物的使用,植物不得不进化出各种各样的生存策略来应对水分不足和高盐胁迫等[12]。研究表明,CYP450基因家族在植物应对各种非生物胁迫和适应各种不利环境条件中扮演重要的生物学角色。
目前,大量植物CYP450基因被鉴定。植物CYP450的爆炸性增长也使得其成为植物代谢过程中数目最大、功能最复杂的一个激酶家族。多项研究表明,植物CYP450基因家族可通过参与植物内源激素的合成和降解,从而调节植物的
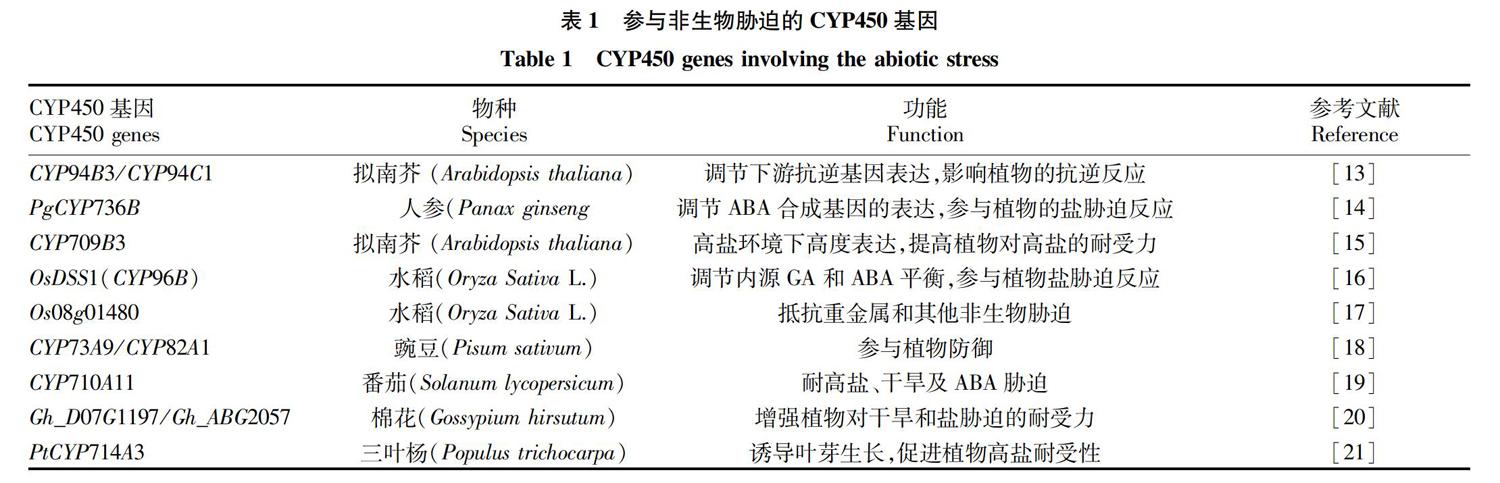
逆境胁迫响应。例如,拟南芥中的CYP94B3和CYP94C1基因分别可以促进 JA-Ile氧化形成12-OH JA-Ile 和12-COOH JA-Ile,使JA-Ile失活,影响茉莉酸代谢途径,调节下游抗逆基因的表达,从而影响植物的逆境胁迫响应[13]。Balusamy等[14]将人参中的PgCYP736B基因转移到拟南芥中,通过超表达发现该基因可以减少过氧化氢(H2O2)的积累,增加类胡萝卜素的含量,增强脱落酸合成基因的表达,从而增大植物对盐胁迫的忍耐力。同样,超表达拟南芥CYP709B3基因后亦可增强植物对盐胁迫的忍耐力[15]。水稻OsDSS1(CYP96B)基因可微量调节赤霉素(GA)与脱落酸(ABA)之间的平衡,从而影响水稻的生长发育和盐胁迫响应[16]。越来越多的CYP450家族成员(表1)被报道在拟南芥[9,13,15]、水稻[16-17]、豌豆[18]、番茄[19]、棉花[20]、三叶杨[21]、大豆[22]和烟草[23]等植物的非生物胁迫中上调表达,这些基因对植物的抗逆境响应具有重要的生物学意义。因此,对植物CYP450基因的非生物胁迫研究将为人们培育各种抗性新品种打下坚实的理论基础。
4 与植物解毒代谢有关的CYP450基因
病害、虫害和细菌等是植物生长过程中常面临的自然威胁,同时,除草剂、杀虫剂及其他化学污染物的使用又是植物不可逃避的人为毒害。当植物受到非生物胁迫和外源有机化合物毒害时,体内的CYP450就会被激活,通过一系列催化代谢反应,使有毒性的化合物转变成不含毒性的化合物,增强其生存竞争力,从而达到适者生存的目的[24]。Frear等[25]在棉花中发现能够代谢灭草隆(monuron)脱去2个N-甲基并生成尿素的CYP450酶系,直接证明了植物CYP450可参与除草剂代谢并具有解毒功能。目前,越来越多的研究相继证明CYP450基因家族可参与植物对除草剂的解毒代谢过程,这类基因在植物面临除草剂的威胁时高量表达,是植物对不同作用机理的除草剂产生抗性的机制之一[26]。随着研究手段的不断进步,植物CYP450的解毒代谢功能已成为科研工作者的研究热点。
植物主要通过代谢、缀合和分离3个过程对除草剂进行降解,而CYP450酶系则在第一个过程中扮演极其重要的角色,通过脱烷基、环氧化和羟基化等催化作用将除草剂转化为低毒或无毒产物[27]。例如,水稻中CYP450酶系降解苄嘧磺隆(bensulfuron-methyl,BSM),通过O-脱甲基作用产生4-OH BSM[28]。小麦和玉米对苯基脲类除草剂绿麦隆(chlorotoluron)的代谢主要表现为CYP450酶系介导的环甲基羟基化和N-脱甲基化作用[29-30]。CYP450酶系所介导的除草剂代谢还包括小麦对禾草灵(diclofop)的芳基羟基化[31],以及玉米对氯磺隆(chlorsulfuron)和醚苯磺隆(triasulfuron)的芳基羟基化作用等[32]。除此之外,植物CYP450还介导杀虫剂及其他外源物质的代谢。例如,日本看麦娘可以分解用于测定动物CYP450酶活性的7-甲氧基试卤灵和7-乙氧基香豆素[33]。
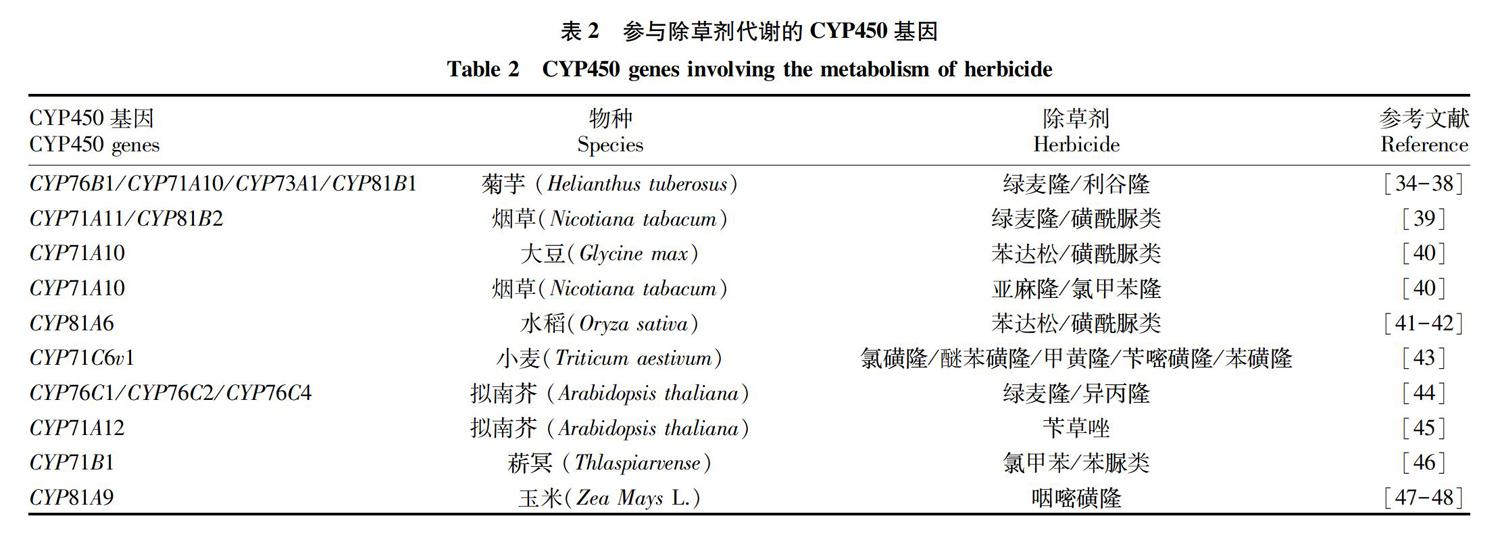
随着研究手段的不断进步,越来越多的植物CYP450基因被克隆和功能鉴定。例如,1994年Pierrel等[34]首次从菊芋块茎中分离得到了具有除草剂抗性的植物CYP450基因CYP73A1,该基因是一个反式肉桂酸4-羟化酶,通过环甲基羟基化过程,对绿麦隆进行降解。Robineau等[35]将菊芋CYP76B1基因过表达在烟草中能够分别增强对绿麦隆和利谷隆(linuron)抗性的10和20倍。目前已经有多个菊芋CYP450基因(表2)被证实对除草剂绿麦隆有降解作用[34-38]。煙草CYP71A11[39]、CYP81B2[39]和大豆CYP71A10[40]也能代谢脲类除草剂。并且在转基因烟草中,CYP71A10的表达也可增强对除草剂代谢活性,从而提高烟草对亚麻隆和氯甲苯隆的耐受性[40]。水稻中分离的CYP81A6与水稻抗苯达松(bentazone)和磺酰脲类除草剂密切相关[41-42]。在酵母中表达的小麦CYP71C6v1 基因,不仅表现出了对氯磺隆(chlorsulfuron)和醚苯磺隆(triasulfuron)的5-羟基化酶活性,同时也能催化甲磺隆(metsulfuron-methyl)、苄嘧磺隆(bensulfuron-methyl)和苯磺隆(tribenuron-methly)代谢[43]。Hfer等[44]研究发现,拟南芥的CYP76家族(表2)能够代谢萜醇类化合物和苯脲类除草剂。Didierjean等[38]通过转基因试验证实,从菊芋中克隆的CYP76B1 和CYP450 还原酶的融合蛋白对除草剂的代谢活性没有单独的CYP76B1高,表明融合蛋白在其高级结构的组装过程中稳定性有所下降,从而导致其功能活性下降。由于人类对除草剂的滥用,自然环境遭到严重的污染,特别是土壤和水污染已成为环境污染的主要来源。因此,细胞色素CYP450对除草剂的代谢功能被广泛研究也显得越来越有价值。近年来,通过基因工程技术,越来越多植物[45-48]和非植物CYP450家族[49-51]被证实具有除草剂代谢功能。总之,细胞色素CYP450是一类与除草剂代谢密切相关的解毒酶,研究其解毒功能和代谢机制对于开发新型除草剂和培育抗除草剂的转基因作物具有深远的意义。
5 展望
目前,随着高通量测序技术和生物信息学的发展,越来越多的植物CYP450被分离,但这仍是科研领域研究的重点和难点。虽然已成功分离数以万计的CYP450,但被功能鉴定的仍是沧海一粟。因此,分离植物CYP450基因是前提,解析CYP450基因的功能才是重点。另外,CYP450基因的表达调控属于转录水平的调控,受一些生物和非生物因子的调节,如光照、水分和组织器官的特异性等因素。因此,植物CYP450基因的表达和调控机制也是未来研究领域的必走之路。对于植物CYP450基因的研究,无论是从理论上探究植物生理代谢、适应性进化以及与生态环境的关系,还是在植物基因工程、生物防治、作物改良及环境修复上的应用,如培育多抗性作物、构建植物雄性不育系、培养能够降解除草剂及农药残留的转基因品种和超级微生物等方面显示出广阔的应用前景。
参考文献
[1] SHIOTA N,KODAMA S,INUI H,et al.Expression of human cytochromes P450 1A1 and P450 1A2 as fused enzymes with yeast NADPH-cytochrome P450 oxidoreductase in transgenic tobacco plants[J].Biosci Biotechnol Biochem,2000,64(10):2025-2033.
[2] XU J,WANG X Y,GUO W Z.The cytochrome P450 superfamily:Key players in plant development and defense[J].Journal of integrative agriculture,2015,14(9):1673-1686.
[3] WERCK-REICHHART D,FEYEREISEN R.Cytochromes P450:A success story[J].Genome Biol,2000,1(6):1-9.
[4] 戴素明,周程爱,谢丙炎,等.细胞色素P450表达在植物防御反应中的作用[J].石河子大学学報(自然科学版),2004,22(S1):184-187.
[5] BOZAK K R,YU H,SIREVG R,et al.Sequence analysis of ripening-related cytochrome P-450 cDNAs from avacado fruit[J].Proc Natl Acad Sci USA,1990,87(10):3904-3908.
[6] GHOSH S.Triterpene structural diversification by plant cytochrome P450 enzymes[J].Front Plant Sci,2017,8:1-15.
[7] NELSON D,WERCK-REICHHART D.A P450-centric view of plant evolution[J].Plant J,2011,66(1):194-211.
[8] NELSON D R,MING R,ALAM M,et al.Comparison of cytochrome P450 genes from six plant genomes[J].Tropical plant biology,2008,1:216-235.
[9] NELSON D R,SCHULER M A,PAQUETTE S M,et al.Comparative genomics of rice and Arabidopsis.Analysis of 727 cytochrome P450 genes and pseudogenes from a monocot and a dicot[J].Plant physiology,2004,135(2):756-772.
[10] 余小林,曹家树,崔辉梅,等.植物细胞色素P450[J].细胞生物学杂志,2004,26(6):561-566.
[11] SUZUKI N,RIVERO R M,SHULAEV V,et al.Abiotic and biotic stress combinations[J].New Phytol,2014,203(1):32-43.
[12] MEENA K K,SORTY A M,BITLA U M,et al.Abiotic stress responses and microbe-mediated mitigation in plants:The omics strategies[J].Frontiers in plant science,2017,8:1-25.
[13] KITAOKA N,MATSUBARA T,SATO M,et al.Arabidopsis CYP94B3 encodes jasmonyl-L-isoleucine 12-hydroxylase,a key enzyme in the oxidative catabolism of jasmonate[J].Plant & cell physiology,2011,52(10):1757-1765.
[14] BALUSAMY S R,RAHIMI S,YANG D C.Characterization of squalene-induced PgCYP736B involved in salt tolerance by modulating key genes of abscisic acid biosynthesis[J].Int J Biol Macromol,2019,121:796-805.
[15] MAO G H,SEEBECK T,SCHRENKER D,et al.CYP709B3,a cytochrome P450 monooxygenase gene involved in salt tolerance in Arabidopsis thaliana[J].BMC Plant Biol,2013,13:1-13.
[16] TAMIRU M,UNDAN J R,TAKAGI H,et al.A cytochrome P450,OsDSS1,is involved in growth and drought stress responses in rice (Oryza sativa L.)[J].Plant Mol Biol,2015,88(1/2):85-99.
[17] RAI A,SINGH R,SHIRKE P A,et al.Expression of rice CYP450-like gene (Os08g01480) in Arabidopsis modulates regulatory network leading to heavy metal and other abiotic stress tolerance[J].PLoS One,2015,10(9):1-17.
[18] WHITBRED J M,SCHULER M A.Molecular characterization of CYP73A9 and CYP82A1 P450 genes involved in plant defense in pea[J].Plant Physiol,2000,124(1):47-58.
[19] 毛萍,黄宝玉,喻宏,等.番茄细胞色素P450基因CYP710A11的抗逆功能[C]//中国的遗传学研究——遗传学进步推动中国西部经济与社会发展——2011年中国遗传学会大会论文
摘要汇编.北京:中国遗传学报,2011.
[20] MAGWANGA R O,LU P,KIRUNGU J N,et al.Knockdown of cytochrome P450 genes Gh_D07G1197 and Gh_A13G2057 on chromosomes D07 and A13 reveals their putative role in enhancing drought and salt stress tolerance in Gossypium hirsutum[J].Genes,2019,10(3):1-21.
[21] WANG C T,YANG Y,WANG H H,et al.Ectopic expression of a cytochrome P450 monooxygenase gene PtCYP714A3 from Populus trichocarpa reduces shoot growth and improves tolerance to salt stress in transgenic rice[J].Plant Biotechnol J,2016,14(9):1838-1851.
[22] GUTTIKONDA S K,TRUPTI J,BISHT N C,et al.Whole genome co-expression analysis of soybean cytochrome P450 genes identifies nodulation-specific P450 monooxygenases[J].BMC Plant Biol,2010,10:1-19.
[23] RALSTON L,KWON S T,SCHOENBECK M,et al.Cloning,heterologous expression,and functional characterization of 5-epi-aristolochene-1,3-dihydroxylase from tobacco (Nicotiana tabacum)[J].Arch Biochem Biophys,2001,393(2):222-235.
[24] DURST F.Biochemistry and physiology of plant cytochrome P-450[M]∥RUCKPAUL K,REIN H.Microbial and plant cytochrome P-450:Biochemical characteristics,genetic engineering and practical implications,frontiers in biotransformation.New York:Taylor and Francis,1991:191-232.
[25] FREAR D S,SWANSON H R,TANAKA F S.N-demethylation of substituted 3-(phenyl) methylureas:Isolation and characterization of a microsomal mixed function oxidase from cotton[J].Phytochemistry,1969,8(11):2157-2169.
[26] 汪思遠,蒋世翠,王康宇,等.植物细胞色素P450 的研究进展[J].吉林蔬菜,2014(4):41-45.
[27] SIMINSZKY B.Plant cytochrome P450-mediated herbicide metabolism[J].Phytochemistry reviews,2006,5(2/3):445-458.
[28] DENG F,HATZIOS K K.Characterization of cytochrome P450-mediated bensulfuron-methyl O-demethylation in rice[J].Pesticide biochemistry and physiology,2002,74(2):102-115.
[29] MOUGIN C,CABANNE F,CANIVENC M C,et al.Hydroxylation and N-demethylation of chlorotoluron by wheat microsomal enzymes[J].Plant science,1990,66(2):195-203.
[30] KREUZ K,FONN-PFISTER R.Herbicide-insecticide interaction in maize:Malathion inhibits cytochrome P450-dependent primisulfuron metabolism[J].Pesticide biochemistry and physiology,1992,43(3):232-240.
[31] PERSANS M W,SCHULER M A.Differential induction of cytochrome P450-mediated triasulfuron metabolism by naphthalic anhydride and triasulfuron[J].Plant Physiol,1995,109(4):1483-1490.
[32] MORELAND D E,CORBIN F T,MCFARLAND J E.Oxidation of multiple substrates by corn shoot microsomes[J].Pesticide biochemistry and physiology,1993,47(3):206-214.
[33] WANG HC,LI J,LV B,et al.The role of cytochrome P450 monooxygenase in the different responses to fenoxaprop-P-ethyl in annual bluegrass (Poa annua L.) and short awned foxtail (Alopecurus aequalis Sobol.)[J].Pesticide biochemistry and physiology,2013,107(3):334-342.
[34] PIERREL M A,BATARD Y,KAZMAIER M,et al.Catalytic properties of the plant cytochrome P450 CYP73 expressed in yeast[J].Eur J Biochem,1994,224(3):835-844.
[35] ROBINEAU T,BATARD Y,NEDELKINA S,et al.The chemically inducible plant cytochrome P450 CYP76B1 actively metabolizes phenylureas and other xenobiotics[J].Plant Physiol,1998,118(3):1049-1056.
[36] CABELLO-HURTADO F,BATARD Y,SALAN J P,et al.Cloning,expression in yeast and functional characterization of CYP81B1,a plant cytochrome P450 that catalyzes in-chain hydroxylation of fatty acids[J].J Biol Chem,1998,273(13):7260-7267.
[37] WERCK-REICHHART D,GABRIAC B,TEUTSCH H,et al.Two cytochrome P-450 isoforms catalysing O-de-ethylation of ethoxycoumarin and ethoxyresorufin in higher plants[J].Biochem J,1990,270(3):729-735.
[38] DIDIERJEAN L,GONDET L,PERKINS R,et al.Engineering herbicide metabolism in tobacco and Arabidopsis with CYP76B1,a cytochrome P450 enzyme from Jerusalem artichoke[J].Plant Physiol,2002,130(1):179-189.
[39] YAMADA T,KAMBARA Y,IMAISHI H,et al.Molecular cloning of novel cytochrome P450 species induced by chemical treatments in cultured tobacco cells[J].Pesticide biochemistry and physiology,2000,68(1):11-25.
[40] SIMINSZKY B,CORBIN F T,WARD E R,et al.Expression of a soybean cytochrome P450 monooxygenase cDNA in yeast and tobacco enhances the metabolism of phenylurea herbicides[J].Proc Natl Acad Sci USA,1999,96 (4):1750-1755.
[41] PAN G,ZHANG X Y,LIU K D,et al.Map-based cloning of a novel rice cytochrome P450 gene CYP81A6 that confers resistance to two different classes of herbicides[J].Plant molecular biology,2006,61(6):933-943.
[42] LIU C,LIU S Q,WANG F,et al. Expression of a rice CYP81A6 gene confers tolerance to bentazon and sulfonylurea herbicides in both Arabidopsis and tobacco[J].Plant cell,tissue and organ culture,2012,109(3):419-428.
[43] XIANG W S,WANG X J,REN T R.Expression of a wheat cytochrome P450 monooxygenase cDNA in yeast catalyzes the metabolism of sulfonylurea herbicides[J].Pestic Biochem Physiol,2006,85(1):1-6.
[44] HFER R,BOACHON B,RENAULT H,et al.Dual function of the cytochrome P450 CYP76 family from Arabidopsis thaliana in the metabolism of monoterpenols and phenylurea herbicides[J].Plant physiology,2014,166(3):1149-1161.
[45] HAYASHI E,FUZIMOTO K,IMAISHI H.Expression of Arabidopsis thaliana cytochrome P450 monooxygenase,CYP71A12,in yeast catalyzes the metabolism of herbicide pyrazoxyfen[J].Plant biotechnology,2007,24:393-396.
[46] LAMB S B,LAMB D C,KELLY S L,et al.Cytochrome P450 immobilisation as a route to bioremediation/biocatalysis[J].FEBS Letters,1998,431(3):343-346.
[47] LI J,YU H,ZHANG F Z,et al.A built-in strategy to mitigate transgene spreading from genetically modified corn[J].PLoS One,2013,8(12):1-8.
[48] HIROSE S,KAWAHIGASHI H,OZAWA K,et al.Transgenic rice containing human CYP2B6 detoxifies various classes of herbicides[J].J Agric Food Chem,2005,53(9):3461-3467.
[49] KAWAHIGASHI H,HIROSE S,OZAWA K,et al.Analysis of substrate specificity of pig CYP2B22 and CYP2C49 towards herbicides by transgenic rice plants[J].Transgenic Res,2005,14(6):907-917.
[50] KAWAHIGASHI H,HIROSE S,INUI H,et al.Enhanced herbicide cross-tolerance in transgenic rice plants co-expressing human CYP1A1,CYP2B6,and CYP2C19[J].Plant science,2005,168(3):773-781.
[51] KAWAHIGASHI H,HIROSE S,OHKAWA H,et al.Phytoremediation of the herbicides atrazine and metolachlor by transgenic rice plants expressing human CYP1A1,CYP2B6,and CYP2C19[J].J Agric Food Chem,2006,54(8):2985-2991.

