基于转录组测序的奶牛产奶性状重要功能基因挖掘
陈一丹 张昱 杨洁 张勤,2 姜力
(1. 中国农业大学动物科学技术学院 畜禽育种国家工程实验室 农业农村部动物遗传育种与繁殖重点实验室,北京 100193;2. 山东农业大学动物科技学院,泰安 271018)
牛奶营养丰富,是人类良好的蛋白质和钙的来源,同时也是生产多种加工食品的原料,对人类生活具有重要意义[1]。产奶性状是奶牛最重要的生产性状,挖掘影响奶牛产奶性状的功能基因或遗传变异一直是奶牛分子育种中的研究热点。如何准确的确定基因型与表型之间的关系是当前畜禽遗传研究的重要挑战之一。而基因组变异与基因表达以及表型关系的整合分析是揭示基因型与表型关系的有效途径[2]。
近年来,快速发展的二代测序技术为揭示复杂性状遗传基础以及表型变异的机制提供了重要工具。RNA-seq技术不但可以检测细胞或组织中所有基因的表达,而且为鉴定转录本中的遗传变异提供了新的机会[3],为深入挖掘奶牛产奶性状功能基因和重要突变位点提供了便利条件。众所周知,转录本中的遗传变异会对基因表达和基因的转录后调控具有调控作用。因此,本研究以中国荷斯坦奶牛为研究对象,选择高产奶牛和低产奶牛泌乳期的血液组织进行转录组分析,通过检测差异表达基因和转录本中的遗传变异位点,并结合生物信息学分析,筛选与产奶性状相关的重要功能基因和遗传变异。本研究旨在进一步挖掘和鉴定影响奶牛产奶性状的遗传标记位点,为提高我国奶牛分子选育的准确性提供重要信息。
1 材料与方法
1.1 材料
本研究试验个体来自于北京三元金银岛牛场,按照305 d产奶量、乳脂量和乳蛋白量3个性状表型信息及育种值信息对所有个体进行筛选,最终选取3头高产荷斯坦奶牛和3头低产荷斯坦奶牛,产奶性状表型信息如表1所示。所有试验奶牛均为健康个体,在同一饲养环境下饲养。试验牛进行尾椎静脉采血,血液收集到抗凝管后立即与RNA保护试剂混合,用于RNA的提取。
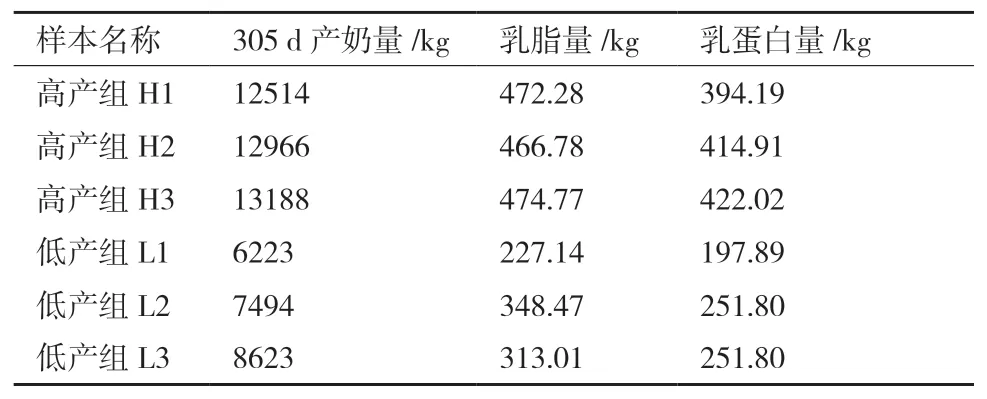
表1 试验群体
1.2 方法
1.2.1 RNA提取及测序 使用Trizol法提取所有样品的总RNA。利用Nanodrop核酸分析仪和Qubit对RNA进行纯度和浓度的检测。使用Agilent 2100对RNA的完整性进行检测。每个样品的浓度均大于100 ng/μL,RIN值>7.5。cDNA 文库的构建参照Illumina TruSeqTMRNA(Illumia,USA)样品制备试剂盒操作说明进行。cDNA文库质检合格后,使用Illumina Hiseq 2500测序平台进行的双末端测序。
1.2.2 测序数据质量控制 测序后的原始序列(Raw reads)需去除带接头、含有 poly-N以及低质量reads形成Clean reads。将Clean reads比对到牛的参考基因组序列(UMD3.1)和相应的基因注释文件(UMD3.1)。
1.2.3 遗传变异检测 使用BWA[4]软件将转录组双端测序数据比对到参考基因组上,然后使用SAMtools[5]与BCFtools[6]软件进行遗传变异的检测。使用vcffilter软件以DP>5、QUAL>30、MQ>40为条件对检测到的变异进行过滤。使用BCFtools注释已存在于dbSNP数据库中的遗传变异。使用snpEff[7]软件对所有变异进行功能性预测分析。
1.2.4 遗传变异筛选及功能基因注释 基于检测到的遗传变异,筛选出突变等位基因频率在高产组中≥1/2,且低产组中≤1/3的遗传变异;以及在低产组中突变等位基因频率≥1/2,且高产组中≤1/3的遗传变异。在这些遗传变异中,进一步筛选出被snpEff预测为影响程度高(HIGH)和中等(MODERATE)的变异,并统计这些变异所在的基因。
1.2.5 基因的差异表达分析 利用 BWA软件进行转录组测序数据的比对。使用featureCounts[8]统计counts数后,利用DESeq2[9]软件进行基因差异表达分析。以P<0.05、差异表达倍数>1.5为显著性阈值筛选高低产组之间的差异表达基因。
1.2.6 基因的富集分析 利用DAVID(https://david.ncifcrf.gov/)数据库对所有差异表达基因进行GO功能注释和KEGG通路分析,以P<0.05为显著性阈值筛选显著的GO条目和富集的通路。
1.2.7 候选功能基因的筛选 将筛选的重要遗传变异涉及的基因与差异表达基因取交集,筛选潜在影响基因表达的遗传变异。利用DESeq2软件输出的标准化表达矩阵,使用R语言对筛选的候选基因进行绘图,并对这些基因进行生物信息学分析。同时将这些基因与Animal QTL数据库(https://www.animalgenome.org/cgi-bin/QTLdb/BT/index)中包含的产奶性状QTLs进行比对,筛选与产奶量、乳脂量、乳蛋白量、乳脂率、乳蛋白率QTLs重叠的候选功能基因,进一步挖掘与奶牛产奶性状相关的功能基因。
2 结果
2.1 测序数据质量检测结果
原始测序数据经质控后最终获得38.1G的Clean reads。将这些Clean reads比对到牛的参考基因组上,每个个体的比对率在 83.6%-92.8%之间,结果如表2所示。这些reads比对到mRNA中的比例在84.5%-94.7%之间。其中大部分reads都比对到基因的编码区,比对率在62.7%-74.9%之间。

表2 测序数据质量评估
2.2 遗传变异检测结果
测序数据经分析后获得6个个体转录本中的所有遗传变异,统计结果如表3所示。所有个体转录本检测到的遗传变异平均数为145 750,其中SNP的数量占总遗传变异数的91.23%。与dbSNP数据库进行比对,结果显示已被数据库收录的遗传变异比例在80.95%到85.28%之间,其余为本研究新发现的遗传变异。
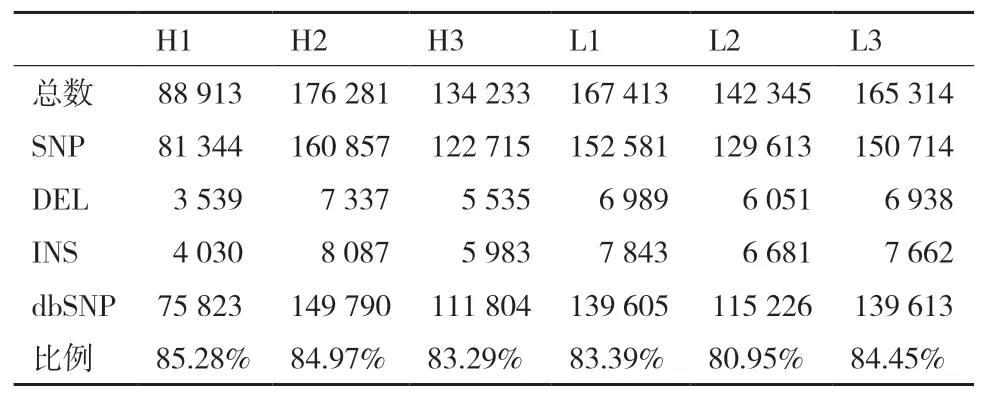
表3 遗传变异数目统计
使用snpEff软件对所有遗传变异进行功能性预测分析,将其影响程度分为HIGH、MODERATE、LOW和MODIFIER四种情况,结果如表4所示。被预测为HIGH的遗传变异对蛋白质产物有高度的破坏性,可能导致蛋白质功能丧失或截断;被预测为MODERATE的遗传变异可能对蛋白质的性质和功能具有影响;被预测为LOW的遗传变异不会使蛋白质发生改变;而被预测为MODIFIER的遗传变异是非编码的遗传变异或影响非编码基因的变异[3]。将这些遗传变异所在的基因组功能区域进行注释(表5),结果显示每个个体在基因的外显子区检测到的遗传变异数平均为15 642个,在3' UTR和5' UTR检测到的变异数量平均为7 883个和942个。一些基因内含子上的遗传变异被预测可能导致新可变剪切体的产生。

表4 遗传变异影响程度的预测结果
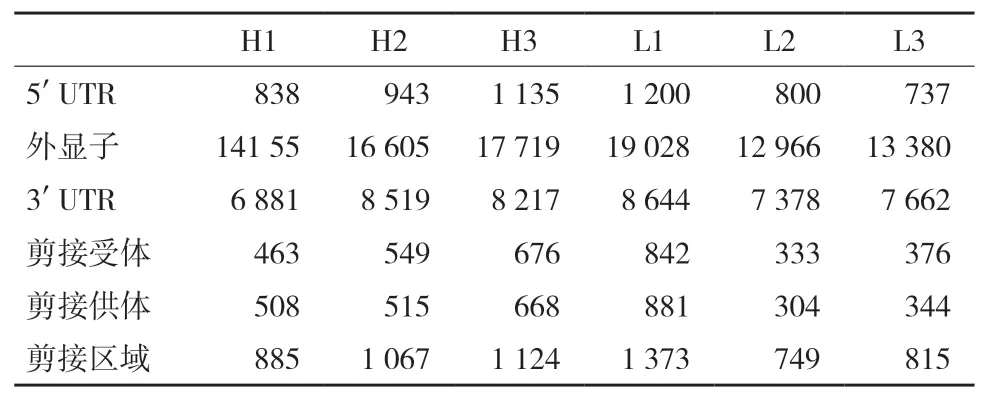
表5 遗传变异在基因组功能区的分布
2.3 遗传变异筛选结果
根据突变位点在高低组中不同的等位基因频率,对所有遗传变异进行筛选。结果显示在高产组中突变位点等位基因频率≥1/2,且在低产组中等位基因频率≤1/3的遗传变异共有27 143个,其中22 301个已存在于dbSNP数据库中(图1),其中被预测为对蛋白功能有重要(HIGH)影响的遗传变异有330个,对蛋白功能有中等程度(MODERATE)影响的遗传变异有1 174个(图1),这些变异共涉及979个基因(图2-A)。
同时,我们筛选到在低产组中突变等位基因频率≥1/2,且在高产组中等位基因频率≤1/3的遗传变异共40 650个,其中33 687个已存在于dbSNP数据库中(图1),其中被预测对蛋白功能具有重要(HIGH)影响的遗传变异有120个,对蛋白功能为中等程度(MODERATE)影响的遗传变异有927个(图1),这些变异共涉及643个基因(图2-B)。

图1 遗传变异筛选结果分析
2.4 差异表达基因分析结果
利用DESeq2软件进行基因的差异表达分析,以P<0.05、|Fold Change|≥ 1.5作为显著性阈值,最终在高低组之间检测到431个差异表达基因,其中190个基因在低产组的表达量高于在高产组的表达量,241个基因在低产组的表达量低于在高产组的表达量(图2)。
2.5 产奶性状重要候选基因的筛选
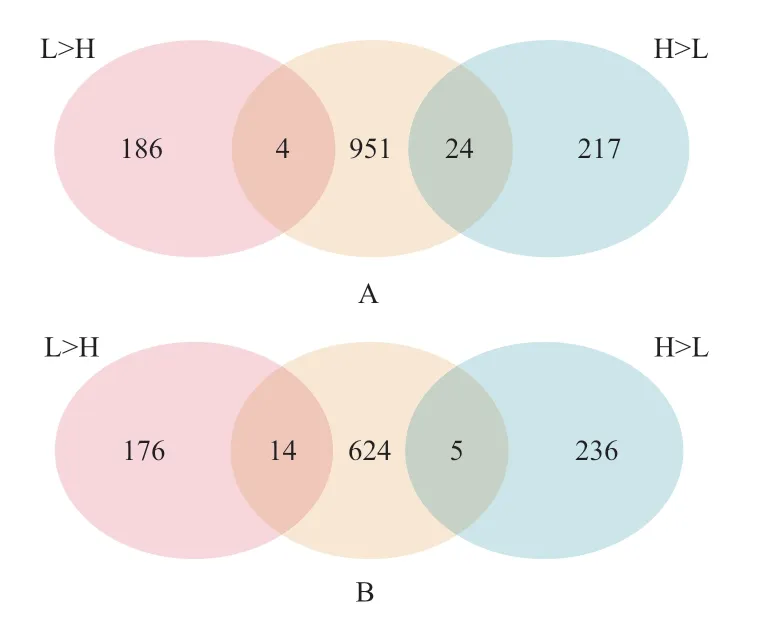
图2 差异表达基因与含有重要遗传变异基因的整合分析
将差异表达基因与上述筛选的含有重要遗传变异的基因取交集,共获得47个基因(图2)。其中在高产组中突变等位基因频率≥1/2,且低产组中等位基因频率≤1/3,同时预测对蛋白功能具有HIGH或MODERATE影响的遗传变异所涉及的基因与差异表达基因取交集,共筛选到28个基因,其中24个基因在高产组中的表达量显著高于在低产组(图2-A)。在低产组中等位基因频率≥1/2,且在高产组中等位基因频率≤1/3,并且预测对蛋白功能具有HIGH或MODERATE影响的遗传变异所涉及的基因与差异表达基因取交集,共获得19个基因,其中14个基因在低产组中的表达量显著高于在高产组(图2-B)。
将筛选到的47个基因进一步进行生物信息学分析,检测到1个显著的GO条目(GO:0004252~serine-type endopeptidase activity)和1条显著的通路(bta01100:Metabolic pathways),共包含9个 基 因:ASS1、CKB、GGT1、UPP1、MGAM、SDSL、HP、LTF和MMP9(表6)。
将47个基因与Animal QTL数据库中产奶性状QTLs进行比对,筛选到4个已报道与产奶性状相关的基因,即DEFB4A、LTF、PGLYRP1、MS4A8。将上述12个重要候选基因内的遗传变异进行汇总,共获得14个遗传变异位点(表7),其中8个突变会引起氨基酸的错义突变。使用DESeq2软件得到的标准化表达量后,对这14个突变位点不同基因型个体进行基因表达量的统计,结果显示不同基因型个体之间基因的表达量存在明显差别,如图3所示。由于本研究试验群体有限,未来需在更大规模的奶牛群体中进一步验证。

表6 筛选基因的GO和KEGG分析
3 讨论
奶牛的产奶性状一直是育种工作者最为关注的性状之一,随着人们生活水平的日益提高,人们对牛奶的需求量不断地增加。准确的遗传评估无疑为加快奶牛产奶性状遗传进展发挥了重要作用。当前,基因组选择是继奶牛育种中的表型选择、育种值选择之后的最先进的分子育种方法。如果可以获得更多的与产奶性状有关的分子标记信息,并将其加入到基因组选择当中,就可以进一步提高奶牛分子选育的准确性。因此,许多科研工作者通过全基因组关联分析、转录组、蛋白质组等多种策略挖掘影响奶牛产奶性状的功能基因和遗传变异[10-13]。目前,利用高低产奶牛的乳腺和肝脏组织的转录组研究已有报道,这些研究着重利用测序数据进行差异表达基因、差异表达非编码RNA的筛选,并利用生物信息学手段对基因和非编码RNA之间的调控关系进行预测[12,14],从而进一步挖掘影响奶牛产奶性状的候选基因。本研究利用二代转录组测序技术对高低产组奶牛血液组织中的差异表达基因进行检测。鉴于奶牛在泌乳过程中许多重要的营养物质是通过血液运送到乳腺组织合成牛奶中的各种乳成分,对血液组织的研究具有重要的意义。此外,为了实现分子标记应用于奶牛育种实践的目标,本研究侧重于高低产奶牛基因组中的遗传变异的检测和分析,同时结合基因在高低组中的表达以及在不同基因型个体中的表达进行重要候选基因及遗传变异的筛选。研究结果为揭示奶牛产奶性状表型差异的遗传基础及分子选育提供了重要信息。

表7 12个重要候选基因的遗传变异情况
本研究共筛选到12个影响产奶性状的候选功能基因,其中ASS1、DEFB4A、GGT1、HP、LTF、MMP9、MGAM、UPP1、PGLYRP1基因内含有在高产组中突变等位基因频率较高,而在低产组中等位基因频率较低的遗传变异。这9个重要候选基因中包含11个重要突变位点,其中7个为错义突变。位于PGLYRP1基因的SNP为同义突变,Wang等[15]的研究显示该突变位点与荷斯坦奶牛的305 d产奶量和体细胞数密切相关,并且突变型奶牛的305 d产奶量相比野生型个体的产奶量显著上升,体细胞评分显著下降,推断该突变可能通过改变mRNA的稳定性影响蛋白质的表达和功能,同时也可能通过密码子的偏好性改变蛋白质的折叠结构,从而影响蛋白功能的发挥。研究发现这些基因中包含多个重要的酶类,与营养物质的吸收、合成及代谢过程紧密相关。例如,ASS1基因所编码的精氨酸琥珀酸合成酶与氨基酸的合成、代谢和尿素循环等都有密切联系[16]。GGT1基因所编码的精-谷氨酰转移酶是一种细胞膜结合酶,在γ-谷氨酰循环中发挥重要作用,与氨基酸的吸收密切相关[17]。MGAM基因编码的麦芽糖-葡萄糖化酶是一种位于小肠绒毛刷状缘的酶,负责将多糖分解为葡萄糖[18],与碳水化合物的消化和吸收、半乳糖、蔗糖和淀粉的代谢有关。此外,位于这些基因上的遗传变异不同基因型下基因的表达量存在明显差异,都表现为突变型纯合子个体基因的表达量最高,野生型纯合子个体的表达量最低,杂合子个体表达量普遍居于两者之间。由于这些基因包含的遗传变异在高产组中的基因频率明显高于低产组,同时显示突变型个体基因的表达量高于野生型个体,并且这些基因在高产组的表达量显著高于低产组。因此,本研究推测这些遗传变异对产奶性状表型有正向作用,突变型为优势基因型,可进一步验证后作为分子标记应用于奶牛的分子育种中。
此外,本研究筛选到重要的一个乳成分基因,即乳铁蛋白基因(LTF)。LTF编码的乳铁蛋白是牛奶中重要的营养成分,在先天免疫系统中起着重要的作用[19-20],参与铁代谢、抗肿瘤、抗细菌等多种免疫调节过程[21-23]。LTF基因在许多研究中被证明与奶牛的生产性能有关。2010年,O' Halloran等[24]发现LTF上一个位于转录起始位点上游-28 bp的SNP突变(A/C)对乳蛋白量有一定影响。2015年,Mao等[25]的研究表明LTF上位于转录起始位点上游-270 bp从T到C的SNP突变对产奶量、乳脂率和乳蛋白率均有正向的影响,而位于-190 bp从G到A的突变则对乳脂率和乳蛋白率有不利的影响。2017年,Viale等[26]发现LTF中一个SNP(rs43765462)与荷斯坦奶牛的乳脂率性状正相关。2018年,Raschia等[27]发现LTF上另外一个SNP(rs43706485)与奶牛的305 d产奶量有潜在的相关性。本研究首次在LTF基因中发现一个可能引起可变剪接的SNP突变(T/A)。该突变位点不同基因型个体LTF基因的表达量表现为突变纯合子基因型个体(AA)远大于突变杂合子基因型个体(AT),大于野生型纯合子个体(TT),并且该突变等位基因频率在高产组明显高于低产组。因此,本研究认为该突变为对奶牛的产奶量、乳脂量和乳蛋白量具有正向影响。由于牛奶中的天然乳铁蛋白含量极低,每毫升牛奶中仅含0.02 mg-0.35 mg[28]。随着乳铁蛋白在婴幼儿免疫调节以及抗肿瘤等多种功能作用的不断解析[23,29-30],目前天然乳铁蛋白做为食品添加剂和营养强化剂的价格仍较为昂贵。因此,培育牛奶中具有较高含量乳铁蛋白成分的奶牛具有很高的经济价值。本研究发现的该分子标记有望将来用于选育牛奶中富含乳铁蛋白的奶牛品系。
同时,本研究分别在DEFB4A基因和MS4A8基因中检测到一个重要的遗传变异。Bagnicka 等[31]的研究结果表明DEFB4A基因上位于转录位点起始位置上游的2 239 bp从C到T的突变对奶牛的乳脂率有不利影响。本研究发现DEFB4A基因上存在一个从C到A的错义突变(rs43108924),该突变位点在高产组中的等位基因频率高于在低产组中,同时该突变位点不同基因型个体中DEFB4A的表达量呈现为突变纯合子基因型个体表达量大于野生型个体,因此推测该突变是对奶牛的产奶量、乳脂量和乳蛋白量有正向作用的有利突变。Cochran等[32]的研究发现MS4A8中的错义突变(rs109761676)对奶牛的产奶量、乳脂率和乳蛋白率有负面影响。而该突变位点同样在本研究中被筛选到。本研究结果显示该突变位点(G/T)在奶牛低产组中的等位基因频率明显高于在高产组中的基因频率,再次证明了该突变对产奶性状是一个潜在的不利突变。对该遗传变异位点进行不同基因型个体表达量的分析,发现在该基因突变型纯合子个体(GG)的表达量低于杂合子基因型(GT)的个体和野生型个体(TT),说明该突变可能是引起MS4A8表达量降低的隐性突变,通过降低基因的表达水平最终导致奶牛产奶生产水平的下降。
值得关注的是,CKB基因编码的肌酸激酶B与肌酸代谢、氨基酸代谢、尿素循环等均有一定的关系[33]。SDSL基因所编码的丝氨酸脱水酶类似物是一种与丝氨酸脱水酶类似的,与氨基酸的合成与代谢有关的蛋白质[34]。本研究发现这两个基因上均存在一个遗传变异在奶牛低产组中突变等位基因频率明显高于高产组中等位基因频率,且突变型纯合子个体基因的表达量高于或接近于突变型杂合子表达量高于野生型表达量。这说明一些突变也可能是通过上调所在基因表达量影响奶牛的重要代谢过程,最终对产奶性能产生不利影响。
4 结论
本研究利用奶牛产奶性状表型两尾极端个体的转录组测序数据进行遗传变异检测和基因的差异表达分析,旨在筛选与奶牛产奶性状相关的重要候选功能基因及遗传变异。通过对突变位点在高低产组中等位基因频率分布的比较、基因差异表达分析、生物信息学分析以及与产奶性状QTL数据库的比对,最终筛选到12个重要的产奶性状候选基因和位于这些基因内的14个重遗传变异。经分析,这些变异包括潜在的有利突变和不利突变,可能通过改变基因的表达调控奶牛产奶性状的表型,这些信息对将来更加准确的进行奶牛分子育种具有重要意义。

