肿瘤基因组图谱之泛癌图谱
何询,胡昊良
·综述·
肿瘤基因组图谱之泛癌图谱
何询,胡昊良
518057 深圳未名新鹏生物医药有限公司(何询);410000 长沙,湖南师范大学动物多肽药物创制国家地方联合工程实验室(胡昊良)
根据肿瘤类型和病理过程不同,肿瘤治疗的传统方法主要包括手术、化疗和放疗等。肿瘤的靶向治疗和免疫治疗虽然具有更好的疗效和更小的副作用,但目前其效果仍受到如肿瘤异质性等多重因素的制约。有趣的是,不同组织和器官的肿瘤在分子水平上具有相似性,这种相似性甚至比同种肿瘤不同分型之间的相似性更大[1]。因此,这也为肿瘤的精准治疗提供了更多的可能性。美国国立卫生研究院(NIH)资助的癌症基因组图谱(TCGA)之一的“泛癌图谱”项目,意旨从分子特征的角度分析多种肿瘤类型。
最近,这项研究的最新成果为我们带来了关于肿瘤研究的全新视野。科学家们对收集的 33 种肿瘤、10000 多个肿瘤病理样本的癌症基因组图谱数据,以及对肿瘤的转录组学、蛋白质组学、甲基化组学、临床数据进行了全面综合的分析,挖掘出其中的有效信息,成功绘制出泛癌图谱(图 1)。该图谱针对人类癌症发生的方式、地点和原因提供了全景视图(主要包括肿瘤的细胞起源模式、致癌过程和信号通路),这为癌症诊断和治疗提供了新思路[2-4]。泛癌图谱数据报告分为三大部分,26 篇科研论文分别发表在《细胞》及其旗下顶尖医学杂志上。
1 癌症的细胞起源模式
在肿瘤发展过程中,基因组突变的多样性、信号通路和致癌过程的改变受到多种因素的影响,包括癌细胞的发展、分化和表观遗传调控。这也就造成我们认为的同一种癌症,可能在分子水平上具有很大的差异。随着诊疗技术的不断发展,传统的组织病理学分析已经不足以描述癌症的类型。部分同种组织的不同类型肿瘤存在较大的异质性,而组织学相关的癌症类型之间却存在分子相似性。泛癌图谱根据分子特征对人类癌症类型进行了重新分类,为科研和临床试验设计带来了新的见解。
研究者们采用一种名为 iCluster 的算法,对来自33 种肿瘤类型的 1.1 万个肿瘤样本的非整倍体、DNA 甲基化、mRNA、miRNA 和蛋白表达数据进行的综合聚类分析,揭示了不同癌症类型之间的细胞起源是肿瘤分类的重要特征[4]。研究者将解剖学上的 33 种癌症重新归类为 28 种亚型,其中 2/3 的肿瘤亚型存在不同程度的异质性;但针对某一种新肿瘤亚型,其中竟又包括了 25 种传统解剖学意义上的癌症[4]。因此,传统的以组织学或解剖学来源的肿瘤分类应以基于不同组织类型肿瘤之间的分子相似性分类作为补充。
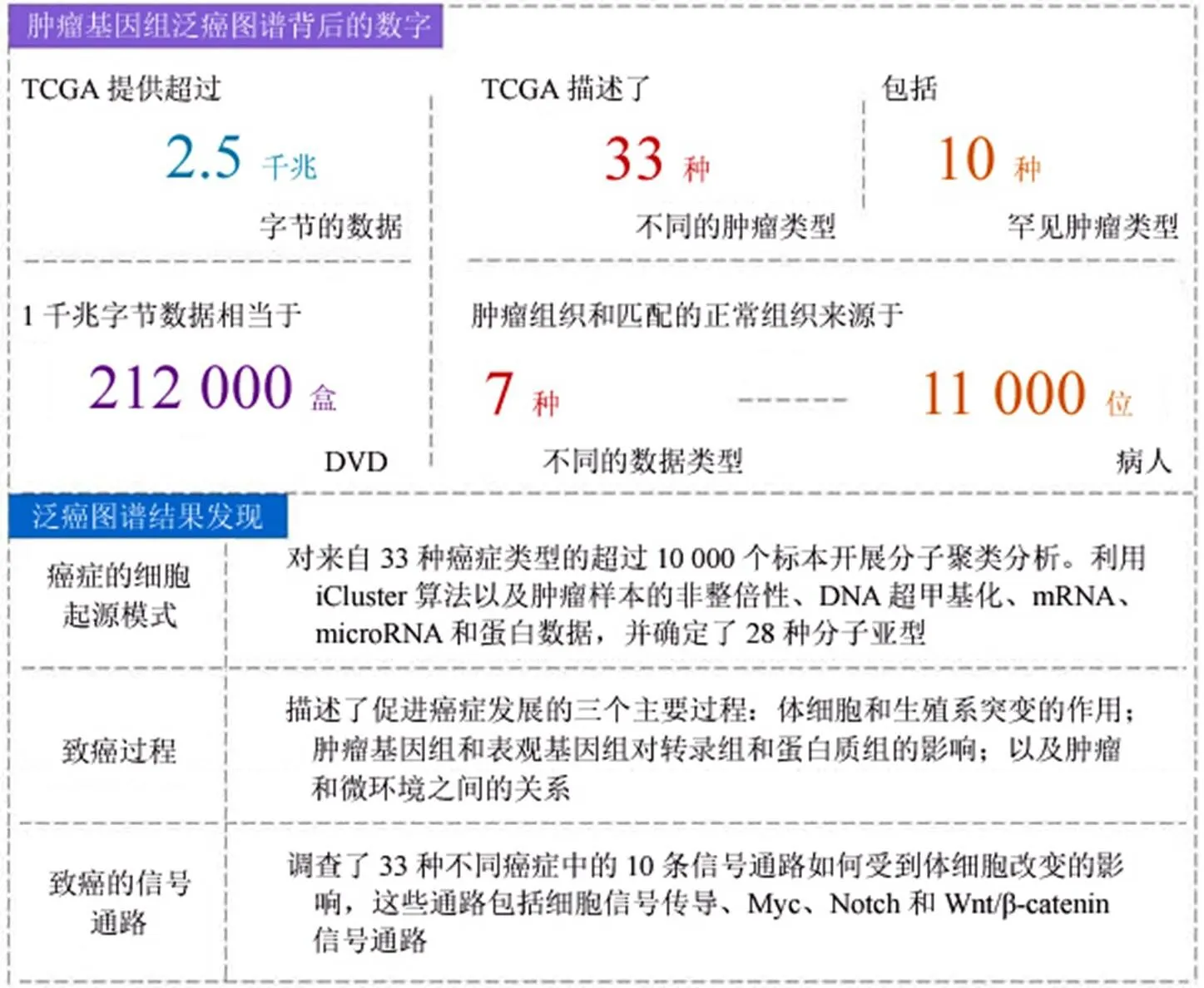
图 1 癌症基因组图谱(TCGA)-泛癌图谱的工作规模和产出
同种癌症有不同的分子特征,不同种癌症却具有同样的分子特征,这可能会导向新的治疗方案。Katherine A. Hoadley 说:“一直以来,肿瘤的位置是我们确定治疗方案的主要依据,而这项研究对我们了解不同肿瘤类型之间的关系是有帮助的。如果肿瘤具有基因多样性,那么我们应该重新思考对待它们的方式。”据研究者估计,以分子特征作为分类基准,至少将会有 1/10 癌症患者需要重新进行肿瘤分类以及提供新的治疗方案。
有 4 篇论文针对胃肠腺癌、乳腺癌、鳞状细胞癌和肾细胞癌的分子特征进行了深入的分析[5-8]。大多数异质性的癌症亚型类型属于四种主要的细胞起源模式:泛胃肠型、泛妇科型、泛鳞状细胞型和泛肾脏型。胃肠道腺癌可分为五种分子亚型[5]。其中,具有染色体不稳定的(CIN)泛胃肠道肿瘤分子特征往往存在基因组片段化;而基因组稳定的(GS)结直肠癌则存在 SOX9 和 PCBP1 特异性突变[6]。五种泛妇科肿瘤亚型包含了 16 个分子类型聚类[7]。其中两种亚型显示出激素受体(ERs,PR/AR)水平升高,这将为激素疗法治疗该亚型妇科肿瘤提供有力依据[7]。在五种鳞状细胞癌的亚型中,泛鳞状细胞癌(SCCs)具有与其他亚型不同的分子特征,并通过对遗传和表观改变的综合分析对其进行了重新的亚分类[8]。肾细胞癌(RCC)亚型的分子特征揭示,在三种肾细胞癌组织学亚型中存在相应的体细胞突变,包括透明细胞癌(ccRCC)、乳头状细胞癌(PRCC)和嗜铬细胞癌(ChRCC)。TP53 突变伴随着 ccRCC 和 PRCC 患者的生存率降低;ccRCC 亚型往往存在 BAP1 突变;而 ChRCC 亚型与 PBRM1 和 PTEN 突变有明显的相关性。而 CDKN2A 突变、DNA 高甲基化以及免疫相关 Th2 基因的表达增加与各 RCC 亚型的生存率降低均存在相关性[8]。
此外,该图谱另一篇研究则集中在:利用机器学习算法测定癌细胞的干细胞样特征,以此鉴定可能的新靶点[9]。在癌症的发展过程中,癌细胞会逐渐通过去分化获得祖细胞和干细胞样特征。研究者开发了一种对数回归(OCLR)机器学习算法,通过对细胞转录组和表观遗传学特征的分析评估癌细胞去分化程度。研究者意外发现,癌细胞的去分化程度与 PD-L1 水平以及免疫微环境有关,而且转移型癌细胞的去分化程度会更高[9]。
2 致癌过程
在长达半个世纪的“抗癌之战”中,DNA 测序给预防和治疗癌症带来了新希望,靶向突变的抑癌因子和融合蛋白以及靶向免疫应答反应等新型疗法逐渐走进视野。控制癌症发展的致癌过程集中在染色体的非整倍体改变、剪接突变、生殖细胞和体细胞突变以及驱动基因融合。癌症图谱对致癌突变与基因、蛋白各分子层面的作用进行了详尽的分析,让我们更清楚地“看见”癌症如何生长。这部分内容揭示了生殖细胞和体细胞突变在癌症进展中的作用,并论述了突变对细胞信号传导和免疫细胞组成的影响。研究者们详细论述了癌症相关的数百种生殖细胞突变、融合基因、免疫组化模式、淋巴细胞浸润模式、增强子活性、染色体非整倍体、长链非编码 RNA(IncRNA)、剪接突变等分子特征与癌症发展的关系[3]。
染色体水平的改变显示出不同癌症类型存在差异性的特征,如鳞状细胞肺癌中会发生 3P 染色体缺失[10]。而在传统的 33 种肿瘤类型中,发现了多达 1964 个剪接位点突变,这将直接导致某一肿瘤类型中出现多种选择性的剪接位点突变[11]。癌症中体细胞剪接因子基因(如 SF3B1、U2AF1 和 SRSF2)热点突变导致的 RNA 剪接能力失调可能是肿瘤发生的一个重要标志[12]。在许多易感基因(包括 BRCA1 和 BRCA2)突变中,生殖细胞突变的比例要高于体细胞突变[13]。这也强调了基因组的完整性在肿瘤易感性中起重要作用。基因融合能够重新排列基因启动子以增强致癌作用,并通常作为特定癌症类型的诊断标记,如融合慢性骨髓性白血病中 BCR 和 ABL1 基因发生融合[14]。在所有癌症类型中,复发率最多的是前列腺腺癌 TMPRSS2-ERG 的基因融合,同时融合驱动因子也存在于特定的肿瘤亚型中(如肺腺癌的 ALK-EML4 融合)[15]。因此,这些复杂的相互作用过程表明,人们应当在更广阔的视野下评估癌症,这将导致临床和科研的巨大变化。针对不同肿瘤类型进行多组学联合分析将成为科研的主流。
另外,肿瘤微环境(TME)和免疫组分的作用对肿瘤发展同样至关重要。由于肿瘤自身的异质性较强,不同免疫组分预后准确性存在明显差异。在绝大多数肿瘤类型中,T 辅助(CD4+)细胞亚型 TH1 细胞及其衍生的细胞因子(如干扰素-γ)都与良好的临床结果紧密相关[16]。在多数浸润型肿瘤的免疫细胞亚型中,CD8+细胞毒性 T 细胞对患者生存影响最大[17]。此外,记忆 T 细胞、TFH 细胞和 B 细胞均与生存时间的延长有关[18-20]。然而,所有肿瘤内的髓样来源抑制细胞(MDSC)存在有效的免疫抑制能力,能够促进癌症的进展、转移和扩散[21]。嗜中性粒细胞则是与预后不良最为相关的免疫细胞亚群[22]。因此,对特定免疫成分的预后和预测能力需要评估多个免疫参数及其组合,这将是一种更准确的评估手段[23]。
3 致癌的信号通路
控制细胞生长、凋亡和细胞周期的信号通路改变是癌症发生的常见标志。而信号通路在不同肿瘤类型的改变频率和共同激活频率是存在差异的,其中包含着复杂的相互作用和通路间的相互串扰。某些关键信号通路(如 RTK-RAS 信号),在不同类型的癌症中经常发生改变,而其他通路则只在某些恶性肿瘤亚型中发生改变,例如:鳞状组织中氧化应激反应通路会反复改变[24]。泛癌图谱对不同癌症中信号通路的改变程度和机制进行了深入分析,揭示了这些过程在不同癌症中的相似性和差异性[2]。该研究总结出在 33 种癌症中基因频繁发生变化的 10 条经典通路,包括细胞周期、Hippo、Myc、Notch、Nrf2、PI3K、RTK-RAS、TGF、p53 和 Wnt 通路。这将有助于开发新的联合疗法和个性化治疗[2]。
研究者对 9125 个肿瘤样本中的突变、拷贝数、mRNA、基因融合和表观遗传数据进行综合分析,探索了其中基因突变的模式和机制。在所有癌症类型中,RTK-RAS 通路是变化频率最高的信号通路(46%),其次是细胞周期通路(45%)和 PI3K 通路(33%)[2]。黑色素瘤是 RTK-RAS 通路变化频率最高(94%)的肿瘤亚型。有趣的是,一些通路(如细胞周期通路、PI3K 通路)的变化分布在多个基因上,而其他通路(如 Wnt、Myc 和 Nrf2 通路)的改变只影响少数一两个基因。
阐明不同肿瘤亚型之间通路改变的协作性和排斥性与潜在的治疗意义密切相关。根据通路变化的情况,研究者把 33 种癌症分为 64 种亚型,准确阐明了不同肿瘤亚型之间信号通路变化的协作性和排斥性。研究者发现,89% 的肿瘤在上述 10 种典型通路中至少有一种通路发生改变。而且根据现有的靶向药物治疗方案,至少 57% 的肿瘤能够得到一种有效的靶向药物治疗(如 BRCA1/2 和 IDH1/2),30% 的肿瘤(如MSI 和 POLE 亚型)则存在两种或两种以上的靶向药物治疗的可能性,这也很可能为肿瘤患者带来新的联合治疗方案[2]。比如说,联合使用 CDK4和 MDM2 抑制剂,通过频繁的放大(78%)CDK4 和 MDM2 两个靶点的信号,而在去分化脂肉瘤中起到协同作用[25]。
4 总结
泛癌图谱是一份大型的、系统性的研究报告,有许多新颖的发现和颠覆性的突破。新的泛癌图谱基于分子相似性对人类肿瘤类型进行了重新分类;揭示了致癌过程中的复杂环境;阐明了致癌信号通路中基因突变的机制和协同性。更有助于准确选择个体化药物和更加综合全面的治疗方案。泛癌症图谱也揭示了不同肿瘤类型的共同细胞起源,为癌症的临床诊断和靶向治疗提供重要的参考。此外,泛癌图谱数据库公开的数据更加方便了科研、医务人员对原始数据进行多角度、多层次的分析,为更多创新型科研成果的产出提供可能。
[1] The Cancer Genome Atlas Research Network. Integrated genomic characterization of oesophageal carcinoma. Nature, 2017, 541(7636): 169-175.
[2] Sanchez-Vega F, Mina M, Armenia J, et al. Oncogenic signaling pathways in the cancer genome atlas. Cell, 2018, 173(2):321-337, e10.
[3] Ding L, Bailey MH, Porta-Pardo E, et al. Perspective on oncogenic processes at the end of the beginning of cancer genomics. Cell, 2018, 173(2):305-320, e10.
[4] Hoadley KA, Yau C, Hinoue T, et al. Cell-of-origin patterns dominate the molecular classification of 10,000 tumors from 33 types of cancer. Cell, 2018, 173(2):291-304, e6.
[5] Ricketts CJ, De Cubas AA, Fan H, et al. The cancer genome atlas comprehensive molecular characterization of renal cell carcinoma. Cell Rep, 2018, 23(1):313-326, e5.
[6] Liu Y, Sethi NS, Hinoue T, et al. Comparative molecular analysis of gastrointestinal adenocarcinomas. Cancer Cell, 2018, 33(4):721-735, e8.
[7] Berger AC, Korkut A, Kanchi RS, et al. A comprehensive pan-cancer molecular study of gynecologic and breast cancers. Cancer Cell, 2018, 33(4):690-705, e9.
[8] Campbell JD, Yau C, Bowlby R, et al. Genomic, pathway network, and immunologic features distinguishing squamous carcinomas. Cell Rep, 2018, 23(1):194-212, e6.
[9] Malta TM, Sokolov A, Gentles AJ, et al. Machine learning identifies stemness features associated with oncogenic dedifferentiation. Cell, 2018, 173(2):338-354, e5.
[10] Taylor AM, Shih J, Ha G, et al. Genomic and functional approaches to understanding cancer aneuploidy. Cancer Cell, 2018, 33(4):676-689, e3.
[11] Jayasinghe RG, Cao S, Gao Q, et al. Systematic analysis of splice-site- creating mutations in cancer. Cell Rep, 2018, 23(1):270-281, e3.
[12] Seiler M, Peng S, Agrawal AA, et al. Somatic mutational landscape of splicing factor genes and their functional consequences across 33 cancer types. Cell Rep, 2018, 23(1):282-296, e4.
[13] Huang KL, Mashl RJ, Wu Y, et al. Pathogenic germline variants in 10,389 adult cancers. Cell, 2018, 173(2):355-370, e4.
[14] Cilloni D, Saglio G. Molecular pathways: BCR-ABL. Clin Cancer Res, 2012, 18(4):930-937.
[15] Gao Q, Liang WW, Foltz SM, et al. Driver fusions and their implications in the development and treatment of human cancers. Cell Rep, 2018, 23(1):227-238, e3.
[16] Fridman WH, Zitvogel L, Sautès-Fridman C, et al. The immune contexture in cancer prognosis and treatment. Nat Rev Clin Oncol, 2017, 14(12):717-734.
[17] Petitprez F, Fossati N, Vano Y, et al. PD-L1 expression and CD8(+) T-cell infiltrate are associated with clinical progression in patients with node-positive prostate cancer. Eur Urol Focus, 2019, 5(2):192-196.
[18] Yuen GJ, Demissie E, Pillai S. B lymphocytes and cancer: a love-hate relationship. Trends Cancer, 2016, 2(12):747-757.
[19] Vinuesa CG, Linterman MA, Yu D, et al. Follicular helper T cells. Annu Rev Immunol, 2016, 34:335-368.
[20] Pagès F, Berger A, Camus M, et al. Effector memory T cells, early metastasis, and survival in colorectal cancer. New Engl J Med, 2005, 353(25):2654-2666.
[21] Anani W, Shurin MR. Targeting myeloid-derived suppressor cells in cancer. Adv Exp Med Biol, 2017, 1036:105-128.
[22] Gentles AJ, Newman AM, Liu CL, et al. The prognostic landscape of genes and infiltrating immune cells across human cancers. Nat Med, 2015, 21(8):938-945.
[23] Bruni D, Angell HK, Galon J. The immune contexture and immunoscore in cancer prognosis and therapeutic efficacy. Nat Rev Cancer, 2020. Online ahead of print.
[24] Cancer Genome Atlas Network. Comprehensive genomic characterization of head and neck squamous cell carcinomas. Nature, 2015, 517(7536):576-582.
[25] Laroche-Clary A, Chaire V, Algeo MP, et al. Combined targeting of MDM2 and CDK4 is synergistic in dedifferentiated liposarcomas. J Hematol Oncol, 2017, 10(1):123.
10.3969/j.issn.1673-713X.2020.06.013
何询,Email:hexun168@sina.com
2020-09-27

