恶臭假单胞菌的正丁醇耐受性驯化及比较基因组学分析
肖 琳, 吴安宁, 许国超, 韩瑞枝, 倪 晔*
(1.江南大学 工业生物技术教育部重点实验室,江苏 无锡214122;2.江南大学 生物工程学院,江苏 无锡214122)
恶臭假单胞菌广泛分布于自然界中,常被用来降解环境中的一些有机溶剂,如对硝基苯酚、水杨酸、烷烃等。刘忠等分离到一株合成L-半胱氨酸的恶臭假单胞菌[1],广东省农科院土壤肥料研究所解开治等人发现一株能高效降解拟除虫菊酯的恶臭假单胞菌XP12[2],这在生物农药领域具有重要应用价值。然而,当有机溶剂含量较高时,菌体本身的存活率就会下降,进而导致生物降解及生物合成的功能受到影响。目前,有关恶臭假单胞菌有机溶剂耐受性元件及改造方面的研究较少[3-4]。作者通过ARTP诱变技术对恶臭假单胞菌进行随机诱变,在高正丁醇压力下进行驯化培养,定向筛选高正丁醇耐受性菌株。
ARTP诱变育种[4]是近年来发展起来的一种新型的育种技术,它受到射频功率的驱动,可以产生等离子射流,进而影响细胞壁和质膜的结构和渗透性,导致DNA损伤,包括错义突变,缺失或移码突变[5-8]。由于ARTP技术具有活性粒子浓度高、操作容易、运行成本低廉、安全性好,正突变率高以及对环境无污染等特点[9],在微生物突变育种及生物医学领域得到广泛应用。南京工业大学姜岷团队利用ARTP诱变策略将大肠杆菌AFP111中木糖转化为琥珀酸代谢途径的关键物质ATP的浓度提高了1.33倍[10];南京农业大学赵耕毛团队通过ARTP技术培育Arthrobactersp.KQ11突变体,使其葡聚糖酶活性明显增加[11];成都大学田敏团队通过ARTP诱变获得一株达托霉素高产菌株[12]。
基因组重测序是高通量测序的一种重要应用,通过对基因组序列已知的个体进行基因组测序[13],并在个体或者群体水平上进行差异性研究。日本学者Kamada等同时利用第三代测序仪PacBio RS sequencer和第二代测序仪Illumina MiSeq对纳豆枯草芽孢杆菌进行高通量测序和比较基因组研究,得到一个更为完整和高质量的全基因组序列,填补了之前对该菌株测序的空白,并发现了330个新的蛋白质编码序列[14-15];中国农业科学院朱振东团队通过基因组重测序鉴定候选基因,并为大豆中的新型疫霉菌抗性基因RpsHC18开发诊断标记[16]。以上结果表明,新一代测序是一种精确、快速且经济有效的研究动植物和微生物多样性的重要手段。因此,作者通过对ARTP随机诱变并驯化获得的高正丁醇耐受性恶臭假单胞菌进行全基因组重测序,根据测序所得的SNV和Indels分布进行KEGG/GO注释并分析,为进一步研究其溶剂耐受性提供了理论依据。
正丁醇是一种较常见的短链醇类有机物,其用途非常广泛,主要用于生产酯类衍生物,如:邻苯二甲酸正丁酯、丙烯酸丁酯及醋酸丁酯等;此外,正丁醇还是一种极具潜力的新型生物燃料。目前,已有许多关于丙酮丁醇梭菌、大肠杆菌等微生物菌株正丁醇耐受性的研究报道,而对于恶臭假单胞菌的正丁醇耐受性的研究较少。宋亮[17-18]等通过蛋白质组学研究发现了恶臭假单胞菌中3-羟基异丁酸脱氢酶、异分支酸水解酶超家族、蛋白延伸因子EF-Ts对大肠杆菌有机溶剂耐受性影响较大;钱晓红[19]等通过构建全局转录因子突变库,最终筛选到两株高有机溶剂耐受菌;张法[20]等筛选到一株耐受更高正丁醇和环己烷的突变株,并通过基因敲除发现对大肠杆菌有机溶剂耐受性影响较大的两个基因;司海明[21]等通过基因芯片技术,发现了一系列与大肠杆菌有机溶剂耐受性相关的关键基因;吴安宁[22]等从分子伴侣出发,发现secB及其突变体T10A可显著提高大肠杆菌正丁醇耐受性。作者旨在对驯化获得的恶臭假单胞菌菌株进行基因组重测序,挖掘与正丁醇耐受性相关的元件,为构建正丁醇耐受性宿主菌提供理论依据。
1 材料与方法
1.1 菌株、培养条件及驯化实验方法
菌株:P.putida901,作者所在实验室保藏。培养于固体或液体营养肉汤培养基(胰蛋白胨10 g/L,NaCl 5 g/L,牛肉浸膏3 g/L)中。诱变后的菌株在涂板后用液体营养肉汤培养基从固体平板上洗脱至液体摇瓶(50 mL)中,振荡培养6 h后加入正丁醇,继续培养18 h后进行转接传代,最后通过计算它们的比生长速率对最终的驯化菌株进行耐受性比较。
1.2 ARTP随机诱变方法
采用思清源生物科技公司的常压室温等离子体诱变系统,实验中以时间为可变参数,设定功率为100 W[1-4]。P.putida901菌株培养至对数期,用无菌生理盐水洗涤两次后稀释至A600nm为0.5~0.7之间,取10μL该稀释菌液滴在载片上,涂布均匀,将它们分别暴露在ARTP粒子流下10、15、20、35 s,处理过后的载片转移至含有1 mL营养肉汤培养基的EP管中于30℃和120 r/min振荡培养2 h,之后将培养液稀释102,103,104倍,取50μL涂布于固体平板上(每个稀释样品做3次平行涂布),在30℃培养24 h。
1.3 全基因组重测序方案
利用Illumina HiSeq测序平台进行全基因组重测序,根据制造商所给方案进行二代测序样品库的构建,首先将基因组DNA随机切割成500 bp的小片段,用End Prep Enzyme Mix处理片段进行末端修复,使其在一个反应中同时进行5′磷酸化和dA-加尾,最后通过T-A连接来在两端添加衔接子。使用AxyPrep Mag PCR Clean-up(Axygen)对衔接子修饰后的DNA片段进行分离纯化,从中回收长度约410 bp的片段(具有约350 bp的插入片段)(见表1)。然后使用P5和P7引物对每个样品扩增8个循环,因为两个引物都携带可以进行桥式PCR的序列,并且P7引物还携带碱基标签,故可以多路复用。使用AxyPrep Mag PCR Clean-up纯化PCR产物,通过Agilent 2100生物分析仪对其进行验证,并通过Qubit2.0荧光计进行定量。然后根据制造商的说明书将具有不同索引的文库多路复用并加载到Illumina HiSeq仪器上。使用2×150双端配置进行测序,图像分析和碱基调用则由HiSeq仪器上的HiSeq控制软件进行。

表1 测序方案Table 1 The Sequencing protocol
1.4 全基因组重测序数据分析软件及方法
数据过滤可以去除原始数据中带有的接头和低 质 量 的reads, 对 于 所 有 原 始reads, 使 用Cutadapt1.9.1去除衔接子的序列、聚合酶链式反应中的引物序列等,使用BWA0.7.12将干净数据绘制到参考基因组,使用PicardV1.119删除重复的映射结果,SNV/InDel的注释则是由Annovar完成,Pindel和CNVnator用于进行基因组结构变异分析。
2 结果与讨论
2.1 高正丁醇耐受性突变体的驯化筛选
为筛选高正丁醇耐受性突变体,利用ARTP诱变育种技术对菌株P.putida901进行随机诱变和正丁醇驯化培养。
首先考察了不同ARTP照射时间与P.putida901致死率的关系曲线(见图1)。图中在15 s诱变时间下,致死率为81.7%,10 s诱变时间下致死率为54.3%,均处于合理水平,因此选用10 s和15 s的照射时间进行诱变处理。在进行传代培养时,正丁醇添加的初始质量浓度为4 g/L,并以质量浓度为0.8 g/L的梯度逐渐增加,每个质量浓度下进行5轮转接,最终在10 s诱变条件下获得耐受12 g/L质量浓度的正丁醇的突变株。经验证发现在9.6 g/L质量浓度的正丁醇下,突变株对正丁醇耐受性提高了约两倍(见图2)。对于15 s诱变时间,在正丁醇质量浓度增加到9.6 g/L时突变体正丁醇耐受性效果反而变差,推测可能是由于该条件下突变率和负突变增多,导致致死率增加。随后,对未经任何驯化的恶臭假单胞菌(WT)、经正丁醇驯化后的恶臭假单胞菌(WTn-butanol)以及经ARTP技术诱变并驯化后的恶臭假单胞菌(ARTP10S)进行基因组重测序。
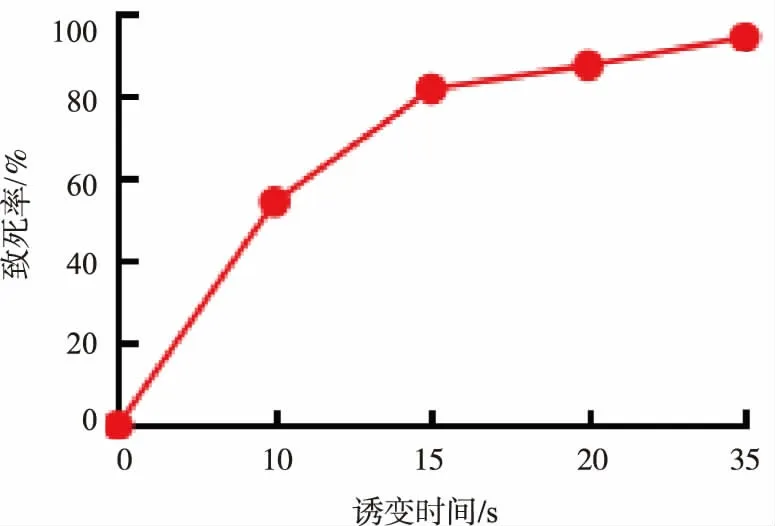
图1 不同ARTP诱变处理时间下P.putida 901的致死率Fig.1 Mortality rate of P.putida 901 under different ARTP mutagenesis time

图2 对照(WT)与经诱变驯化后菌株的比生长速率Fig.2 Specific growth rate of control strain(WT)and adapted strain
2.2 突变体的基因组重测序结果和分析
为探究突变株与野生型菌株之间的差异,利用第二代测序技术,构建插入片段长度为350 bp的文库,采用测序平台Illumina HiSeq对文库进行测序,根据参考基因组比对结果进行注释,得到表2中的数据。结果显示3个测序样本之间WTn-butanol的PF数据(pass filtering data)和干净数据(Clean data)与其他两个样本的数据量差异较大,因为干净数据是在PF数据基础上进一步删除质量不好的reads后得到的数据,这说明测序过程中WTn-butanol样本测序所得到的质量不好的reads数较少,因此其干净数据与其他两者相差较大。而所有样品的SNV和Indels数相差在15~50个之间,这表明相对于参考基因组来说,3个样品各自都有特异的突变,同时还可能有共同的突变。

表2 测序结果中的SNV和Indel数据统计Table 2 SNV and Indel statistics in sequencing results
此外,根据物种的已知基因,将物种的基因组划分为不同的功能单位,然后依据突变所发生的位置,将每个突变位点进行功能区域分类,并对各样本所检测到的突变位点依据功能区进行分类统计。从表3发现,大部分突变位点分布在基因的外显子区,即CDS区或者mRNA区,而 在ncRNA区、3'UTR区及一些剪切位点区域的突变个数是0,这说明突变发生在基因编码区中比较多,在非编码区突变很少,一方面可能是因为这些区域在基因组上所占比例较小,发生突变的概率较小;另一方面可能是因为突变发生在这些区域会影响到不同RNA之间的结合,因为突变位点若位于mRNA 3′UTR区,会影响microRNA对其结合,导致mRNA降解受阻,mRNA表达会上升,进而影响到一些关键蛋白的表达,如与耐受性相关的外排泵蛋白及其他的膜蛋白。
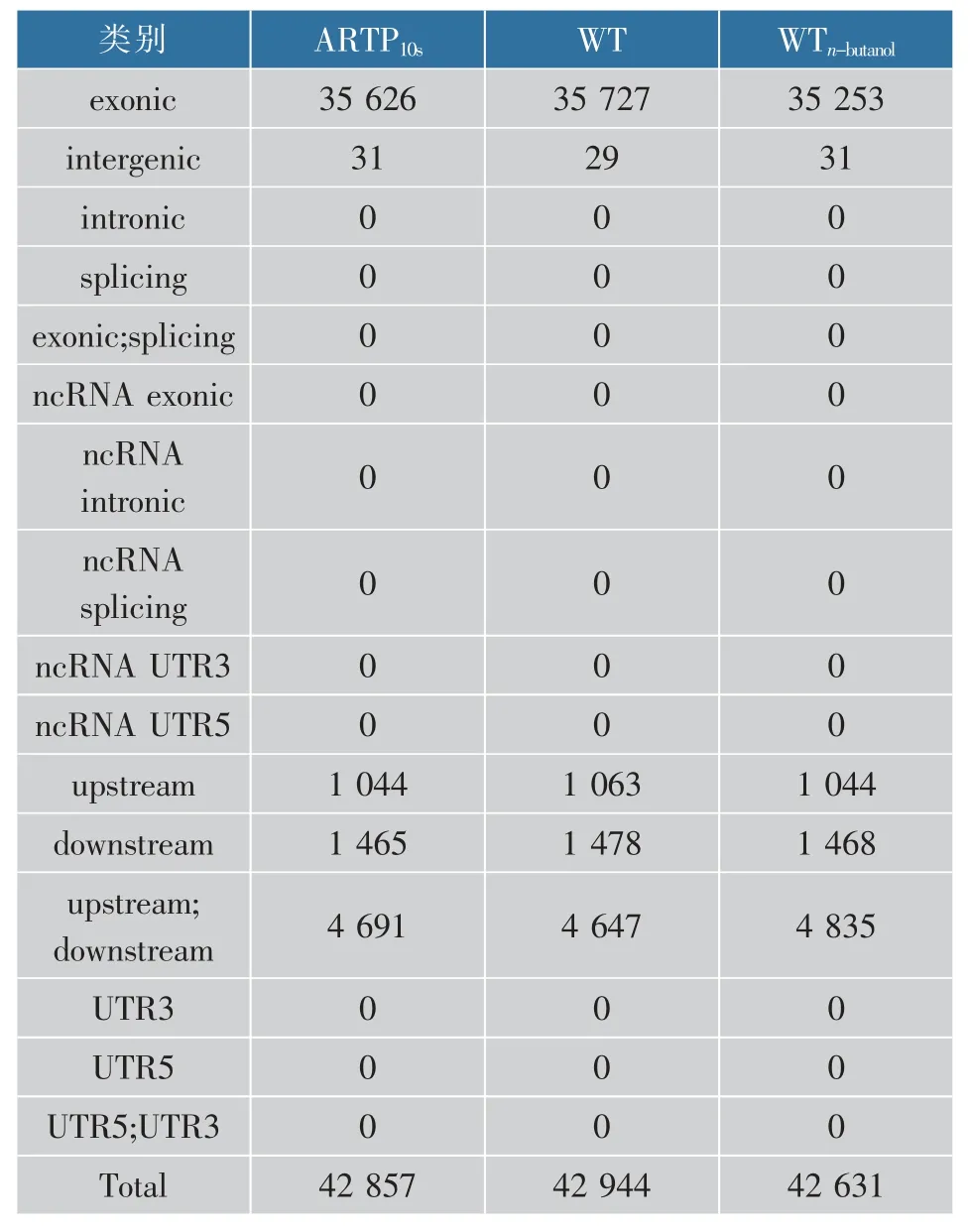
表3 SNV/Indels在基因组不同功能区域的分布状况Table 3 Distribution of SNV/Indels in different functional regions of the genome
接着,对各个样本之间进行分组(WT/ARTP10S、WT/WTn-butanol、ARTP10S/WTn-butanol)和比较基因组学研究,以期获得它们之间共有和特有的突变数目(见图3)。分析发现它们彼此之间相对于参考基因组来说,共有突变基因数目相近(均为41 000左右),其中最多的是WT,并通过两两之间的比较发现它们的特有突变基因数目相差至少有1 100个。在WT/WTn-butanol分组中,WT的特有突变占总突变数的3.7%,WTn-butanol的特有突变比例为3.0%;而在WT/ARTP10S分 组 中,WT的 特 有 突 变WTn-butanol的特有突变比例为2.9%,ARTP10S特有突变比例为2.7%;在WTn-butanol/ARTP10S分组中,ARTP10S的特有突变比例为3.3%,WTn-butanol的特有突变比例为2.7%,这说明在同样的正丁醇胁迫条件下,经过ARTP诱变系统处理过的菌株的突变个数多于未经诱 变 的菌株。 而相对 于WT来说,WTn-butanol和ARTP10S两者之间的特有突变差异为0.3%,这些结果说明ARTP诱变技术及正丁醇驯化都不同程度地对基因组序列突变产生了特有的影响,并且正是由于这些差异的突变而导致细胞的耐受性产生变化,后期将对这些差异突变进行更深层次的研究,以期获得更多参与正丁醇耐受性相关的细胞元件。

图3 不同样本中突变基因的Venn图分布状况Fig.3 Venn plot of mutations in different samples
2.3 基因组重测序结果的功能注释
根据总的Indels分布情况(见图4)发现Indels长度集中分布在1~50 bp区域,并且数量最多的还是1 bp大小的片段插入,且随着插入片段越长,Indels数目也越来越少。相对于参考基因组来说,样本WTn-butanolIndels数目最多(2 205个),其次是WT(2 171个),最少的是ARTP10S(2 153个),这与图3反映的结果一致。上述结果说明ARTP处理10 s并没有引起基因序列的大面积缺失或者插入,仅仅是一些小片段的插入或者缺失,通过进一步分析发现Indels插入位置在treZ、fimV、LOR等基因上,其中treZ基因所编码的麦芽糖寡糖基海藻糖水解酶能够水解海藻糖,而海藻糖是能够稳定生物膜和蛋白质结构的物质,破坏其水解酶基因能够提高海藻糖的含量,进而稳定细胞生物膜,提高细胞的耐受性;fimV基因编码细胞运动蛋白,LOR基因编码细胞膜组件蛋白,这与细胞的生理性应激反应相关。
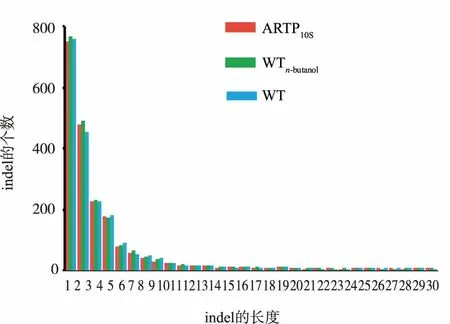
图4 不同样本Indel的长度分布状况Fig.4 Distribution of Indel length
将测序结果中的SNV及Indels使用GO数据库进行本体功能分析,同时通过在线软件链接KEGG数据库对测序样本分组的SNV/Indels进行信号通路分析。根据KEGG数据库功能注释发现其含有人类疾病、有机体系、代谢、细胞过程、环境应激、遗传信息处理等6个功能大类,每一大类中又可以分为不同的代谢功能类别,共有59条代谢通路。分别 统 计 了WT/ARTP10S、WT/WTn-butanol、ARTP10S/WTn-butanol之间所有突变基因的功能,重点比较了WT/ARTP10S的注释结果,发现最多的共有突变主要涉及代谢相关的基因,占总突变的98.6%(见图5),而这一大类中数目最多的是氨基酸代谢相关的基因突变,占14.2%的比例,其次是糖代谢相关,占比12.2%,说明正丁醇胁迫下恶臭假单胞菌细胞中糖代谢、氨基酸代谢相关物质含量发生了改变,正丁醇胁迫下菌株的糖酵解途径和三羧酸循环可能被抑制,氨基酸代谢和糖代谢相关基因的突变使细胞内有足够的能量供应,维持细胞生长,以抵抗外界不利因素,使其更进一步适应正丁醇环境,进而提高耐受性。同时比较了WT/ARTP10S突变之间的差异基因注释结果,从图6可以看到,突变总数目最多的依然是代谢类相关基因,占比为78.6%。突变差异数目最多的基因是环境应激大类中的膜转运相关基因,占总差异突变数的14.3%。由于膜转运蛋白与细胞耐受性紧密联系,当环境中正丁醇进入细胞内对其生长产生抑制时,细胞则通过膜转运蛋白将正丁醇排出细胞外,进而提高其自身的正丁醇耐受性。根据GO数据库注释结果发现所有突变涉及分子功能、细胞组件、生物学过程等多种生理代谢工程。通过统计WT/ARTP10S这一组的结果(图7),发现其中涉及分子功能的比例是44.2%,其突变基因数目从高到低依次为催化活力、结合相关基因、核酸结合转录因子活力、转运活力、分子转导活力、电子载体活力、抗氧化活力与蛋白结合转录因子活力、结构分子活力相关基因。涉及生物过程的基因比例是48.2%,其突变数目从高到低依次是代谢过程、细胞的过程、生物调节、单一的生物过程、定位、运动、再生相关基因。而细胞组分这一类比例较少,为7.6%,它的基因突变数目从高到低依次是细胞组分、膜组分、膜、大分子复合物、细胞器组分及拟核相关基因。综上,三大类功能中基因突变数较多的有蛋白或者膜结合相关的基因、代谢过程相关基因及膜转运和膜组分相关基因,而这些基因与细胞的正丁醇耐受性紧密联系,各司其职。首先,膜组分相关基因合成的细胞膜能够阻挡正丁醇进入细胞,产生防护的第一道屏障;其次,进入细胞的正丁醇,可通过膜转运蛋白将其输送至细胞外;最后,若膜转运蛋白被破坏,细胞可通过自身的糖代谢、氨基酸代谢相关基因的调节来抵抗环境中压力。

图5 WT/ARTP10S共有基因突变的KEGG数据库注释结果统计Fig.5 KEGG database annotation results of WT/ARTP10S shared gene mutation
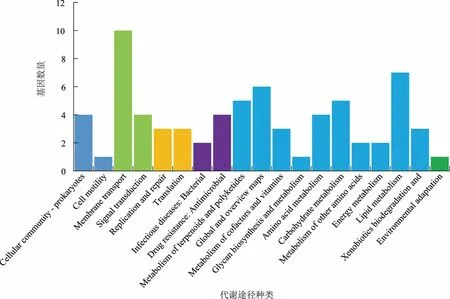
图6 WT/ARTP10S差异突变基因的KEGG数据库注释结果统计Fig.6 KEGG database annotation results of WT/ARTP10S differential gene mutation
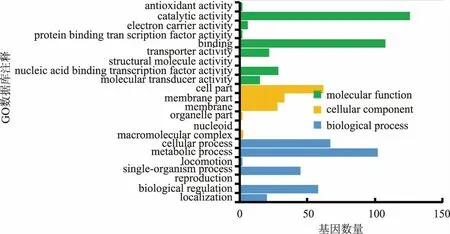
图7 WT/ARTP10S相对参考基因组的所有突变在GO数据库注释结果统计Fig.7 GO database annotation results of WT/ARTP10S mutant genes
3 结语
采用ARTP技术对P.putida901进行随机诱变和驯化筛选,使其对正丁醇的耐受质量浓度从4 g/L提高至12 g/L。在9.6 g/L正丁醇质量浓度下,突变株的比生长速率提高了2倍以上。运用高通量测序技术对经过ARTP诱变后传代驯化的恶臭假单胞菌进行测序,发现经ARTP诱变后的样本基因效果比野生型有所减少。通过KEGG数据库对差异基因进行代谢通路分析[23-25],发现突变最多的是氨基酸等代谢相关基因。通过GO数据库对突变基因进行注释发现基因突变主要集中于分子功能相关、结合相关及拟核相关基因群[25-27]。本研究为恶臭假单胞菌的溶剂耐受性元件挖掘和菌株表型改造等研究提供了一定的依据。

